Introducción
El camarón blanco Litopenaeus vannamei es una especie de importancia comercial que se distribuye a lo largo del Pacífico oriental tropical (Holthuis, 1980) y junto con el camarón café (Farfantepenaeus californiensis) y azul (Litopenaeus stylirostris), constituyen una pesquería en esteros y mar abierto que se mantiene en el nivel máximo sostenible (Sagarpa, 2012), con una producción promedio de aproximadamente 50 mil toneladas para el período 2003-2013 (Conapesca, 2013). A pesar de ello, el sector pesquero reclama el declive de la producción, lo que ha originado la necesidad de implementar nuevas estrategias que contribuyan a su recuperación. De manera particular, en el estado de Sinaloa, México, la repoblación mediante la liberación de larva producida en el laboratorio ha sido vista por autoridades y pescadores como una de estas alternativas (Saucedo-Barrón, 2015).
La liberación de larva o juveniles producidos en cautiverio para repoblación, ya sea para la recuperación del tamaño de la población desovante o para el incremento del tamaño poblacional al nivel permisible por la capacidad de carga del ecosistema para recuperar los niveles de captura, es una de las medidas con potencial (Bell et al., 2006). Sin embargo, persiste una preocupación inherente al potencial impacto negativo sobre las poblaciones silvestres, particularmente en términos genéticos (Perez-Enriquez et al., 2013). Miller y Kapuscinsky (2003) señalan como potenciales impactos genéticos la reducción de la variabilidad genética en la población silvestre a nivel intrapoblacional y diferenciación interpoblacional, entre otros.
La pérdida de variabilidad genética que se da cuando poblaciones silvestres son suplementadas con poblaciones de cultivo, ocurre debido al efecto “Ryman-Laikre” (ver Miller y Kapuscinsky, 2003) en el cual hay una reducción del tamaño efectivo de la población total (Ne), debido a una proporción desigual entre la cantidad de individuos reclutas respecto al número efectivo de reproductores de cada grupo (silvestre y de cultivo). Este fenómeno sucede cuando la larva producida proviene de un número reducido de padres, situación que ocurre comúnmente en especies en cultivo (Taniguchi, 2004). La reducción en Ne puede generar un incremento de la deriva génica y/o de la endogamia, y por consecuencia, una mayor probabilidad de depresión por endogamia; esto a su vez, tiene el potencial de afectar negativamente la eficacia biológica de la población, así como su viabilidad y su potencial evolutivo (Hallerman, 2003).
La introducción de larvas de un mismo origen, en subpoblaciones genéticamente diferenciadas, tiene el potencial de causar disrupciones en la conformación de complejos de genes coadaptados que se expresan ante ambientes en particular, dando como resultado una menor eficacia biológica de dichas subpoblaciones (Miller y Kapuscinsky, 2003). Bajo estas consideraciones, la instrumentación de un programa de repoblación requiere, como punto de partida, la caracterización genética de las poblaciones objetivo, en términos de su diversidad genética, nivel de estructuración en subpoblaciones y flujo genético, entre otros parámetros.
En el caso particular de L. vannamei, la caracterización genética de las poblaciones silvestres en un amplio rango geográfico (miles de kilómetros) desde México hasta Ecuador ha mostrado resultados contrastantes (Sunden y Davis, 1991; Valles-Jiménez et al., 2004), probablemente debido a la naturaleza de los marcadores genéticos. Sunden y Davis (1991) encontraron bajos niveles de diversidad y baja diferenciación genética interpoblacional con aloenzimas, mientras que Valles-Jiménez et al. (2004) utilizando microsatélites reportaron una mayor diversidad genética y diferencias significativas entre las regiones de estudio.
A nivel regional (decenas a cientos de kilómetros) no existen estudios que determinen el grado de diferenciación genética en L. vannamei entre localidades del golfo de California. De acuerdo con un período larvario planctónico de aproximadamente 3 semanas en mar abierto (Hendrickx, 2001) y corrientes estacionales intensas en la parte sureste del golfo capaces de transportar partículas por largas distancias en períodos cortos de tiempo (Marinone et al., 2011), la hipótesis es que las poblaciones de Sinaloa no presentan diferenciación genética significativa. Sin embargo, en otras especies de peneidos (e.g., Penaeus (Litopenaeus) stylirostris, F. californiensis) varios autores indican evidencias de diferenciación genética dentro del propio golfo (Aubert y Lightner, 2000; De la Rosa-Vélez et al., 2000; Ramos-Paredes y Grijalva-Chon, 2003).
El objetivo del presente estudio fue caracterizar la composición genética de la población del camarón blanco (L. vannamei) de Sinaloa, México, determinar si existe diferenciación genética entre las localidades de estudio y definir recomendaciones de manejo, incluyendo la información genética, en función de potenciales actividades de repoblación de la especie.
Materiales y métodos
Los sitios elegidos para la recolecta de ejemplares se ubicaron en 5 sistemas lagunares costeros de Sinaloa que cubren la mayor parte de la zona costera del centro-norte del estado y en los cuales se registra más del 70% de la producción de pesca ribereña (Delegación de Pesca de Sinaloa, datos no publicados). La distancia entre los puntos más alejados es de aproximadamente 320 km. Dentro de cada laguna se colectaron individuos en 2 sitios: en la boca y en la parte más interna. Se realizaron lances con red suripera (luz de malla de 1”) en octubre de 2011 en las localidades siguientes: Agiabampo (Jitz-Agia) y Banderas (Jitz-Ban) en la bahía de Jitzamuri, estero criadero (Colo-Cria) y boca (Colo-Boca) en la bahía del Colorado, San Ignacio (Nava-Igna) y Huitussi (Nava-Hui) en la bahía de Navachiste, Reforma (StaMa-Ref) y Yameto (StaMa-Yam) en bahía Santa María y Altata (Pab-Alt) y Las Aguamitas (Pab-Agua) en la ensenada del Pabellón-Altata (Fig. 1). Se colectaron 50 ejemplares por sitio, de los cuales se cortaron 2 pleópodos para preservarlos en etanol al 80%.
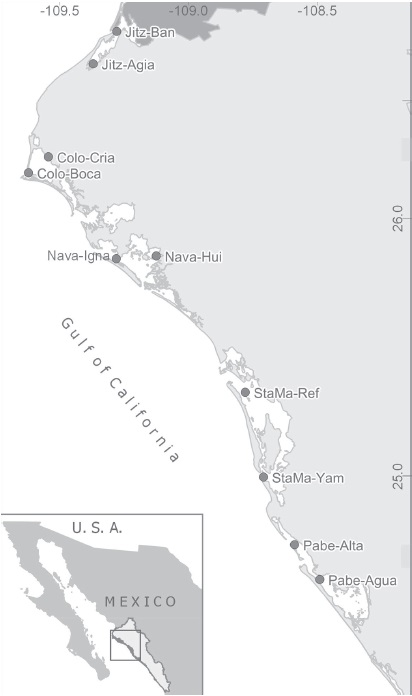
Figura 1 Sitios de muestreo de camarón Litopenaeus vannamei en 5 lagunas costeras de Sinaloa, México. Bahía de Jitzamuri: Las Banderas (Jitz-Ban) y Agiabampo (Jitz-Agia); bahía del Colorado: boca (Colo-Boca) y estero criadero (Colo-Cria); bahía de Navachiste: Huitussi (Nava-Hui) y San Ignacio (Nava-Igna); bahía Santa María: La Reforma (StaMa-Ref) y Yameto (StaMa-Yam); Altata - ensenada del Pabellón: Altata (Pabe-Alta) y Aguamitas (Pabe-Agua).
Se realizaron extracciones de ADN mediante el kit comercial de extracción y purificación de ADN (Wizard® Genomic DNA Purification-Promega). Se analizaron 6 microsatélites (Pvan1758, Pvan1815, TumxLv8.256, TumxLv10.312, TumxLv9.103 y Lv5) marcados con flourescencia (6-FAM, VIC, NED o PET), siguiendo el protocolo de amplificación reportado por Perez-Enriquez et al. (2009). La genotipificación se realizó mediante el analizador genético ABI3130 (Applied Biosystems), los alelos fueron leídos y los tamaños clasificados con el programa GeneMapper® version 4.0, homogenizando las lecturas entre localidades con el estándar de tamaño GeneScan 500 LIZ (Applied Biosystems). Las lecturas de alelos se confirmó al menos una vez en aquellas muestras que presentaron patrones de genotipos no esperados. Se calcularon las frecuencias alélicas y se estimó la diversidad genética [número de alelos por locus (na), número efectivo de alelos por locus (ne), heterocigosidad observada (Ho) y heterocigosidad esperada (He)] con el programa Arlequin 3.5 (Excoffier et al., 2005). Con el mismo programa se estimó la desviación del equilibrio de Hardy-Weinberg (EHW) para cada locus y cada población, y se verificó la presencia de alelos nulos mediante el programa Microchecker (Van Oosterhout et al., 2004).
La estructura genética de la población de Sinaloa se probó mediante el valor de Fst obtenido mediante un análisis de varianza molecular (Amova) utilizando Arlequin 3.5. Se realizó un análisis de Fst pareado por localidades usando el paquete Adegenet de R (Jombart, 2008). Los valores de Fst se visualizaron como distancias genéticas en un árbol “vecino más cercano” de Saitou y Nei (1987) estimado con el paquete Ape de R (Paradis et al., 2004). Para visualizar la distribución de genotipos por individuo/localidad en 2 dimensiones se corrió un análisis de componentes principales (PCoA) utilizando el paquete Adegenet de R (Jombart, 2008). La estructura genética también se infirió mediante el método de agrupamiento instrumentado en el programa STRUCTURE 2.3 (Pritchard et al., 2000). Este modelo asume la presencia de un número K de grupos, cada uno de ellos caracterizado por un set de frecuencias alélicas y los individuos son asignados a cada grupo por probabilidad. Las condiciones de las corridas del método Monte Carlo de la Cadena de Markov fueron 1,000,000 pasos de precalentamiento y 100,000 simulaciones para cada número posible de grupos (K = 1 hasta 8). El valor de K más probable se calculó mediante el método de Evanno (Evanno et al., 2005) implementedo en el programa STRUCTURE HARVESTER (Earl y von Holdt, 2012).
Resultados
Las frecuencias de los alelos más comunes fueron en general similares entre localidades, con algunas diferencias particulares (tabla 1). Sobresale una elevada cantidad de alelos en cada microsatélite, con diferencias de una sola base. La diversidad genética en las localidades fue relativamente elevada (tabla 2) con valores promedio de número de alelos por locus na = 16.1, número efectivo de alelos por locus ne = 8.8, y heterocigosidad esperada He = 0.87, aunque con una menor heterocigosidad observada (Ho = 0.69). El comparativo entre localidades mostró homogeneidad y sin ningún patrón de variación acorde a la posición geográfica (Fig. 2). El sitio con la menor diversidad fue Nava-Hui y la diferencia entre los sitios con mayor y menor variabilidad fue del 19% en na (Nava-Igna vs. Pabe-Agua) (tabla 2). Se detectaron desviaciones del EHW en todas las localidades (tabla 1). Los loci Pvan1758 y TUMXLv9.103 presentaron desequilibrio significativo en todas las localidades. El análisis con Microchecker indicó que estas desviaciones pueden estar asociadas a la ocurrencia de alelos nulos. En otros loci (p. ej. Pvan1815 y TUMXLv8.256) también se registró la probabilidad de alelos nulos en más de 5 localidades.
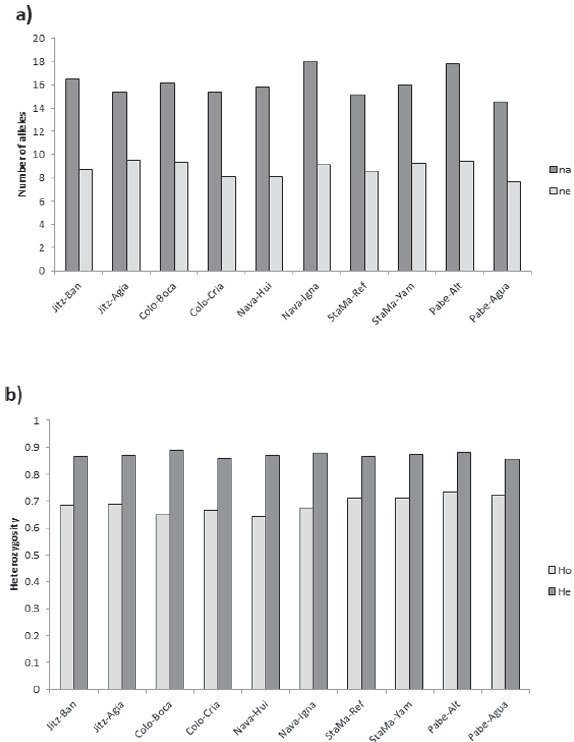
Figura 2 Diversidad genética por sitio de muestreo. a) Número de alelos por locus total (na) y efectivo (ne); b) heterocigosidad observada (Ho) y esperada (He).
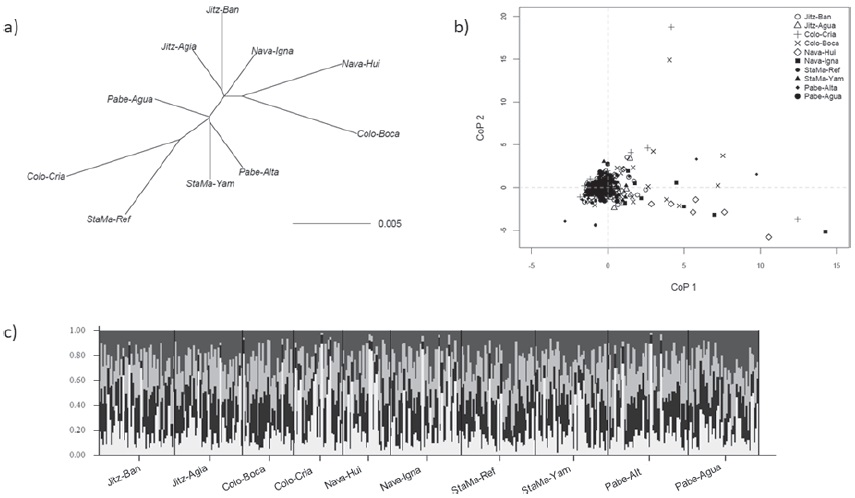
Se obtuvo un valor significativo de diferenciación genética global entre las 10 localidades (Fst = 0.012, p < 0.001). El análisis de Amova dio diferencias significativas entre las lagunas costeras (Fst = 0.004; p < 0.05) y no arrojó diferencias significativas entre los sitios dentro de cada laguna costera (bahía de Jitzamuri, bahía del Colorado, bahía de Navachiste, bahía Santa María y ensenada del Pabellón-Altata; Fig. 1), (Fst = 0.005; p = 0.196). El análisis pareado de Fst indicó que 11 de las 45 comparaciones simultáneas fueron estadísticamente significativas (Fig. 3). Sin embargo, ninguno de los análisis de diferenciación [i.e. distancias genéticas (Fig. 4a), PCoA (Fig. 4b), método de grupos (Fig. 4c)] mostraron una agrupación acorde a la ubicación geográfica de las localidades y las lagunas costeras. Un segundo análisis de Amova agrupando los datos por laguna costera (bahía de Jitzamuri, bahía del Colorado, Bahía de Navachiste, bahía Santa María y ensenada del Pabellón-Altata; Fig. 1), reveló que no hubo diferencias genéticas significativas entre estos cuerpos de agua (Fst = 0.005; p = 0.062).
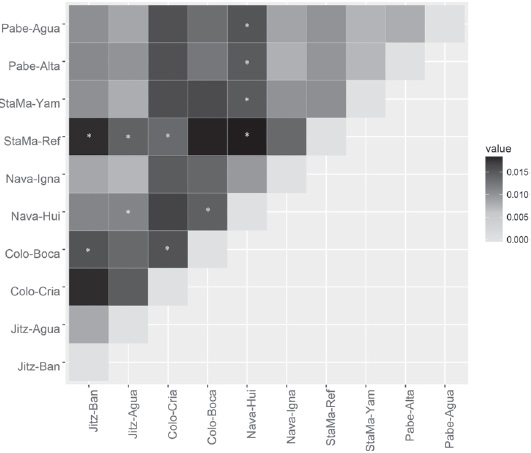
Figura 3 Análisis de Fst pareado entre las localidades de muestreo. El asterisco muestra diferencias significativas valor p < 0.05.
Discusión
La variabilidad genética en todas las bahías recolectadas en Sinaloa fue elevada y superior a la reportada previamente para una localidad silvestre (Yameto; Valles-Jiménez et al., 2004), lo que pudiera indicar que el potencial de viabilidad de la población en el largo plazo se conserva aun considerando los cambios en el medio ambiente.
El análisis de estructura genética de la población silvestre mostró un patrón poco consistente, ya que aun cuando el índice Fst indicó diferencias genéticas significativas entre localidades, dichas diferencias no fueron acordes con su ubicación geográfica. Sin embargo, dado que el análisis en donde se agruparon muestras de las 2 localidades de cada sistema estuarino como una sola localidad no indicó valores pareados de Fst significativos, se concluye que el camarón que habita las 5 lagunas costeras del estudio constituyen una sola población panmíctica, con flujo genético entre ellas.
Si bien, para el camarón blanco Litopenaeus vannamei no hay estudios de diferenciación genética de corta escala geográfica, los análisis a amplia escala indican niveles de diferenciación significativos, derivados de barreras geográficas u oceanográficas (Valles-Jiménez et al., 2004). Supungul et al. (2000) con Penaeus monodon en Tailandia obtienen resultados similares con lo inferido para L. vannamei, con una población genéticamente diferenciada respecto a su área de distribución, pero homogeneidad local.
La homogeneidad genética de L. vannamei a nivel regional pudiera ser explicada por la dispersión larvaria de organismos entre lagunas costeras debido a las corrientes y a la carencia de barreras al flujo. La combinación de un periodo larvario de aproximadamente 25 días (FAO, 2006) con los patrones de corrientes de la parte sur del golfo de California (la cual contiene al área de estudio), favorecen el transporte de partículas en una escala de cientos de kilómetros (Marinone et al., 2011), y soportarían la hipótesis de la falta de diferenciación genética regional.
Sin embargo, los resultados contrastan con lo reportado para otras especies de peneidos dentro de la región de estudio con periodos larvarios de duración similar (e.g., L. stylirostris y F. californiensis), los cuales indican flujo genético limitado y, por ende, diferenciación genética en algunas regiones del golfo de California (Aubert y Lightner, 2000; De la Rosa-Vélez et al., 2000; Ramos-Paredes y Grijalva-Chon, 2003). Este contraste en las conclusiones podría ser explicado, al menos en parte, por la naturaleza de los marcadores genéticos utilizados. Los estudios con aloenzimas (De la Rosa et al., 2000; Ramos-Paredes y Grijalva-Chon, 2003) pudieran revelar una diferenciación originada por presiones selectivas, ya que los loci utilizados pudieran no ser neutrales a la selección (Freeland, 2005; p 45-46). Por lo que respecta a los marcadores tipo RAPD utilizados por Aubert y Lightner (2000), se ha demostrado que pueden tener poca reproducibilidad por lo cual se debe demostrar en primera instancia que tienen un modelo de herencia mendeliana antes de su aplicación (Novelo et al., 2010).
De cualquier manera, no se puede descartar que exista un fenómeno de corta escala asociado a la diferenciación genética, tal como el aislamiento y la presencia de remolinos en el Alto golfo (Marinone et al., 2011), que sirvan como barreras oceanográficas y/o geográficas. Esto puede ser similar a lo reportado en L. schmitii por Borell et al. (2007), peneido del Atlántico con un periodo larvario de duración similar a L. vannamei (Boschi, 1977), quienes reportaron diferenciación genética en un rango geográfico relativamente corto en Cuba, probablemente asociado a un aislamiento ocasionado por este tipo de barreras.
Por otra parte, una posible alternativa para explicar la desviación del EHW aun habiendo panmixia entre localidades, es el efecto Wahlund, el cual establece que la mezcla de poblaciones con diferentes frecuencias genotípicas tiende a presentar un déficit de heterocigotos (ver Hedrick, 2000). En este sentido, considerando que el cultivo de camarón en el estado de Sinaloa es una actividad de alta relevancia (Conapesca, 2013), se propone la hipótesis de la mezcla de poblaciones de cultivo con silvestres. Esta hipótesis podría ser soportada por el hecho de que la composición genética de las poblaciones de cultivo es diferente a las silvestres (Mendoza-Cano et al., 2013; Perez-Enriquez et al. 2009), y a que se ha observado la presencia de ejemplares con perfiles genéticos de camarón cultivado en muestras de poblaciones silvestres del estado de Sinaloa (Perez-Enriquez et al., 2018).
Las implicaciones de los resultados del presente estudio para efectos de programas potenciales de repoblación de la especie van en 3 vertientes: 1) dado que no hay elementos que indiquen que cada localidad deba ser tratada, desde el punto de vista genético, de manera independiente, la constitución de un lote de reproductores de origen silvestre puede tener como origen cualquiera de los cuerpos de agua comprendidos en el estudio; 2) dado el potencial de disrupción de la composición genética derivada de las actividades acuícolas, se sugiere que se evite la liberación de larvas proveniente de laboratorios dedicados a la producción comercial de larva; 3) el manejo por repoblación requiere de manera adicional la consideración de factores ecológicos (e.g., razones que explican la disminución de las poblaciones, sitios adecuados para la liberación, calidad del hábitat, etc.) y pesqueros (distribución del esfuerzo, cuotas de captura, etc.), entre otros (ver Miller y Kapuscinsky, 2003; Perez-Enriquez et al., 2013) para incrementar la probabilidad de éxito.











 nova página do texto(beta)
nova página do texto(beta)