La Ecología Molecular es una disciplina científica propuesta originalmente por Paul Weiss para describir «el continuo de todas las interacciones bióticas entre los niveles molecular, celular y organísmico hasta el ambiente» (Lambert, 1995). Más recientemente, el término se ha utilizado para hacer referencia a la disciplina caracterizada por la implementación de técnicas de genética molecular para investigar problemas ecológicos (Carvalho, 1998, Eguiarte et al., 2007). El avance tecnológico en la genética molecular, desde la electroforesis de proteínas hasta la secuenciación masiva de nueva generación (Davey y Blaxter, 2010, Lewontin y Hubby, 1966, Shendure y Hanlee, 2012), ha determinado el surgimiento y el desarrollo de esta disciplina. Considerando estos avances, la Ecología Molecular ha sido definida actualmente como «una ciencia interdisciplinaria en la que las herramientas y métodos de la biología molecular, la genómica y la bioinformática se han fusionado con la teoría, conceptos y enfoques de la biología organísmica, incluyendo la ecología, la evolución, la conservación y la conducta» (Andrew et al., 2013).
Un momento importante en el desarrollo de esta disciplina fue la aparición de la revista Molecular Ecology en 1992, que incluía como sus principales líneas de interés la biología molecular de las poblaciones, la genética molecular ambiental, la adaptación molecular y los desarrollos tecnológicos de la genética molecular aplicados a la ecología (Burke, Seidler y Smith, 1992). Veinte años después de la publicación del primer número de Molecular Ecology, Andrew et al. (2013) identificaron avances importantes en 3 grandes direcciones: a) el desarrollo de las técnicas de identificación genética (genotipado o genotipificación); b) el desarrollo de nuevos métodos analíticos como la aplicación de la teoría de la coalescencia, y c) el desarrollo de aplicaciones bioinformáticas para analizar grandes grupos de datos.
Como resultado de estos avances, las agendas de investigación se han modificado y es posible anticipar mayores progresos en líneas como: a) la ecología trófica basada en secuencias de ADN; b) el análisis de la diversidad microbiana al interior de organismos multicelulares; c) la filogeografía; d) la filogeografía de comunidades; e) la genómica del paisaje; f) la genómica ecológica y la adaptación molecular; g) la especiación y la hibridación, y h) el análisis del parentesco y la conducta (Andrew et al., 2013).
Las diferentes subdisciplinas de la Ecología Molecular son particularmente significativas para el estudio de las especies nativas en países megadiversos, como lo es México (MacNeely, Miller, Reid, Mittermeier y Werner, 1990). Este país incluye una parte importante del centro de biodiversidad mesoamericano y la mayor parte de la zona de transición mexicana, una región biogeográfica caracterizada por el contacto entre las biotas Neártica y Neotropical. En conjunto, los impresionantes niveles de diversidad de especies y endemismo, así como la compleja historia biogeográfica del territorio mexicano, configuran un escenario en el que las investigaciones en Ecología Molecular son fundamentales para comprender la historia evolutiva de la biota de esta región. De manera no menos importante, la Ecología Molecular puede proveer herramientas importantes e información para el manejo y la conservación de los ecosistemas.
En este trabajo se presenta una revisión de los artículos de investigación en Ecología Molecular de especies distribuidas en México publicados desde 1990 hasta el año 2016 para conocer el desarrollo y el estado actual de la investigación en este campo en el país. En particular, deseamos responder 4 preguntas principales: a) ¿qué tanta investigación existe en el campo de la Ecología Molecular en México?; b) ¿en qué áreas se ha investigado más intensamente?; c) ¿qué sistemas biológicos han atraído el mayor interés?, y d) ¿cuáles son las tendencias de investigación de la Ecología Molecular en el país? Esta revisión concluye con una evaluación de los retos y perspectivas de la investigación de la Ecología Molecular en México.
La búsqueda de literatura se realizó en diciembre del 2016 utilizando la base de datos Scopus® para el área de ciencias de la vida. Como palabras clave se utilizaron las siguientes: «Molecular Ecology», «Population Genetics», «Phylogeography», «Phylogenetics», «Molecular Genetics», «Conservation Genetics», «Genomics», «Metagenomics», «Genetic Diversity», «Genetic Structure», «Genome», «Molecular Markers», «DNA», «RNA», «Gene». Como criterios geográficos se utilizaron las palabras: «Mexico», «Middle America», «Central America», «Neotropics», «North America», «America». El periodo de búsqueda abarcó desde 1990, y se seleccionaron artículos de investigación y revisiones, tanto en inglés como en español.
Se encontraron 1,057 estudios que encajaron con los criterios de la revisión, pero tras realizar una segunda selección manual en la que se eliminaron las publicaciones centradas en la evolución molecular, la biología molecular y la ecología evolutiva, al igual que aquellas que no incluían a México como área de estudio total o parcial, se compiló un total de 656 publicaciones. Al agrupar los estudios por año, se observó una clara tendencia al incremento continuo en el número de estudios publicados hasta el año 2012, cuando se alcanza el pico de productividad con 95 estudios. Entre 2013 y 2016 el número de artículos publicados se redujo considerablemente (Fig. 1), lo cual podría indicar cambios en las tendencias de investigación (i.e., nuevos enfoques) y el incremento en los estándares técnicos para la publicación de estudios.

Figura 1 Número de estudios publicados por año sobre Ecología Molecular realizados total o parcialmente en México de acuerdo con la búsqueda realizada a través de Scopus®.
Las subdisciplinas de la Ecología Molecular mejor representadas fueron la genética de poblaciones (35.3% de los estudios), filogeografía (30.3%), filogenética (11%) (en el contexto de un análisis biogeográfico o de diversidad de comunidades), genética de la conservación (9.2%), genética del paisaje (4.1%), especiación/hibridación (3.5%), ecología genómica y adaptación (3.1%), diversidad microbiana al interior de organismos multicelulares (2.1%), ecología trófica basada en secuencias de ADN (0.7%), parentesco y comportamiento (0.3%), filogeografía de comunidades (0.3%) y genómica del paisaje (0.1%) (Fig. 2).
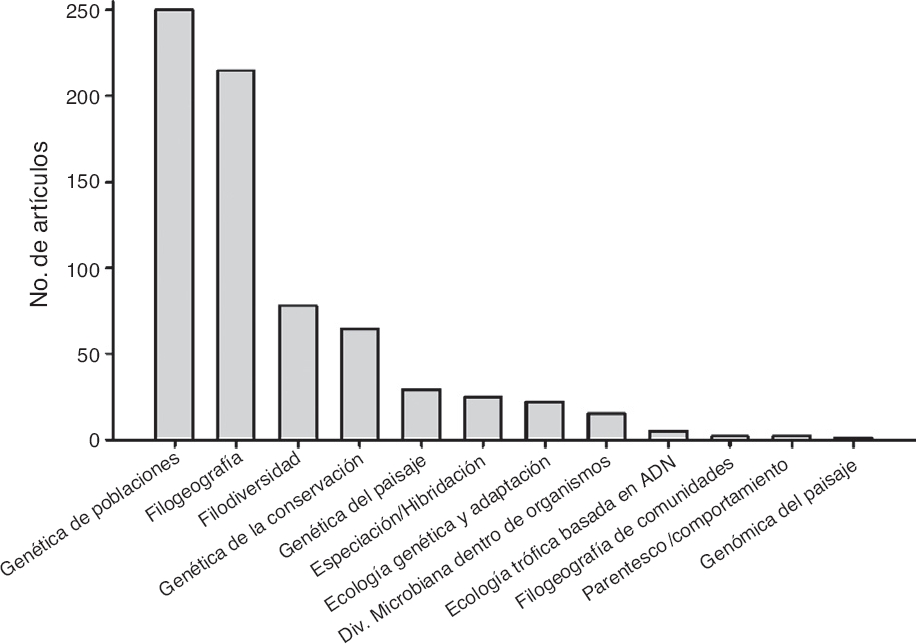
Figura 2 Número total de estudios publicados realizados en México por área de la Ecología Molecular entre 1990 y 2016 de acuerdo con la búsqueda realizada a través de Scopus®.
En lo que respecta a los organismos estudiados, la mayoría de las publicaciones se han centrado en animales (58.5%) y plantas (32.5%), mientras que hongos (2.3%), bacterias (1.5%), arqueas, protozoarios y virus (menos del 1%) están escasamente representados (Fig. 3). Para las especies de animales, la mayor parte de los estudios se han centrado en los fila Chordata (72.1%) y Arthropoda (18.5%) (Fig. 3). En el caso de las plantas, la mayor parte de los estudios han sido sobre angiospermas (82.2%) y gimnospermas (13.7%), con una mínima representación de las pteridofitas y las antocerofitas (Fig. 3).
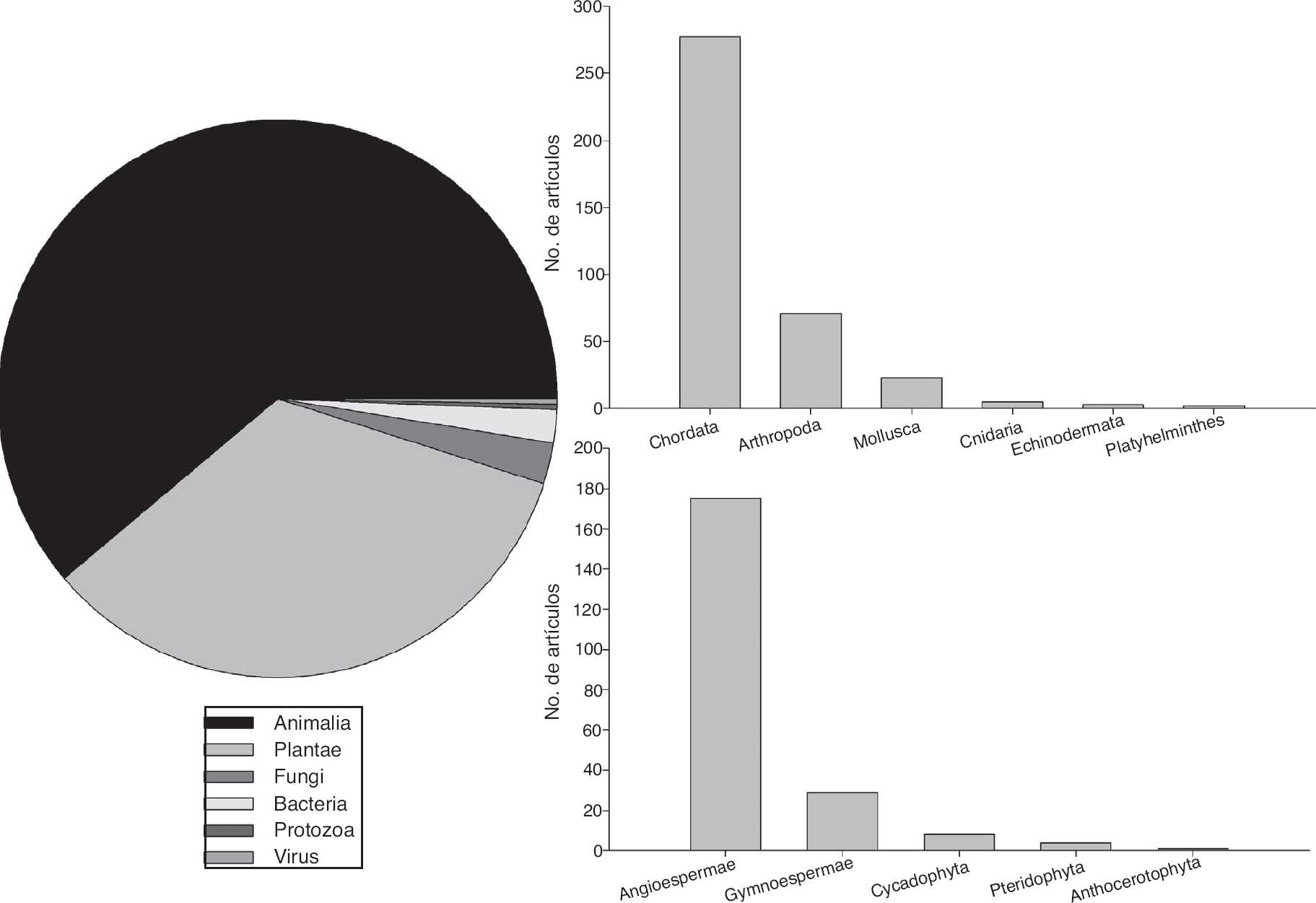
Figura 3 Proporción de los estudios sobre Ecología Molecular realizados en México por grupo de organismos, detallando aquellos correspondientes a animales y plantas de acuerdo con la búsqueda realizada a través de Scopus®.
La mayoría de los estudios recopilados utilizaron las secuencias de fragmentos de ADN como principal fuente de información, mientras que en segundo lugar aparecen los microsatélites (Fig. 4). Los otros tipos de marcadores, incluyendo la secuenciación de nueva generación, han sido utilizados en una proporción menor. Por otro lado, un análisis de acumulación de sistemas estudiados (i.e., géneros en los que se han centrado los estudios de Ecología Molecular) (Fig. 5) muestra que la gran mayoría de las publicaciones se centran en sistemas que son estudiados por primera vez, mientras que relativamente pocos géneros han sido objeto de 2 estudios. Esta tendencia es precisamente la que cabría esperar en un país con una altísima diversidad biológica. Sin embargo, es de esperar que en el futuro aumente el número de especies y géneros que son objeto de estudios recurrentes.

Figura 4 Número de estudios publicados por año sobre Ecología Molecular en México según el tipo de marcador molecular utilizado de acuerdo con la búsqueda realizada a través de Scopus®.
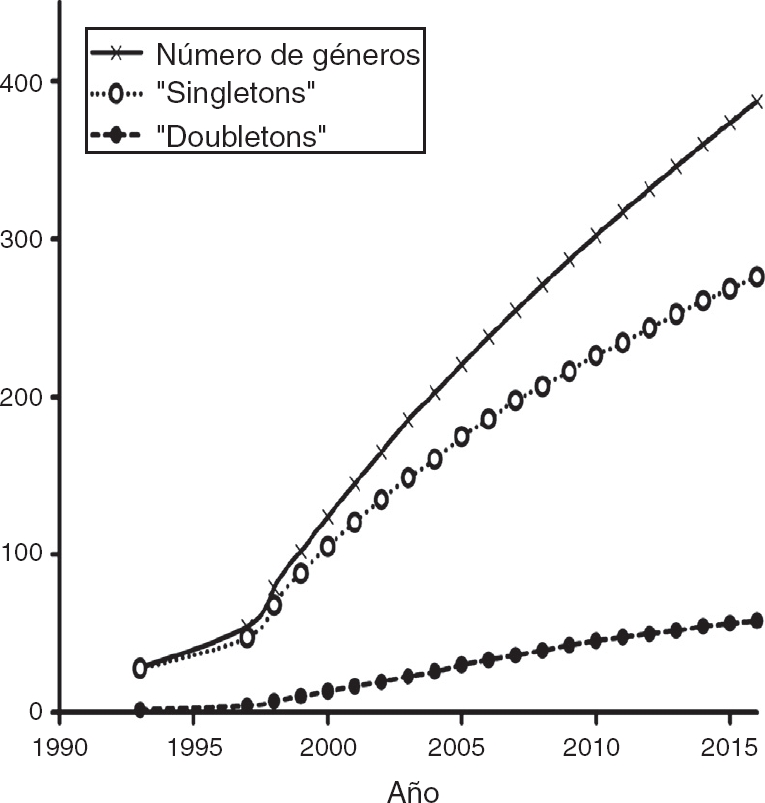
Figura 5 Curva de acumulación de sistemas estudiados en los trabajos sobre Ecología Molecular en México reportados en este estudio.
A continuación presentamos una breve revisión por área y algunos ejemplos representativos de trabajos sobre Ecología Molecular realizados en México. Presentamos primero los campos bien establecidos, como la genética de poblaciones, la filogeografía, la genética de la conservación y la genética del paisaje, y posteriormente discutimos las áreas emergentes de la Ecología Molecular.
Genética de poblaciones, genética de la conservación y genética del paisaje
Indudablemente la Ecología Molecular debe gran parte de sus bases teóricas y analíticas al desarrollo que tuvo la genética de poblaciones durante el siglo xx. En particular, los trabajos de Fisher, Haldane y Wright sentaron las bases matemáticas para el estudio empírico de la variación genética a nivel molecular (Charlesworth y Charlesworth, 2017). Posteriormente, durante el desarrollo de este campo se presentaron discusiones importantes sobre el carácter neutral o adaptativo de la variación genética, que pudieron resolverse gracias a los datos proporcionados por la electroforesis de enzimas y la secuenciación de ADN. A la vez, los avances teóricos permitieron establecer los modelos de sustitución neutral en secuencias, el modelo de sitios infinitos y el modelo de alelos infinitos, así como el reloj molecular (Charlesworth y Charlesworth, 2017). De forma subsiguiente, se propusieron métodos estadísticos que derivaron en la prueba de hipótesis e inferencias evolutivas a partir del análisis de muestras de una población, como lo propone la teoría de coalescencia. De igual forma, se reconoció la importancia de las asociaciones no aleatorias entre diferentes loci o sitios en la secuencia de ADN (desequilibrio de ligamiento) para, más recientemente, formalizar todo un marco conceptual sobre el papel de los procesos de selección en el origen y mantenimiento de la diversidad a nivel molecular (para una discusión detallada, ver Castillo-Cobián, 2007, Charlesworth y Charlesworth, 2017).
En el caso de la genética de poblaciones en México, se han llevado a cabo numerosos estudios que tienen un enfoque predominantemente descriptivo al cuantificar la variación y la estructura genéticas de los organismos de interés (estos estudios representan un 35.3% del total considerado en el presente trabajo), lo que representa un reto en el momento de reseñar aquellos más relevantes. Más recientemente, la genética de poblaciones se ha aplicado en estudios dirigidos a la conservación de diversos taxones o bajo un enfoque paisajístico. Por este motivo, en la presente sección presentamos una breve reseña de trabajos en los que el enfoque genético poblacional para caracterizar la diversidad y el flujo génico ha sido relevante para responder hipótesis en áreas como la genética del paisaje y de la conservación.
En particular, el estudio de los efectos de la configuración del paisaje sobre el flujo génico en las poblaciones naturales se ha desarrollado de forma importante, vinculando a la genética de poblaciones con la ecología del paisaje (Holderegger y Wagner, 2008, Manel et al., 2003). Esta visión paisajística ha permitido incrementar el entendimiento de cómo la topografía y las variables ambientales determinan la variación genética a nivel individual y poblacional, y por lo tanto también representa un área del conocimiento fundamental para definir estrategias de manejo y conservación de especies (Segelbacher et al., 2010). La relación entre los impactos antropogénicos y los patrones de variación y flujo génico ha sido estudiada de forma amplia en diferentes especies mexicanas, incluyendo especies representativas, bien por su carácter endémico, como el ajolote Ambystoma mexicanum (Ambystomidae; Parra-Olea et al., 2011, Recuero et al., 2010) y el pez cola de espada Xiphophorus gordoni (Carson, Espinosa-Pérez y Souza, 2013), o por estar bajo alguna categoría de amenaza, como es el caso del quetzal Pharomacrhus mocinno (Trogonidae; Solórzano, Baker y Oyama, 2004) y la tortuga verde del Pacífico Chelonia mydas (Cheloniidae; Chassin-Noria, Abreu-Grobois, Dutton y Oyama, 2004).
Diferentes autores han enfocado sus estudios en evaluar el efecto de la fragmentación del hábitat sobre la diversidad genética y el flujo de genes en especies de plantas tropicales de bosques secos y bosques lluviosos (Cuartas-Hernández et al., 2015, González-Astorga y Núñez-Farfán, 2001, Peñaloza-Ramírez et al., 2016, Quesada et al., 2013, Rosas et al., 2011, Vargas et al., 2006) y especies de bosques templados, como Quercus castanea (Fagaceae; Herrera-Arroyo et al., 2013). Estos estudios en genética del paisaje también han evaluado el efecto de la fragmentación considerando relaciones interespecíficas como la polinización, la dispersión y los sistemas de apareamiento en plantas (Figueroa-Esquivel et al., 2010, Quesada et al., 2004, Quesada et al., 2013), lo cual contribuye al entendimiento de los efectos a nivel ecosistémico de la degradación y transformación del hábitat. Los estudios de fragmentación del hábitat también se han aplicado a diversos animales, como las abejas nativas Euglossa dilema y E. viridissima (Apidae; Zimmermann et al., 2011), el roedor Habromys simulatus (Cricetidae, Castañeda-Rico, León-Paniagua, Ruedas y Vázquez-Domínguez, 2011), el gorrión Spizella wortheni (Emberizidae, Canales-Delgadillo, Scott-Morales y Korb, 2012), la tarántula Brachypelma vagans (Theraphosidae; Machkour-M’Rabet, Henaut, Calmé y Legal, 2012) y el cocodrilo Crocodrylus moreletii (Crocodylidae), una especie para la cual se han reportado procesos de hibridación asociados con cambios en la configuración del hábitat por influencia humana (González-Trujillo et al., 2012).
Otros estudios (que se podrían incluir en la categoría de genética de la conservación) no incluyen componentes explícitos del paisaje, pero sí utilizan diferentes marcadores moleculares con el fin de caracterizar atributos como la diversidad genética, la estructura genética y el tamaño efectivo poblacional en especies que presentan un grado de amenaza, como por ejemplo Tillandsia achyrostachys (Bromeliaceae; González-Astorga, Cruz-Angón, Flores-Palacios y Vovides, 2004), Dioon sonorense (Zamiaceae; González-Astorga, Vovides, Cabrera-Toledo y Nicolalde-Morejón, 2009), Cyprinodon julimes (Cyprinodontidae; Carson et al., 2014), Gambusia clarkhubbsi (Poeciliidae; Echelle et al., 2013), Odocoileus virginianus (Cervidae; Hernández-Mendoza, Parra-Bracamonte, de la Rosa-Reyna, Chassin-Noria y Sifuentes-Rincón, 2014). Algunos otros trabajos basados en marcadores moleculares han tenido el fin de definir unidades de manejo sostenible y conservación para especies nativas de México como Jacaratia mexicana (Caricaceae; Arias et al., 2012), Pteronotus davyi (Moormopidae; Guevara-Chumacero et al., 2013), Cichlasoma urophthalmus (Cichlidae; Harrison et al., 2014) y Swietenia macrophylla (Melliaceae; Alcalá, Salazar, Guitérrez-Granados y Snook, 2014). Finalmente, los métodos moleculares han sido utilizados con el fin de documentar procesos críticos (y desafortunados) para la preservación de la diversidad biológica, como la extinción de especies (List et al., 2010, Martínez-Méndez et al., 2015, Sandoval-Castillo y Beheregaray, 2015).
Los estudios de grupos de plantas se encuentran dirigidos a una gran variedad de taxones, incluyendo especies de alto valor ecosistémico como el encino Quercus castanea (Fagaceae; Herrera-Arroyo et al., 2013) y especies distribuidas en ecosistemas bajo un alto grado de amenaza, como Cyclopogon luteoalbus en el bosque mesófilo (Orchidaceae; Juárez, Montaña y Ferrer, 2011) y Cestrum miradorense (Solanaceae; Reyes-Zepeda, González-Astorga y Montaña, 2013). También destacan los estudios sobre especies microendémicas como Guaiacum unijugum (Zygophyllaceae; MacCauley, Cortés-Palomec y Oyama, 2010) o Fagus grandifolia var. mexicana (Fagaceae; Montiel-Oscura, Ramírez-Herrera, Ángeles-Pérez, López-Upton y Antonio-López, 2013) y por lo tanto simbólicas considerando que presentan distribuciones extremadamente restringidas y pocas poblaciones remanentes. En otros casos destacan seguimientos temporales para evaluar los procesos de erosión genética debidos a cambios demográficos en poblaciones de especies como Ferocactus histrix (Cactaceae; Castro-Félix et al., 2014) y el pinabete espinoso (Picea chihuahuana: Pinaceae; Wehenkel y Sáenz-Romero, 2012). Por último, vale la pena destacar algunos estudios que reportan niveles considerables de flujo génico aún bajo fuertes presiones resultado de la pérdida y fragmentación del hábitat (Winkler, Koch y Hietz, 2011).
Procesos de domesticación
En este apartado es fundamental destacar aportes numerosos e importantes relacionados con el estudio de los procesos de manejo y domesticación que caracterizan a un país megadiverso a nivel cultural como México, en su mayoría para especies de plantas nativas (Aguirre-Dunga et al., 2012, Blair et al., 2012, Chávez-Pesqueira et al., 2014, Martínez-Castillo et al., 2012, Pacheco-Olvera et al., 2012, Parra et al., 2010, Salazar et al., 2010, Van Heerwaarden et al., 2010), los cuales abarcan diferentes especies de importancia cultural, biológica y económica. Por ejemplo, resalta la identificación de procesos de domesticación y selección artificial a nivel fenotípico con evidencia de diferenciación genética aún con intercambio genético con poblaciones silvestres en el frijol tépari Phaseolus acutifolius (Fabaceae; Blair et al., 2012), la pitaya Stenocereus pruinosus (Cactaceae; Parra et al., 2010) y la jícara Crescentia cujete (Bignoniaceae; Aguirre-Dunga et al., 2012). De igual forma, se han realizado estudios comparando los niveles de diversidad genética en accesiones del frijol lima (Phaseolus lunatus, Fabaceae), a partir de los cuales se ha sugerido la existencia de procesos de erosión genética (Martínez-Castillo et al., 2012).
Entre los grupos con mayor cantidad y enfoques de estudio relacionados con la genética de poblaciones, manejo y domesticación en México resaltan las múltiples variedades del maíz (Zea mays, Poaceae). Los estudios relacionados con esta especie abarcan desde la descripción de los procesos de domesticación por poblaciones humanas precolombinas (Matsuoka et al., 2002) hasta el estudio de procesos de flujo génico entre poblaciones cultivadas y sus parientes silvestres (Pineda-Hidalgo et al., 2013, Van Heerwaarden et al., 2010). Algunos otros ejemplos notables incluyen al chicle o chicozapote, Manilkara zapota (Sapotaceae; González-Hernández, García-Pérez y Guntin-Marey, 2012), a los magueyes Agave cupreata y A. potatorum (Asparagaceae; Aguirre-Dugua y Eguiarte, 2013), a la palma Brahea dulcis (Arecaceae; Ramírez-Rodríguez, Mussali-Galante, Quero y Tovar-Sánchez, 2012) y al cactus Escontria chiotilla (Cactaceae; Tinoco, Casas, Luna y Oyama, 2005).
Finalmente, y relacionados de forma indirecta con los procesos de domesticación, es relevante mencionar estudios que han determinado el grado de flujo génico entre parientes silvestres y organismos genéticamente modificados de la calabaza Cucurbita argyrosperma ssp. sororia (Cucurbitaceae; Cruz-Reyes, Ávila-Sakar, Sánchez-Montoya y Quesada, 2015) en su centro de origen.
Filogeografía y filogeografía comparada
La filogeografía se enfoca en estudiar la distribución geográfica de los linajes de genes, en particular a nivel intraespecífico, o entre especies emparentadas. Esta disciplina ha presentado un amplio crecimiento desde su inicio formal en 1987 (Avise, 2009), el cual se ve reflejado en el número de estudios realizados en México (Fig. 2), superado tan solo por la genética de poblaciones. Junto a los estudios filogeográficos, también es importante destacar a los estudios filogenéticos con un enfoque biogeográfico explícito y a los estudios filogenéticos dirigidos a describir la diversidad biológica de comunidades, categorías para las cuales se encontró un número significativo de estudios. Para el caso de México (y Centroamérica), la prevalencia de estudios filogeográficos se ha visto potenciada por la compleja historia geológica (y por tanto evolutiva) de las áreas de distribución de las especies que se distribuyen aquí.
En este sentido, las preguntas e hipótesis de investigación a nivel intraespecífico e interespecífico se han enfocado en describir cómo cambios climáticos y geológicos han moldeado la historia evolutiva de especies y linajes completos (e.g., Bryson et al., 2011b, Bryson et al., 2011a, Maldonado-Sánchez et al., 2016), proponer escenarios generales a nivel evolutivo en ecosistemas clave como los bosques de niebla (Ramírez-Barahona y Eguiarte, 2013), estudiar procesos de variación y flujo génico en especies bajo expansiones demográficas recientes (e.g., Trujillo-Sierra, Delgado-Valerio, Ramírez-Morillo, Rebolledo-Camacho y Pérez-Nasser, 2013), describir procesos de divergencia genética entre áreas biogeográficas (e.g., Eberhard, Iñigo-Elias, Enkerlin-Hoeflich y Cun, 2015) y describir patrones comunes de evolución entre diferentes grupos biológicos (Gutiérrez-García y Vázquez-Domínguez, 2013). La integración y el análisis comparado de diferentes grupos biológicos para describir patrones generales de los procesos evolutivos en México y Centroamérica (filogeografía comparada) representan un adelanto importante en la dinámica de la filogeografía en México.
Un estudio destacado en esta última área corresponde al trabajo de Ornelas et al. (2013), quienes describen el efecto de elementos geográficos como el istmo de Tehuantepec en México sobre la estructura genética, los procesos de flujo génico y la demografía histórica para 15 especies distribuidas en los bosques mesófilos de México (7 especies de aves, 3 especies de roedores y 5 especies de plantas). Ornelas et al. (2013) sugieren que, aunque el istmo ha favorecido eventos de diferenciación intraespecífica, cuando se comparan los patrones entre especies se observa que el periodo durante el que se desarrollaron estos eventos de diferenciación varía. Por lo tanto, los autores concluyen que la historia evolutiva de la biota de los bosques mesófilos es de linaje específico y está definida por atributos como la capacidad de dispersión de las especies y diferencias en el nicho ecológico de las mismas.
De forma similar, a partir de la gran cantidad de estudios filogeográficos realizados en México y en la región neotropical, Gutiérrez-García y Vázquez-Domínguez (2013) describieron patrones comunes a nivel filogenético y filogeográfico. Dichos patrones fueron agrupados por las autoras en 3 grupos con historias evolutivas comunes. El primero corresponde al grupo Maya, definido por la heterogeneidad geológica que ha favorecido una estructuración y divergencia intraespecífica fuerte además de la presencia de refugios. El grupo Medio-Centroamericano se caracteriza por diferenciación genética en las especies entre tierras altas y bajas asociada con actividad volcánica intensa. Finalmente, el grupo Panameño se caracteriza por haber presentado migraciones latitudinales bidireccionales a través del Istmo de Panamá que promovieron la divergencia de especies y procesos de especiación.
Los ejemplos descritos anteriormente representan iniciativas importantes para caracterizar la historia evolutiva a nivel filogeográfico de diferentes grupos de especies. Este tipo de enfoques, comparando los patrones demográficos a nivel histórico de especies diferentes que comparten una distribución similar, representa un campo de estudio novedoso y prometedor, bien sea a partir de disciplinas como la filogeografía comparada o bien la filogeografía de comunidades. Para México, además de los trabajos mencionados, destacan los aportes de Riddle, Hafner, Alexander y Jaeger (2000) y Pfeiler, Johnson, Richmond y Markow (2013). Para el caso de Riddle et al. (2000), los autores describen procesos de vicarianza en diferentes grupos de aves, mamíferos, anfibios y reptiles asociados a cambios peninsulares e intrusiones del mar de Cortés en Baja California entre el Plioceno y el Pleistoceno. Por otro lado, Pfeiler et al. (2013) describieron patrones de estructuración genética e historia demográfica en especies simpátricas de escarabajos (Coleoptera: Histiridae y Staphylinidae) en parches necróticos de cactus columnares del desierto de Sonora; los autores reportaron niveles altos de flujo génico atribuido a un alto potencial de dispersión de las especies y diferentes niveles de expansión demográfica entre las especies analizadas.
Especiación e hibridación
El estudio de la especiación se ha desarrollado con la finalidad de entender cómo el aislamiento entre poblaciones se desarrolla como producto de un proceso evolutivo a nivel ecológico y geográfico, y se complementa a partir del estudio de cómo el aislamiento se mantiene en presencia de hibridación potencial (Andrew et al., 2013). En este sentido, las nuevas tendencias en el estudio de ambos procesos, y en particular la hibridación, pretenden determinar la base genética de la adecuación de los híbridos en poblaciones naturales, así como estimar la magnitud y el origen de la introgresión, y el papel de elementos móviles del ADN en los procesos de hibridación (Andrew et al., 2013). Para el caso de los estudios mexicanos, los estudios de hibridación y especiación responden a algunas de estas tendencias globales, como se detallará más adelante. Por ejemplo, varios grupos importantes de organismos se caracterizan por haber experimentado una divergencia reciente en México, lo que tiene consecuencias significativas para el reconocimiento y la delimitación de especies. En un trabajo reciente, Flores-Rentería et al. (2013) demostraron para un complejo de pinos piñoneros (Pinus discolor, P. johannis, P. culminicola y P. cembroides) que las secuencias completas del ADN de cloroplasto proveen una herramienta adecuada para la resolución de las relaciones filogenéticas en este tipo de casos problemáticos, particularmente cuando se usan en combinación con datos morfológicos y ecológicos, brindando evidencia robusta para contrastar hipótesis taxonómicas.
Por su parte, el proceso de hibridación ha sido claramente documentado en especies del género Quercus en México (Albarrán-Lara et al., 2010, Eaton et al., 2015, Peñaloza-Ramírez et al., 2010). En estos estudios se han identificado algunos patrones interesantes, como la hibridación simultánea entre más de 2 especies en la sierra Tarahumara (Peñaloza-Ramírez et al., 2010), así como el incremento en la asimetría fluctuante foliar en los híbridos en el volcán de Tequila, Jalisco (Albarrán-Lara et al., 2010). Mientras tanto, los estudios realizados en animales se han centrado en el papel de la hibridación en los procesos de diversificación, especiación y adaptación, particularmente en varios linajes de peces (Carson y Dowling, 2006, Culumber et al., 2011, Hulsey y García-de León, 2013, Jones et al., 2012, Strecker, 2006, Strecker et al., 2012).
Campos emergentes
La mayoría de las nuevas tendencias de estudio en Ecología Molecular propuestas por Andrew et al. (2013) tuvieron una representación baja entre los estudios que han sido realizados en México (en conjunto acumulan el 10.1% de los estudios). Estas áreas corresponden a: a) ecología trófica basada en secuencias de ADN; b) diversidad microbiana al interior de los organismos; c) genómica del paisaje; d) ecología genómica y adaptación molecular, y e) parentesco y conducta. Andrew et al. (2013) describen cada una de estas tendencias y plantean diferentes retos, de los cuales es fundamental resaltar el desarrollo de métodos precisos para identificar y cuantificar las especies usadas como recursos en las relaciones tróficas; desarrollar bancos de códigos de barras de la diversidad microbiana, librerías de transcriptomas y genomas para especies focales, y desarrollar métodos para diferenciar contaminantes ambientales de los organismos de interés; identificar loci asociados al ambiente, describir el papel de la introgresión sobre la estructura genética, desarrollar simulaciones que permitan explorar la importancia a nivel evolutivo de los procesos genéticos propios a nivel de paisaje; determinar las funciones ecológicas de los genes, determinar la base genómica de la relación fenotipo-ambiente y describir procesos de adaptación local.
Indudablemente estas áreas y preguntas de investigación, aunque están pobremente representadas en los estudios mexicanos, experimentarán un notable desarrollo durante los próximos años gracias al desarrollo tecnológico y reducción de costos en técnicas de secuenciación masiva. A continuación se mencionarán algunos estudios de las áreas descritas anteriormente en los que ya se han producido resultados importantes.
Ecología trófica basada en secuencias de ADN y diversidad microbiana al interior de los organismos
La diversidad microbiana al interior de organismos multicelulares es una de las áreas emergentes más fascinantes identificadas por Andrew et al. (2013). Recientemente, autores como Montoya, Bandala y Garay-Serrano (2015) describieron la asociación entre especies de micorrizas e individuos del género Alnus en bosques montanos de México a partir de monitoreos por un periodo de 4 años. Morales-Jiménez, Zúñiga, Ramírez-Saad y Hernández-Rodríguez (2012) utilizaron secuencias de los genes del ARN ribosomal 16S para analizar la composición de la comunidad bacteriana del tracto digestivo en el escarabajo descortezador Dendroctonus rhizophagous, una especie que ataca y mata plántulas e individuos juveniles de 11 especies de pinos en la Sierra Madre Oriental, lo que permitió identificar varias especies de bacterias con actividad celulolítica, rasgo que es clave en la historia de vida del insecto. Otro ejemplo con implicaciones prácticas importantes fue la utilización de secuencias de ADN para el análisis de la diversidad y prevalencia de parásitos hemosporidios en la sangre de varias especies de palomas de la isla Socorro, sitio en el que se planea reintroducir a la especie Zenaida graysoni, que era originaria de la isla pero se extinguió en estado silvestre (Carlson et al., 2013). Por lo tanto, este estudio permitirá tomar medidas para reducir la infección de estas aves una vez que sean liberadas.
Amato et al. (2013) utilizaron secuenciación de nueva generación para determinar la riqueza, diversidad y composición de la microbiota gastrointestinal de monos aulladores (Alouatta pigra) en un gradiente de calidad de hábitat, desde bosque tropical perennifolio hasta condiciones de cautiverio. Se encontró una correlación de la calidad del hábitat con la diversidad de especies de plantas consumidas por los individuos, y a su vez con la diversidad de la microbiota. Una comunidad microbiana menos diversa tiene también una menor capacidad digestiva, lo cual tiene un impacto en la salud del hospedero. Un estudio que podemos ubicar en la categoría de ecología trófica basada en secuencias de ADN es la utilización del código de barras de ADN para relacionar los adultos y las larvas de acantocéfalos parásitos que se encuentran en diferentes hospederos (aves y peces, respectivamente), que dio como resultado que los especies dulceacuícolas del centro de México se encuentran parasitadas por una sola especie de acantocéfalo, Polymorphus brevis (Alcántara-Escalera, García-Varela, Vázquez-Domínguez y Pérez-Ponce de León, 2013).
Genómica del paisaje, ecología genómica y adaptación molecular
Uno de los pocos estudios publicados a la fecha en el campo de la genómica del paisaje describió la variación en el tamaño del genoma en variedades de maíz criollo y poblaciones silvestres de teosinte a lo largo de un gradiente altitudinal (Díez et al., 2013). Se encontró que el tamaño del genoma fue menor en las variedades de maíz en comparación con el teosinte, así como una correlación negativa entre el tamaño del genoma y la altitud en el maíz, mientras que en el teosinte la correlación fue con la temperatura y la precipitación. En el caso de la ecología genómica y procesos de adaptación molecular, diferentes estudios han utilizado herramientas de secuenciación de última generación con el fin de describir los procesos poblacionales asociados a eventos de paralelismo o convergencia a nivel molecular en especies del género Astyanax en México (Bradic, Teotónio y Borowsky, 2013); de igual forma se han descrito procesos de selección local definidos por el balance entre la selección natural y el flujo génico (Pespeni y Palumbi, 2013).
Parentesco y conducta
Para el tema del estudio del parentesco y la conducta encontramos pocos estudios de caso en México que se hayan apoyado en el uso de marcadores moleculares. Entre los ejemplos disponibles destacan el análisis de la divergencia en los patrones de canto del colibrí formador de leks Campylopterus curvipennis (González y Ornelas, 2014), la cual se debe a las condiciones sociales locales y al aprendizaje de los individuos, aun cuando existe alto flujo génico entre leks y poblaciones. En otro caso, se analizó la estructura genética y el parentesco entre individuos de 8 grupos sociales en una población de mono aullador (Alouatta pigra) en Palenque, Chiapas (Belle, Estrada, Strier y Fiore, 2012).
Conclusiones y perspectivas
El panorama de la Ecología Molecular en México indica un desarrollo considerable de la disciplina en nuestro país. Los sistemas de estudio han sido diversos, aunque predominan los vertebrados y las plantas, mientras que otros grupos están extremadamente subrepresentados. Además de los sistemas de estudio tradicionales para la ecología, es importante resaltar líneas novedosas de investigación encaminadas a la descripción de procesos demográficos históricos en las poblaciones humanas de América (Raghavan et al., 2015) y de ancestría para las poblaciones humanas en México (Gravel et al., 2013, Johnson et al., 2011, Rangel-Villalobos et al., 2008). Estos estudios han aplicado los fundamentos y métodos propios de la Ecología Molecular con el fin de describir patrones genético-poblacionales, filogeográficos, de parentesco y conducta a partir de fragmentos de ADN mitocondrial (Figueiro, Hidalgo y Sans, 2011 González-Martín et al., 2015, Guardado-Estrada et al., 2009), microsatélites (Camacho-Mejorado et al., 2015, Quinto-Cortés et al., 2010) y a nivel genómico (Moreno-Estrada et al., 2014). Por otra parte, todas las áreas de la Ecología Molecular están presentes con trabajos en México, aunque existe una predominancia de la genética de poblaciones y la filogeografía. La mayoría de los estudios están basados en secuencias de fragmentos de ADN y microsatélites.
En general, nuestra revisión sugiere la necesidad de desarrollar trabajos en las áreas emergentes de la Ecología Molecular identificadas por Andrew et al. (2013), a la vez que se continúa con la caracterización de la impresionante diversidad biológica del país utilizando los enfoques más tradicionales. Por ejemplo, se puede esperar el rápido desarrollo de la genómica del paisaje, conforme la implementación de los métodos de secuenciación de nueva generación permitan una evaluación más precisa de hipótesis ecológicas y evolutivas (e.g., Bonilla-Rosso et al., 2012, Centeno et al., 2012, Rebollar et al., 2012).
Igualmente, el estudio de la diversidad microbiana al interior de los organismos pluricelulares, la ecología trófica, el parentesco y el comportamiento, y la filogenia de las comunidades, son temas que tienen un importante potencial de crecimiento considerando los pocos, pero conceptualmente novedosos, estudios que han sido publicados en estas áreas (e.g., Carlson et al., 2013, Morales-Jiménez et al., 2012, Nguyen et al., 2013, Ramírez-Puebla et al., 2010, Tapia-Torres et al., 2015, Tovar-Sánchez et al., 2013). Para lograr estos objetivos será necesario un esfuerzo de modernización de los centros de investigación del país, expandiendo sus capacidades técnicas y académicas y fortaleciendo el apoyo a los proyectos de investigación.











 text new page (beta)
text new page (beta)


