Introducción
El género Alouatta representa primates neotropicales con una amplia distribución desde el sureste de México hasta el norte de Argentina (Cortés-Ortiz, Rylands y Mittermeier, 2015). Este género contiene 9 especies: Alouatta palliata, Alouatta macconelli, Alouatta pigra, Alouatta seniculus, Alouatta arctoidea Alouatta sara, Alouatta caraya, Alouatta belzebul, y Alouatta guariba (Cortés-Ortiz et al., 2015). La especie A. palliata está constituida por 5 subespecies: A. p. palliata, A. p. mexicana, A. p. aequatoriales, A. p. coibensis y A. p. trabeata (Cortés-Ortiz et al., 2015).
En México, la subespecie A. palliata mexicana se distribuye en los estados de Veracruz, Tabasco, Chiapas y Oaxaca (Oropeza-Hernández y Rendón-Hernández, 2012) y es una de las subespecies que está catalogada por la Unión Internacional para la Conservación de la Naturaleza como críticamente amenazada (Cuarón, Shedden, Rodríguez-Luna, de Grammont y Link, 2008) y por la Norma Oficial Mexicana NOM-059 (Semarnat, 2010) como en peligro de extinción. A pesar del grado de vulnerabilidad de A. p. mexicana, existe escasa información sobre la diversidad genética en las poblaciones remanentes (Dunn et al., 2014). La información existente indica que esta subespecie presenta bajos niveles de variación, que pueden ser producto ya sea de un evento de cuello de botella o un efecto fundador; ambos relacionados con la historia de colonización y expansión de este primate en Centroamérica (Dunn et al., 2014; Ellsworth y Hoelzer, 2006).
La variación genética en diferentes poblaciones del género Alouatta se ha examinado utilizando diversos marcadores moleculares tales como isoenzimas, aloenzimas, microsatélites y secuencias de ADN mitocondrial (fig. 1). De estos marcadores, los microsatélites son los más utilizados principalmente en las especies A. palliata, A. caraya y A. pigra (fig. 1). Los resultados obtenidos de diversidad genética en diferentes especies de Alouatta indican que A. belzebul y A. seniculus son las especies con niveles altos de diversidad con distintos marcadores moleculares (tabla 1). La especie A. caraya tiende a mostrar niveles intermedios de diversidad genética (isoenzimas) aunque existen casos en los que se han documentado valores cercanos a los obtenidos para A. belzebul o A. seniculus, utilizando microsatélites y ADN mitocondrial (tabla 1). En cambio, A. pigra y A. palliata son las especies que presentan niveles bajos de diversidad genética medida a través de ADN mitocondrial, isoenzimas y microsatélites (tabla 1).
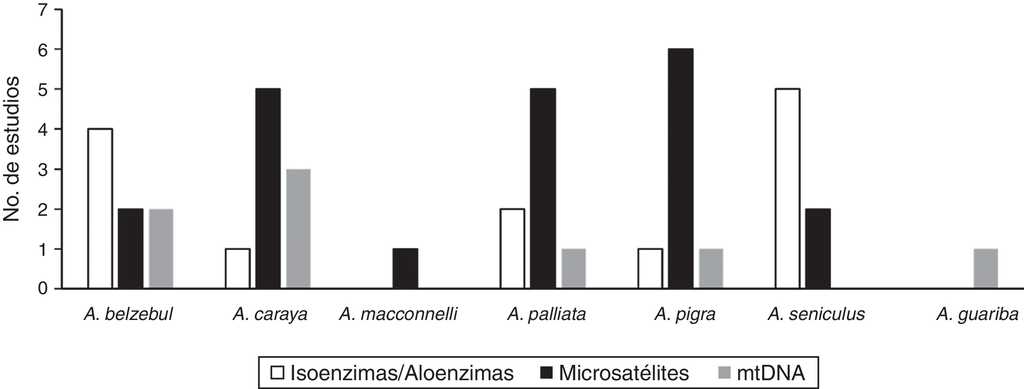
Figura 1 Marcadores moleculares utilizados en estudios que reportan variabilidad genética en diferentes especies de monos aulladores del género Alouatta. Referencias: A. belzebul (Schneider et al., 1991; Melo, Sampaio, Schneider y Schneider, 1992; Sampaio, Schneider y Schneider, 1996; Zaldívar et al., 2003; Gonçalves et al., 2004; Nascimento, Bonvicino, da Silva, Schneider y Seuanez, 2005; Nascimento, Bonvicino, de Oliveira, Schneider y Seuánez, 2008; Bastos, Gonçalves, Ferrari, Silva y Schneider, 2010). A. caraya (Szapkievich, Comas, Zunino y Mudry, 1998; Nascimento et al., 2005; Ascunce, Hasson, Mulligan y Mudry, 2007; Nascimento et al., 2007; Oklander, Zunino, Di Fiore y Corach, 2007; Ruiz-García et al., 2007; Oklander et al., 2010; Collevatti, Souza-Neto, Silva-Jr y Telles, 2013; Oklander y Corach, 2013). A. macconnelli (Ruiz-García et al., 2007). A. palliata (Malmgren y Brush, 1978; Zaldívar et al., 2003; Winkler et al., 2004; Ellsworth y Hoelzer, 2006; Ruiz-García et al., 2007; Milton et al., 2009; Cortés-Ortiz et al., 2010; Dunn et al., 2014). A. pigra (James, Leberg, Quattro y Vrijenhoek, 1997; Winkler et al., 2004; del Valle, Estrada, Espinoza, Lorenzo y Naranjo, 2005; Ellsworth y Hoelzer, 2006; Améndola-Pimenta, 2009; Cortés-Ortiz et al., 2010; Van Belle, Estrada, Strier y di Fiore, 2012). A. seniculus (Lima et al., 1990; Pope, 1992; Sampaio et al., 1996; Pope, 1998a; Zaldívar et al., 2003; Ellsworth y Hoelzer, 2006; Ruiz-García et al., 2007). A. guariba (Harris, Gifalli-Iughetti, Braga y Koiffmann, 2005).
Tabla 1 Diversidad genética estimada con diferentes marcadores moleculares en monos aulladores del género Alouatta.
| DNA mitocondrial (h) |
Isoenzimas/aloenzimas (H) |
Microsatélites (H) |
Referencias | |
|---|---|---|---|---|
| A. belzebul | 0.95 | 0.05-0.09 | 0.65-0.74 | Sampaio et al. (1996); Zaldívar et al. (2003); Gonçalves et al. (2004); Nascimento et al. (2008); Bastos et al. (2010) |
| A. seniculus | 0.096-0.110 | 0.64-0.65 | Lima et al. (1990); Zaldívar et al. (2003); Ellsworth y Hoelzer (2006); Ruiz-García et al. (2007) | |
| A. caraya | 0.877-0.946 | 0.04 | 0.54-0.60 | Szapkievich et al. (1998); Ascunce et al. (2007); Nascimento et al. (2007); Oklander et al. (2010); Collevatti et al. (2013) |
| A. pigra | 0.02 | 0.3-0.59 | James et al. (1997); del Valle et al. (2005); Van Belle et al. (2012) | |
| A. palliata | 0.486 | 0-0.01 | 0.16-0.56 | Zaldívar et al. (2003); Winkler et al. (2004); Ruiz-García et al. (2007); Milton et al. (2009); Dunn et al. (2014) |
h: diversidad haplotípica; H: heterocigocidad promedio.
Estas diferencias en los valores de diversidad genética entre las especies del género Alouatta se han atribuido a eventos relacionados con su historia biogeográfica (Cortés-Ortiz et al., 2003; Ellsworth y Hoelzer, 2006; Zaldívar et al., 2003), a los cambios en el tamaño de las poblaciones por huracanes o epidemias (fiebre amarilla), a la pérdida y fragmentación del hábitat (Oklander, Kowalewski y Corach, 2010; Pope, 1996; Ruiz-García et al., 2007), así como a diferencias en los patrones de dispersión debido a las condiciones ecológicas en las cuales se encuentran las poblaciones de aulladores (Nascimento, Bonvicino y Seuánez, 2007).
La pérdida y fragmentación del hábitat tienen consecuencias importantes en la diversidad genética de las poblaciones, ya que puede causar reducción de los tamaños poblacionales, aislamiento entre poblaciones y limitar la dispersión de los individuos entre parches; estos factores provocan una pérdida de diversidad genética, altos niveles de endogamia, y por consiguiente las poblaciones o especies corren el riesgo de extinción (Frankham, Ballou y Briscoe, 2010). Un caso particular es la región de Los Tuxtlas, Veracruz, en las selvas húmedas del sureste de México, ya que cerca del 80% de la vegetación original ha sido deforestada, principalmente por la conversión de selva a pastizales de ganadería (Ricker, López-Vega y Mendoza-Márquez, en prensa). Esta situación ha tenido un impacto negativo en las poblaciones de mono aullador de manto que habitan en la región, ya que se han reducido drásticamente las poblaciones y algunas permanecen aisladas en fragmentos de selva (Arroyo-Rodríguez y Mandujano, 2006; Estrada, Juan-Solano, Martínez y Coates-Estrada, 1999). Sin embargo, existe escasa información sobre aspectos genéticos de estas poblaciones.
Por lo tanto, este estudio tiene como objetivos estimar la diversidad genética en A. p. mexicana en la región de Los Tuxtlas, y comparar la diversidad genética en grupos de monos aulladores de manto que habitan en selva continua y fragmentos de selva. Lo anterior, con la finalidad de generar información sobre aspectos genéticos en monos aulladores que habitan en un paisaje fragmentado en dicha región. Para ello, se analizó la variación de microsatélites a partir de ADN extraído de muestras fecales de monos aulladores de manto que habitan en la Reserva de la Biosfera Los Tuxtlas, en Veracruz. Considerando la historia biogeográfica de la especie, se espera que A. p. mexicana en la región de los Tuxtlas presente una baja diversidad genética. Por otro lado, los grupos de monos aulladores en fragmentos de selva presentarán bajos niveles de diversidad y altos niveles de endogamia en comparación con los grupos de selva continua, como resultado de la pérdida y fragmentación de la selva.
Materiales y métodos
El presente estudio se llevó a cabo en un hábitat continuo y 3 fragmentos de selva que se encuentran en la Reserva de la Biosfera Los Tuxtlas al sureste de Veracruz (18°42’-18°15’ N, 95°17’-94°40’O). La reserva cubre una superficie total de 155,122.46 ha, divididas en una de zona de amortiguamiento (125,403 ha) y 3 zonas núcleo: volcán San Martín Pajapan (1,883 ha), volcán Santa Marta (18,031 ha) y volcán San Martín Tuxtla (9,805 ha) (Diario Oficial de la Federación, 1998). La vegetación dominante en la reserva es selva alta perennifolia, aunque en el caso de las áreas fragmentadas presentan un alto grado de perturbación encontrándose vegetación secundaria (Ibarra-Manríquez, Martínez-Ramos, Dirzo y Núñez-Farfán, 1997). El clima es cálido y húmedo con temperatura promedio anual de 24.1° C y precipitación promedio anual de 4,201 mm; marzo (92 mm) y abril (77 mm) son los meses más secos, y la época de lluvia comprende de junio a febrero (Gutiérrez-García y Ricker, 2011).
El hábitat continuo se localiza en la selva protegida de la Estación de Biología Tropical Los Tuxtlas del Instituto de Biología de la UNAM (18°34’-18°46’ N, 95° 04’-95°09’ O). Este sitio cubre una superficie total de 640 ha (fig. 2a) y forma parte del área de selva del volcán San Martín Tuxtla. En relación a los 3 fragmentos de selva, el primer fragmento (F1) tiene un área de 4 ha (18°36’N y 95°04’ O) y el segundo fragmento (F2) de 42 ha (18°37’N y 95°05’O), los cuales se ubican en los terrenos del ejido Balzapote (fig. 2a). El tercer fragmento (F3) de selva de 93 ha (18°38’N y 95°05’O) se ubica en el cerro del Borrego en el ejido de Monte Pío (fig. 2a). La distancia promedio entre fragmentos es de 2 km, y de los fragmentos con el hábitat continuo es de 3.7 km.
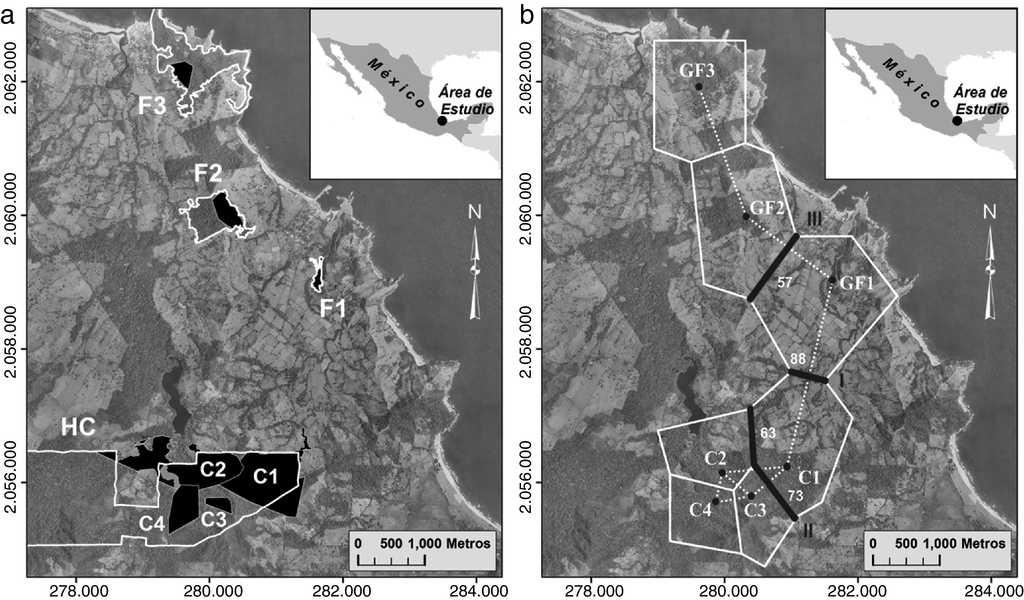
Figura 2 a) Mapa del sitio de estudio. Los polígonos de color negro muestran el ámbito hogareño de los grupos de monos aulladores de manto en el hábitat continuo (C1, C2, C3 y C4) y en los fragmentos de selva (F1, F2 y F3). F1=fragmento de 4ha. F2=fragmento de 42ha. F3=fragmento de 93ha. El hábitat continuo (HC) de la estación de Biología Tropical Los Tuxtlas y los tres fragmentos de selva se encuentran delimitados con las líneas blancas. b) Barreras genéticas detectadas en el programa Barrier v2.2. Las barreras se muestran con líneas negras y enumeradas de acuerdo al orden de importancia (I, II y III). Los valores a un lado de las barreras es el porcentaje de bootstrap que apoyan a cada segmento de la barrera. La línea punteada representa la triangulación de Delaunay y la línea sólida blanca indica la teselación de Voronoi.
Durante los periodos de marzo a octubre de 2009, abril a noviembre de 2010 y mayo de 2011 se recolectaron muestras fecales de individuos pertenecientes a 4 grupos de monos aulladores en hábitat continuo, de los cuales 2 fueron grupos vecinos (C3 y C4) con respecto a los 2 grupos principales de estudio (C1 y C2; fig. 2a). También se obtuvieron muestras fecales de 3 grupos en fragmentos de selva; cada grupo en un fragmento de tamaño diferente (GF1: grupo del fragmento de 4 ha, GF2: grupo del fragmento de 42 ha y, GF3: grupo del fragmento de 93 ha; fig. 2a). El tamaño y composición de los grupos se muestran en la tabla 2. Cada uno de los miembros del grupo se identificaron por edad (adultos, subadultos, juveniles e infantes) siguiendo la clasificación de Balcells y Baró (2009); por sexo (macho y hembra) en adultos ya que en infantes y juveniles no es posible detectarlo a simple vista, y por características físicas tales como, tamaño, manchas sin pigmento en piel y pelaje de diferentes partes del cuerpo (cola, manos y patas), y cicatrices faciales.
Tabla 2 Tamaño y composición de los grupos de monos aulladores de manto (A. palliata mexicana) en el hábitat continuo y en los fragmentos de selva de la Reserva de la Biosfera Los Tuxtlas. En paréntesis se indica el número de individuos que se genotiparon.
| Hábitat continuo | Hábitat fragmentado | ||||||
|---|---|---|---|---|---|---|---|
| C1 | C2 | C3a | C4a | GF1 | GF2 | GF3 | |
| MA | 5-6 (5) | 2-4 (3) | 3 (1) | 6 (2) | 3 (3) | 6-7 (3) | 5-6 (3) |
| HA | 2-3 (3) | 3-5 (3) | 4 (2) | 2 (2) | 3 (3) | 5-6 (3) | 5-6 (2) |
| SA | 0 | 0-2 (1) | 0 | 0 | 0-2 | 1-4 | 0-3 (2) |
| J | 0-1 (1) | 1-3 (2) | 3 | 1 | 2-4 (2) | 0-3 (3) | 2-3 (2) |
| I | 1-2 (2) | 2-4 (1) | 3 | 1 | 1-3 (2) | 1-2 (2) | 2-5 (3) |
| Total | 8-12 (11) | 10-16 (10) | 13 (3) | 10 (4) | 11-13 (10) | 16-19 (11) | 15-22 (12) |
C1 : grupo 1; C2 : grupo 2; C3 : grupo 3; C4 : grupo 4; GF1 : grupo del fragmento de 4 ha; GF2 : grupo del fragmento de 42 ha; GF3 : grupo del fragmento de 93 ha; HA : hembra adulta; I : infante; J : juvenil; MA : macho adulto; SA : subadulto.
a Grupos vecinos en el hábitat continuo.
En total se recolectaron 61 muestras fecales de individuos pertenecientes a los 7 grupos estudiados (tabla 2). El excremento se colectó fresco ya que se siguió a cada uno de los individuos, previamente identificados, hasta el momento de la defecación. Cada muestra se tomó con guantes y cucharas desechables nuevas para evitar cualquier contaminación con ADN humano o de las heces de otro mono. Las excretas se almacenaron inmediatamente en frascos de plástico esterilizados y se mantuvieron en una hielera mientras permanecían en campo con el fin de evitar la degradación del ADN por incrementos en la temperatura del ambiente (Nsubuga et al., 2004). Después del trabajo de campo, las muestras fueron procesadas siguiendo el método de conservación de «dos pasos» (two-step) de Roeder, Archer, Poiner y Morin (2004) ya que permite extraer una mayor cantidad de ADN y de mejor calidad para los análisis genéticos. A este método se le aplicaron las siguientes modificaciones: a cada muestra se le añadió etanol al 100% y se colocaron en un refrigerador (4° C) durante 24 a 26 h; posteriormente el etanol fue retirado, se le añadió sílica gel y se mantuvieron en refrigeración (4 °C) hasta el momento de extracción de ADN.
Extracción y amplificación del ADN
El ADN genómico de las heces de los aulladores fue extraído con el kit comercial QIAamp DNA Stool Mini Kit (Qiagen) siguiendo las instrucciones del protocolo de extracción del kit y las modificaciones sugeridas por Nsubuga et al. (2004). También se extrajo ADN de tejido hepático y músculo de una hembra adulta (A. palliata) con el kit de extracción DNeasy Blood & Tissue (Qiagen). Estas muestras se utilizaron como control positivo para las extracciones y amplificaciones de ADN. Los tejidos fueron proporcionados por la Colección Nacional de Mamíferos del Instituto de Biología, Universidad Nacional Autónoma de México. El ADN extraído de heces y tejido fue almacenado a una temperatura de -20 °C.
El número de microsatélites seleccionados fueron 13 por ser identificados como polimórficos para A. palliata, de los cuales 5 fueron caracterizados para A. palliata (Ap68 y Ap74, Ellsworth y Hoelzer, 1998; Apm01, Apm04 y Amp09, Cortés-Ortiz, Mondragón y Cabotage, 2010), 3 fueron descritos para humanos (D6S260, D8S165 y D14S51, Ellsworth y Hoelzer, 1998) y 5 fueron diseñados para A. belzebul (Ab04, Ab06, Ab20, Ab17 y Ab16, Gonçalves, Silva, Barbosa y Schneider, 2004).
El extremo 5’ del oligo «forward» de cada microsatélite se marcó con fluorescencia FAM (Ap74, Ab06, Ap68, D6S260, Ab16, Apm04, D8S165), HEX (Apm09, D14S51, Ab20 y Ab04) y NED (Apm01, Ab17). Los 13 microsatélites fueron amplificados con los reactivos del kit Multiplex PCR Kit (Qiagen). Cada PCR fue realizada en un volumen final de 15 μL, manteniendo las concentraciones de cada reactivo sugeridas por el protocolo de amplificación del Kit (PCR Multiplex con solución Q). Además, se agregó 1 μL de albúmina de suero de bovino para eliminar inhibidores (ej. sustancias de plantas, alcaloides, polisacáridos) de la TaqDNA polimerasa que puedan encontrarse en las heces de los monos aulladores.
Las condiciones de amplificación en el termociclador fueron las siguientes: desnaturalización inicial a 95 °C durante 15 min seguido de 40 ciclos de desnaturalización a 95 °C por 1 min, alineamiento (ver tabla 3 para la temperatura específica de cada locus) por 1 min y extensión a 72 °C por 1 min, y una extensión final a 72 °C por 30 min. Los productos de cada PCR fueron visualizados mediante electroforesis en un gel de agarosa al 2% para verificar la calidad y el tamaño de las muestras amplificadas así como para confirmar que no existiera contaminación en los controles negativos.
Tabla 3 Loci de microsatélites junto con su unidad repetida, temperatura de alineamiento (Ta) y el secuenciador en el cual fueron procesados (*).
| Locus | Secuencia repetida en tándem | Ta | Secuenciador ABI PRISM |
|||
|---|---|---|---|---|---|---|
| 310 | 3100 | 3500 | 3730 | |||
| Ap68 | (TG)17 | 50.2 °C | * | * | * | * |
| Ap74 | (TG)19 | 56 °C | * | * | * | |
| Apm01 | (TG)25 | 60 °C | * | * | ||
| Apm04 | (AC)21 | 62 °C | * | * | ||
| Apm09 | (CA)16 | 56 °C | * | * | ||
| Ab04 | (GA)2AA(GA)12AG | 60 °C | * | * | * | |
| Ab06 | (CT)9TTT(CT)11GTCTGTCTTAT(AC)16 | 55 °C | * | * | * | |
| Ab16 | (CAAA)11GAAGAAAGAAGGAAAGAA(GAAA)2 | 60 °C | * | * | ||
| Ab17 | (CAAA)2G(GAAA)2GAGAAAAA(GAAA)14 | 62 °C | * | * | ||
| Ab20 | (CAAA)16 | 63 °C | * | * | * | |
| D6S260 | (CA)23 | 55 °C | * | * | ||
| D8S165 | (AC)16 | 55 °C | * | * | * | |
| D14S51 | (GT)19 | 56 °C | * | * | * | |
Los genotipos determinados a partir del análisis de microsatélites con ADN de muestras fecales están propensos a asignarlos de manera incorrecta debido a errores ligados a la secuencia de ADN (alelos nulos) y errores estocásticos (ej. falso alelo y amplificación preferencial de un alelo: «allelic dropout»; Bonin et al., 2004; Pompanon, Bonin, Bellemain y Taberlet, 2005). Por lo tanto, para incrementar la confiabilidad en la asignación de genotipos, se repitieron las PCR en el 33% (promedio) de las muestras para cada locus (tabla 4), y de las cuales se hicieron 3 repeticiones para homocigotos y 2 para heterocigotos (Bayes, Smith, Alberts, Altmann y Bruford, 2000).
Tabla 4 Características de los 13 loci de microsatélites amplificados en 61 muestras de monos aulladores de manto (Alouatta palliata mexicana) de la Reserva de la Biosfera Los Tuxtlas, Veracruz.
| Locus | N | Muestras repetidas (%) |
Tamaño alelos (pb) |
N a | N e | H o | H e | EHW | ||
|---|---|---|---|---|---|---|---|---|---|---|
| x2 | df | P | ||||||||
| Ap68 | 59 | 34 | 191 | 1 | 1.00 | 0.00 | 0.00 | -- | -- | -- |
| Ap74 | 34 | 37 | 146-152 | 3 | 1.16 | 0.15 | 0.14 | 0.00 | 4 | 1.00 |
| Apm01 | 50 | 27 | 208-222 | 5 | 1.20 | 0.10 | 0.17 | 8.31 | 4 | 0.08 |
| Apm04 | 39 | 29 | 246-250 | 2 | 1.98 | 0.23 | 0.49 | 16.78 | 12 | 0.16 |
| Apm09 | 22 | 61 | 176 | 1 | 1.00 | 0.00 | 0.00 | -- | -- | -- |
| Ab04 | 52 | 38 | 181-183 | 2 | 1.08 | 0.08 | 0.07 | -- | -- | -- |
| Ab06 | 38 | 41 | 272-276 | 3 | 1.14 | 0.08 | 0.12 | -- | -- | -- |
| Ab16 | 55 | 36 | 170 | 1 | 1.00 | 0.00 | 0.00 | -- | -- | -- |
| Ab17 | 20 | 33 | 162 | 1 | 1.00 | 0.00 | 0.00 | -- | -- | -- |
| Ab20 | 52 | 17 | 237 | 1 | 1.00 | 0.00 | 0.00 | -- | -- | -- |
| D6S260 | 55 | 19 | 176-180 | 3 | 2.03 | 0.42 | 0.51 | 13.24 | 14 | 0.51 |
| D8S165 | 30 | 32 | 137-139 | 2 | 1.18 | 0.03 | 0.15 | 6.63 | 4 | 0.16 |
| D14S51 | 35 | 25 | 135-143 | 3 | 1.19 | 0.00 | 0.16 | 15.28 | 6 | 0.02 |
| Promedio (± EE)(todos los loci) | 41.62 (3.63) | 33 | 2.15 (0.34) | 1.23 (0.10) | 0.08(0.03) | 0.14 (0.05) | 60.24 | 48 | 0.11 | |
| Promedio (± EE)(loci variables) | 41.63 (3.66) | 2.88 (0.35) | 1.37 (0.14) | 0.14 (0.05) | 0.23 (0.06) | 60.24 | 48 | 0.11 | ||
EHW: equilibrio de Hardy-Weinberg; He: heterocigocidad esperada; Ho: heterocigocidad observada; N: número de muestras genotipificadas; Na: número de alelos; Ne: número efectivo de alelos;. -: parámetros no estimados por el programa GENEPOP.
Genotipificación
Los productos de PCR visualizados en el gel de agarosa fueron separados mediante electroforesis capilar utilizando 4 diferentes secuenciadores (tabla 3). El uso de diferentes equipos se debió a que las muestras fueron analizadas en diferentes periodos de tiempo, y a la disponibilidad de estos en el laboratorio. Se utilizó el secuenciador ABI PRISM 3730 junto con GeneScan 500 ROX como marcador de peso estándar, y los secuenciadores ABI PRISM 310, 3500 y 3100 con GeneScan 350 ROX como marcador de peso estándar. Considerando que las muestras se procesaron en diferentes equipos, se utilizó el programa Allelogram v2.2. (Morin, Manaster, Mesnick y Holland, 2009) para identificar las diferencias en el número de pares de bases de los alelos resultantes y normalizar los alelos diferentes. Para normalizar los alelos se utilizaron muestras control, que en este caso fueron aquellas que se procesaron en los diferentes secuenciadores.
Análisis genéticos
El programa GENEPOP v4.2 (Rousset, 2008) se usó para detectar aquellos loci que podrían presentar desviaciones del Equilibrio Hardy-Weinberg y evidencia de desequilibrio de ligamiento (DL). Este análisis se realizó a través del método exacto de Fisher y usando los parámetros preconfigurados de GENEPOP para el protocolo de la cadena de Markov (1,000 dememorizaciones, 100 lotes y 1,000 iteraciones por lote). El nivel de significancia (α) fue ajustado mediante la corrección de Bonferroni para comparaciones múltiples (p = 0.05/k, donde k es el número de grupos). El programa MICRO-CHEKER v2.2.3 (Van Oosterhout, Hutchinson, Wills y Shipley, 2004) se utilizó para detectar errores debido al «tartamudeo» de la polimerasa (stutter), a la amplificación preferencial de un alelo (allelic dropout) y alelos nulos. Además, se utilizó para determinar la frecuencia de alelos nulos en cada uno de los loci. El programa GenAlEx v6.5 (Peakall y Smouse, 2012) se usó para estimar el número de alelos (Na), número efectivo de alelos (Ne), heterocigocidad observada (Ho) y heterocigocidad esperada (He) para cada locus.
Con el objetivo de comparar la diversidad genética entre los grupos de monos aulladores y entre tipos de hábitat (continuo vs. fragmentado), se estimaron diferentes parámetros de diversidad genética por grupo y por tipo de hábitat. Para lo cual, 5 loci polimórficos fueron seleccionados (Apm01, Apm04, Ab04, Ab06 y D6S260) por presentar un mayor número de muestras amplificadas (tabla 4) y menos del 30% de datos faltantes por locus (promedio: 14.8% de datos faltantes por locus). También se seleccionaron aquellas muestras (50) que presentaron datos genotípicos en más de 2 loci de los 5 loci polimórficos.
Para cada grupo de monos aulladores y tipo de hábitat se estimó la riqueza alélica (Ar: número promedio de alelos por locus, corregido por tamaño de muestra) utilizando el método de rarefacción con el programa Fstat v2.9.3.2 (Goudet, 1995). También se determinó el número promedio de alelos por locus (A), He y Ho con el programa GenAlEx v6.5 (Peakall y Smouse, 2012) y el coeficiente de endogamia (Fis) en el programa Genetix v4.05 (Belkhir, Borsa, Goudet y Bonhomme, 2004). Las pruebas Kruskal-Wallis y U Mann-Whitney (Zar, 2010) se utilizaron para determinar diferencias en los parámetros de diversidad genética entre grupos y tipo de hábitat, respectivamente. Estos análisis estadísticos se realizaron en el programa IBM SPSS Statistics 22.
Para determinar si las poblaciones de monos aulladores han experimentado una reciente reducción en su tamaño efectivo poblacional (cuello de botella) se utilizó el programa BOTTLENECK v1.2.02 (Piry, Luikart y Cornuet, 1999). Este programa compara la He de la población con la heterocigocidad esperada en equilibrio de mutación-deriva génica (Heq) para cada locus, y determina si las poblaciones presentan un exceso de heterocigocidad (He > Heq) (Cornuet y Luikart, 1996). La prueba de Wilcoxon fue utilizada, debido al pequeño número de loci (5), para determinar si la población presenta un exceso de heterocigotos bajo el modelo de mutaciones de dos fases (TPM, por sus siglas en inglés; Di Rienzo et al., 1994) con 95% de SMM, varianza de 12% (Piry et al., 1999) y 10,000 repeticiones.
El nivel de diferenciación genética se determinó entre grupos y entre tipos de hábitat, estimando los valores de Fst a partir del análisis de varianza molecular (AMOVA, por sus siglas en inglés). Este análisis se realizó en el programa GenAlEx v6.5 (Peakall y Smouse, 2012) activando la opción de interpolar datos faltantes. La significancia estadística de las pruebas se estimó a partir de 9,999 permutaciones y se aplicó la corrección Bonferroni para comparaciones múltiples.
El programa Barrier v2.2 (Manni, Guerard y Heyer, 2004) se utilizó para identificar la posible presencia de barreras genéticas entre los grupos. Para este análisis se utilizó una matriz de valores de Fst y la ubicación geográfica de cada grupo determinada por coordenadas geográficas. La confiabilidad de cada barrera se determinó a partir de 100 réplicas bootstrap de la matriz de Fst, utilizando el paquete diveRsity (Keenan, McGinnity, Cross, Crozier y Prodöhl, 2013). La robustez de cada barrera es proporcional al número de veces que la barrera es apoyada por las réplicas bootstrap.
Resultados
El análisis de 13 loci de microsatélites indicó que 5 loci fueron monomórficos y 8 polimórficos (tabla 4). No se encontró evidencia significativa de desequilibrio de Hardy-Weinberg (p > 0.05) en ninguno de los loci variables, incluyendo el locus D14S51, después de aplicar la corrección Bonferroni (α = 0.007; tabla 4). En ninguno de los loci se encontró evidencia de DL (p > 0.05). La frecuencia de alelos nulos presentó un valor de 0.2 para los loci Apm04 y D14S51, y 0.1 para Apm01. Dakin y Avise (2004) sugieren que la presencia de alelos nulos con una frecuencia menor o igual a 0.2 no causa algún problema en los análisis genéticos. Por tal razón, dichos loci no se descartaron de los análisis subsecuentes.
El análisis de los 8 loci polimórficos indicó que el promedio de Ho es de 0.14 y de He es de 0.23. El valor de A fue de 2.88 y el promedio de Ne fue de 1.37 (tabla 4). El análisis de diversidad por tipo de hábitat indicó que los monos aulladores en hábitat continuo presentaron valores ligeramente superiores en A (2.40), Ar (2.23), Ho (0.19) y He (0.28), comparado con los grupos en el hábitat fragmentado para cada uno de los parámetros de diversidad (A = 2.00; Ar = 1.92; Ho = 0.15; He = 0.24; fig. 3). El análisis por grupo indicó que el grupo GF2 presentó valores inferiores en todos los parámetros de diversidad genética (A = 1.60; Ar = 1.37; Ho = 0.06; He = 0.19) mientras que el grupo C4 presenta valores superiores en Ar (1.58) y Ho (0.25) y el C2 en A (2.00) y He (0.28) (fig. 3). Sin embargo, no se encontraron diferencias significativas en los parámetros de diversidad genética entre hábitats (U Mann Whitney; A: U = 8.5 p = 0.42; Ar: U = 10 p = 0.69; Ho: U = 11 p = 0.84; He: U = 10 p = 0.59) ni entre grupos (Kruskal Wallis; A: H = 1.90 p = 0.93; Ar: H = 1.03 p = 0.99; Ho: H = 3.59 p = 0.73; He: H = 1.18 p = 0.98).
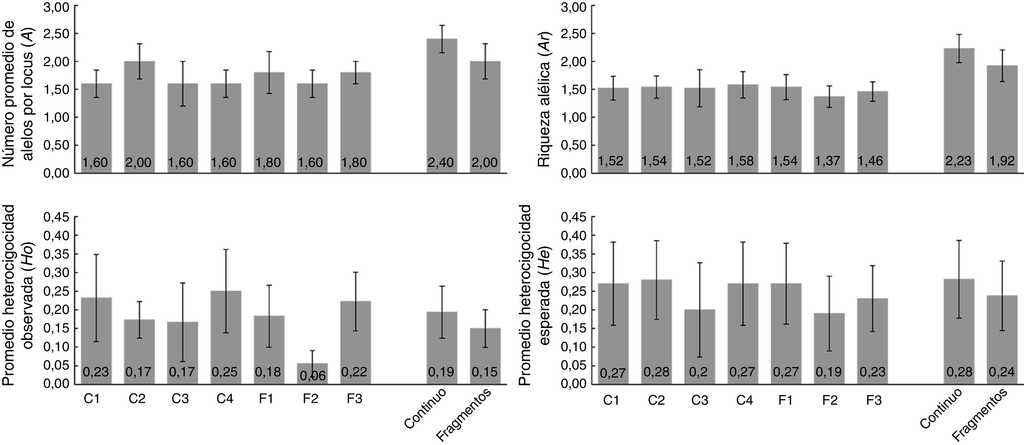
Figura 3 Diversidad genética por grupo de monos aulladores de manto (A. palliata mexicana) y por tipo de hábitat. El número de individuos y loci utilizados para estimar cada uno de los parámetros de diversidad se muestran en la tabla5. Los números dentro de las barras representan los valores promedio. Las líneas en la parte superior de las barras indican el error estándar.
Los valores de FIS resultaron positivos para todos los grupos de aulladores y por tipo de hábitat. FIS fue significativamente diferente de cero para los grupos C2 y GF2, y para el hábitat continuo y fragmentado (tabla 5). El análisis de cuello de botella indicó que no hay un exceso significativo de heterocigocidad (tabla 5) bajo el modelo mutacional TPM en ninguno de los grupos ni en los tipo de hábitat.
Tabla 5 Índice de endogamia y valores de p obtenidos en la prueba de Wilcoxon del modelo mutacional de dos fases (TPM) calculado a través del programa Bottleneck.
| N | Loci | FIS | IC 95% | Valores de p para TPM | |
|---|---|---|---|---|---|
| Grupos | |||||
| C1 | 8 | 5 | 0.24 | -0.50 a 0.61 | 0.06 |
| C2 | 10 | 5 | 0.42 | 0.02 a 0.71 | 0.56 |
| C3 | 3 | 5 | 0.38 | -1.00 a 0.50 | 0.88 |
| C4 | 4 | 5 | 0.25 | -0.77 a 1.00 | 0.13 |
| GF1 | 7 | 5 | 0.39 | -0.27 a 0.79 | 0.19 |
| GF2 | 9 | 5 | 0.75 | 0.18 a 1.00 | 0.81 |
| GF3 | 9 | 5 | 0.18 | -0.57 a 0.59 | 0.84 |
| Hábitat | |||||
| Continuo | 25 | 5 | 0.33 | 0.08 a 0.53 | 0.89 |
| Fragmentado | 25 | 5 | 0.39 | 0.06 a 0.66 | 0.84 |
C1 : grupo 1 del hábitat continuo; C2 : grupo 2 del hábitat continuo; C3 : grupo 3 del hábitat continuo; C4 : grupo 4 del hábitat continuo; FIS: índice de endogamia; GF1 : grupo del fragmento de 4 ha; GF2 : grupo del fragmento de 42 ha; GF3 : grupo del fragmento de 93; IC : intervalo de confianza para FIS; N: número de muestras.
El análisis de AMOVA mostró que no existe una diferenciación genética entre los grupos estudiados (Fst = 0.01, p = 0.33). Los valores de Fst entre grupos variaron desde -0.098 (GF2 vs. GF3) a 0.135 (GF1 vs. C1) (tabla 6). Ningún valor de p, entre comparaciones pareadas, fue significativo después de aplicar la corrección Bonferroni (p ≤ 0.002). En cambio, cuando se realizó el análisis por tipo de hábitat, AMOVA proporcionó un valor de Fst de 0.043 y significativo (p = 0.01) lo cual indica una baja diferenciación genética entre el hábitat continuo y fragmentado. El análisis de Barrier detectó la existencia de 3 posibles barreras genéticas (fig. 2b), de las cuales la primera barrera (I) es apoyada con 88% de los bootstrap, mientras que la segunda (II) y la tercera barrera (III) son apoyadas con menos del 73% de los bootstrap. La barrera I separa a los grupos del hábitat continuo (C1, C2, C3 y C4) del fragmentado (GF1, GF2 y GF3) (fig. 2b).
Tabla 6 Valores de Fst (debajo la diagonal) para cada comparación pareada entre los grupos de monos aulladores.
| C1 | C2 | C3 | C4 | GF1 | GF2 | GF3 | |
|---|---|---|---|---|---|---|---|
| C1 | - | 0.245 | 0.083 | 0.397 | 0.012 | 0.059 | 0.019 |
| C2 | 0.017 | - | 0.388 | 0.398 | 0.385 | 0.377 | 0.386 |
| C3 | 0.109 | -0.051 | - | 0.423 | 0.412 | 0.068 | 0.051 |
| C4 | -0.020 | -0.085 | -0.018 | - | 0.401 | 0.394 | 0.412 |
| GF1 | 0.135 | 0.000 | -0.011 | -0.038 | - | 0.244 | 0.410 |
| GF2 | 0.075 | -0.036 | 0.115 | -0.063 | 0.019 | - | 0.394 |
| GF3 | 0.097 | -0.029 | 0.104 | -0.043 | -0.040 | -0.098 | - |
Los valores de Fst (debajo de la diagonal) y valores de p (arriba de la diagonal) se estimaron a partir de 9,999 permutaciones implementado en GenAlEx v6.5.
Discusión
Diversidad genética en A. palliata mexicana
Los resultados de este estudio sugieren que los grupos de A. p. mexicana estudiados en la Reserva de la Biosfera Los Tuxtlas, presentan una baja diversidad genética comparado con lo reportado en otros estudios con microsatélites para la misma especie. En este estudio se obtuvo en promedio 2.88 alelos por locus, una heterocigocidad observada de 0.14 y una heterocigocidad esperada de 0.23 considerando 8 loci polimórficos. En cambio, en otros trabajos para monos aulladores de manto (A. palliata) se ha reportado una diversidad alélica de 3 (Winkler et al., 2004), 4.7 (Ruiz-García et al., 2007), 5.7 (Milton, Lozier y Lacey, 2009) y hasta de 6.3 (Ellsworth y Hoelzer, 2006) alelos por locus, analizando 5, 9, 13, y 8 microsatélites, respectivamente. En el caso de la heterocigocidad esperada, se reportan valores de 0.37 (Ellsworth y Hoelzer, 2006), 0.45 (Ruiz-García et al., 2007) y 0.58 (Milton et al., 2009), y para la heterocigocidad promedio observada de 0.16 (Winkler et al., 2004), 0.35 (Ellsworth y Hoelzer, 2006) y 0.56 (Milton et al., 2009).
La baja diversidad genética encontrada en los grupos de monos aulladores de manto en comparación con los estudios mencionados anteriormente, podría ser un reflejo de la historia de colonización y expansión en Centroamérica de esta subespecie (Dunn et al., 2014; Ellsworth y Hoelzer, 2006). Por lo anterior, es de esperarse que las poblaciones de monos aulladores de manto que se distribuyen en Norteamérica presenten una menor diversidad genética que aquellos que se encuentren en Centroamérica (Ellsworth y Hoelzer, 2006). Este supuesto coincide con lo reportado en este trabajo, ya que se analizaron muestras de monos aulladores de una localidad de México y se obtuvieron valores de diversidad genética menores en comparación con los estudios que analizaron muestras de Centroamérica (Ellsworth y Hoelzer, 2006; Milton et al., 2009; Ruiz-García et al., 2007; Winkler et al., 2004). Adicionalmente, dicho supuesto se corroboró al realizar un análisis de correlación entre los patrones de diversidad (Ho, He, A, Ar) y latitud, considerando los estudios realizados en Centroamérica (Ellsworth y Hoelzer, 2006; Milton et al., 2009; Winkler et al., 2004) y México (Cortés-Ortiz et al., 2010; Ellsworth y Hoelzer, 2006 y el presente estudio). Los resultados indican una relación negativa entre diversidad y latitud. Es decir, que a medida que aumenta la latitud (México) hay una disminución del número promedio de alelos (Rho de Sperman = -0.971, n = 6, p= 0.001), heterocigocidad observada (Rho de Sperman = -0.883, n = 6, p = 0.002) y heterocigocidad esperada (Rho de Sperman = -0.949, n = 5, p = 0.05). Este patrón de baja diversidad es consistente con lo reportado en trabajos que consideran muestras de poblaciones mexicanas de monos aulladores (A. p mexicana) utilizando microsatélites (Ellsworth y Hoelzer, 2006; Cortés-Ortiz et al., 2010) y ADN mitocondrial (Dunn et al., 2014).
Diversidad genética y fragmentación del hábitat
Se conoce que la pérdida de diversidad genética y aumento de endogamia resultan de la fragmentación del hábitat (Frankham et al., 2010). Los resultados de este estudio no apoyan de manera contundente la existencia de una pérdida de diversidad genética en los grupos de monos aulladores estudiados. No se encontraron diferencias significativas en los diferentes parámetros de diversidad genética (Ho, He, A, Ar) entre grupos del hábitat continuo y fragmentos de selva ni entre tipos de hábitats (continuo vs. fragmentado). Estos resultados son similares a lo documentado para el títi plateado (Mico argentatus) y mono araña muriqui (Brachyteles arachnoides), en los cuales no se detectaron variaciones significativas en los niveles de diversidad genética reportados en los grupos estudiados en fragmentos de selva (Gonçalves et al., 2009; Pope, 1998b). Los resultados obtenidos en este estudio podrían sugerir que la baja diversidad se mantiene en los grupos a pesar de que el hábitat de los monos aulladores ha sido fragmentado. Sin embargo, es importante confirmar esta información incrementando el tamaño de muestra y número de microsatélites ya que al realizar el análisis por tipo de hábitat, se puede identificar una ligera tendencia de que en el hábitat fragmentado parecen tener en promedio menos alelos por locus (A), riqueza alélica (Ar), He y Ho, en comparación con el hábitat continuo. Estos resultados podrían ser un indicio de variaciones en la diversidad genética entre diferentes condiciones ecológicas. Diferencias en los patrones de diversidad entre sitios continuos y fragmentos de selva se ha reportado en otras especies de primates no humanos, en los cuales han encontrado una disminución en riqueza alélica (Holmes et al., 2013), número de alelos por locus, y heterocigocidad observada (Grativol, Ballou y Fleischer, 2001) en sitios fragmentados.
El análisis de cuello de botella realizado en este estudio indicó que los monos aulladores no han experimentado una reducción reciente o fuerte en el tamaño efectivo poblacional. Existe evidencia sobre la reducción en el tamaño de las poblaciones (cuello de botella) de lémures ratón (Microcebus ravelobensis, Microcebus bongolavensis y Microcebus danfossi; Olivieri, Sousa, Chikhi y Radespiel, 2008) y tamarinos (Saguinus bicolor, Farias, Santos, Gordo y Hrbek, 2015) atribuido a la fragmentación del hábitat. El número limitado de loci considerados en este estudio podría estar reduciendo la detección de un cuello de botella en la población (Cornuet y Luikart, 1996).
Endogamia y diferenciación genética
El índice de endogamia (Fis) obtenido en este estudio revela un exceso de homocigotos tanto en los grupos del hábitat continuo como en los fragmentos de selva. Resultados similares fueron reportados por Gonçalves et al. (2003) quienes encontraron valores positivos de Fis en grupos de monos tití plateado (Mico argentatus) en selva fragmentada y sugieren la presencia de endogamia. Los valores de Fis de este estudio podrían indicar cierto nivel de endogamia en los grupos de monos aulladores estudiados. Sin embargo, el efecto Wahlund podría también estar influyendo en los resultados obtenidos de Fis.
Los valores de Fst revelan una baja diferenciación genética (Fst = 0.01) entre el hábitat continuo y fragmentado. Estos resultados difieren a lo reportado en una especie hermana de mono aullador (A. caraya), para la cual se documentó una diferenciación genética mayor (Fst = 0.10) entre selva continua y fragmentada (Oklander et al., 2010). Patrones similares se han reportado en otras especies de primates como Leontopithecus rosalia (Grativol et al., 2001) y Lepilemur edwardsi (Craul et al., 2009), en las cuales también se encontró una alta diferenciación genética entre sitios continuos y fragmentados.
La baja diferenciación genética entre los tipos de hábitats de este estudio sugiere que podría existir flujo genético entre las diferentes condiciones ecológicas. Sin embargo, este flujo podría ser afectado debido a la barrera detectada que separa a los grupos del hábitat continuo de los fragmentos de selva. En particular, esta barrera se ubica entre los grupos C1 y el GF1, y en una zona ausente de vegetación por la presencia de potreros y cultivos. Esta barrera podría limitar la dispersión de los individuos entre sitios. Existe evidencia de que en zonas perturbadas, la tasa de dispersión puede ser afectada teniendo como consecuencia una reducción en el flujo génico entre los grupos (Améndola-Pimenta, 2009; Oklander y Corach, 2013; Oklander et al., 2010).











 nova página do texto(beta)
nova página do texto(beta)


