Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista mexicana de biodiversidad
versión On-line ISSN 2007-8706versión impresa ISSN 1870-3453
Rev. Mex. Biodiv. vol.85 no.1 México mar. 2014
https://doi.org/10.7550/rmb.36116
Ecología
Diversidad genética y abundancia relativa de Didelphis marsupialis y Didelphis virginiana en Chiapas, México
Genetic diversity and relative abundance of Didelphis marsupialis and Didelphis virginiana in Chiapas, Mexico
Bárbara Cruz-Salazar1*, Lorena Ruiz-Montoya1, Darío Navarrete-Gutiérrez1, Eduardo E. Espinoza-Medinilla2, Ella Vázquez-Domínguez3 y Luis Bernardo Vázquez1
1 El Colegio de La Frontera Sur, Unidad San Cristóbal de las Casas. Carretera Panamericana s/n. Barrio de María Auxiliadora, 29200 San Cristóbal de Las Casas, Chiapas, México. * bcruz@ecosur.edu.mx.
2 Investigación y Posgrado, Universidad de Ciencias y Artes de Chiapas. 1a. Sur Poniente Núm. 1460, 29290 Tuxtla Gutiérrez, Chiapas, México.
3 Departamento Ecología de la Biodiversidad, Instituto de Ecología, Universidad Nacional Autónoma de México Ciudad Universitaria. Apartado postal 70-275, 04510 México D. F., México.
Recibido: 27 febrero 2013
Aceptado: 24 septiembre 2013
Resumen
En especies de amplia distribución, como Didelphis marsupialis y Didelphis virginiana, se predicen tamaños poblacionales grandes, alta diversidad y poca diferenciación genética. En este estudio se estimó la abundancia relativa de D. marsupialis y D. virginiana mediante la captura de organismos en la región de Los Altos y la Depresión Central de Chiapas. Se estimó también la diversidad y estructura genética a partir de 7 loci microsatélites. La abundancia relativa fue baja, entre 0.008-0.06 individuos trampas noches y la mayor incidencia de tlacuaches se observó en la Depresión Central. La proporción de sexos se ajustó significativamente a la relación 1:1 en ambas especies. La mayor diversidad genética se registró en la Depresión Central en D. virginiana (He= 0.58) con altos niveles de endogamia. Se observó diferenciación genética moderada entre las especies (Frt= 0.13) y mínima entre poblaciones de cada especie (Fsr= 0.01). A pesar de que los valores de diversidad genética fueron moderados, D. marsupialis y D. virginiana tienen abundancias relativas bajas con altos niveles de endogamia, lo que puede tener consecuencias negativas a futuro sobre la variabilidad genética de estas especies en las poblaciones estudiadas.
Palabras clave: tlacuaches, estructura poblacional, microsatélites, loci, heterocigosidad, variación genética, estructura genética.
Abstract
In wide distribution species, as D. marsupialis and D. virginiana, large population sizes, high diversity and little genetic differentiation are predicted. In this study, the relative abundance of D. marsupialis and D. virginiana by capturing organisms in the Highlands and the Central Depression regions, in Chiapas, was estimated. Also the diversity and structure genetic from 7 microsatellite loci were estimated. The relative abundance was low (between 0.008-0.06 individuals traps nights) and the highest incidence of opossums was observed in the Central Depression. The sex ratio was significantly adjusted to the relation 1:1 in both species. The greater genetic diversity was recorded in the Central Depression in D. virginiana (He= 0.58), with high levels of inbreeding. Genetic differentiation moderate was observed between species (Frt= 0.13) and minimum among populations of each species (Fsr= 0.01). Although the values of genetic diversity were moderate, D. marsupialis and D. virginiana have low abundances with high levels of inbreeding, which may have negative consequences in the future on the genetic variability of these species in the studied populations.
Key words: opossums, population structure, microsatellites, loci, heterozygosity, genetic variation, genetic structure.
Introducción
Las poblaciones presentan variación en tamaño, densidad y distribución en escalas tanto temporales como espaciales (Maurer, 2000; Hey y Machado, 2003). A escala espacial local los patrones de distribución están determinados principalmente por competencia, depredación, disponibilidad de recursos alimenticios y de refugios, mientras que a escala regional también intervienen la migración, dispersión y extinción (Steiner y Catzeflis, 2004; Stropp et al., 2009). Los cambios en tamaño y distribución de las poblaciones influyen, a su vez, en los patrones y niveles de diversidad genética (Vázquez-Domínguez et al., 2012).
Los marsupiales son mamíferos considerados ancestrales, ya que en el Cretácico superior y principios del Cenozoico tuvieron una notable radiación adaptativa, sobre todo en cuanto a hábitos alimenticios y características morfológicas en Australia y parte del continente Americano, dando lugar a los mamíferos placentarios (Clemens, 1968). En México se distribuyen 8 especies de marsupiales, dentro del orden Didelphimorphia, en 7 géneros (Marmosa, Tlacuatzin, Caluromys, Chironectes, Didelphis, Metachirus y Philander) (Medina-Romero et al., 2012). De ellos, el género Didelphis es uno de los más ampliamente distribuidos (Sunquist et al., 1987), con 2 especies: D. virginiana y D. marsupialis (Ringier, 1961; Gardner, 1973; Sunquist et al., 1987). Estas especies son consideradas generalistas (Cabello, 2006), oportunistas y exitosas, capaces de habitar distintos ambientes, incluso con incidencia de perturbaciones antropogénicas (Sunquist et al., 1987; Adler et al., 1997). Los individuos son capaces de desplazarse a grandes distancias en poco tiempo y pueden cambiar de hábitos alimenticios con relativa facilidad, lo que probablemente ha ayudado a que las especies ocupen grandes áreas en su distribución geográfica (Ringier, 1961; Gardner, 1973; Sunquist et al., 1987). Las hembras no dan atención individual a las crías y tienen 2 camadas por año hasta con 13 crías por camada (MacManus, 1970, 1974).
Los patrones de distribución, hábitos alimenticios, alta capacidad de reproducción y resistencia o flexibilidad a perturbaciones antropogénicas de D. marsupialis y D. virginiana, permiten suponer que son especies cuyas poblaciones son relativamente grandes y con poca diferenciación genética. Sin embargo, no se ha estudiado su diversidad genética ni su dinámica demográfica en México. Se conocen aspectos generales, su taxonomía, comportamiento y hábitos alimenticios y relaciones filogenéticas (Ceballos y Oliva, 2005; Medina-Romero et al., 2012). En otros países se han estudiado aspectos poblacionales (Tyndale-Biscoe y MacKenzie, 1976; Holmes, 1991; Adler et al., 1997; Gehrt et al., 1997; Cáceres y Monteiro-Filho, 1998; Kelly y Caro, 2003; Cabello, 2006), influencia del paisaje y el efecto de variables microclimáticas en su supervivencia (MacManus, 1971; Fleck y Harder, 1995; Gehrt et al., 1997; Orjuela y Jiménez, 2004; Kanda et al., 2005); la relación de la estructura del hábitat, abundancia de recursos y áreas urbanas con la abundancia (Lambert et al., 2005; Markovchick-Nicholls et al., 2007) y sobre filogeografía de 5 especies de la Región de Guayana (Steiner y Catzeflis, 2004).
En el presente estudio se exploró el tamaño poblacional de D. marsupialis y D. virginiana en México, así como la diversidad y estructura genética. Los objetivos específicos fueron: i) estimar la abundancia relativa, proporción de sexos, peso y talla de los individuos de las 2 especies en las poblaciones de Los Altos y la Depresión Central de Chiapas y ii) determinar la diversidad genética y el grado de diferenciación genética intra e interespecífica.
Materiales y métodos
Área de estudio. El estudio se realizó en 2 regiones de Chiapas, en Los Altos y la Depresión Central. Se eligieron estas 2 regiones dado que ambas especies son relativamente abundantes (Naranjo et al., 2005). Estas 2 regiones son espacialmente contiguas y representan una superficie geográfica amplia (Rangel-Salazar et al., 2005; Rodiles-Hernández, 2005), por lo que permiten incluir un intervalo adecuado de variación en parámetros climáticos, precipitación, temperatura, humedad, suelo; de asociaciones vegetales y áreas de cultivo activas o abandonadas (González-Espinosa et al., 2005, 2006). Lo anterior es importante porque dichas variaciones ambientales pueden determinar la estructura poblacional o demográfica de Didelphis (Adler et al., 1997; Cáceres y Monteiro-Filho, 1998), lo que a su vez influye en la diversidad genética. De esta manera, se incrementó la probabilidad de tener poblaciones grandes y con una distribución geográfica amplia de Didelphis.
En la Depresión Central de Chiapas se ubicó un sitio de muestreo en el cerro Coquelexquitzán (CC), al sureste del municipio de Tuxtla Gutiérrez (Cuadro 1). La vegetación predominante es selva baja caducifolia, con temperatura media anual de 20 a 29º C, con una estación de secas, noviembre-mayo, y otra de lluvias, junio-octubre, con una precipitación media anual de 922 mm.
En Los Altos de Chiapas se ubicaron 5 sitios de muestreo: i) Reserva Biológica Cerro Huitepec (HU); ii) Amatenango del Valle (AV); iii) Villa de las Rosas (VR), Corral de Piedra (CP) y iv) Reserva Biológica Moxviquil (MO) (Cuadro 1). La vegetación que predomina en los sitos son asociaciones de pino-encino, en diferentes estados sucesionales. El bosque más viejo se encuentra en HU (> 40 años) (Ramírez-Marcial et al., 2001). En MO son bosques que desde hace 15 años dejaron de ser explotados intensivamente, desde entonces están en un proceso de regeneración natural, aunque en algunas zonas persiste la extracción de leña (González et al., 2012). VR, CP y AV son sitios con bosques perturbados por la extracción de leña y productos no maderables, y también hay áreas que han sido convertidas en cultivos y pequeños asentamientos humanos (B. Cruz-Salazar, obs. pers.). Se consideró la diferencia en el número de sitios de muestreo en cada región para el análisis e interpretación de resultados obtenidos.
Estimación de características poblacionales. En cada sitio se ubicaron 2 puntos de muestreo y en cada uno de éstos se colocaron 24 trampas Tomahawk cebadas con sardina, dispuestas de manera radial y separadas por 20 m; durante 4 noches consecutivas, 8 noches por sitio (Lambert et al., 2005). El resultado fue un esfuerzo de muestreo por sitio de 192 trampas noches. Cada punto de muestreo tuvo un área de 4.5 ha y por sitio de muestreo un área total de 9 ha. La determinación taxonómica a nivel de especie de cada individuo se basó en la coloración de las mejillas, el largo de la zona obscura de la cola y el color del almizcle. Las mejillas en D. marsupialis son de color crema a amarillas y en D. virginiana son blancas; el largo de zona obscura de la cola alcanza más de la mitad en D. marsupialis, mientras que en D. virginiana es menor y el almizcle es café ocre en D. marsupialis y verde en D. virginiana (Aranda, 2000; Ceballos y Oliva, 2005). Se tomaron las siguientes medidas de cada individuo capturado: peso, longitud total (cabeza-cola), sexo, así como una muestra de tejido de la oreja.
Debido a la baja captura en los sitios de Los Altos de Chiapas y a que sólo se tuvo un sitio de muestreo en la Depresión Central de Chiapas, se agruparon los datos de Los Altos y entonces se hicieron las estimaciones de abundancia relativa, proporción de sexos, talla y peso por región. La abundancia relativa se obtuvo como el número de individuos capturados por trampas noches (Adler et al., 1997), que corresponden a 192 trampas noches para la Depresión Central y a 960 para Los Altos de Chiapas.
Se realizó un análisis de independencia mediante la prueba de G (Sokal y Rholf, 2003) para evaluar si la abundancia relativa es independiente de la región y de la especies. Mediante un ji-cuadrada (χ2), por especie, se comparó si la proporción de sexos se ajusta a una relación de 1:1. También se realizó una prueba de U Mann-Whitney para probar si había diferencias en el peso y la longitud de los individuos entre regiones para cada especie.
Diversidad y estructura genética. Las muestras de tejido de oreja se conservaron en alcohol y posteriormente en refrigeración a -4° C hasta su procesamiento. De cada muestra se extrajo ADN nuclear siguiendo el protocolo de fenol-cloroformo-alcohol isoamílico (Hamilton et al., 1999). Se probaron 11 microsatélites, 5 de los cuales fueron diseñados para D. marsupialis (Dm1, Dm 2, Dm 3, Dm 4 y Dm 5) (Lavergne et al., 1998); mientras que los 6 restantes fueron diseñados para Caluromys philander, Didelphis aurita y Metachirus nudicaudatus (Dias et al., 2009) (Cuadro 2). Sólo con 7 marcadores se pudieron obtener resultados claramente legibles, reproducibles en el 96% de las muestras y por lo tanto, interpretables (Mnud 20, Mnud 41, Dm 1, Dm 2, Dm 3, Dm 5 y Daur 08).
La amplificación de los microsatélites se hizo con la reacción en cadena de la polimerasa (PCR) con la mezcla maestra (mastermix; PROMEGA), utilizando un termociclador de gradiente MJ mini, personal termal cycler de BIO-RAD. Las condiciones de la amplificación se basaron según Lavergne et al. (1998) y Dias et al. (2009), con modificación de la temperatura de alineamiento (TA) para 5 de los 7 marcadores analizados (Dm 1= 54° C; Dm 2= 49° C; Dm 3= 52° C; Mnud 20= 51° C y Daur 08= 52° C). El producto de la PCR se visualizó en geles de poliacrilamida al 10%, teñidos con bromuro de etidio y observados con luz ultravioleta.
Se estimaron las medidas de diversidad genética de heterocigosidad observada y esperada en equilibrio de Hardy-Weinberg (EHW), número de alelos promedio y porcentaje de alelos polimórficos por especie y región. Se realizó una combinación de probabilidades de pruebas independientes para obtener un análisis de EHW por población (Sokal y Rholf, 2003). La diferenciación entre especies y regiones se determinó, con base en los estadísticos Fst, Fis y Fit mediante un análisis de varianza molecular, con 999 iteraciones (Peakall y Smouse, 2006). Todos los parámetros genéticos se obtuvieron con el programa GenAlEx, versión. 6.4. También se evaluó la presencia de alelos nulos con el programa Micro-Checker, versión. 2.2.3 (Van Oosterhout et al., 2004), con el cual se hizo una corrección de las frecuencias alélicas y genotípicas, usando el 95% de confianza y 1 000 repeticiones (ver Chakraborty et al., 1992). Con el programa Free-NA (Chapuis y Estoup, 2007) se estimó la Fst corregida por presencia de alelos nulos. Asimismo, se estimó la distancia genética de Cavalli-Sforza y Edwards (1967) con el método ENA (Dc-ENA) e INA (Dc-INA), con el mismo programa, y se obtuvieron los respectivos dendrogramas con el programa PHYLIP, versión 3.69 (Felsenstein, 2009).
Resultados
Se capturó un total de 35 individuos, 16 de D. marsupialis y 19 de D. virginiana; no se capturó ninguna otra especie de marsupial (Cuadro 3). A pesar del menor esfuerzo de muestreo en la Depresión Central, para ambas especies hubo una mayor captura en esta región (16 individuos D. marsupialis: 10 machos y 6 hembras; y 19 individuos D. virginiana: 10 machos y 9 hembras), que en los Altos de Chiapas (Cuadro 3). La abundancia relativa estimada fue entre 0.008-0.05 individuos trampas noches para D. marsupialis y 0.01-0.06 individuos trampas noche para D. virginiana (Fig. 1). Sin embargo, el análisis de independencia de G mostró que la abundancia relativa, número de individuos capturados por trampas noches, es independiente de la especie y región (G= 0.35, p= 0.55). En las 2 regiones y para las 2 especies se encontró una proporción de sexos acorde con una relación de 1:1. En Los Altos para las 2 especies el valor observado fue igual al esperado y en la Depresión Central para D. marsupialis un valor de χ2= 2.7, p= 0.10 y para D. virginiana χ2= 0.1, p= 0.73.

Los organismos de la Depresión Central tuvieron en promedio, mayor peso (2 371.43 g) y longitud (80.86 cm) que en Los Altos (1 472.73 g y 78.45 cm) (Cuadro 4). La prueba de U Mann-Whitney no mostró diferencias significativas entre regiones en el peso y el tamaño de D. virginiana (tamaño: U= 8.00, p= 0.09; peso: U= 14.0, p= 0.14), mientras que para D. marsupialis no hubo diferencias en el tamaño entre regiones (U= 17.00, p= 0.63), pero sí en el peso (U= 1.00, p= 0.002).

Diversidad y estructura genética. En D. marsupialis 2 loci estuvieron fuera EHW en Los Altos de Chiapas (Mnud 41: χ2= 3.938; p= 0.05; g. l.= 1, y Dm 1: χ2= 7.00; p= 0.008; g. l.= 1); la combinación de probabilidades indicó que no hay EHW en esta población (Cuadro 5). En la Depresión Central, para esta misma especie, se encontraron 2 marcadores fuera de equilibrio: Dm 1 y Dm 2 (χ2= 8.00, p= 0.005, g. l.= 1; χ2= 8.89, p= 0.03, g. l.= 3, respectivamente) y nuevamente la prueba combinada detectó que no hay EHW (Cuadro 5).
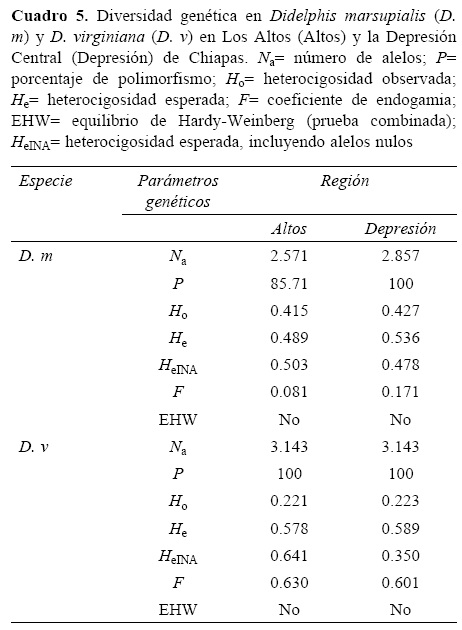
En cuanto a D. virginiana 4 loci estuvieron fuera de EHW en Los Altos de Chiapas: Mnud 20 (χ2= 4.15, p= 0.04, g. l.= 1); Dm1 (χ2= 22, p= 0.00, g. l.= 3); Dm3 (χ2= 22; p= 0.00; g. l.= 3), y Dm5 (χ2= 16.75; p= 0.001; g. l.= 3). En la Depresión Central 6 loci estuvieron fuera de EHW, con excepción de Daur 08 (χ2= 5.90, p= 0.43, g. l.= 10) (Fig. 2). En ambas poblaciones la prueba combinada mostró que las poblaciones no están en EHW.
El análisis de varianza molecular mostró una diferenciación genética significativa entre especies (Frt= 0.13, p= 0.001) y entre las poblaciones; 4 en total, 2 poblaciones de D. marsupialis y 2 de D. virginiana; Fst= 0.14, p= 0.001. Entre poblaciones de cada especie no hubo diferencia genética significativa (Fsr= 0.01, p= 0.26). Los coeficientes de endogamia Fit y Fis fueron significativamente distintos de cero (Fit= 0.58, p= 0.001; Fis= 0.51, p= 0.001).
La mayor diferenciación genética se encontró entre poblaciones de distintas regiones y especies, y la menor se observó entre poblaciones de una misma especie. Dicho patrón se mantiene con y sin la consideración de alelos nulos (Cuadro 6).
El análisis de alelos nulos con el algoritmo EM (Dempster et al., 1977), indicó una frecuencia de alelos nulos entre 0.00-0.40. Mediante el método de Chakraborty et al. (1992), en D. virginiana se detectaron alelos nulos en 4 loci (Dm 1, Dm 2, Dm 3 y Dm 5) en Los Altos, con frecuencia promedio de 0.52; en la Depresión Central se obtuvo una frecuencia promedio de 0.49. En D. marsupialis se detectó presencia de alelos nulos en Dm 1, en Los Altos, con frecuencia promedio de 0.13. En la Depresión Central, los loci Dm 2 y Dm 5 presentaron alelos nulos y la frecuencia promedio para esta población fue de 0.21. La heterocigosidad esperada considerando la presencia de alelos nulos (HeINA) fue de 0.503 en Los Altos y 0.478 en la Depresión Central en D. marsupialis y en D. virginiana de 0.641 y 0.350 en las mismas poblaciones, respectivamente (Cuadro 5). Finalmente, los valores de distancia genética de Cavalli-Sforza y Edwards (1967) excluyendo alelos nulos (DcENA) mostraron mayor distancia entre poblaciones de diferentes especies y la menor entre poblaciones de una misma especie (Fig. 3A). Al considerar los alelos nulos (DcINA), se mantiene la misma tendencia (Fig. 3B).
Discusión
Las especies capaces de explotar una alta variedad de recursos suelen colonizar una amplia gama de hábitats y presentan intervalos de distribución (Krabbe y Rahbek, 2010) y tamaños poblacionales localmente grandes (Gaston et al., 2000). Los tlacuaches D. marsupialis y D. virginiana son considerados organismos comunes, flexibles a las perturbaciones y con tasas de reproducción altas (Sunquist et al., 1987; Adler et al., 1997; Cabello, 2006). Los resultados del presente estudio sugieren que D. marsupialis y D. virginiana en Chiapas pueden desarrollarse en hábitats con ambientes como los que se observan en Los Altos (frescos) y en la Depresión Central (cálidos) (González-Espinosa et al., 2005); sin embargo, en ambas regiones sus poblaciones fueron relativamente pequeñas (D. marsupialis: 0.008-0.05 individuos trampas noches, D. virginiana: 0.01-0.06 individuos trampas noches). No obstante, es importante mencionar la posibilidad de sesgo en los resultados debido a la diferencia de esfuerzo de muestreo entre regiones. La abundancia relativa, un indicador del tamaño poblacional (Lambert et al., 2005), observada en el presente estudio fue independiente de la región, con valores que se encuentran dentro de los registrados en investigaciones previas. Por ejemplo, un estudio demográfico con D. marsupialis en el norte de Colombia registra una abundancia relativa de 0.010 individuos trampas noches (Adler et al., 1997). Asimismo, en el sur de Brasil y en Las Cuevasde Belice, se registró una densidad poblacional entre 1.4 y 2.26 individuos/ ha, respectivamente (Cáceres y Monteiro-Filho, 1998; Kelly y Caro, 2003). Para D. virginiana se ha observado una densidad de 0.23 individuos/ ha en Las Cuevas de Belice (Kelly y Caro, 2003). La abundancia relativa de estos didélfidos es comparativamente baja con respecto a otras especies de animales que también se consideran generalistas, de amplia distribución, tolerantes a la perturbación y con alta capacidad reproductiva, como los mapaches (Procyon lotor), cuyas poblaciones se favorecieron con las actividades humanas (Bozek et al., 2007). Procyon lotor presenta una densidad poblacional entre 3.4 y 13.6 individuos/ km2 en Ontario, Canadá (Rosatte et al., 2010). Es importante considerar que, como sucede cuando se tienen tamaños poblacionales pequeños, sobre todo, de animales de talla mediana, los cambios de uso de suelo pueden impactar negativamente las poblaciones de estas especies.
La abundancia relativa está determinada por las tasas de natalidad y mortalidad, las cuales dependen de los recursos disponibles, del grado de competencia intra e interespecífica y de la depredación (Gaston et al., 2000). No es posible determinar con nuestro estudio los factores específicos que pueden causar una abundancia relativa baja en D. marsupialis y D. virginiana. Para conocer los factores es necesario realizar un análisis de la fluctuación temporal de la abundancia relativa, así como la evaluación directa de factores potenciales de afectación, tanto aquellos asociados a actividades humanas como ambientales y demográficos
La proporción de sexos observada, de 1:1, puede considerarse una condición idónea para el crecimiento poblacional, en particular para especies en las que la aptitud no difiere entre sexos (Ehrlich y Roughgarden, 1987). Sin embargo, en especies polígamas, como D. marsupialis y D. virginiana, se espera un mayor número de machos, especialmente en áreas con mayor disponibilidad de recursos y menor competencia (Dickman, 1988; Hardy, 1997), por lo que la proporción de sexos observada podría estar asociada al tamaño poblacional bajo. Por otro lado, se observó mayor talla y peso en organismos capturados en la Depresión Central; no obstante, sólo se detectaron diferencias significativas en cuanto al peso entre regiones y únicamente para D. marsupialis. Dicha diferencia podría ser atribuida a la mayor disponibilidad en cuanto a cantidad y calidad de recursos alimenticios y de refugio (Rocha-Loredo et al., 2010), aspecto que debería evaluarse en estudios futuros.
Diversidad y estructura genética. Los alelos nulos son aquellos que no se detectan porque hay modificaciones en las secuencias de alineación del ADN blanco y por lo tanto, no se obtiene producto durante la PCR (Pompanon et al., 2005). Los alelos nulos pueden resultar en una estimación sesgada de la diferenciación poblacional, debido a la reducción de la diversidad genética dentro de las poblaciones (Chapuis y Estoup, 2007). En este estudio se detectó la presencia de alelos nulos, sin embargo, los valores de heterocigosidad y distancias genéticas no se modificaron al considerarlos, y las tendencias de diversidad y agrupación fueron las mismas.
Las especies D. marsupialis y D. virginiana son consideradas comunes, generalistas, con altas tasas de reproducción y con cierta resistencia a la perturbación (Kasparian et al., 2004), por lo que se esperaba que las poblaciones estudiadas presentaran niveles altos de diversidad genética y poca diferenciación entre sus poblaciones. No se conocen estudios de diversidad genética nuclear para D. marsupialis y D. virginiana, además de los 2 trabajos enfocados al diseño de microsatélites (Lavergne et al., 1998; Fike et al., 2009). Para D. virginiana se diseñaron 21 microsatélites para un población del norte de Indiana, EUA, donde se encontró un valor promedio de He de 0.768 (Fike et al., 2009), mientras que para D. marsupialis se diseñaron 5 microsatélites para una población de la Guayana Francesa (Brasil), donde la diversidad promedio fue de He= 0.93 (Lavergne et al., 1998). En estos 2 casos los valores son mayores que los encontrados en este trabajo (0.548).
Se observaron también valores positivos del coeficiente de endogamia para todas las poblaciones, donde resaltan los valores de las poblaciones de D. virginiana: valores positivos indican que los organismos están estrechamente emparentados (Hartl y Clark, 1997). La endogamia puede darse porque los apareamientos no suceden al azar y/o por un aislamiento de las poblaciones por la presencia de barreras físicas o ecológicas (Smith y Wayne, 1996; Sexton et al., 2009). En este caso, existe una diferencia de altitud de 2 000 m entre las poblaciones de Los Altos y la Depresión Central de Chiapas, asociado a un cambio radical en las condiciones ambientales, distintos usos de suelo; agrícola, ganadero, forestal y asentamientos humanos, todo lo cual puede representar barreras físicas y ecológicas para el movimiento de los tlacuaches entre las regiones y potencialmente disminuir o anular el flujo genético. Asimismo, bajo condiciones de aislamiento y tamaño poblacional pequeño, la población puede ser forzada a un apareamiento entre organismos emparentados.
La distribución heterogénea de las frecuencias alélicas entre regiones puede deberse al efecto de la deriva genética, que tiene un efecto más significativo que la selección natural cuando las poblaciones son pequeñas (Castillo, 2007), como en el caso de D. marsupialis y D. virginiana en Chiapas. La acción de la deriva genética y la endogamia puede promover la fijación y pérdida de alelos en las poblaciones, y causar una diferenciación genética entre ellas (Vázquez-Domínguez et al., 2012). En este estudio, sin embargo, los niveles de Fst observados entre poblaciones de la misma especie sugieren una diferenciación reducida y moderada entre especies. Estudios filogenéticos con marsupiales sugieren poca diferenciación entre las especies, por ejemplo, Jansa y Voss (2000) en un estudio con marsupiales de América mostraron que la divergencia entre especies del género Didelphis oscila entre 0.95 y 1.3% con el gen IRBP; y de 5.7 a 12.6% con el citocromo b. Cervantes et al. (2010) encontraron una diferenciación genética de 7.8 a 9.3% entre D. marsupialis y D. virginiana con el citocromo c oxidasa subunidad I (Cox1). La diferenciación observada entre D. marsupialis y D. virginiana en este estudio también puede considerarse baja (13.4%), en tanto que los microsatélites tienen tasas de mutación mayores que el citocromo b, el gen IRBP y el Cox1 (Rentaría, 2007). La baja diferenciación entre estas especies puede deberse a que son especies cercanamente relacionadas (Voss y Jansa, 2009; Medina-Romero et al., 2012) y a que sus atributos ecológicos les han permitido sobrevivir a los cambios ambientales y con pocas modificaciones genéticas a lo largo de su evolución. El análisis de distancia genética también mostró mayor diferenciación entre especies que entre poblaciones, mientras que dentro de las poblaciones fue D. marsupialis la que presentó valores más altos. Ello puede estar asociado, entre otras posibilidades, al hecho de que D. virginiana es más generalista que D. marsupialis y que se encuentra más frecuentemente asociada a zonas con actividades humanas (Bozek et al., 2007).
Las condiciones geográficas de Los Altos y la Depresión Central de Chiapas sugieren un limitado flujo genético entre las poblaciones, junto con los niveles de endogamia y la heterogeneidad de las frecuencias alélicas sugerirían una diferenciación entre las poblaciones de cada especie. Inesperadamente, el valor de Fst entre poblaciones indica mínima diferenciación genética (1%). Esto puede ser debido a que las poblaciones se originaron de una misma población y las diferencias en las frecuencias alélicas y niveles de endogamia sean resultado de deriva genética. Es importante hacer notar que no se descarta que la diferenciación observada sea resultado de un sesgo en las frecuencias alélicas asociado a un tamaño de muestra pequeño para cada especie por población.
La tasa de evolución moderada de los marsupiales (Jansa y Voss, 2000; Steiner y Catzeflis, 2004) probablemente ha contribuido al mantenimiento de la diversidad genética y a la escasa diferenciación entre poblaciones y especies. No obstante, si se confirman tamaños poblacionales pequeños de D. marsupialis y D. virginiana se anticipa que estas especies pueden requerir un cambio de estatus hacia algún grado de vulnerabilidad en Los Altos y la Depresión Central (Frankham et al., 2004). Así, la conservación de D. marsupialis y D. virginiana es de alta prioridad por pertenecer a un grupo de mamíferos poco representado en México, por la relación que guardan con radiaciones adaptativas en Australia (Medina-Romero et al., 2012) y por su papel ecológico en diversos ecosistemas.
Agradecimientos
Expresamos nuestro agradecimiento a Jorge Luis Cruz-Burguete, Jorge Bolaños-Citalán, Eugenia Sántiz-López, Alfonso Ortiz-Moreno, Wenceslao Bonifaz, Trinidad Alejandro Guillén-Díaz y Claudia Macías por su apoyo en campo. A Maricela García-Bautista por su ayuda en el trabajo de laboratorio. Este trabajo es parte de la tesis de doctorado de BCS (Beca Conacyt-número 175336).
Literatura citada
Adler, G. H, J. J. Arboledo y B. L. Travi. 1997. Population dynamics of Didelphis marsupialis in Northern Colombia. Studies on Neotropical Fauna and Envirnoment 32:7-11. [ Links ]
Aranda, M. 2000. Huellas y otros rastros de los mamíferos grandes y medianos de México. Comisión Nacional para el Conocimiento y Uso de la Biodiversidad. Instituto de Ecología, A. C., Xalapa, Veracruz. 212 p. [ Links ]
Bozek, C. K., S. Prange y S. D. Gehrt. 2007. The influence of anthropogenic resources on multi-scale habitat selection by raccons. Urban Ecosystems 10:413-425. [ Links ]
Cabello, D. R. 2006. Reproduction of Didelphis marsupialis (Didelphimorphia: Didelphidae) in the Venezuelan Andes. Acta Therologica 51:427-433. [ Links ]
Ceballos, G. y G. Oliva. 2005. Los mamíferos silvestres de México. Comisión Nacional para el Conocimiento y Uso de la Biodiversidad: Fondo de Cultura Económica. México, D. F. 986 p. [ Links ]
Cervantes, F. A., J. Arcangeli, Y. Hortelano-Moncada y A. V. Borisenko. 2010. DNA barcodes effectively identify the morphologically similar Common Opossum (Didelphis marsupialis) and Virginia Opossum (Didelphis virginiana) from areas of sympatry in Mexico. Mitochondrial DNA 21:44-50. [ Links ]
Cáceres, N. C. y E. L. A. Monteiro-Filho. 1998. Population dynamics of the Common opossum, Didelphis marsupialis (Mammalia, Marsupialia), in southern Brazil. International Journal of Mammalian Biology 63:169-172. [ Links ]
Castillo, C. A. 2007. La selección natural a nivel molecular. In Ecología molecular, L. E. Eguiarte, V. Souza y X. Aguirre (eds.). Instituto Nacional de Ecología, Semarnat, México, D. F. p. 11-48. [ Links ]
Cavalli-Sforza, L. L. y A. W. F. Edwards. 1967. Phylogenetic analysis: models and estimation procedures. American Journal of Human Genetics 19:233-257. [ Links ]
Chakraborty, R., M. De Andrade, S. P. Daiger y B. Budowle. 1992. Apparent heterozygote deficiencies observed in DNA typing data and their implications in forensic applications. Annals of Human Genetics 56:45-57. [ Links ]
Chapuis, M. P. y A. Estoup. 2007. Microsatellite null alleles and estimation of population differentiation. Molecular Biology and Evolution 24:621-631. [ Links ]
Clemens, W. A. 1968. Origin and early evolution of marsupials. Evolution 22:1-18. [ Links ]
Dempster, A. P., N. M. Laird y D. R. Rubin. 1977. Maximum likelihood from incomplete data via the EM algorithm. Journal of the royal Statistical Society 39:1-38. [ Links ]
Dias, I. M. G., G. Amato., H. M. Cunha, R. DeSalle, A. P. Paglia, J. K. Peterson y C. G. Fonseca. 2009. Isolation, characterization and cross-species amplification of new microsatellite markers for three oposum species of the Didelphidae family. Conservation Genetics Resources 1:405-410. [ Links ]
Dickman, C. R. 1988. Variation in response to interspecific competition. American Society of Naturalists 132:289-297. [ Links ]
Ehrlich, P. R. y J. Roughgarden. 1987. The science of ecology. Macmillan Publishing Company y Collier Macmillan Canada, New York. 710 p. [ Links ]
Frankham, R., J. D Ballou y K. H. McInnes. 2004. A primer of conservation genetics. University of Cambridge, Cambridge. 220 p. [ Links ]
Felsenstein, J. 2009. Phylip: phylogeny inference package. Department of Genome Sciences and Department of Biology, University of Washington. http://evolution.genetics.washington.edu/phylip.html; última consulta: 20.II.2013. [ Links ]
Fike, J. A., J. C. Beasley y O. E. Rhodes Jr. 2009. Isolation of 21 polymorphic microsatellite markers for the Virginia opossum (Didelphis virginiana). Molecular Ecology Resources 9:1200-1202. [ Links ]
Fleck, D. W. y J. D. Harder. 1995. Ecology of marsupials in two Amazonian rain forest in northeastern Peru. Journal of Mammalogy 76:809-818. [ Links ]
García, E. 1973. Modificaciones al sistema de clasificación climática de Köpen (para adaptarlo a las condiciones de la República Mexicana). Instituto de Geografía, Universidad Nacional Autónoma de México, México, D. F. 246 p. [ Links ]
Gardner, A. L. 1973. The systematics of the genus Didelphis (Marsupialia: Didelphidae) in North and Middle America. Special Publications of the Museum Texas Tech University 4:1-81. [ Links ]
Gaston, K. J., T. M. Blackburn, J. J. D. Greenwood, R. D. Gregory, R. M. Quinn y J. H. Lawton. 2000. Abundance-occupancy relationships. Journal of Applied Ecology 37:39-59. [ Links ]
Gehrt, S. D., D. E. Clark y E. K. Fritzell. 1997. Dynamics and ecology of Virginia opossums in Sourthern Texas. The Southwestern Naturalist 42:170-176. [ Links ]
González, D. P., E. D. J. Martínez, M. P. A. López y S. Gómez. 2012. Estrategia para la restauración de la cuenca del Valle de Jovel. Fondo Mexicano para la Conservación de la Naturaleza, A. C. y Fundación Gonzalo Río Arronte, I. A. P., México, D. F. 55 p. [ Links ]
González-Espinosa, M., N. Ramírez-Marcial, G. Méndez-Dewar, L. Galindo-Jaimes y D. Golicher. 2005. Riqueza de especies de árboles en Chiapas: variación espacial y dimensiones ambientales asociadas al nivel regional. In Diversidad biológica en Chiapas, M. González-Espinosa, N. Ramírez-Marcial y L. Ruiz-Montoya (eds.). Ecosur, Cocytech, Plaza y Valdez Editores, México, D. F. p. 81-125. [ Links ]
González-Espinosa, M., Ramírez-Marcial N. y L. Galindo-Jaimes. 2006. Secondary succesion in montane pine-oak forests of Chiapas, México. In Ecology and conservation of Neotropical montane oak forests, M. Kappelle (ed.). Ecological Studies 185:209-221. [ Links ]
Hamilton, M., E. Pincus, A. Di Fiore y R. C. Fleischer. 1999. Universal linker and ligation procedures for construction of genomic DNA libraries enriched for microsatellites. BioTechniques 27:500-507. [ Links ]
Hardy, I. C. W. 1997. Opossum sex ratios revisited: significant or nonsignificant? The American Naturalist 150:420-424. [ Links ]
Hartl, D. L. y A. G. Clark. 1997. Principles of population genetic. Sinauer, Sunderland. 542 p. [ Links ]
Hey, J. y C. A. Machado. 2003. The study of structured populations - new hope for a difficult and divided science. Nature Reviews Genetics 4:535. [ Links ]
Holmes, D. J. 1991. Social behavior in captive Virginia opossums, Didelphis virginiana. Journal of Mammalogy 72:402-410. [ Links ]
Jansa, S. A y R. S. Voss. 2000. Phylogenetic studies on Didelphid marsupials I. Introduction and Preliminary Results from Nuclear IRBP Gene Sequences. Journal of Mammalian Evolution 7:43-77. [ Links ]
Kanda, L. L., T. K. Fuller, P. R. Sievert y K. D. Friedland. 2005. Variation in winter microclimate and its potential influence on Virginia opossum (Didelphis virginiana) survival in Amherst, Massachusetts. Urban Ecosystems 8:215-255. [ Links ]
Kasparian, M. A., E C. Hellgreen, S. M. Ginger, L. P. Lavesque, J. E. Clark y D. L. Winkerman. 2004. Population characteristics of Virginia opossum in the cross timbers during raccoon reduction. American Midland Naturalist 151:154-163. [ Links ]
Kelly, M. J. y T. Caro. 2003. Low density of small mammals at Las Cuevas, Belize. Mammalian Biology 68:372-386. [ Links ]
Krabbe, B. M. y C. Rahbek. 2010. Causality of the relationship between geographic distribution and species abundance. The Quarterly Review of Biology 85:3-25. [ Links ]
Lambert, T. D., J. R. Malcolm y B. L. Zimmerman. 2005. Variation in small mammal species richness by trap height and trap type in southeastern Amazonia. Journal of Mammalogy 86:982-990. [ Links ]
Lavergne, A., C. Douady y D. M. Catzeflis. 1998. Isolation and characterization of microsatellite loci in Didelphis marsupialis (Marsupialia: Didelphidae). Molecular Ecology 8:517-518. [ Links ]
MacManus, J. J. 1970. Behavior of captive opossums, Didelphis marsupialis virginiana. American Midland Naturalist 84:144-169. [ Links ]
MacManus, J. J. 1971. Activity of captive Didelphis marsupialis. Journal of Mammalogy 52:846-848. [ Links ]
MacManus, J. J. 1974. Didelphis virginiana. Mammalian Species 40:1-6. [ Links ]
Maurer, B. A. 2000. Macroecology and consilience. Global Ecology y Biogeography 9:275-280. [ Links ]
Markovchick-Nicholls, L., H. M. Regan, D. H. Deutschman, A. Widyanata, B. Martin, L. Noreke y T. A. Hunt. 2007. Relationships between human disturbance and wildlife land use in urban habitat fragments. Conservation Biology 22:99-109. [ Links ]
Medina-Romero, M., I. Goyenechea y J. Castillo-Cerón. 2012. Phylogenetic measures applied to the conservation of Mexican marsupials. Revista Mexicana de Biodiversidad 83:1216-1220. [ Links ]
Naranjo, P. E., C. Lorenzo M. y A. Horváth. 2005. La diversidad de mamíferos en Chiapas. In Diversidad biológica en Chiapas, M. González-Espinosa. N. Ramírez-Marcial y L. Ruiz-Montoya (eds.). El Colegio de la Frontera Sur, Cocytech, Plaza y Valdés, S. A. de C. V. México, D. F. p. 221-264. [ Links ]
Orjuela, C. O. J. y G. Jiménez. 2004. Estudio de la abundancia relativa para mamíferos en diferentes tipos de coberturas y carretera, finca Hacienda Cristales, área Cerritos-La Virginia, municipio de Pereira. Universitas Scientiarum 9:87-96. [ Links ]
Peakall, R. y P. E. Smouse. 2006. GENALEX 6: genetic analysis in Excel. Population genetic software for teaching and research. Molecular Ecology Notes 6:288-295. [ Links ]
Pompanon, F., A. Bonin, E. Bellemain y P. Taberlet. 2005. Genotyping errors: causes, consequences and solutions. Nature Reviews Genetics 6:847-859. [ Links ]
Ramírez-Marcial, N., M. González-Espinosa y D. Williams-Linera. 2001. Anthropogenic disturbance and tree diversity in montane rain forests in Chiapas, México. Forest Ecology and Management 154:311-326. [ Links ]
Rangel-Salazar, J. L., P. L. Enríquez y T. Will. 2005. Diversidad de aves en Chiapas: prioridades de investigación para su conservación. In Diversidad biológica en Chiapas, M. González-Espinosa. N. Ramírez-Marcial y L. Ruiz-Montoya (eds.). El Colegio de la Frontera Sur, Cocytech, Plaza y Valdés, S. A. de C. V. México, D. F. p. 265-324. [ Links ]
Ringier, H. J. 1961. Review of Oligocene Didelphid marsupials. Journal of Paleontology 35:218-228. [ Links ]
Rentaría, A. M. 2007. Breve revisión de los marcadores moleculares. In Ecología molecular, L. E. Eguiarte. V. Souza y X. Aguirre (eds.). Instituto Nacional de Ecología, Semarnat, México, D. F. p. 541-566. [ Links ]
Rodiles-Hernández, R. 2005. Diversidad de peces continentales en Chiapas. In Diversidad biológica en Chiapas, M. González-Espinosa. N. Ramírez-Marcial y L. Ruiz-Montoya (eds.). El Colegio de la Frontera Sur, Cocytech, Plaza y Valdés, S. A. de C. V. México, D. F. p. 195-220. [ Links ]
Rosatte, R., M. Ryckman, K. Ing, S. Proceviat, M. Allan, L. Bruce, D. Donovan y J. C. Davies. 2010. Density, movements, and survival of raccoons in Ontario, Canada: implications for disease spread and management. Journal of Mammalogy 91:122-135. [ Links ]
Sexton, J. P., P. J. McIntyre, A. L. Angert y K. J. Rice. 2009. Evolution and ecology of species range limits. Annual Reviews of Ecology, Evolution, and Systematics 40:415- 436. [ Links ]
Smith, T. B. y R. K. Wayne. 1996. Molecular genetic approaches in conservation. Oxford University Press, Nueva York.
483 p. [ Links ]
Sokal, R. R. y F. J. Rholf. 2003. Biometry. The principles and practice of statistics in biological research. W. H. Freeman and Company, New York. 887 p. [ Links ]
Steiner, C. y F. M. Catzeflis. 2003. Mitochondrial diversity and morphological variation of Marmosa murina (Didelphidae) in French Guiana. American Society of Mammalogists 84:822-831. [ Links ]
Steiner, C. y F. M. Catzeflis. 2004. Genetic variation and geographical structure of five mouse-sized opossum (Marsupialia, Didelphidae) trhroughout the Guiana Region. Journal of Biogeograpy 31:959-973. [ Links ]
Stropp, J., H. T. Steege, Y. Malhi, ATDN y RAINFOR. 2009. Disentangling regional and local tree diversity in the Amazon. Ecography 32:46-54. [ Links ]
Sunquist, M. E., S. N. Austad y F. Sunquist. 1987. Movemnt patterns and home range in the common oposum (Didelphis marsupialis). Journal of Mammalogy 68:173-176. [ Links ]
Tyndale-Biscoe, H. y R. B. MacKenzie. 1976. Reproduction in Didelphis marsupialis and D. albiventris in Colombia. Journal of Mammalogy 57:249-265. [ Links ]
Van Oosterhout, C., W. F. Hutchinson, D. P. M. Willis y P. Shipley. 2004. Micro-checker: software for identifying and correcting genotyping error in microsatellite data. Molecular Ecology Notes 4:535-538. [ Links ]
Vázquez-Domínguez, E., M. Suárez-Atilano, W. Booth, C. González-Baca y A. D. Cuarón. 2012. Genetic evidence of a recent successful colonization of introduced species on islands: Boa constrictor imperator on Cozumel Island. Biological Invasions 14:2101-2116. [ Links ]
Voss, R. S. y S. A. Jansa. 2009. Phylogenetic relationships and classification of Didelphid marsupials, an extant radiation of new world metatherian mammals. Bulletin of the Americam Museum of Natural History 322:1-177. [ Links ]














