Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Similars in
SciELO
Similars in
SciELO
Share
Revista mexicana de biodiversidad
On-line version ISSN 2007-8706Print version ISSN 1870-3453
Rev. Mex. Biodiv. vol.81 n.3 México Dec. 2010
Taxonomía y sistemática
Análisis del tamaño del genoma y cariotipo de Agave aktites Gentry (Agavaceae) de Sonora, México
Genome size and karyotype analysis of Agave aktites Gentry (Agavaceae) from Sonora, Mexico
Guadalupe Palomino1*, Javier Martínez1 e Ignacio Méndez2
1 Laboratorio de Citogenética, Jardín Botánico, Instituto de Biología, Universidad Nacional Autónoma de México. Apartado postal 70–614, 04510 México, D.F., México. *Correspondencia: palomino@ibiologia.unam.mx
2 Instituto de Matemáticas Aplicadas y Sistemas, Universidad Nacional Autónoma de México. Apartado postal 20–726, 01000 México D.F., México.
Recibido: 02 diciembre 2008
Aceptado: 13 abril 2010
Resumen
Se determinó el tamaño del genoma y la estructura del cariotipo de 2 poblaciones silvestres de Agave aktites Gentry de Las Bocas y San Carlos, Sonora, México. El contenido de ADN nuclear en tejido foliar se determinó por citometría de flujo y los cromosomas se observaron en metafase mitótica de meristemos radiculares. Las plantas en ambas poblaciones son diploides (2n= 2x= 60). El contenido promedio 2C de ADN nuclear fue de 8.404 pg; 1Cx= 4 120 millones de pares de nucleótidos. El cariotipo bimodal fue similar en las 2 poblaciones y consistió de 10 cromosomas grandes y 50 pequeños y correspondió a 46m+6st+8t: también mostró un par de cromosomas telocéntricos grandes con constricción secundaria. El cociente de los brazos cromosómicos fue diferente en los pares 7, 8, 14 y 16 del grupo de cromosomas pequeños que presentan diferencias morfológicas entre las 2 poblaciones. Estos rearreglos cromosómicos podrían deberse a intercambios cromosómicos heterocigóticos espontáneos y son evidencia de que los genomas de distintas poblaciones de A. aktites se encuentran en un activo proceso de diferenciación que podría llevar a la especiación. Los análisis son básicos para conocer la diversidad genética intraespecífica de A. aktites y para establecer estrategias de conservación in situ y ex situ para esta especie.
Palabras clave: contenido nuclear de ADN, citometría de flujo, estructura del cariotipo.
Abstract
Genome size and karyotype structure of 2 wild populations of Agave aktites Gentry from Las Bocas and San Carlos, Sonora, Mexico were determined. Nuclear DNA content of leaf tissue was measured through flow cytometry, and chromosomes were observed in mitotic metaphase of root tips. All individual plants studied in both populations are diploids (2n= 2x= 60). The mean 2C nuclear DNA content was 8.404 pg; 1Cx= 4 120 million of base pairs. All plants of the 2 populations of A. aktites show a bimodal karyotype consisting of 10 large + 50 small chromosomes and corresponded to 46m+6st+8t; they also have a pair of large telocentric chromosomes with secondary constriction. The arm ratio was different in pair numbers 7, 8, 14 and 16 of the small chromosome group and there are morphological differences in these chromosomes between the 2 populations studied. These chromosome rearrangements may be due to spontaneous heterozygous chromosomal exchange and they are evidence that the genome of A. aktites is in active process of differentiation among populations that could lead to speciation. These analyses are fundamental for understanding the intraspecific genetic diversity in A. aktites and establishing in situ and ex situ conservation strategies for this species.
Key words: nuclear DNA content, flow cytometry, karyotype structure.
Introducción
El género Agave L. es endémico del continente americano e incluye aproximadamente 200 especies y 247 taxa infraespecíficos, de los cuales 150 especies se distribuyen en México (García–Mendoza, 2002; García–Mendoza y Chiang, 2003). En el estado de Sonora el género Agave se encuentra representado por 26 especies, 4 subespecies y 4 variedades nativas (Gentry, 1982). Agave aktites Gentry se encuentra ubicado en el subgénero Agave, grupo Rigidae y es una especie endémica de la región sudoeste del estado de Sinaloa y áreas adyacentes del estado de Sonora. Es la única especie de Agave que crece de manera natural y regularmente en las dunas cercanas a la playa en vegetación costera, aunque también se encuentran plantas a varios kilómetros de la costa. Se han colectado individuos de esta especie en algunas islas cercanas, como la de Lechuguilla al norte de Topolobampo, Sinaloa.
Las plantas de A. aktites son pequeñas en comparación con otras especies de Agave y se reconocen por presentar rosetas entre 40–70 cm de altura y 60–110 cm de ancho, hojas lineares, glaucas y de color azul grisáceo, espinas cortas, (12–20 mm de longitud), inflorescencias paniculadas de 3–4 m de altura con 10–15 ramas pequeñas y flores verde pálido de 60–70 mm de longitud en pequeñas umbelas (Gentry, 1982).
El género Agave tiene un número cromosómico básico x = 30. Este valor ha sido corroborado con la observación de 30 bivalentes en metafase I de la meiosis en varias especies de Agave (Brandham, 1969; Pinkava y Baker, 1985; Ruvalcaba–Ruiz y Rodríguez–Garay, 2002). El valor de x = 30 del género Agave podría corresponder a un paleopoliploide, un poliploide secundario, o haberse originado de un poliploide intragenérico (Pinkava y Baker, 1985).
Las especies de Agave presentan cariotipos bimodales altamente conservados, formados por un grupo de 5 pares de cromosomas grandes y 25 de pequeños, en los cuales se ha llevado a cabo un proceso de selección natural llamado orto–selección cariotípica, que es la tendencia de que el mismo tipo de rearreglo se repita una y otra vez en cromosomas diferentes de la misma especie (White, 1978). Este proceso da como resultado en Agave un número básico (x) uniforme y cariotipos donde se mantiene el grupo de cromosomas grandes y pequeños (Brandham y Doherty, 1998). También se ha corroborado que en el proceso de evolución en Agave están involucrados rearreglos estructurales en el grupo de cromosomas grandes y pequeños (Brandham, 1969; Castorena–Sánchez et al., 1991; Ruvalcaba–Ruiz y Rodríguez–Garay, 2002; Palomino et al., 2005; Moreno–Salazar et al., 2007).
Se han realizado estudios sobre el contenido de ADN nuclear en diferentes especies de Agave (Cavallini et al., 1996; Zonneveld, 2003; Palomino et al., 2003; 2005; Moreno–Salazar et al., 2007). El contenido de ADN nuclear en plantas de Agave diploides es de 8 pg aproximadamente (Zonneveld, 2003; Palomino et al., 2007).
En este trabajo se determinó el contenido nuclear de ADN en picogramos (pg), y en millones de pares de bases de nucleótidos el nivel de poliploidía y la estructura del cariotipo de 2 poblaciones silvestres de Agave aktites de Sonora, México, con el objetivo de evaluar la variación inter– e intraespecífica en sus genomas; esta información es básica para planear estrategias de mejoramiento, biotecnología y conservación in situ y ex situ para esta especie.
Materiales y métodos
Material vegetal y sitios de muestreo. Las plantas estudiadas se obtuvieron de 2 poblaciones silvestres de A. aktites; una en Las Bocas, sitio localizado a 26° 35' N, 115° 20' O, y 30 m snm de altitud, en la costa del estado de Sonora y próximo a los límites con el estado de Sinaloa; la otra, cerca de San Carlos, en dunas próximas a la playa, a 31° 25' N, 112° 48' O y una altitud de 30 m snm, al noroeste de Guaymas, Sonora. Se colectaron 10 individuos de cada sitio, se colocaron en macetas con una mezcla de suelo orgánico, arena y vermiculita y se mantuvieron durante 3 años en los invernaderos del Jardín Botánico, Instituto de Biología, UNAM.
Determinación del tamaño del genoma y nivel de ploidía. Se utilizaron 5 plantas de A. aktites de cada población y se realizaron 3 repeticiones por cada planta. Se utilizaron plantas de maíz (Zea mays cv. CE–777) 2C de ADN = 5.433 pg (Dolezel et al., 1998) como especie de referencia. Se determinó el contenido total de ADN nuclear, el tamaño del genoma en pg y en millones de pares de bases de nucleótidos (Mpb) y los niveles de ploidía mediante citometría de flujo.
Se utilizó un citómetro de flujo (CF) Partec CA II (Partec GMBH) calibrado con núcleos de eritrocitos de pollo (Galbraith et al., 1998). La potencia del CF se ajustó para que el histograma que representa los núcleos de la planta de referencia apareciera en el canal 50 de la pantalla del aparato y asegurar las condiciones óptimas del CF. Los núcleos aislados se obtuvieron de acuerdo a Palomino et al. (2003; 2005).

De los genomas se obtuvo 1pg de ADN = 980 Mpb (Bennett et al., 2000). 1pg = 2 x 109 pares de nucleótidos.
Conteos cromosómicos, cariotipo e idiograma. Para la observación del número cromosómico 2n y elaboración del cariotipo en plantas de A. aktites, se utilizaron 15 a 23 células en metafase mitótica de 5 a 6 plantas de cada población. Los ápices de raíces secundarias fueron colocados en una solución de 8–hidroxiquinoleína 0.002 M por 6 h a 18° C en oscuridad. Posteriormente, se fijaron las puntas de raíz en una solución Farmer (3 partes de alcohol absoluto: 1 parte de ácido acético glacial) por 24 h. Las puntas de raíz se hidrolizaron con HCl (1N) 15 min a 60° C y los cromosomas se tiñeron con reactivo de Schiff por 1 h y después se colocaron en ac. propiónico y orceína al 1.8% (Moreno–Salazar et al., 2007).
Las preparaciones se congelaron con hielo seco (Conger y Fairchield, 1953) y se montaron en bálsamo de Canadá. Las mejores células (15 a 23 ) se fotografiaron en un foto–microscopio Zeiss modelo II. Las fotografías se utilizaron para medir los brazos cromosómicos y la longitud total del genoma. La posición del centrómero se obtuvo siguiendo a Levan et al. (1964); el cociente de los brazos cromosómicos (r = brazo largo/corto; q/p) se calculó para cada cromosoma.
La homología cromosómica se estableció de acuerdo con las similitudes en longitud y posición centromérica. Adicionalmente, la constricción secundaria fue utilizada para distinguir pares homólogos entre las poblaciones. Los idiogramas se construyeron de acuerdo con los valores promedio del brazo corto y largo en cada par de cromosomas y se agruparon en cromosomas metacéntricos (m), submetacéntricos (sm), subtelocéntricos (st) y telocéntricos (t). El número de los cromosomas homólogos se asignó siguiendo la reducción en la longitud del cromosoma, para un total de 30 pares. El índice de asimetría (TF %) se obtuvo conforme a Sinha y Roy (1979).
Análisis estadístico.Las diferencias en el contenido de ADN en pg y su composición en Mpb entre los genomas de las 2 poblaciones de A. aktites, se evaluaron con un ANOVA anidado de 2 niveles. El modelo consideró las localidades dentro de las poblaciones y las repeticiones de núcleos dentro de las plantas. Este modelo también se aplicó a la longitud total del genoma (LTG) y al índice de asimetría (TF %). Los análisis se realizaron con JMP 7.0 SAS.
Resultados
La figura 1 muestra los histogramas característicos obtenidos en el análisis del contenido nuclear de ADN en las plantas de A. aktites de Sonora. El primero se ubicó en el canal 50 y correspondió a núcleos en fase G0/G1 del contenido nuclear 2C de ADN para Zea mays (planta de referencia) y otro menor en el canal 100, que representó los núcleos en fase G2, con un contenido de 4C de ADN. El histograma comprendido en el canal 75 corresponde a núcleos G0/G1 de A. aktites. En todas las muestras la cantidad de núcleos de A. aktites en fase G2 observados en el canal 150 fue escasa, y no se observaron núcleos con contenido de ADN mayores a 4C, lo que indica ausencia de endopoliploidía. El análisis de la distribución mostró que aproximadamente el 99 % de los núcleos de las hojas de Zea mays y de A. aktites estuvieron en fase G0/G1. En los histogramas de las muestras analizadas se obtuvieron valores de coeficiente de variación (CV) con valores de 1.28 a 4.17 %, lo que validó las repeticiones obtenidas en este estudio (Dolezel et al., 1997).
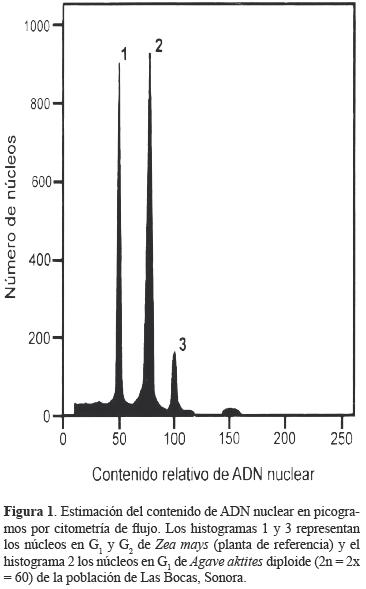
Las cantidades 2C de ADN calculado con base en el cociente del valor de la muestra respecto a la media de los histogramas se observan en el Cuadro 1. El promedio del tamaño del genoma 2C de ADN fue = 8.408 pg con una composición del genoma 1Cx (grupo de cromosomas monoploide; Greilhuber et al., 2005) = 4120 Mpb para la población de A. aktites de Las Bocas. En la población de San Carlos se observaron valores 2C de ADN = 8.400 pg y un valor 1Cx = 4119 Mpb.
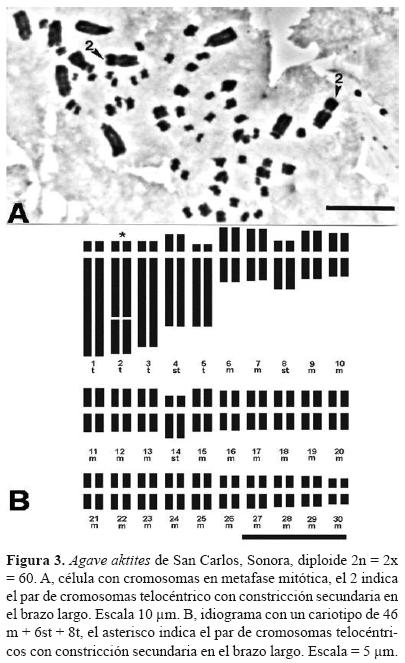
Las 2 poblaciones de Agave aktites estudiadas fueron diploides con 2n = 2x = 60 y mostraron un cariotipo bimodal formado por 5 pares de cromosomas grandes y 25 pares de cromosomas pequeños (Cuadro 2; Figuras 2 y 3). Las 2 poblaciones de A. aktites presentaron el mismo cariotipo formado por 46m + 6st + 8t (Cuadro 1).

Discusión
El ANOVA mostró que las diferencias observadas en la cantidad 2C de ADN de los individuos de las 2 localidades no fueron significativas (p = 0.3607). El valor promedio de 2C ADN = 8. 404 pg y 1Cx = 4120 Mpb, es el primer registro de estos parámetros para A. aktites. De 200 especies que pertenecen al género Agave, solamente en 27 se ha analizado el tamaño del genoma, y la mayoría son diploides con una cantidad 2C de ADN que va de 6.0 pg en A. fourcroydes a 9.6 pg en A. desertii (40 % de variación en el tamaño del genoma), Bennett y Leitch, 2005; Palomino et al., 2007.
El contenido de ADN para las plantas diploides de A. aktites (2C ADN = 8.404 pg) es cercana a los valores obtenidos para A. angustifolia 2C ADN = 8.45 y 8.49 pg (Palomino et al., 2005; Moreno–Salazar et al., 2007) y A. rhodacantha (8.415 pg; Moreno–Salazar et al., 2007), ambas pertenecientes al grupo Rigidae de Agave, al igual que A. aktites. La ausencia de endopliploidía también ha sido observada en A. tequilana y A. angustifolia (Palomino et al., 2003; 2005; Moreno–Salazar et al., 2007).
Todas las plantas de las 2 poblaciones de A. aktites estudiadas son diploides (2n = 2x = 60) y en ellas se observaron cariotipos bimodales característicos del género Agave, también presentes en otras especies silvestres y cultivadas del género (Pinkava y Baker, 1985; Castorena–Sánchez et al., 1991; Palomino et al., 2005; 2008; Moreno–Salazar et al., 2007).
En los individuos de las 2 poblaciones de A. aktites se observó el mismo cariotipo formado por 46m + 6st + 8t; sin embargo, al analizar el cociente del brazo largo (q) entre el brazo corto (p) en los cromosomas de A. aktites de las 2 poblaciones, se evidenciaron diferencias grandes en algunos pares cromosómicos. En el grupo de cromosomas grandes, las variaciones son notables en los pares de cromosomas telocéntricos 1, 2 y 3 al comparar las poblaciones de Las Bocas y San Carlos (Figs. 2 B, 3 B; Cuadro 3). En el grupo de cromosomas pequeños, las diferencias en el cociente q/p son evidentes en los pares 7, 8, 14 y 16, y también hay diferencias en la morfología y el tipo de cromosoma (Cuadro 3).
En las plantas de Las Bocas, el par de cromosomas número 7 fue subtelocéntrico y en la de San Carlos metacéntrico; el par 8 en los individuos de Las Bocas es metacéntrico y subtelocéntrico en los de San Carlos. En los individuos de Las Bocas el par 14 corresponde a un metacéntrico y en los de San Carlos es subtelocéntrico. En los individuos de Las Bocas el par 16 es subtelocéntrico, pero metacéntrico en las plantas de San Carlos.
La media de la longitud de los cromosomas grandes es similar en las 2 poblaciones; sin embargo, se encontraron diferencias significativas para este parámetro entre las 2 poblaciones en los pares 17 (p = 0.026), 18 (p = 0.023), 26 (p = 0.039) y 28 (p = 0.037), y marginalmente significativa (p = 0.078) en el par 16 del grupo de cromosomas pequeños (Cuadro 2). En las plantas de A. aktites de Las Bocas, la constricción secundaria se encontró en el par 3 de los cromosomas telocéntricos, mientras que en las plantas de San Carlos se evidenció en el par 2 de los cromosomas telocéntricos (Figs. 2 B y 3 B). Los valores de índice de asimetría (TF %) fueron similares en ambas poblaciones y la diferencia resultó no significativa (p = 0.497; Cuadro 1).
Se encontraron diferencias entre el cariotipo de las 2 poblaciones de A. aktites en el cociente de los brazos cromosómicos (q/p). La longitud total de los cromosomas y la posición de la constricción secundaria entre pares de cromosomas grandes evidencian cambios estructurales en algunos cromosomas grandes y pequeños del complemento cromosómico de estas plantas (Cuadros 2, 3; Figs. 2B, 3B).
La distancia entre las 2 poblaciones y las barreras físicas, como la vegetación, son factores que podrían limitar el flujo genético entre ellas, causar aislamiento geográfico o reproductivo y facilitar la diferenciación de las poblaciones debido a deriva genética. Moreno–Salazar et al. (2007), al estudiar el contenido de ADN y el cariotipo de poblaciones silvestres de Agave angustifolia y A. rhodacantha, encontraron que todas las plantas examinadas eran diploides con 2n = 2x = 60. Agave angustifolia mostró 2 diferentes citotipos estructurales en 3 poblaciones analizadas y A. rhodacantha tuvo el mismo cariotipo en las 2 poblaciones estudiadas. El cariotipo de las plantas de ambas especies fue bimodal. Los citotipos estructurales registrados para Agave angustifolia presentaron también un par de cromosomas grandes con constricción secundaria, al igual que el observado en el cariotipo de A. rhodacantha. En A. angustifolia, los citotipos estructurales observados muestran diferentes cariotipos en términos de proporción y tipo de cromosomas, y se encontraron tanto en el grupo de cromosomas grandes como pequeños.
Estos cambios en la estructura de los cromosomas son ocasionados por intercambios heterocigóticos espontáneos que ya se han registrado en varias especies de Agave (Palomino et al., 2005; Moreno–Salazar et al., 2007). En el caso de A. aktites, aunque la diferencia entre los cromosomas de las 2 poblaciones estudiadas es notable, no se encontraron citotipos estructurales diferentes, como se observó en poblaciones de A. angustifolia (Moreno–Salazar et al., 2007); sin embargo, es evidente que los genomas de las plantas de las poblaciones de A. aktites se encuentran en un proceso activo de diferenciación que podría finalmente llevar a la especiación.
Agradecimientos
A la Red de Agaváceas SAGARPA, SINAREFI, por el financiamiento parcial de estas investigaciones. Al Dr. Alejandro Ortega Corona, por la identificación y colecta de las plantas; al Dr. Fernando Chiang, por la revisión del manuscrito; a Miriam Ladd, por su asistencia técnica y a Felipe Villegas, por la preparación de la figura 1.
Literatura citada
Bennett, M. D., P. Bhandol e I. J. Leitch. 2000. Nuclear DNA amounts in angiosperms and their modern uses–807 new estimates. Annals of Botany 86:859–909. [ Links ]
Bennett, M. D. e I. Leitch. 2005. Plant DNA C– values database http://www.rbgkew.org.uk/cval/homepage.html; (última consulta: 4.X.2005). [ Links ]
Brandham, P. E. 1969. Inversion heterozygosity and sub–chromatid exchange in Agave stricta. Chromosoma (Berlin) 26:270–286. [ Links ]
Brandham, P. E. y M. J. Doherty. 1998. Genome size variation in the Aloaceae, an angiosperm family displaying karyotypic orthoselection. Annals of Botany 82 (Supplement A):67–73. [ Links ]
Cavallini, A., L. Natalli, G. Ciononi e I. Castorena–Sánchez. 1996. Cytophotometric and biochemical analyses of DNA in pentaploid and diploid Agave species. Genome 39:266–271. [ Links ]
Castorena–Sánchez, I., R. M. Escobedo y A. Quiroz. 1991. New cytotaxonomical determinants recognized in six taxa of Agave in the section Rigidae and Sisalane. Canadian Journal of Botany 69:1257–1264. [ Links ]
Conger, D. D. y L. M. Fairchield. 1953. A quick freeze method for making smear slides permanent. Stain Technology 28:281–283. [ Links ]
Dolezel, J. 1997. Aplication of flow cytometry for study of plant genomes. Journal of Applied Genetics 38: 285–302. [ Links ]
Dolezel, J., J. Greilhuber, S. Lucretti, A. Meister, M. A. Lysak, L. Nardi y R. Obermayer. 1998. Plant genome size estimation by flow cytometry: inter–laboratory comparison. Annals of Botany 82 (Supplement A):17–26. [ Links ]
Galbraith, D. W., G. M. Lambert, J. Macas y J. Dolezel. 1998. Analysis of nuclear DNA content and ploidy in higher plants. In Current protocols in cytometry, J. P. Robinson, Z. Darzynkiewicz, P. N. Dean, L. G. Dressler, A. Orfao, P. S. Rabinovitch, C. C. Stewart, H. J. Tanke y L. L. Wheeless (eds.). Wiley, New York. p. 1–22. [ Links ]
García–Mendoza, A. 2002. Distribution of Agave (Agavaceae) in México. Cactus and Succulent Journal 74:177–188. [ Links ]
García–Mendoza, A. y F. Chiang. 2003. The confusion of Agave vivipara L. and A. angustifolia Haw., two distinct taxa. Brittonia 55:82–87. [ Links ]
Gentry, H. S. 1982. Agaves of continental North America. The University of Arizona Press, Tucson. 670 p. [ Links ]
Greilhuber J., J. Dolezel, M. Lysak y M. D. Bennett. 2005. The origin, evolution and proposed stabilization of the terms "genome size" and "C value" to describe nuclear DNA contents. Annals of Botany 95:255–260. [ Links ]
Levan, A., K. Fredga y A. A. Sandberg. 1964. Nomenclature for centromeric position on chromosome. Hereditas 52:201–220. [ Links ]
Moreno–Salazar, S. F., M. Esqueda. J. Martínez y G. Palomino. 2007. Tamaño del genoma y cariotipo en Agave angustifolia y A. rhodacantha de Sonora, México. Revista Fitotecnia Mexicana 30:13–23. [ Links ]
Palomino, G., J. Dolezel, I. Méndez y A. Rubluo. 2003. Nuclear genome size analysis of Agave tequilana Weber. Caryologia 56:37–46. [ Links ]
Palomino, G., J. Martínez e I. Méndez. 2005. Citotipos en Agave angustifolia Haw. determinados por citometría de flujo y análisis de sus cariotipos. Revista Internacional de Contaminación Ambiental 21 (Suplemento 1):49–54. [ Links ]
Palomino, G., J. Martínez e I. Méndez. 2007. Variación inter e intraespecífica en especies de Agave por citometría de flujo y análisis de sus cromosomas. In En lo ancestral hay futuro: del tequila, los mezcales y otros Agaves, P. C. García–Marín, A. L. Saavedra, L. E. Eguiarte y D. Z. Villareal (eds.). Centro de Investigación Científica de Yucatán / Consejo Nacional de Ciencia y Tecología / Comisión Nacional para el Conocimiento y Uso de la Biodiversidad / Secretaría del Medio Ambiente y Recursos Naturales / Instituto Nacional de Ecología, Mérida, Yucatán, p. 41–65. [ Links ]
Palomino, G., J. Martínez e I. Méndez. 2008. Karyotype studies in cultivars of Agave tequilana Weber. Caryologia 61:144–153. [ Links ]
Pinkava, D. J. y M. A. Baker. 1985. Chromosome and hybridization studies of Agaves. Desert Plants 7:93–100. [ Links ]
Ruvalcaba–Ruiz, D. y B. Rodríguez–Garay. 2002. Aberrant meiotic behavior in Agave tequilana Weber var. Azul. BioMed Central Plant Biology 2:1–4. [ Links ]
Sinha, S. S. N. y H. Roy. 1979. Cytological studies in the genus Phaseolus I. Mitotic analysis in fourteen species. Cytologia 44:191–199. [ Links ]
White, M. J. D. 1978. Modes of speciation. Freeman, San Francisco, California. p. 455. [ Links ]
Zonneveld, B. J. M. 2003. The systematic value of nuclear DNA content in Agave L. and some related genera of the Agavaceae. Bradleya 21:121–125. [ Links ]














