INTRODUCCIÓN
La periodontitis es la principal causa relacionada con la pérdida de dientes en adultos, se inicia a partir de las bolsas periodontales que forman una biopelícula polimicrobioma que interactúa con células del huésped y puede conllevar la inflamación crónica del periodonto, la destrucción del hueso alveolar, la pérdida de la masa ósea,1-3 e incluso se ha asociado con varias patologías sistémicas.2-4
En principio, la composición cualitativa de la microbiota oral está asociada con el estado de salud del huésped, el periodonto se mantiene sano, en gran parte debido a los numerosos mecanismos de protección del huésped que operan en la cavidad oral y la simbiosis de las bacterias comensales que inducen una respuesta inmune proporcionada a la biomasa escasa, resolviendo la inflamación;4 sin embargo, algunas bacterias patógenas, como las que forman parte del complejo rojo (Porphyromonas gingivalis, Tan-nerella forsythia y Treponema denticola), evaden el sistema inmune y se ocasiona una respuesta inmune desregulada a causa de los factores de virulencia y los patrones moleculares asociados a daño (DAMP), lo que ocasiona la disbiosis franca patogénica e hiperinflamación por la alteración de procesos como estrés oxidativo, secreción de citocinas proinflamatorias, prostaglandinas y metaloproteinasas (MMP), eso provoca fallo en la resolución de la inflamación y la pérdida de la homeóstasis, lo que conlleva un daño al tejido conectivo, el hueso y la periodontitis crónica.1,4,5
El papel que juegan la comunidad de la biopelícula polimicrobiana y la perpetuidad del proceso inflamatorio es de vital importancia,5-7 por lo que la identificación y clasificación bacteriana es importante para definir la microbiota en pacientes sanos y/o enfermos, lo que ayudará a instaurar un tratamiento adecuado.8 Dentro de las bacterias odontopatógenas involucradas en la periodontitis, se encuentra Porphyromonas gingivalis, que orquesta el desarrollo de la enfermedad periodontal convirtiendo una comunidad microbiana benigna en una disbiótica.9 Desafortunadamente, algunas de esas especies bacterianas Gram negativas, anaeróbicas subgingivales, Aggregatibacter actinomycetemcomitans, son resistentes a algunos antibióticos como la clindamicina.7 Por ello, la eliminación mecánica habitual de todos los depósitos bacterianos de las superficies bucales no descamativas es un requisito fundamental para prevenir enfermedades.10
Por lo anteriormente expuesto, el estudio de la comunidad microbiana involucrada en la periodontitis ha tenido gran avance durante los últimos años,4,6,10 incluyendo las técnicas de cultivos estandarizados y el desarrollo de cultivos anaeróbicos, la microscopia de fuerza atómica11 y las técnicas de diagnóstico molecular como la reacción en cadena de la polimerasa (PCR), la pirosecuenciación y secuenciación de Sanger;3,10,12 esta última es una técnica de diagnóstico molecular sensible y específica que analiza la secuencia del ARN ribosómico 16S que detecta gran cantidad de especies bacterianas presentes en las muestras de pacientes sanos o enfermos.1,3,10,12
El objetivo de este trabajo fue identificar bacterias asociadas con la enfermedad periodontal empleando un marcador genético para 16S de la subunidad ribosomal bacteriana.
MATERIAL Y MÉTODOS
Población de estudio
El presente estudio fue realizado en las clínicas interdisciplinarias en la Unidad de Especialidades Odontológicas de la Secretaría de la Defensa Nacional, en el periodo comprendido de septiembre de 2015 a junio del 2016. El estudio incluyó a 80 pacientes entre 20 y 70 años de edad, todos los participantes autorizaron ser parte del estudio y firmaron un consentimiento informado. El Comité Institucional de Investigación y Ética de la Unidad de Especialidades Odontológicas y de la Escuela Militar de Graduados de Sanidad aprobaron el protocolo de estudio y consentimiento informado. Los pacientes fueron divididos en tres grupos de estudio, dos grupos que presentaron enfermedad periodontal, clasificados con base en el diagnóstico clínico utilizando la escala de Russell, un grupo incluyó 30 pacientes diagnosticados clínicamente con bolsa periodontal de 4 a 6 mm (periodontitis moderada), otro incluyó 20 pacientes diagnosticados clínicamente con bolsa periodontal > 7 mm (periodontitis severa) y un grupo de 30 pacientes periodontalmente sanos (grupo control).
Examinación clínica y toma de muestra
La examinación clínica se llevó a cabo por un periodoncista calibrado, quien determinó la profundidad de las bolsas periodontales sin generar sangrado de forma espontánea, con base en la escala de Russell y empleando sonda periodontal sencilla Williams calibrada como se muestra en la Figura 1 . Una vez evaluada la profundidad de la bolsa, se tomó la muestra de las bolsas periodontales más profundas. La toma de muestra para pacientes sanos se obtuvo con un Microbrush estéril considerando la muestra suficiente de la profundidad del surco gingival. Una vez tomadas las muestras, fueron colocadas en un tubo de polipropileno con 1 mL de buffer de fosfatos y se almacenaron a -70 oC en el Laboratorio de Biología Molecular hasta su análisis.
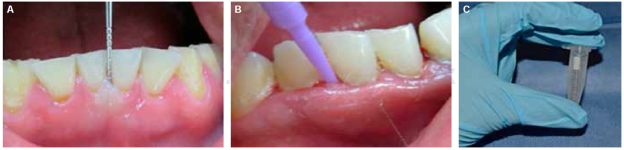
Figura 1 Examinación clínica y toma de muestra. A) Exploración clínica intraoral con sonda periodontal. B) Toma de muestra en pacientes con bolsas periodontales. C) Almacenamiento de la muestra con PBS1X.
Los criterios de inclusión fueron los siguientes:
Pacientes con periodontitis, clasificados con base en los criterios del índice periodontal de Russell; es de decir, la presencia de bolsas periodontales con una profundidad de sondeo > 4 mm, así como pacientes periodontalmente sanos (o que presenten bolsas periodontales).
Pacientes derechohabientes de la Unidad de Especialidades Odontológicas.
Los criterios de exclusión fueron los siguientes:
Extracción de ADN, amplificación de ARNr 16S, secuenciación e identificación
La extracción de ADN se realizó utilizando un kit comercial (DNeasy Blood and Tissue Kit, Qiagen®). La concentración y la pureza del ADN fueron determinadas con el espectrofotómetro NanoDrop 1000 (Thermo Scientific). La evaluación de la integridad del ADN se realizó con la amplificación del gen p-globina. La amplificación de la ADNr 16S se llevó a cabo utilizando los cebadores universales DG74: 5' AGGAGGT-GATCCAACCGCA3' y RW01 5' AACTGGAGGAA-GGTGGGGAT3',13 en un volumen final de 25 ¿vL que contenía 5 ¿vL de ADN (aproximadamente a 50 ng/i/L), 0.5 U de Taq polimerasa (Invitrogen), 1 mM de cada cebador, 3.75 mM de MgCl2, 0.2 mM de cada dNTP y buffer de Taq polimerasa. La amplificación se llevó a cabo en un termociclador Amp PCR System 9700 (Applied Biosystems) usando las siguientes condicio nes de reacción: una desnaturalización inicial de 5 min a 94 oC, seguida de 30 ciclos de desnaturalización a 94 oC por 30 s, alineamiento a 62 oC por 30 s y extensión a 72 oC por 30 s. Finalmente, se llevó a cabo una extensión a 72 oC por 5 min. Se utilizó como control positivo de bacteria el ADN de Helicobacter pylori (ATCC 43504). Los amplicones se purificaron usando ExoSAP-IT (USB) y se secuenciaron con un secuenciador automático ABI PRISM 3130 (Applied Biosystems) usando el kit de secuencia ABI PRISM BigDye Terminator v3.1 (Applied Biosystems). Las secuencias fueron comparadas usando la aplicación del BLAST de la base de datos GenBank http://www.ncbi.nlm.nih.gov/blast/Blast.cgi.
RESULTADOS
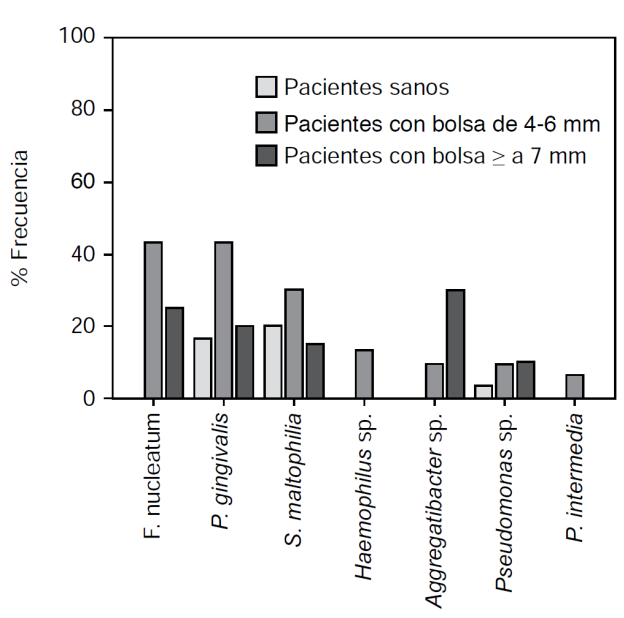
Los datos moleculares permitieron identificar bacterias con identidad alta (mayor del 80%) en las muestras analizadas como se observa en la Figura 2 .

Figura 2. Resultados moleculares representativos. A) Gel de agarosa al 2%, M representa el marcador de tamaño molecular 1 Kb, (-) blanco de reactivos, (+) control positivo de H. pylori (ATCC 43504) y 1-11 muestras representativas de pacientes con periodontitis. B) Electroferograma de secuenciación. C) Identidad reportada en NCBI/ BLAST Assembled Genomes del 97% para S. maltophilia.
Las bacterias detectadas en este estudio fueron: Stenotrophomonas maltophilia, Porphyromonas gingivalis, Pseudomonas sp., Fusobacterium nucleatum, Haemophilus sp., Aggregatibacter sp. y Prevotella intermedia, las cuales tuvieron una distribución diferente entre los grupos de estudio (Cuadro I y Figura 3), encontrando asociación entre la presencia de P. gingivalis y el grupo de periodontitis moderada (OR = 3.8, IC = 1.2-12.7, p = 0.02) y periodontitis severa (OR = 11.6, IC = 3.0-45.2, p < 0.001). Igualmente, se asoció la presencia de F. nucleatum con periodontitis moderada y (OR = 47.1, IC = 2.6-841, p = 0.008) y periodontitis severa (OR = 21.6, IC 1.1-417, p = 0.04). Asimismo, se asoció la presencia de Aggregatibacter sp. con periodontitis severa (OR = 27.3, IC 1.4-519, p = 0.02).
Cuadro I Frecuencia y distribución de las bacterias en los grupos de estudio.
| Bacteria | Sanos n = 30 (%) | Periodontitis moderada n = 30 (%) | Periodontitis severa n = 20 (%) | p |
| Stenotrophomonas maltophilia | 6 (20) | 9 (30) | 3 (15) | 0.423 |
| Porphyromonas gingivalis | 5 (16.6) | 13 (43.3) | 4 (20) | 0.047* |
| Pseudomonas sp. | 1 (3.3) | 3 (10) | 2 (10) | 0.548 |
| Fusobacterium nucleatum | 0 | 13 (43.3) | 5 (25) | < 0.001* |
| Haemophilus sp. | 0 | 4 (13.3) | 0 | 0.03* |
| Aggregatibacter sp. | 0 | 3 (10) | 6 (30) | 0.004* |
| Prevotella intermedia | 0 | 2 (6.6) | 0 | 0.181 |
* Significancia estadística entre los grupos.
DISCUSIÓN
La comunidad microbiana oral representa la mejor asociación con la especie humana, ya que se ha encontrado una fuerte correlación entre la composición cualitativa de la microbiota oral y los pacientes sanos o enfermos.4
Normalmente, el periodonto se mantiene saludable debido a los numerosos mecanismos de protección del huésped que operan en la cavidad oral, habiendo una relación simbiótica entre las bacterias y el huésped.1
Los resultados en la identificación bacteriana en general mostraron bacterias reportadas por otros autores tanto en pacientes sanos como enfermos;6 sin embargo, la frecuencia de ellas y sus asociaciones fueron características en cada grupo de estudio, sugiriendo como está descrito en la patología que un desbalance de microorganismos puede conducir a enfermedad.4
Cabe destacar que en los tres grupos de estudio fue detectada P. gingivalis, una bacteria que conforma el denominado complejo rojo con Tannerella forsythia y Treponema denticola, el cual se ha asociado con periodontitisagresiva;3 sin embargo, la frecuencia de esta bacteria fue más alta en el grupo de pacientes con periodontitis moderada y su presencia se asoció con los dos grupos de periodontitis.
Curtis y Darveau, en 2011, mencionan que en pacientes sanos se ha encontrado a P. gingivalis, lo que indica que su presencia sola no es la responsable de la enfermedad,3 otros grupos han descrito que P. gingivalis puede interaccionar con Strepto-coccus gordonii, y favorecer la colonización de otros patógenos conduciendo a la enfermedad.14 En este estudio, se observó que P. gingivalis y F. nucleatum fueron detectadas con mayor frecuencia en pacientes con periodontitis moderada y se asociaron con los dos tipos de periodontitis. Esta evidencia sugiere que la asociación de estas bacterias podría estar favoreciendo la colonización de otros patógenos, tales como Aggregatibacter sp., la cual predominó y se asoció a periodontitis severa, como se ha documentado en otros reportes.15 En periodontitis severa también predominó y se asoció F. nucleatum, lo que también podría sugerir que la asociación entre Aggregatibacter sp. y F. nucleatum es idónea para sobrevivir y florecer en el periodonto y progresar a una periodontitis crónica destructiva en la que se da una desregulación del proceso inflamatorio que a su vez proporciona nuevos nutrientes para las bacterias, los cuales son derivados de la destrucción del tejido, como lo han reportado otros autores.9,16
Aggregatibacter actinomycetemcomitans es una bacteria que produce leucotoxina, la cual lisa los neutrófilos humanos, lo que ocasiona un desbalance en la respuesta inmune innata y, por otro lado, la respuesta inflamatoria persiste en el periodonto;17 sin embargo, el estado de salud en general del paciente, factores de riesgo como la falta de higiene oral, el tabaquismo, la obesidad, el estrés y las posibles asociaciones genéticas son importantes para el desarrollo de la patología.18
Con respecto al resto de las bacterias detectadas aquí, se reporta la presencia en este padecimiento, pero no como el principal causante, sino como bacterias que manejan una simbiosis con otras, o bien como oportunistas de otras bacterias responsables de la periodontitis como es el caso de Stenotrophomonas maltophilia que se ha reportado incluso como oportunista en gingivitis en pacientes con leucemia.19 Respecto a Pseudomonas sp., Haemophilus sp. y Prevotella intermedia, se han encontrado también en cavidad bucal de pacientes sanos; sin embargo, su frecuencia es mayor en pacientes con periodontitis.20,21
Los hallazgos presentados aquí resaltan la importancia de realizar la exploración en las bolsas periodontales debido a la microbiota bacteriana relacionada con la patología, lo que conllevará dar un diagnóstico y tratamiento antimicrobiano más específico en pacientes con periodontitis.











 nueva página del texto (beta)
nueva página del texto (beta)