INTRODUCCIÓN
El desarrollo y la utilización de técnicas no invasivas para la identificación de biomarcadores es una herramienta promisoria para el diagnòstico tanto de enfermedades de la cavidad bucal como del estado de salud general.1 En este sentido la saliva rápidamente ha emergido como una fuente no invasiva con importante información biológica;2,3 ésta se forma luego de la filtración de la sangre en las glándulas salivales, conteniendo agua, electrolitos, proteínas, microorganismos y material genético, incluyendo tanto ácido desoxirribonucleico (ADN) y ácido ribonucleico (ARN) en la porción celular y en las células libres.4 Como producto de la filtración sanguínea, el material genético de las células libres en saliva se origina de múltiples fuentes del cuerpo, mientras que el proveniente de la fracción celular es derivado casi exclusivamente de la mucosa oral.5
El ARN puede estar en la cavidad bucal desde varias fuentes, incluyendo la secreción de saliva desde las glándulas salivales mayores y menores, fluido gingival crevicular y de la descamación de las células epiteliales orales.6 Probablemente varias secreciones de las glándulas salivales de micro- y macromoléculas y ARN podrían originarse desde las células acinares o por la circulación. Frecuentemente el proceso de descamación de las células epiteliales de la cavidad bucal podría ser otra fuente de ARN.7-9
La saliva es un componente importante para el habla, digestión y como medio de protección contra microorganismos.10 Adicionalmente biomarcadores para infecciones bacterianas, víricas y fúngicas han sido reportados en saliva tanto para enfermedades sistémicas como para las locales.11,12 Estos marcadores pueden ser de varias especies moleculares incluyendo desde proteínas y anticuerpos hasta ADN y ARN.6 Recientemente, el ARN obtenido de células libres en la saliva humana mostró que puede ser utilizada para estudios de biomarcadores de cáncer bucal,13-15 mediante técnicas de PCR, asimismo se ha sugerido que la células libres en la saliva de individuos saludables contienen más de 3,000 especies de ARNm.16
Poco se conoce acerca de la naturaleza molecular y las propiedades del ARN de la saliva humana;17 uno de los principales problemas es la cantidad de compuestos seleccionados en la saliva en comparación con la sangre, otra dificultad es la capacidad de distinguir entre los transcriptomas de origen humano y microbiano; además, la limitada cantidad de ARN salival requiere de métodos más sensibles y específicos.18 Por otro lado, se ha sugerido que la saliva contiene ribonucleasas de varias fuentes, lo que posiblemente podría dificultar el análisis de ARN en saliva,6 aún no está totalmente claro cómo el ARN y las ribonucleasa pueden coexistir en la saliva, una posible explicación es que el ARN salival endógeno es protegido de la degradación igualmente como pasa con el ARN del plasma;19,20 asimismo se ha reportado que la saliva contiene cadenas fraccionadamente degradadas de mARN18 y que la degradación de ARN en la saliva es relativamente lento en comparación con la degradación del ARN endógeno,6 lo que sugiere que existen mecanismos de estabilización de ARN salival. Todos estos obstáculos se están abordando en la actualidad, y debido a este hecho, se podría esperar un creciente interés en la investigación con ARN salival.
Entre las ventajas que tiene el uso de saliva en comparación con las muestras de sangre se han propuesto que el método de recolección es seguro, indoloro y no traumático, la técnica de recolección es fácil de manipular, no requiere de personal entrenado, el equipo de recolección es simple y la muestra de saliva puede ser tomada varias veces sin causarle incornodidad al paciente.21,22 Por tal motivo se hace necesario la estandarización de técnicas de biología molecular que utilicen como medio de estudio la saliva, lo que puede contribuir con el diagnóstico y pronóstico de algunas enfermedades, asimismo podría contribuir con la realización de investigaciones en el área de genómica que ayuden a comprender de una mejor manera la fisiopatologías de estas enfermedades desde una perspectiva molecular.
El objetivo de esta investigación fue estandarizar un protocolo de extracción de ARN en muestras de saliva en niños.
MATERIAL Y MÉTODOS
Participantes
Todos los 60 niños participantes eran atendidos en las clínicas odontológicas de la Universidad de Cartagena. La edad promedio fue de 6.8 (DE = 4.6) años. Los participantes no tenían antecedentes personales de enfermedades neoplásicas, inmunodeficiencias, desordenes autoinmunes o hepatitis. Para que todos los niños pudieran participar era necesario que algunos de sus padres aceptaran a partir de la firma de su consentimiento informado por escrito. Este estudio fue aprobado por el Comité de Ética de la Universidad de Cartagena.
Obtención de las muestras de saliva
Las muestras de saliva humana no estimulada se colectaron en tubos de polipropileno en hielo, en una cantidad de 3 mL. A todos los niños se les indicó el lavado de los dientes y la no ingesta de bebidas ni comida una hora antes de la colección salival. Todas las muestras se obtuvieron siguiendo el protocolo de salivación autónoma dentro del laboratorio de Bioquímica de Universidad de Cartagena, donde fueron almacenadas a -80 °С hasta su posterior análisis.
Conteo celular con trypan blue
Para este ensayo se tuvo en cuenta procedimientos reportados previamente.23 Se tomaron 20 μL saliva y se mezclaron con 20 μL de solución de trypan blue en un tubo Eppendorf de 0.6 mL. Se pipetearon 1 о μL de esta nueva suspensión en cada lado de un hemocitómetro. Luego en un microscopio óptico (Nikon SE) se observaron en un aumento de 10x y se cuantificaron las células de los cuatro cuadrantes ubicados en las esquinas de la cámara de Neubauer (BOECO), obteniendo un promedio del número de células, el cual fue multiplicado por el factor de dilución y por 10,000 para calcular el numero total de células en 1 mL de saliva.
Extracción de ARN en saliva
Se utilizaron y evaluaron los siguientes protocolos de extracción de ARN en saliva:
Protocolo A: en este método se utilizó el reactivo de lisis QIAzol® (TRIzol) de la marca Qiagen siguiendo las recomendaciones de los fabricantes con algunas modificaciones, inicialmente se añadió 500 μL de TRI-zol a 500 μL de muestra de saliva en tubos Eppendorf de 2 mL, se incubó a 25 °С por 10 min, luego se agregó 100 μL de una mezcla de cloroformo: isoamílico (24:1), haciendo vórtex por 15 segundos, se incubó a temperatura ambiente durante 5 min y se centrifugó a 10,000 rpm durante 10 min a 4 °С, luego se recolectó el sobrenadante en un nuevo tubo Eppendorf de 1.5 mL, al cual se le agregó 250 μL de isopropanol, haciendo vórtex por 15 segundos, se incubó overnight a 4 °С; después se centrifugó a 10,000 rpm durante 15 min a 4 °С, se eliminó el sobrenadante por inversión y se hicieron dos lavados al precipitado con 500 μL de etanol al 75%, vórtex por 15 segundos y centrifugación a 8,800 rpm durante 5 min a 4 °С, eliminando el sobrenadante por inversión, luego se secó el precipitado a temperatura ambiente. Por último, éste se resuspendió en 20 μL de agua libre de ribonucleasas y almacenó a -20 °С hasta su posterior análisis.
Protocolo B: en éste se empleó el RNeasy® Protect Saliva Mini Kit de la marca Qiagen, siguiendo las recomendaciones del fabricante, con algunas modificaciones, inicialmente al momento de la recolección se tomaron 200 μL de saliva y se les adicionó un 1 mL de ARN Protect Saliva Reagent en un tubo Eppendorf de 2 mL, se hicieron vórtex por 30 segundos, luego se centrifugò esta mezcla por 10 min a 12,000 rpm a 25 °С, se removió el sobrenadante utilizando puntas y micropipetas, al precipitado se le adicionaron 350 μL de Buffer RLT haciendo vórtex dos veces por 30 segundos, luego se agregaron 350 μL de etanol al 70% y después de una homogenizacion por pipeteo se transfirió este contenido a una columna RNeasy® MinElute Spin de 2 mL, centrifugación a 10,000 rpm por 15 segundos a 25 °С, se descarto el líquido que atravesó la columna y se adicionaron en ésta 350 μL de Buffer RW1, centrifugando a 10,000 rpm por 15 segundos a 25 °С, nuevamente se descartó el líquido que pasó a través de la columna y se adicionaron 500 μL de Buffer RPE, centrifugación a 10,000 rpm por 15 segundos a 25 °С, se descarto el líquido que atravesó la columna y se adicionaron en ésta 500 11L de etanol al 80%, centrifugación a 10,000 rpm por 2 min a 25 °С, se descartó el líquido que atravesó la columna y se realizó una centrifugación con la columna vacía a 12,000 rpm por 5 min a 25 °С, se descartó el líquido residual y luego se reemplazó el tubo de colección por uno nuevo, se adicionaron 14 μL de agua libre de ribonucleasas en todo el centro de la membrana de la columna y se centrifugó a 10,000 rpm por 1 min a 25 °С, se recolectó el ARN aislado y se guardó a -20 °С hasta su posterior análisis.
Características operativas evaluadas
Para ambos protocolos se midieron los siguientes parámetros:
Concentración de ARN: la valoración de la concentración del ARN fue realizada por espectrofotometría a 230, 260 y 280 nm en el equipo NanoDrop 2000 UV-Vis Spectrophotometer (Thermo Scientific). El valor de la concentración era proporcionada por el equipo, sin embargo, ésta era rectificada multiplicando por 40 la absorbencia a 260 nm. Estas mediciones se realizaron por duplicados para cada muestra.
Calidad de ARN: se tuvo en cuenta el grado de pureza obtenido de la relación de absorbencias A260/ A280 en el equipo NanoDrop 2000 UV-Vis Spectrophotometer (Thermo Scientific) del Laboratorio de Inmunología de la Universidad de Cartagena. Se consideró una relación de 1.5 a 2 como un indicativo de un buen grado de pureza y por tanto este ARN estaba entre un rango de calidad aceptable.
Rendimiento: fue determinado teniendo en cuenta la relación entre la concentración de ARN en saliva y el número de células para cada muestra. Considerado como la cantidad en ng de ARN entre el número de células en 1 mL de saliva.
Análisis de la expresión genica mediante RT-PCR
Luego con el protocolo que presentó mejores resultados, el ARN total se utilizó para obtener el cDNA, el cual se utilizó para la amplificación por PCR convencional utilizando los «primers» (5'-ATCACCGTCAC-CACCAGCGT-373'-GTCCCATTCGCCACGACTTT-GT-5') correspondientes al gen de la glucuronidasa beta (GUSB), dNTPs (10 mM), MgCI2 (25 mM), y una unidad de la polimerasa Ecotaq. La reacción de PCR se realizó en un termociclador (BIO-RAD T100™) bajo las siguientes condiciones: 94 °С durante cinco minutos, 40 ciclos a: 94 °С durante 30 segundos, 62.5 °С durante 30 segundos, 72 °С durante un minuto y un tiempo de elongación final a 72 °С durante 10 minutos.
Los productos de PCR se visualizaron mediante electroforesis en geles de agarosa al 1.0% en tampón TAE 1x (Tris 40 mM; 0.1% ácido acético glacial; EDTA 1 mM) conteniendo EZ-Vision® (Amresco®) y exposición a la luz uv en el equipo ChemiDoc™ XRS+ System. Todas las muestras fueron analizadas por triplicado.
Análisis estadístico
Fue iniciado a través del diseño de una base de datos en Excel Microsoft® Office 2010, luego ésta fue transportada al programa STATA® (StataCorp LP, Coliege Station, TX, USA). Inicialmente se evaluó el supuesto de normalidad de los datos a partir del test de Shapiro Wilk. Para el análisis descriptivo fueron usadas las medidas de tendencia central, dispersión y las proporciones. Para la comparación entre los protocolos se utilizó la prueba t-Student para variables cuantitativas y la prueba χ2 para las variables cualitativas. Para todas las pruebas se asumió una probabilidad límite de decisión de 0.05.
RESULTADOS
Al analizar la cantidad de células por mL de saliva se encontró una media de 564,977.8 (DE = 246,678.6) células/mL, con un rango que osciló entre un valor mínimo de 242,500 células/mL y máximo 1,487,500 células/mL (Figura 1).
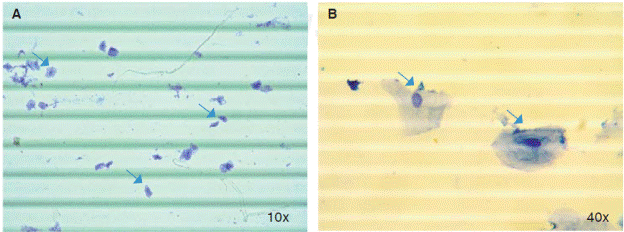
Figura 1 Células epiteliales en saliva. Cuantificación celular en suspensión de trypan blue. А) у В) Ildentificación lde células epiteliales en muestras de saliva (flechas) utilizando microscopio óptico con objetivos de 10х y 40x respectivamente.
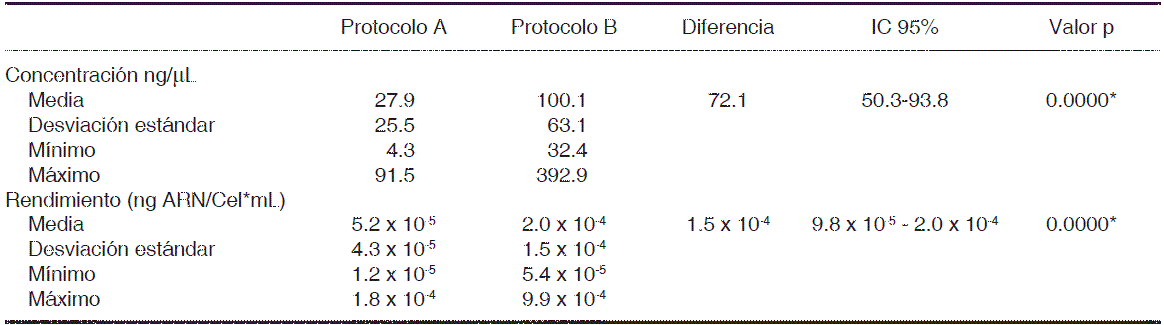
Respecto a las características operativas de los dos protocolos evaluados para la extracción de ARN en saliva se encontró que la media global de concentración de ARN aislado de muestras de saliva fue de 71.0 (DE = 62.4) ng/μL en donde el valor mínimo fue de 4.3 ng/μL y el maximo de 392.9 ng/μL, para el protocólo A (QIAzol®) la media de concentración fue de 27.9 (DE = 25.5) ng/μL y para el protocolo B (RNeasy® Protect Saliva Mini Kit-Qiagen) fue de 100.1 (DE = 63.1) ng/μL; para el rendimiento la media global fue de 1.4 X 10 -4 (DE = 1.3 X 10-4) ng ARN/Cel*mL con un valor mínimo de 1.2 X 10-5 ng ARN/Cel*mL y un máximo de 9.9 X 10-4 ng ARN/CeľmL, para el protocolo A fue de 5.2 X 10-5 (DE = 4.3 X 10-5) ng ARN/Cel*mL y para el protocolo В de 2.0 X 10-4 (DE = 1.5 X 10-4) ng ARN/ Cel*mL (Cuadro I). Al comparar los protocolos de extracción de ARN en muestras de saliva se encontraron diferencias estadísticamente significativas para la concentración de ARN (p = 0.0000) y rendimiento (p = 0.0000) (Figura 2), en donde el protocolo B mostro mejores resultados que el protocolo A.
Protocolo A (QIAzol®) у B (RNeasy® Protect Saliva Mini Kit-Qiagen). *Estadísticamente significativo (p = 0.0000), prueba t-Student.
Cuadro I Comparación de protocolos para extracción de ARN en saliva.
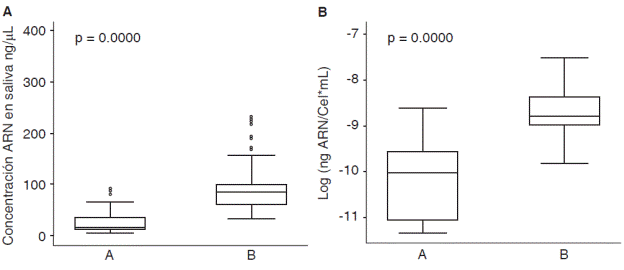
Figura 2 Concentración y rendimiento de los protocolos. A) Concentración de ARN en saliva por protocolos. B) Rendimiento de los protocolos de extracción de ARN en saliva. Protocolo A (QIAzol®) y B (RNeasy® Protect Saliva Mini Kit-Qiagen). Prueba t-Student.
Con respecto a la calidad del ARN extraído en saliva el 41.1% se ubicó entre los rangos óptimos de 1.5 a 2.0 (A260/280), para el protocolo A el cumplimiento de esta condición fue del 62.3% y para el protocolo в de 44.1%; al comparar esta característica con el tipo de protocolo no se observaron diferencias estadísticamente significativas entre éstos (p = 0.146) (Figura 3).
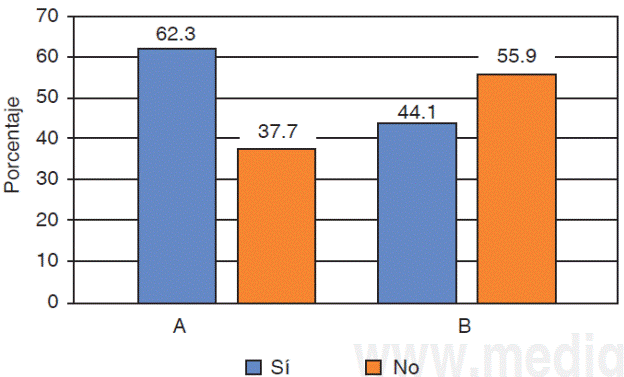
Figura 3 Calidad de ARN según protocolos de extracción. Protocolo A (QIAzol®) у B (RNeasy® Protect Saliva Mini Kit-Qiagen). Prueba χ2 (ρ = 0.146).
Al evaluar el comportamiento de los protocolos según sus características operativas en conjunto se observó que el protocolo в mostró un mejor comportamiento que el protocolo A, ya que en términos generales se obtuvieron mayores concentraciones de ARN en ng/μL empleando una menor cantidad de células/ mL de saliva; asimismo con este protocolo se evidenció la expresión génica del gen normalizador GUSB (Figura 4).
DISCUSIÓN
Respecto a los procedimientos para extracción de ARN en muestras de saliva se logró estandarizar un método, sugiriendo que la saliva es un excelente medio para estudios de biología molecular, lo que concuerda con lo reportado por Park6 quien caracterizó el ARN salival. Asimismo previamente Li16 reportó la presencia de mARN humano en muestras de saliva; de igual forma Chiang,24 además, reconoció que la estabilización y el procesamiento del transcriptoma es un desafío crítico para el estudio de biomarcadores salivales debido a la naturaleza ubicua de nucleasas y proteasas, así como la inestabilidad inherente de estos biomarcadores. Por estos motivos está estandarización se considera un avance y aporte a la investigación, lo que contribuirá a la realización de otras investigaciones que evalúen biomarcadores salivales y estudios de mecanismos moleculares asociados con enfermedades sistémicas y bucales.
Los resultados sugieren que el método que utilizó RNeasy® Protect Saliva Mini Kit-Qiagen mostró mejores características de concentración de ARN y rendimiento al compararlos con el que utilizó QIAzol®, lo que se podría explicar teniendo en cuenta que el RNeasy® Protect Saliva Mini Kit-Qiagen contiene una solución protectora que estabiliza el ARN salival, considerada por Fabryova18 como el mejor reactivo de estabilización de ARN salival disponible comercialmente en la actualidad, esto se determinó mediante la comparación de los valores de Ct de las muestras de ARN tratadas con soluciones estabilizadoras y almacenadas durante 10 semanas a temperatura ambiente.25 La utilización de estas soluciones de estabilización es el primer paso fundamental para el posterior análisis de extracción de ARN salival, debido a que se ha reportado la existencia de ribonucleasas salivales que siguen activas después de la recolección de la saliva, lo que justifica su inhibición antes de procesar dichas muestras.18 Otra posible explicación sería que este método utiliza entre sus reactivos el carrier de poly-А, el cual contiene una proteína de unión al ARN que se une a la cola poli (A) del ARNm, ubicada en el extremo 3', éste es un mecanismo de protección de ARN ampliamente conocido, se ha sugerido que en eucariotas muchas proteínas se unen directa o indirectamente al ARN para lograr la estabilización del mismo. Más del 30% del ARNm que se encuentra en la saliva humana contiene áreas ricas en adenina y uridina, lo que incrementa la estabilidad del ARN, siendo cinco veces más que el encontrado en otros mARNs del organismo humano;26 asimismo áreas ricas en A y Т en el extremo 3' lo estabilizan antes de la traducción, teniendo en cuenta estos mecanismos Khabar27 afirma que el ARNm salival promedio conserva el 42% de su longitud original.
Por otro lado, Pandit28 quien también comparó dos métodos de extracción de ARN en saliva utilizando de igual forma uno con QIAzol® y el otro con un kit comercial afirma que el método con QIAzol® produce un ARN total de alto rendimiento a partir de saliva, arrojando una buena relación de la absorbancia medida a 260 nm y 280 nm, además, afirma que el kit comercial produjo un rendimiento de ARN 10 veces inferior. Por tanto, sugiere incluso la utilización de reactivo de lisis QIAzol® para aislar el ARN de muestras de saliva almacenadas sin inhibidores de ribonucleasas a -80 °С durante más de dos años. Asimismo Dietz4 quien comparó el Qiagen ARN Protect® Saliva Mini Kit y el Kit QIAamp Viral ARN Mini para la extracción de ARN de células libres de sobrenadante de la saliva neonatal, concluyó que aunque en los dos métodos el ARNm se extrajo y se amplificó a partir de todas las muestras de sobrenadante de la saliva, el método de Kit QIAamp Viral ARN Mini mostró mejores resultados en cuanto a la cantidad y concentración de ARN. En este sentido Maron29 comparó el rendimiento de ARN, la calidad, la estabilidad y el rendimiento de RT-qPCR para los sistemas Qiagen RNeasy® Protect Saliva Mini Kit y el ADN Genotek Oragene ARN® en saliva de recién nacidos; sugiriendo que aunque el ensayo de Qiagen puede reducir el tiempo de extracción general, el rendimiento de ARN y el rendimiento en el análisis de transcriptómica posterior es más robusto utilizando el ensayo de ADN Genotek; sin embargo, aclara que la integridad del ARN no difirió entre estos métodos.
Cabe aclarar que una posible explicación de estas diferencias se deba a las condiciones propias de cada experimento, operador y laboratorio; por lo cual se sugiere la utilización del método estandarizado en este estudio teniendo en cuenta las características y las condiciones donde se vaya a aplicar; en este sentido Grabmüller30 comparó cinco kits disponibles en el mercado de uso común para la extracción de ARN (kit de aislamiento mirVana™ miARN Ambion; Trizol® reactivos, Invitrogen; NucleoSpin® miARN Kit Macherey-Nagel; AllPrep DNA/ARN Mini Kit y RNeasy® Mini Kit ambos de Qiagen) para evaluar su eficacia relativa de producir ARN de buena calidad utilizando muestras de pequeñas cantidades de células entre las cuales está la saliva y la mucosa bucal; sugiriendo que aunque existen diferencias considerables entre los rendimientos de los valores de calidad de ARN y los niveles de expresión, en general, no existía un método mejor para satisfacer todas las demandas de los diferentes análisis de ARN y ADN, por tanto, al parecer cada método tiene méritos y desventajas específicas, por lo cual recomienda elegir con cautela los métodos disponibles y ajustar sus características con las necesidades del entorno experimental que se disponga. Del mismo modo, Sellin31 comparo el rendimiento de cinco kits disponibles comercialmente para la extracción de ARN total a partir de muestras con cantidades pequeñas de células, concluyendo que cada kit era generalmente capaz de extraer la cantidad de ARN requerida para aplicaciones de expresión génica и otros ensayos. Sin embargo, las diferencias en la calidad del ARN extraído a través de cada uno de los kits indican que éstos pueden diferir en su capacidad para producir ARN aceptable para algunas aplicaciones, sugiriendo que hay diferencias practicas entre los kits de extracción de ARN disponibles en el mercado que se deben tomar en cuenta al seleccionar los métodos de extracción para ser utilizado en el aislamiento de ARN que conducirá al análisis de la expresión génica.
En este sentido a través del método estandarizado en el presente estudio se logró realizar el análisis de expresión génica del gen normalizador GUSB, lo que sugiere la efectividad del método para extracción de ARN y la posibilidad de emplear la saliva con muestra de estudio de diferentes bio-marcadores en la población infantil, lo que tiene muchos beneficios entre los cuales está la fácil recolección, método indoloro y no invasivo. Asimismo Li13 en muestras salivales de personas saludables logró identificar la expresión genica de 185 tipos de transcriptos, los cuales fueron encontrados en cada individuo, del mismo modo se ha reportado que los transcriptos de B-actina, RPS9, gliceraldehído-3-fosfato deshidrogenase (GAPDH), IL8, espermidina/espermina y N1-acetiltransferasa están presentes en altas concentraciones en muestras saliva.16
En síntesis, el ARN salival es una herramienta prometedora debido a que puede proporcionar informa ción no sólo de la presencia de genes, sino también de su expresión, esto se podría explicar al hecho de que la saliva es un ultrafiltrado del plasma, lo que indica que también podría utilizarse para el diagnóstico de enfermedades no relacionadas con la cavidad bucal.18 En este sentido la saliva puede ser considerada como un medio viable para estudios moleculares, ya que se logró estandarizar un método de extracción de ARN en saliva de niños, específicamente el método que utilizó el RNeasy® Protect Saliva Mini Kit-Qiagen mostró mejores características de concentración de ARN y rendimiento con las condiciones propias del operador y el laboratorio, por lo cual se sugiere su utilización para estudios de señalización bioquímica y genómica poblacional que ayuden a comprender mecanismos moleculares, así como también la búsqueda de biomarcadores salivales de diferentes enfermedades sistémicas y bucales.











 text in
text in 



