Introducción
Actualmente existe una gran cantidad de microorganismos capaces de adquirir resistencia a múltiples antibióticos, lo cual propicia que la efectividad de los tratamientos para inhibir a las bacterias patógenas causantes de enfermedades transmitidas por alimentos (ETAS) y a bacterias de importancia en salud pública se vea disminuida.1
Lo anterior ha generado una problemática mundial que impacta directamente en el diseño y aplicación de tratamientos para mejorar la conservación de alimentos y tratar de minimizar el desarrollo y transmisión de organismos patógenos, además de incidir en los procedimientos médicos para tratar las enfermedades causadas por bacterias resistentes o multi- resistentes a diversos agentes antimicrobianos.2
La proliferación de cepas resistentes como consecuencia del uso de antibióticos es un proceso natural que se ha acelerado por la aplicación inadecuada de ciertos fármacos, y pese a ser un problema inevitable, diversas organizaciones como la Organización Mundial de la Salud (OMS) y los Centros para el Control y Prevención de Enfermedades (CDC) a través de los años se han propuesto implementar numerosas estrategias para prevenir y solucionar, o al menos tratar de retrasar este proceso.3
En respuesta a la mencionada problemática se ha impulsado una intensa labor de investigación para la búsqueda, identificación y desarrollo de nuevos agentes antimicrobianos y la modificación o implementación de novedosos protocolos y/o técnicas para evaluar adecuadamente su efectividad.4,5
Durante décadas, las investigaciones se han enfocado en determinar cuantitativamente la relación entre la dosis aplicada y el efecto de los antibióticos o los agentes suministrados individualmente o combinados, para conocer específicamente cuál combinación es la adecuada que muestre un efecto sinérgico.6 Sin embargo, todos los sistemas biológicos muestran una particular y extrema complejidad que ha suscitado el desarrollo de incontables modelos experimentales, y al mismo tiempo diversos y polémicos enfoques y conjeturas, de igual manera una constante controversia sobre las metodología y la interpretación de los resultados derivados de la combinación de antibióticos y otros compuestos antimicrobianos como infusiones herbales provenientes de plantas, iones metálicos y nanopartículas, endolisinas y principalmente bacteriocinas. Las bacteriocinas son péptidos antimicrobianos naturales sintetizados y secretados por las bacterias7 y son de los metabolitos microbianos considerados más prometedores para aplicarse como bioterapeúticos y atenuar la problemática de la resistencia a los antibióticos.4
Antibióticos: Mecanismos de acción y el inconveniente de la resistencia
Los antibióticos son moléculas derivadas del metabolismo de bacterias y hongos principalmente, pero también pueden ser compuestos obtenidos por síntesis química para inhibir el crecimiento o destruir a microorganismos causantes de infecciones. Selman Waksman fue el primero en acuñar el término "antibiótico" para designar a la estreptomicina en 1943, y desde entonces se aplica a los agentes que se utilizan para combatir las infecciones causadas por bacterias.8
Los antibióticos pueden ejercer su acción antimicrobiana a través de alguno de los cinco mecanismos que utilizan para actuar y causar daño en alguna de las rutas metabólicas esenciales para la sobrevivencia de las bacterias. Estos mecanismos incluyen: (1) la inhibición de la replicación, (2) la interrupción de la transcripción, (3) la inhibición de la síntesis de proteínas o traducción, (4) la inhibición la síntesis de peptidoglucano, el monómero estructural de la pared celular, (5) pueden ejercer su acción para modificar y/o interrumpir la permeabilidad de la membrana celular. Sin embargo, cuando un antibiótico aplicado en la dosis habitual es incapaz de causar daño en los microorganismos, se dice que ha adquirido resistencia al agente antimicrobiano.9
La resistencia antimicrobiana es la capacidad de las bacterias u otros microorganismos para contrarrestar el efecto de algún antibiótico; esta resistencia sobreviene cuando la bacteria sufre algún cambio que reduce o elimina la efectividad de antibiótico, compuestos químicos o cualquier otro agente destinado para curar o prevenir alguna infección. La resistencia puede ser una consecuencia evolutiva vía la selección natural, pero también es causada por el uso indiscriminado de los agentes antimicrobianos.
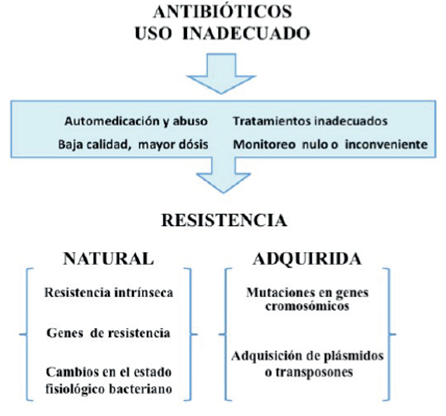
La OMS estima que aproximadamente el 50% de los antibióticos son administrados innecesariamente, lo cuál contribuye al aumento de la resistencia bacteriana que a su vez, incrementa la mortalidad por enfermedades infecciosas generando un grave problema de salud pública (Figura 1). Además, el uso inadecuado de los antibióticos representa un riesgo para la salud y un despilfarro de recursos económicos en los servicios de salud.10
Entre las bacterias más resistentes se reportan al Streptococcus pneumoniae que causa neumonía y es resistente a los antibióticos β-lactámicos, macrólidos y tetraciclinas, también se reportan a los agentes causales de enfermedades diarreicas como son Shigella dysenteriae, Salmonella typhi y Vibrio cholerae, además a Mycobacterium tuberculosis causante de la tuberculosis multirresistente a las drogas (MT-MRD) o tuberculosis extremadamente resistente a las drogas (MT-XRD).11,12
Las bacterias de mayor interés en el área de la salud pública son las que pueden transmitirse de persona a persona e incluyen bacterias resistentes a meticilina como Staphylococcus aureus (SARM), Enterococcus resistente a Vancomicina (ERV), bacterias gramnegativas multirresistentes (MR) como Klebsiella, Pseudomonas, Acinetobacter y algunas especies de Escherichia coli.13
Mecanismos de resistencia a los antibióticos
Las bacterias de manera natural pueden ser intrínsecamente resistentes a ciertos antibióticos pero también pueden adquirir resistencia a través de mutaciones en genes cromosómicos y por transferencia horizontal de genes14 (Figura 1).
Las bacterias pueden adquirir resistencia a los antibióticos a través de varios mecanismos moleculares14 que se dividen en tres grupos:
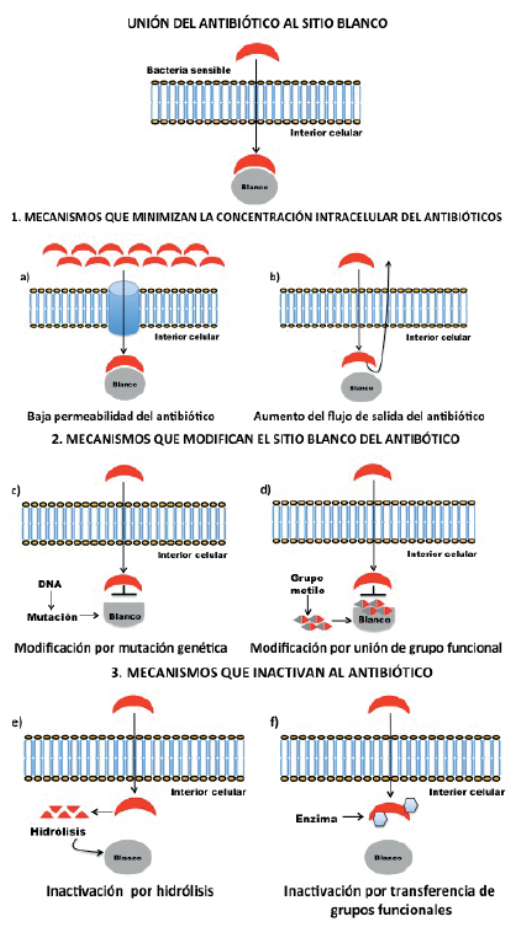
1) Mecanismos que minimizan las concentraciones intra- celulares del antibiótico. Se caracterizan principalmente por una baja penetración del antibiótico a la bacteria blanco debido a una baja permeabilidad, por ejemplo, en las bacterias Gramnegativas los antibióticos hidrofóbicos penetran la membrana externa por difusión a través de las proteína de membrana conocidas como porinas (Figura 2a). Cuando ocurre una baja expresión de las porinas en Pseudomonas spp. y Acinetobacter spp. disminuye la penetración del antibiótico y esto contribuye a la resistencia a nuevas drogas como las cefalosporinas.15 También el aumento del flujo de salida de un antibiótico contribuye a reducir su concentración intracelular al transportarlo fuera de la célula (Figura 2b), y por lo tanto, si ocurre una sobreexpresión de las bombas de eflujo puede promover altos niveles de resistencia a los antibióticos clínicamente útiles.
2) Mecanismos que modifican el sitio blanco del antibiótico. La modificación por una mutación genética altera el sitio de unión a un antibiótico, evitando así una unión eficiente, pero que le permite realizar su función normal, confiriendo resistencia en la bacteria (Figura 2c). Otra mutación causada por transformación bacteriana que forma genes "mosaico" puede conferir resistencia a los antibióticos como la penicilina en Streptococcus pneumoniae.16 También se puede modificar el sitio de unión del antibiótico para protegerlo, pero no por una mutación genética (Figura 2d). El ejemplo clásico son los aminoglucósidos, antibióticos que inhiben la síntesis de proteínas uniéndose al ribosoma, pero si se produce una metilación del sitio de unión al ribosoma además de proteger al antibiótico también genera la resistencia.
3) Mecanismos que inactivan al antibiótico por hidrólisis o modificación.13 La inactivación de los antibióticos a través de la hidrólisis es mediada por enzimas y es el principal mecanismo causante de resistencia antibacteriana. La inactivación enzimática es causada por la acción de hidrolasas y transferasas sintetizadas por ciertos patógenos como una estrategia de defensa bacteriana (Figura 2e). Se han identificado cientos de enzimas que degradan o modifican antibióticos β-lactámicos, aminoglucósidos, fenicoles y macrólidos.17
Los antibióticos también pueden inactivarse por la transferencia enzimática de grupos químicos (acilo, fosfato, nucleotidil y ribitoil) (Figura 2f), siendo los aminoglucósidos un grupo de antibióticos muy susceptible a la inactivación mediada por acetil-transferasas, fosfo-transferasas y nucleotidil-transferasas.18
Terminología básica y determinación de la susceptibilidad a los antibióticos
A nivel mundial diversos centros de referencia han publicado la terminología más idónea y las normas para realizar los ensayos pertinentes en relación al estudio de los agentes antimicrobianos y sus combinaciones. Estos centros incluyen al Instituto de Normas Clínicas y de Laboratorio (Clinical and Laboratory Standards Institute: CLSI), la Sociedad Británica de Quimioterapia Antimicrobiana (British Society for Antimicrobial Chemotherapy: BSAC) y el Comité Europeo de Pruebas de Susceptibilidad Antimicrobiana (European Committee on Antimicrobial Susceptibility Testing: EUCAST), los cuales además de estandarizar la terminología y las guías necesarias, proporcionan los valores de referencia que permiten una correcta y acertada interpretación de los resultados de las pruebas de susceptibilidad.19,20,21
Los métodos que permiten estudiar la sensibilidad bacteriana a los agentes antimicrobianos son las técnicas de dilución y de difusión. Las técnicas de dilución proporcionan resultados cuantitativos como el valor de la concentración mínima inhibitoria (CMI), y las técnicas de difusión proporcionan resultados basados en el efecto que causan en la bacteria blanco (resistencia, susceptibilidad intermedia o susceptibilidad)19 (Figura 3). Ambos métodos son comparables ya que existe una correlación directa entre el diámetro del halo de inhibición del crecimiento microbiano medida por difusión y la CMI determinada por dilución.20
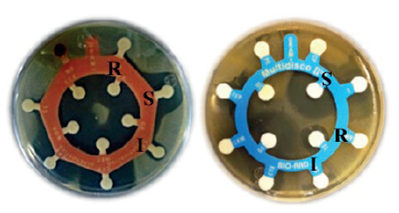
Figura 3 Antibiograma para bacterias Grampositivas (izquierda) y Gramnegativas (derecha) indicando Resistencia (R), Susceptibilidad (S) o Susceptibilidad Intermedia (I).
La resistencia (R) ocurre cuando un microorganismo es incapaz de verse afectado por elevadas dosis de algún antibiótico al que previamente era sensible. Las bacterias, los virus y algunos parásitos pueden desarrollar resistencia como un mecanismo de defensa o de competencia y ser inmunes a los efectos de los antibióticos, los antivirales o a los antipalúdicos respectivamente.1
Las cepas que presentan susceptibilidad intermedia (I), tienen una respuesta variable a un antimicrobiano y son clínicamente susceptibles y/o resistentes, pero pueden ser eliminadas si el antibiótico se concentra en el sitio de la infección o se aumenta la dosificación.
Una cepa susceptible (S) o sensible se refiere a las bacterias que se ven afectadas por los antimicrobianos como consecuencia de la pérdida o inactivación de sus mecanismos de defensa. De acuerdo a este término se puede inferir que un tratamiento con la dosis habitual del antimicrobiano puede detener la infección causada por la bacteria.22
Un concepto estandarizado en la terminología adoptada para el estudio de los distintos agentes antimicrobianos es el de concentraciones mínimas. La Concentración Mínima Inhibitoria (CMI) se considera la concentración más baja, expresada en mg/L necesaria para inhibir el crecimiento de un microorganismo bajo condiciones específicas in vitro en un período de tiempo establecido. Un derivativo de este término es la Concentración Mínima Inibitoria 50 (CMI 50) y representa la concentración de antimicrobiano que inhibe el crecimiento del 50% de las bacterias en un cultivo.23
Otro término común es la Concentración Bactericida Mínima (CBM) es la concentración más baja de un antibiótico, expresada en mg/L, que en condiciones definidas in vitro reduce el 99,9% (3 logaritmos) del número de microorganismos presentes en un medio de cultivo inoculado con un número conocido de bacterias en un determinado periodo de tiempo. La reducción se expresa como la proporción de inóculo o número de unidades formadoras de colonias (UFC) vivas que son incapaces de reproducirse en las condiciones del subcultivo en cierto período de tiempo, y esta proporción se puede expresar gráficamente con las curvas de tiempo de muerte.23
Mezclas binarias de antimicrobianos para combatir la resistencia
Una de las estrategias empleadas para combatir el alarmante fenómeno de la resistencia y multi-resistencia a los antibióticos comprende el desarrollo de esquemas terapéuticos innovadores para mejorar la eficacia de los tratamientos a través de la combinación de antibióticos tradicionales con nuevos compuestos antimicrobianos.
El efecto de la combinación entre agentes antimicrobianos puede ser aditivo, sinergismo, antagonismo o indiferencia.24,25
La indiferencia expresa que el efecto o actividad de una mezcla de antimicrobianos no es diferente a la actividad del agente de mayor actividad cuando actúa independientemente. El efecto de la combinación es igual a la suma de las actividades de dos antimicrobianos que actúan por separado, representa un efecto de adición.
El sinergismo indica que la actividad conjunta de dos antimicrobianos es mayor que la suma de las actividades individuales de los antimicrobianos.
El efecto contrario al sinergismo, el antagonismo, es la actividad que presentan dos antimicrobianos juntos y es significativamente menor que la suma de las actividades de los dos antimicrobianos cuando actúan separados.
El propósito de estudiar las combinaciones de antimicrobianos es determinar si existe sinergismo, lo que permitiría potenciar su aplicación para contrarrestar el desarrollo de resistencia en los microorganismos.26
Dentro de las aplicaciones más promisorias están los tratamientos terapéuticos contra enfermedades infecciosas en la salud pública y en la medicina veterinaria, además para el control de patógenos zoonóticos resistentes a los antimicrobianos presentes en los alimentos, los cuales constituyen un riesgo para la salud humana.27,28,29
Es importante resaltar que los efectos de las interacciones entre antimicrobianos se efectúan in vitro y la significancia clínica que pueden representar es todavía incierta. Sin embargo, la búsqueda de la combinación "perfecta" es la única esperanza para el tratamiento de pacientes que sufren infecciones difíciles de erradicar, o bien, para combatir a las bacterias resistentes a un antibiótico, pero sensibles a determinada concentración de mezclas binarias de antimicrobianos. Además, para la administración clínica de dos o más antimicrobianos combinados es preciso evaluar previamente los probables efectos a nivel de laboratorio y en sistemas in vivo.30
De acuerdo a lo anterior, el perfeccionamiento de considerables investigaciones básicas han realizado un gran esfuerzo para el desarrollo de variadas técnicas in vitro, que puedan evidenciar de manera muy sencilla, las interacciones entre mezclas binarias de agentes antimicrobianos como los antibióticos, y éstos últimos combinados con otras moléculas biológicas con actividad antimicrobiana y con menor posibilidad de generar resistencia.31,32
Sin embargo, los estudios in vitro no consideran las fluctuaciones de las concentraciones del fármaco en el organismo o el curso temporal de la actividad antibacteriana de la droga in vivo, y se requiere conocer la farmacocinética (PK) y la farmacodinámica (PD) de los antimicrobianos.33 La PK estudia la evolución de los fármacos y en los diferentes fluidos y tejidos del organismo con respecto al tiempo, así como el régimen de dosificación y las concentraciones plasmáticas resultantes, y la PD cuantifica la actividad del agente antimicrobiano en las concentraciones que se alcanzan en el lugar de acción, dependiendo del comportamiento farmacocinético, y de la sensibilidad del microorganismo al antibiótico, expresada como la concentración mínima inhibitoria (CMI).34
Durante décadas el análisis farmacocinético-farmacodinámico se ha considerado una excelente herramienta para evaluar un tratamiento antimicrobiano, utilizando modelos para determinar los indices de PK/PD validados experimentalmente in vitro e in vivo, y los resultados son la clave para el desarrollo de nuevos antibióticos.
Pruebas in vitro
Una de las pruebas más utilizadas in vitro para estudiar el comportamiento de sistemas binarios de agentes antimicrobianos es la titulación de tablero de ajedrez. Este ensayo debe su uso extensivo esencialmente a que se realiza de manera sencilla en el laboratorio y no requiere de cálculos matemáticos elaborados, se representan los resultados con una gráfica solamente35 (Figura 4a).
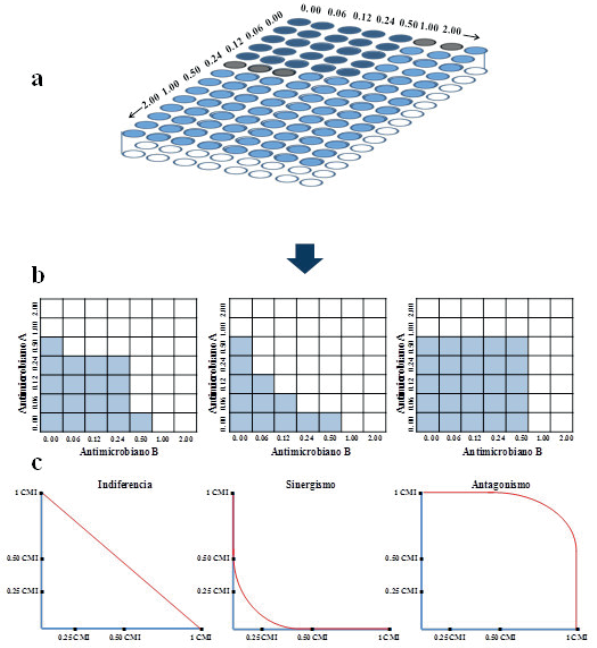
Figura 4 Técnica del tablero de ajedrez para determinar in vitro las interacciones entre mezclas binarias de antimicrobianos. a) Diluciones de los antimicrobianos A y B, agregando el cultivo de microorganismo sensible (~104 UFC/mL) excepto en la primera casilla (control); b) Representación gráfica de las diluciones del antimicrobiano A y B (0, 0.06, 0.12, 0.25, 0.50, 1CMI y 2CMI); c) Representación de la interacción entre dos antimicrobianos mediante un isobolograma.
El ensayo se realiza en placas de microtitulación en cuyos pocillos se introducen el cultivo de estudio, sea la cepa indicadora o microorganismo a determinar susceptibilidad a una determinada concentración (~104 UFC/mL), además de las diluciones de los antimicrobianos en estudio (A y B) (Figura 4a), incubando la placa a las condiciones de temperatura y tiempo óptimas de crecimiento (30-35 °C/16-24h). Estas deben contener concentraciones (mg/L) que incluyan valores mayores y valores menores a la CMI determinada previamente para cada agente por separado contra la bacteria indicadora o microorganismo a estudiar (Figura 4b). Gráficamente en el eje de las abscisas se ubican las diluciones del antimicrobiano A (0, 0.06, 0.12, 0.25, 0.50, 1 y 2 veces la CMI) y en el eje de las ordenadas se distribuyen las mismas diluciones del antimicrobiano B, de forma que la primera casilla es la correspondiente a una valor de cero (control) que multiplica al valor de la CMI de A y B (Figura 4c).
Una vez que se titulan los pocillos con antimicrobianos de concentración conocida (1/n) y se determinan aquellas CMI's que ejercen el efecto esperado en presencia de A con B y viceversa, se determina la Concentración Fraccional Inhibitoria (CFI) de cada componente basándose en las CMI's individuales. Esta es una expresión matemática utilizada para determinar cual de los efectos mencionados anteriormente posee la combinación de los antimicrobianos, de acuerdo a la suma de las CFI individuales (Figura 5) y en la cual se pueden obtener valores de ≤ 0.5 para un efecto sinérgico, de 0.5-1.0 para un efecto aditivo, >1 a 2 para un efecto de indiferencia y ≥ 2 cuando se presenta un antagonismo, de acuerdo a algunos de las directrices de referencia anteriormente mencionadas.32
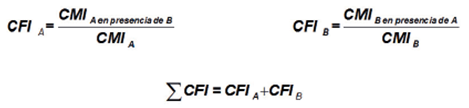
Figura 5 Cálculo de la concentración fraccional inhibitoria (CFI) mediante la sumatoria de las CFI's individuales
Pruebas in vivo
Los modelos experimentales han sido una parte fundamental de la investigación de las enfermedades infecciosas por más de un siglo y se han utilizado para estudiar la relación entre la exposición al fármaco y la eficacia in vivo. En estos modelos la eficacia de los antimicrobianos solos o combinados se puede medir directamente en los focos de infección, y también permiten estudiar la influencia de la terapia antimicrobiana sobre las consecuencias fisiopatológicas de la infección.36
Se han reportado una gran variedad de modelos in vivo para caracterizar la farmacodinámica de los antimicrobianos, pero en general se dividen en modelos humanos que utilizan grupos de sujetos voluntarios, y los modelos no humanos que utilizan diversos animales como las ratas, cerdos, ratones, cobayos y conejos.37
Un ejemplo de la aplicación de modelos experimentales en humanos se reportó para modelar la distribución tisular del antibiótico azitromicina (500 mg) administrado oralmente a seis varones voluntarios sanos durante tres días. Ese estudio incluyó la deteminación de la concentración libre del antibiótico en fluido del espacio intersticial de tejido muscular y en grasa subcutánea, así como la concentración total en plasma y leucocitos a los 1, 3, 5 y 10 días después de la administración. Posteriormente los datos se modelaron simultáneamente en un programa (NONMEM 7.2) usando un modelo de distribución de tejido que representa la unión no lineal a proteína y la ionización estado a pHs fisiológicos, para estimar el rendimiento del antibiótico.38
En la actualidad, los modelos in vivo con animales son críticos en investigación para establecer los valores PK/PD que se relacionan con la eficacia terapéutica. Estos modelos tienenlaventajadedeterminarlaeficaciaantimicrobianaen sitios específicos del cuerpo tales como el muslo en ratones, el peritoneo en ratones y ratas, el pulmón en ratones, ratas y cobayas, endocarditis en conejos y ratas, y la meningitis en conejos.37
El modelo de ratón neutropénico de infección en muslo es el modelo animal más común para examinar las relaciones PK/ PD de los antibióticos, porque es reproducible y relativamente barato. Los ratones neutropénicos por tratamiento con ciclofosfamida (1-4 días) son inyectados en los muslos con las células bacterianas que se van a analizar y que se encuentran en fase logarítmica. Después de esta inoculación y considerando la diferencia de tiempo entre la inoculación y el comienzo de la terapia, se administra el compuesto antimicrobiano ensayado por más de 24 horas con múltiples regímenes de dosificación dependiendo de la vida media y los índices PK/ PD bajo investigación. La eficacia del agente antibacteriano se determina a las 24 horas restando el log10 de las UFC/ muslo de los ratones tratados a las de los ratones control a las 0 horas. Ejemplos notables de la aplicación de este modelo incluyen a las cefalosporina PPI-0903 y linezolid. En el estudio del linezolid, una alternativa para el tratamiento de bacterias grampositivas resistentes a vancomicina, se reveló que un régimen de dosificación de 600 mg dos veces al día sería eficaz contra los patógenos con una CMI de 2-4 mg/L.39
Actualidad y expectativas de las combinaciones binarias de antimicrobianos
Actualmente se justifica la aplicación de las combinaciones de antimicrobianos para extender el espectro de actividad y lograr un sinergismo que mejore el efecto terapéutico para minimizar la resistencia.25
Además de las combinaciones entre antibióticos, se ha tenido un importante auge en la experimentación in vivo utilizando combinaciones de antimicrobianos de naturaleza muy diversa, obteniéndose resultados prometedores que podrían acelerar su implementación en tratamientos destinados a la medicina humana y veterinaria, la agricultura y la industria alimentaria para combatir a las bacterias patógenas.
Algunas de las combinaciones de antimicrobianos más estudiadas son aquellas que involucran antibióticos con otros compuestos derivados de plantas. Durante milenios las plantas
han sido una de las principales fuentes para la obtención de nuevas drogas y se ha comprobado que las infusiones herbales y los aceites esenciales han contribuido enormemente a preservar la salud humana.40
Otra de las combinaciones ensayada en la investigación de antimicrobianos es la que emplea iones metálicos y nanopartículas combinados con antibióticos, la cual ha aportado información muy valiosa al demostrar efectos sinérgicos además de los mecanismos moleculares involucrados en el proceso.41 Las nanopartículas de plata (nano-Ags) son reconocidas ampliamente por sus propiedades antimicrobianas, se han combinado con varios antibióticos tradicionales como la ampicilina, el cloranfenicol y la kanamicina, y después de determinar su CMI y CFI y establecer los efectos sinérgicos de la muestra se estableció la susceptibilidad bacteriana a dicho sistema. Estos estudios confirmaron el gran potencial de la mezcla binaria nano-Ags/ antibiótico como un esquema terapéutico muy prometedor para aplicarse como un tratamiento efectivo contra las enfermedades causadas por infecciones bacterianas.41
Una combinación muy novedosa y sorprendente es el sinergismo establecido entre las enzimas líticas de bacteriófagos y antibióticos.42,43 Las enzimas líticas son producidas por los bacteriófagos al final del ciclo replicativo cuya propiedad particular es la capacidad de digerir la pared bacteriana en segundos. Un claro ejemplo se observa en la muramidasa Cpl-1, la cual rompe específicamente el enlace glucosídico entre el N-acetil ácido murámico y la N-acetil glucosamina (NAM-NAG) de la pared celular de S. pneumoniae resistente a la penicilina.44 Diversas pruebas realizadas con las combinaciones Cpl-1/gentamicina demuestran un efecto sinérgico que se incrementa conforme disminuye la CMI de la penicilina, mientras que la combinación Cp1-1/penicilina muestra sinergismo contra una cepa de S. pneumoniae extremadamente resistente a penicilina, confirmándose el éxito del tratamiento in vitro.
Recientemente se ha considerado a las endolisinas codificadas por fagos para el biocontrol de patógenos en alimentos. Un ejemplo es la endolisina codificada por el bacteriófago estafilocócido phi-SauS-IPLA88, la endolisina LysH5 combinada con Nisina para contrarrestar el crecimiento de S. aureus, bacteria causante de infecciones e intoxicaciones cuando se transmite en leche pasteurizada. La combinación binaria entre Nisina y LysH5 representa uno de los primeros estudios in vitro que confirman a través del sinergismo la efectividad de la mezcla para el control de uno de los patógenos causantes de numerosas intoxicaciones alimentarias en todo el mundo.45
Finalmente, las combinaciones binarias de antimicrobianos más estudiadas en los últimos años se efectúan con moléculas de naturaleza protéica, las bacteriocinas.4
La Nisina es el péptido antimicrobiano natural más exitoso dentro de las bacteriocinas; es producido por Lactococcus lactis subsp lactis y es la bacteriocina más estudiada y con la mayor aplicación en diversas áreas para el control microbiano, y la única bacteriocina con estatus GRAS.46 Esta bacteriocina ha probado ser efectiva cuando se aplica combinada en tratamientos veterinarios realizados in vivo para tratar agentes zoonóticos emergentes.47
Las bacteriocinas más estudiadas y ensayadas en investigaciones in vitro e in vivo además de la Nisina, la Pediocina y la Enterocina producidas por bacterias ácido lácticas,48 no obstante, se ha confirmado que la mayoría de los microorganismos son capaces de sintetizar bacteriocinas. Por ejemplo la bacteria entomopatógena Bacillus thuringiensis (Bt), sintetiza bacteriocinas con un amplio espectro de actividad y su potencial aplicación para el control de patógenos es enorme. Las bacteriocinas de Bt pueden inhibir el crecimiento de Bacillus spp., B. thuringiensis, B. cereus, Listeria innocua, L. monocytogenes, Vibrio cholerae, Staphylococcus xylosus, S. aureus, Shigella flexneri, Salmonella sp., Salmonella choleraesuis, Salmonella enteritidis, Streptococcus pyogenes, Escherichia coli, Pseudomonas putida, Pseudomonas stutzeri, entre otras.49
El mejor ejemplo del éxito potencial de estas bacteriocinas en el área de la medicina se ha reportado para la Thuricina CD, conformada por dos péptidos (Trn-α y Trn-β) producida por B. thuringiensis DPC 6431 y cuyo espectro de actividad es específico contra Clostridium difficile, el agente causal de numerosos brotes de enfermedades diarreicas recurrentes en todo el mundo.50 Este péptido antimicrobiano se ha ensayado in vitro con el método de microdilución en tablero de ajedrez para determinar la actividad de la Thuricina CD sola o en combinaciones pareadas con los antibióticos vancomicina, metronidazol, ramoplanina, y también combinada con otra bacteriocina, la actagardina. La combinación ramoplanina- actagardina fue particularmente eficaz, mostrando un sinergismo parcial con efectos aditivos contra el 61.5% de las cepas de C. difficile probadas.51 Además, se ha estudiado la biodisponibilidad esta bacteriocina in vivo en cerdos como modelo del intestino humano administrándola oralmente en leche, sin embargo los resultados indican que uno de los componenentes de la bacteriocina (Trn- β) es degradado por las enzimas y por lo tanto la administración oral no es muy adecuada. También se ha confirmado la efectividad de la Thuricina CD cuando es administrada intraperitonealmente en ratones inoculados con C. difficile.52
Las aplicaciones de combinaciones de antibióticos con otros compuestos como las bacteriocinas con el propósito de controlar la proliferación de patógenos causantes de enfermedades en humanos y en animales con resultados exitosos, son una evidencia muy fuerte para ser consideradas como los metabolitos bacterianos con mayor futuro como agentes bioterapéuticos. La aplicación de las mezclas antibiótico/bacteriocina permitirá desarrollar numerosas y novedosas terapias que pudieran contrarrestar la grave problemática de la resistencia bacteriana a los antibióticos tradicionales.
Conclusiones
Es difícil vislumbrar a corto plazo un panorama optimista para poder disminuir y contener eficazmente la resistencia a los antimicrobianos. Primero debido a que diariamente se incrementan las infecciones causadas por bacterias resistentes y/o multi-resistentes a antibióticos y segundo, porque son muy pocas las opciones farmacológicas que permiten la aplicación de tratamientos adecuados y efectivos para preservar la salud. Se requiere urgentemente implementar estrategias de investigación y desarrollo para generar nuevos agentes antimicrobianos y estrategias efectivas para su aplicación, porque paradójicamente, la otrora solución a la propagación de las enfermedades infecciosas encontrada en los antibióticos, resulta ser ahora la causa principal de una nueva problemática: la resistencia antimicrobiana.
A nivel mundial existe la conciencia y la voluntad para alentar el desarrollo de múltiples innovaciones e inversiones sin precedentes para generar nuevos agentes terapéuticos, sin embargo, ni las estrategias desplegadas por la OMS para la contención de la resistencia, ni las investigaciones realizadas hasta el día de hoy han logrado un avance significativo para el control de este fenómeno intrínseco del metabolismo bacteriano.











 nueva página del texto (beta)
nueva página del texto (beta)