Introducción
Las infecciones de las vías respiratorias bajas son la principal causa infecciosa de defunción. Tan solo 1 de cada 100 muertes corresponde a niños menores de 15 años. En los países de ingresos bajos, aproximadamente 4 de cada 10 muertes ocurren en niños menores de 15 años, y solo 2 de cada 10 muertes corresponden a personas de 70 años o más. La causa predominante de defunción son las enfermedades infecciosas: en conjunto, las infecciones de las vías respiratorias bajas, la infección por el VIH/sida, las enfermedades diarreicas, el paludismo y la tuberculosis causan casi una tercera parte de las muertes en esos países.1
Según un informe de la Organización Panamericana de la Salud, que actúa como Oficina Regional de la Organización Mundial de la Salud en las Américas hay una elevada resistencia por parte de algunos microorganismos. Ejemplo de ello, Escherichia coli resulta resistente a las cefalosporinas de tercera generación y a las fluoroquinolonas, dos clases importantes y muy utilizadas de fármacos antibacterianos. La resistencia de Klebsiella pneumoniae a las cefalosporinas de tercera generación también es elevada y generalizada. En algunos entornos, hasta un 90 % de las infecciones por Staphylococcus aureus son resistentes a la meticilina, lo cual significa que el tratamiento con los antibióticos habituales no funciona. En este último caso, se calcula que las personas infectadas por S. aureus resistentes a la meticilina tienen una probabilidad de morir un 64 % mayor que las infectadas por cepas no resistentes.2
En México las infecciones causadas por bacterias son una de las principales causas de enfermedades en la población, lo cual ha provocado que se destaque por la prevalencia de las mismas. Se calcula que 450 000 casos de infección relacionada con la atención sanitaria causan 32 muertes por cada 100 000 habitantes por año.3,4
Otro padecimiento frecuente es la micosis superficial en piel y uñas,5 la mayoría causada por dermatofitos. De éstos las especies más frecuentemente encontradas son: Trichophyton rubrum, T. tonsurans, T. mentagrophytes, Microsporum canis y Epidermophyton floccosum, destacándose a T. rubrum como el microorganismo de mayor incidencia en nuestro país.6 A lo anterior se suma que algunos microorganismos han desarrollado resistencia a distintos fármacos, además de que en México existen sesgos en la prevalencia de las inmunodeficiencias primarias debido al subregistro de los casos, producto del desconocimiento de los médicos sobre estos trastornos.2,7 Con lo anterior, surge la necesidad de la búsqueda de alternativas para el control de estas enfermedades; una de ellas es el empleo de plantas medicinales.
Aloe vera es una de las especies vegetales medicinales más importantes en la medicina tradicional mexicana para el tratamiento de diversos padecimientos como: tos, diabetes, erisipela, inflamación e infección de la piel, dolor de estómago y como vermífugo.8 Esta planta es comúnmente conocida como sábila y ha capturado el interés por sus múltiples actividades biológicas.
Recientemente se ha incrementado el estudio de la química y actividades farmacológicas presentadas por las proteínas, péptidos y glicoproteínas provenientes de fuentes naturales.9,10 Koike et al. (1995) reportaron una lectina aislada del A. vera de 35 kDa con actividad de estimulación mitótica de linfocitos, activación del complemento, alternativa, antiinflamatoria, antiúlcera y antitumoral, además de presentar actividad mitogénica.11 Yagi et al. (1997) reportaron una glicoproteína de 29 kDa que aumenta la proliferación de las células dérmicas normales en el humano.12 Choi et al. (2001) reportaron una glicoproteína que nombraron G1G1M1DI2 de 5.5 kDa aislada de A. vera con actividad proliferativa y de migración celular13 y Das et al. (2010) reportaron una proteína de 14 kDa aislada de A. vera, la cual mostró actividad antifúngica contra C. paraprilosis, C. krusei y C. albicans.14 El et al. (2010) reportaron un octapéptido de A. vera con actividad antitumoral y antioxidante.15
El propósito de este estudio fue evaluar la actividad antimicrobiana de extractos proteicos del liofilizado de gel de A. vera.
Material y métodos
Material biológico
Planta: Liofilizado del gel de hoja Aloe vera proporcionado por la empresa Aloe Jaumave, S.A. de C.V. Jaumave, Tamaulipas México. Bacterias: Staphylococcus aureus (ATCC 25923), Bacillus subtilis (ATCC 20), Bacillus cereus (ATCC 14579), Salmonella typhimurium (ATCC 13311), Salmonella typhi (ATCC 6539) y Pseudomonas aeruginosa (ATCC 25619).
Hongos: aislados clínicos de Candida albicans, Trichophyton rubrum, T. mentagrophytes y T. tonsurans.
Extracción de proteínas del liofilizado de gel de A. vera
La extracción de las proteínas se llevó a cabo a partir del liofilizado de gel de A. vera mediante dos métodos. Para el método 1 (M1) se siguió la técnica reportada por Costa et al., (1998)16 con modificaciones: 5 g del liofilizado de A. vera se resuspendieron en 250 mL de una solución amortiguadora A (NaNO3, KH2PO4 y NaN3 [CTR, J.T. Baker y Spectrum respectivamente] y a pH 7) bajo agitación constante a 180 rpm por 6 h en baño de hielo. La suspensión resultante se pasó a través de una membrana de poro de 1 y 0.45 μm y posteriormente se concentró a presión reducida. El concentrado obtenido se centrifugó a 3600 rpm × 30 min a 4 oC y el sobrenadante recuperado se precipitó con (NH4)2SO4 (J.T. Baker) al 80 % (p/v). El precipitado se lavó con acetona (CTR) y se resuspendió en la solución amortiguadora B (Urea, NH4HCO3 y EDTA [J.T. Baker, CTR y CTR respectivamente]). El sobrenadante producto de la resuspensión, fue ultrafiltrado a través de una membrana de corte de 3 kDa y se obtuvo el extracto proteico 1 (EP1). Este procedimiento se repitió 5 veces.
Para la obtención del extracto por el método 2 (M2)17 se utilizaron 5 g de liofilizado de gel de A. vera, que se resuspendieron en 250 mL de una solución amortiguadora C (Tris-HCl [J.T. Baker], sacarosa, SDS y de β-mercaptoetanol [Sigma], a pH 8) y se mantuvieron en agitación constante a 180 rpm por 6 h en baño de hielo, para posteriormente mantenerlo a 4 °C por 12 h. Al término se realizó una centrifugación para eliminar material insoluble, posteriormente se hizo una extracción fenólica con una solución saturada en una relación 1:3 solución A. vera: fenol (Sigma) y posteriormente se centrifugó a 3600 rpm a 4 °C × 30 min. La solución fenólica se recuperó y se precipitó con CH3COONH4 (Monterrey, Reactivo Analítico) 0.1 M en MeOH:H2O (50:50), se agitó y se dejó en reposo durante la noche a −20 °C, después se centrifugó nuevamente. El precipitado se recuperó y se lavó 3 veces con acetona y finalmente se resuspendió en la solución amortiguadora B, obteniendo así el extracto proteico (EP2). Se realizaron 5 réplicas.
La concentración de proteínas de los extractos proteicos EP1 y EP2 se cuantificó por espectrofotometría UV-Vis en el equipo Nadodrop (ThermoScientific) a una longitud de onda de 280 nm. Los EPs se conservaron a -20 °C hasta su uso.
Determinación de la masa molecular
A los extractos proteicos EP1 y EP2s se les realizó una hidrólisis enzimática con tripsina, que consistió en cuatro etapas consecutivas. En la primera etapa, a 100 μL del EP1 o EP2 se les adicionaron 5 μL de Ditiotreitol (DTT; Pro-Pure]) y se incubaron a 65 oC × 45 min; en la segunda y tercera etapas se añadieron 4 μL de C2H4INO (Pro-Pure) y 20 μL de DTT, respectivamente y se incubaron a temperatura ambiente × 45 min; finalmente a ambas digestiones se les adicionó tripsina, en una relación 1:20 (proteína: tripsina [Promega]) y se incubaron a 37 oC × 18 h.
De los hidrolizados de EP1 y EP2 se inyectaron 2 μL en el sistema de nanocromatografía modelo nanoAcquity (Waters) utilizando como fase estacionaria una columna BHE (75 μm × 150 mm y 1.7 μm de tamaño de partícula); como fase móvil se utilizó un gradiente de Agua:ACN (97:3 a 40:60 en 30 min).
La detección de los péptidos se realizó en un espectrómetro de masas (ortogonal cuadrupolo-tiempo de vuelo modelo Q-TOF Premier), configurado con una interfase nano electrospray. El proceso de bioinformática para la secuenciación e identificación se llevó a cabo con el software ProteinLynx Global server ver 2.5, en el cual se usó el algoritmo MASCOT para la obtención de la secuencia de aminoácidos de los péptidos y para la alineación con las secuencias proteicas se usó la base de datos de Arabidosis thaliana en formato FASTA.
Evaluación de actividad antimicrobiana
Actividad antibacteriana18
Los inóculos bacterianos se prepararon por resiembra desde cultivos en medio sólido C. Rivas (Patente IMPI MX/10892) a tubos de ensayo que contenían 5 mL de caldo de cultivo C. Rivas. Estos tubos se incubaron a 37 oC × 18−24 h.
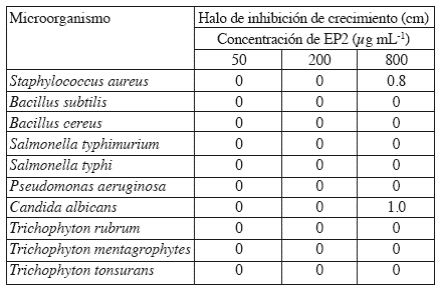
En placas de Petri conteniendo medio sólido C. Rivas se inocularon con 100 μL de una suspensión bacteriana de 1 × 106 UFC mL-1 (que corresponde 0.5 de la escala de Mc Farland). Posteriormente se realizaron orificios con una pipeta Pasteur en el agar, en los cuales se les depositaron 50, 200 y 800 μg mL-1 de EP1 o EP2 en medio C. Rivas. Como control negativo y positivo se utilizaron 25 μL de medio C. Rivas y gentamicina (32 mg mL-1) respectivamente; las placas se incubaron a 37 oC × 48 h, posteriormente se midieron los halos inhibición con ayuda de un Vernier y los resultados se expresaron en cm.15
Actividad antifúngica19
La preparación del inóculo de C. albicans se llevó a cabo de acuerdo a lo marcado anteriormente para actividad antibacteriana, en tanto que para los hongos filamentosos, una asada de micelio se depositó en una caja Petri con medio solido C. Rivas y se incubó a 30 oC de 7−14 d. Posteriormente se recuperaron las conidias por arrastre de superficie con ayuda de un hisopo, se filtraron y se transfirieron a un tubo con 3 mL de H2O destilada estéril. Se realizó el conteo de conidias mL-1en una cámara de Neubauer y se ajustó la densidad a 1−2 × 104 UFC mL-1.
Para el ensayo contra hongos, se prepararon cajas Petri con medio sólido C. Rivas y se inoculó con 100 μL de cada cepa en su respectiva caja. Se hicieron orificios con una pipeta Pasteur y se colocaron en cada pocillo 50, 200 y 800 μg mL-1 de EP1 o EP2 en medio C. Rivas. Como control positivo y negativo se colocaron 25 μL de ketoconazol disuelto en H2O estéril (32 mg mL-1) y medio C. Rivas respectivamente. Las placas se incubaron a 30 oC por 24 h para C. albicans y de 7−14 d para los hongos filamentosos. Posteriormente se midieron los halos de inhibición con un Vernier y los resultados se expresaron en cm.20
Resultados y discusión
Extracción de proteínas del liofilizado de gel de A. vera
En la extracción por el M1 se obtuvo un rendimiento promedio de 6.2 mg (DE ± 1.26) de EP1 que corresponde a un porcentaje de recuperación de 0.1 % y por el M2 se obtuvo un rendimiento medio de 14.9 mg de EP2 (DE ± 1.24) y que corresponde a un porcentaje de 0.3 %; de tal forma el M2 resultó ser 2.4 veces más eficiente que el M1. Murillo et al., reportaron un rendimiento de 146.70 y 96.92 mg g-1 de proteína total para colénquima y parénquima de A. vera respectivamente sin someterse a estrés salino con NaCl.21 El porcentaje de recuperación obtenido en este trabajo se encuentra por debajo de lo reportado por Murillo et al., lo anterior atribuido a que la extracción y determinación de proteínas realizadas por dichos autores fue a partir de tejido seco que fue homogeneizado en una solución amortiguadora de extracción (KH2PO4, EDTA, polivinilpirrolidona) para posteriormente centrifugar la mezcla y recuperar el sobrenadante en donde se cuantificó proteínas totales por el método de Bradford. En este trabajo es importante señalar que la cuantificación de proteínas totales fue realizada para el extracto, resultado de un proceso de purificación.
En el espectro de UV de los extractos proteicos de A. vera que se muestran en la Figura 1, en EP1 se observa una señal característica de la presencia de proteínas a 280 nm, con el punto máximo de absorción desplazado y una mayor área baja la curva mientras que EP2 mostró la señal más próxima a 280 nm y una banda más estrecha lo cual indica mayor pureza.

Figura 1 Comparación de la pureza de los extractos proteicos del gel liofilizado de Aloe vera EP1 (M1) y EP2 (M2).
Cabe señalar que el equipo NanoDrop (ThermoScientific) permite determinar el rendimiento en mg mL-1 de las proteínas extraídas empleando como estándar ASB y además está adaptado para determinar la pureza de proteínas fraccionadas, estableciendo un radio ~1.8-2.00 para indicar la misma.
El incremento en la "pureza" del espectro UV-Vis del EP1 vs EP2 utilizando como precipitante al fenol es debido a que el fenol favoreció una mayor remoción de sustancias interferentes disminuyendo la interacción molecular entre las proteínas y otros componentes como los azúcares17 brindando la ventaja al M2 en cuanto a su porcentaje de recuperación y de rendimiento proteico.
Determinación de la masa molecular
En el EP1 se identificaron por espectrometría de masas, péptidos los cuales fueron ontológicamente alineados con la enzima eterol O-aciltransferasa 1 y una glicoproteína de matriz M2, respectivamente (Figura 2). La primera de ellas producida por células hepáticas, relacionada con el metabolismo de lípidos, particularmente en el catabolismo de esteroles por lo que se dice que topológicamente tiene un dominio transmembrana. La base de datos relacional reporta interacción directa con proteínas de la familia de Lipasa 3 y con la peptidil-prolil-cis-trans- isomerasa de cloroplasto. Chen et al., han reportado el aislamiento de esta enzima de Arabidopsis thaliana demostrando una notable actividad en el metabolismo de esteroles en la mejora de la calidad de las semillas.22,23 Cabe mencionar que no existen reportes de su actividad antimicrobiana, sin embargo se infiere que la enzima participa en los procesos inflamatorios en la respuesta inmune a la infección por ciertos microorganismos como Pseudomonas aeruginosa.
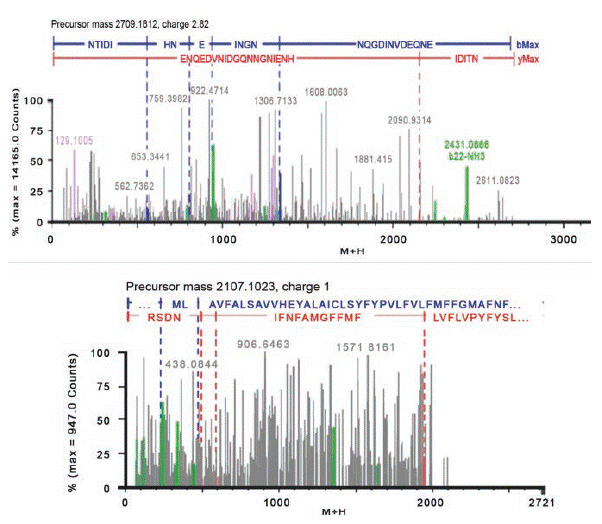
Figura 2 Espectros de masas de los péptidos de 31 y 5kDa (superior e inferior respectivamente) obtenidos del EP1 de A. vera.
La segunda proteína, reportada como una glicoproteína de matriz 2, también alinea fuertemente con una supuesto activador RAB GTPasa (NP_001118517.1) de Arabidopsis thaliana, la cual participa ampliamente en los mecanismos de transporte a través de la membrana y en el tráfico intracelular. Funciona como canal iónico formando parte de las proteínas estructurales de la membrana, pero no se ha reportado ninguna actividad antimicrobiana directa. 23
El método de extracción (M2) con fenol ha permitido la identificación de proteínas, sin embargo no se detectaron los fragmentos necesarios relacionados con los precursores o péptidos detectados para asignar la secuencia de acuerdo a la base de datos MASCOT. Dado el muy bajo contenido de proteína de la planta resulta necesario la concentración de las muestras para lograr identificar un grupo mayor de proteínas o de secuencias peptídicas y lograr el fraccionamiento de proteínas a las cuales podamos atribuir la actividad antimicrobiana observada.
Cabe señalar que aunque han sido identificadas varias secuencias, las reportadas son las de confiabilidad superior al 90%.
Evaluación de actividad antimicrobiana
Nuestros resultados muestran que el EP1 no mostró actividad contra las bacterias y hongos evaluados, en tanto que EP2 mostró actividad inhibitoria del crecimiento contra S. aureus y C. albicans a 800 μg mL-1 con un halo de inhibición de 0.8 y 1 cm, respectivamente y no exhibió actividad inhibitoria para el resto de las bacterias y los hongos filamentosos (Tabla 1). Para el EP2 bioactivo vs S. aureus y C. albicans no se lograron identificar sus componentes mediante el análisis por EM realizado. En tanto que para el EP1 no bioactivo, se logró identificar la presencia de un par de péptidos que presentan una actividad relacionada con el metabolismo de lípidos y con los mecanismos de transporte a través de la membrana y el tráfico intracelular.
EP2 mostró actividad inhibitoria sobre S. aureus y C. albicans, no existen reportes previos con actividad antiestafilocócica del extracto proteico de A. vera, pero sí de actividad anti-C. albicans, por lo cual resulta necesario corroborar si la actividad mostrada por EP2 se trata de la proteína reportada y definir a quién se le atribuye la actividad antiestafilocócica, por lo cual el extracto proteico de sábila resulta una fuente prometedora de moléculas con actividad contra S. aureus y C. albicans.
Conclusiones
El método de extracción M2 permitió la obtención de un extracto proteico del liofilizado de gel de A. vera con mayor rendimiento y pureza EP2 comparado con el EP1 obtenido por el método M1. Se identificó en EP1 la proteína esterol-o-aciltransferasa1 (ATSAT1), esta enzima se ha aislado de Arabidopsis thaliana y la glicoproteína de matriz 2 alínea fuertemente con un activador RAB GTPasa (NP_001118517.1) de A. thaliana. EP2 mostró actividad sobre Staphylococcus aureus y Candida albicans y no existen reportes previos con actividad antiestafilocócica del extracto proteico de A. vera. El extracto proteico de sábila resulta una fuente prometedora de moléculas con actividad anti-S. aureus y anti-C. albicans.











 nueva página del texto (beta)
nueva página del texto (beta)