Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
REB. Revista de educación bioquímica
versión impresa ISSN 1665-1995
Rev. educ. bioquím vol.32 no.3 Ciudad de México sep. 2013
Otras comunicaciones
Respuestas al problema bioquímico
Elizabeth Lira Silva y Ricardo Jasso Chávez
Departamento de Bioquímica, Instituto Nacional de Cardiología. Correo: eli_lira_sil@hotmail.com.
Se ha demostrado que la actividad de las enzimas varía respecto al pH, debido a que en su sitio activo pueden tener aminoácidos con varios grupos ionizables (ácido aspártico, ácido glutámico, cisteína, histidina, arginina y lisina) involucrados en la unión y catálisis. A bajos valores de pH la enzima esta protonada (En+1) mientras que a altos valores estará desprotonada (En−1), de acuerdo con el siguiente modelo para dos grupos ionizables:
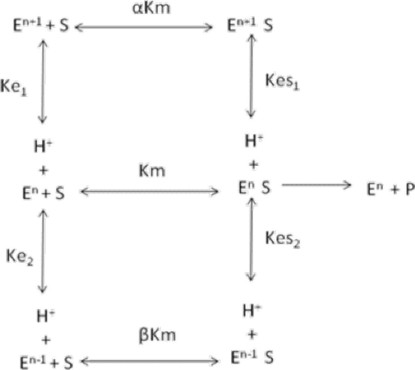
Donde se asume que las tres formas de la enzima son capaces de unir al sustrato pero solo la forma EnS es catalíticamente activa: n es el número de grupos ionizables, En es la forma de la enzima catalíticamente activa, En+1 y En−1 son las formas no catalíticas, Ke1 y Ke2 representan las constantes de disociación de protón de la enzima libre, Kes1 y Kes2 las constantes de disociación de protón de los complejos enzima-sustrato (En+1S y En−1S), α y β son factores que modifican la afinidad de la enzima por el sustrato.
La ecuación de velocidad de Michaelis-Menten que describe el efecto del pH sobre la enzima es:

Donde:

Los valores de los parámetros cinéticos son aparentes, lo cual se indica con asteriscos pues están afectados por la concentración de protones [H+].
Con los valores de la Tabla 1 se construyó la gráfica de actividad de la proteasa alcalina a diferentes concentraciones de caseína y diferentes valores de pH (Fig. 1); con los mismos datos se realizó el gráfico de dobles recíprocos ó gráfico de Lineweaver-Burk (Fig. 2), donde se puede observar que la Km, la Vmax y la eficiencia catalítica dependen del valor del pH del ensayo. Los valores de Km, Vmax y Vmax /Km se pueden obtener a partir de los valores de la ordenada al origen (1/Vmax), abscisa al origen (−1/Km) y de la pendiente (Km/Vmax) de la figura 2. En la Tabla 2 se muestran los parámetros cinéticos calculados a cada valor de pH utilizando el método de Lineweaver-Burk. Se determinó que a valores de pH extremos (pH 5 y 12) la velocidad máxima de la proteasa fue muy baja en comparación con las actividades determinadas en el intervalo de pH 9-10 (el valor de la ordenada al origen b= 1/Vmax disminuye); la afinidad de la enzima (Km) en el intervalo de pH analizado es variable (el valor de la abscisa al origen a= −1/Km), mientras que la eficiencia catalítica de la enzima se incrementa a pH 10.
Considerando el doble reciproco de la ecuación 2, la ordenada al origen es:
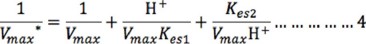
A valores de pH bajos (1-6 ó [H+] alta), el término Kes2/(VmaxH+) puede despreciarse, por lo que la ecuación 4 se reduce a:
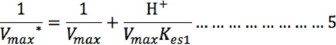
De forma similar, el término H+/(VmaxKes1 ) puede despreciarse a valores de pH altos (8-14 ó [H+] baja), por lo que la ecuación 4 se reduce a:
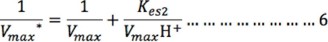
La pendiente del doble recíproco de la ecuación (3) es:
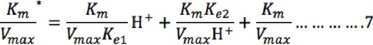
El término (KmKe2)/(VmaxH+) puede ser despreciado a valores de pH bajos (1-6 ó [H+] alta), por lo que la ecuación 7 se reduce a:
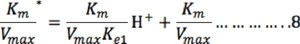
De modo similar, a pH básico, el término Km/(Vmax Ke1) H+ puede ser despreciado a valores de pH altos (8-14 ó [H+] baja); por lo que la ecuación 7 se reduce a:
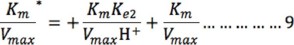
Considerando las ecuaciones anteriores, es posible regraficar los valores de las ordenadas al origen (1/Vmax) y las pendientes (Km/Vmax) vs [H+] ó 1/[H+] dependiendo del intervalo de pH (ácido o básico, respectivamente). En estos regráficos es posible obtener el valor de las constantes de disociación Ke1, Ke2, Kes1 y Kes2, a partir del cruce con las abscisas (Fig. 3). Los valores de pKa se obtuvieron calculando el – logaritmo de las constantes de disociación y fueron pKe1= 5.09, pKe2=11.7, pKes1=5.3 y pKes2 =11.6. En los regráficos es importante notar que solo se deben tomar en cuenta aquellos puntos que caen dentro de la recta, ya que los que no lo hacen se encuentran fuera del intervalo de pH estudiado.
Otra forma de calcular los valores de pKa con mayor precisión es realizar el ajuste por regresión no lineal utilizando las ecuaciones 2 y 3 descritas anteriormente (Fig. 4). La ecuación 2 se editó para obtener los valores de Kes1 y Kes2; mientras que a partir de las ecuaciones 2 y 3 se obtuvo la ecuación 10 para obtener Ke1 y Ke2:
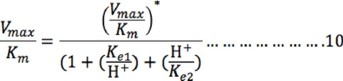
La edición de estas ecuaciones se realizó utilizando el programa de computo Origin 5.0. Al realizar el análisis a pH ácidos se observó la presencia de un tercer grupo ionizable que probablemente esté participando en la estabilidad de la enzima (Fig. 4). La ventaja al utilizar este método es que se obtienen los valores de pKa de unión y de catálisis directamente esto es, sin regráficos (Tabla 3).

Una tercera opción para obtener los valores de pKa es mediante el uso del gráfico de Dixon Webb, obtenido a partir de la forma logarítmica de las ecuaciones 2 y 10. Para la ecuación 2, se obtiene la siguiente ecuación en forma logarítmica:
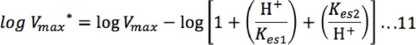
A pH ácido, los términos 1 y Kes2/H+ se pueden despreciar debido a que son muy pequeños respecto a H+/Kes1 , por lo que la ecuación simplificada es:

A pH básico, los términos 1y H+/Kes1 se pueden despreciar debido a que son muy pequeños respecto a Kes2/H+, por lo que la ecuación simplificada es:
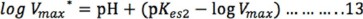
De manera similar se obtiene la ecuación logarítmica a partir de la ecuación 10 para intervalos de pH ácido y la ecuación que resulta es:
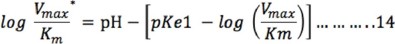
y para pH básico, la ecuación es:
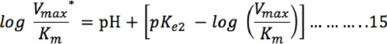

El gráfico de Dixon Webb (Fig. 5) se obtuvo graficando el log Vmax* o el log (Vmax/Km)* versus el pH, en esta gráfica se puede trazar una línea recta tangente a la curva de pendiente = 1 y otra de pendiente= −1 (debido a que el valor de la pendiente de la variable independiente en las ecuaciones 14 y 15 es 1, ver ecuación 16) hasta que intersecten con el valor de Vmax y (Vmax /Km) reales que se obtuvieron a partir del gráfico de Lineweaver Burk y están marcados con líneas horizontales.
Entonces los valores de pKa serán iguales al valor de pH en el punto de intersección de estas dos líneas. Así se puede obtener directamente los valores de pKes1, pKes2, pKe1 y pKe2. Es importante mencionar que los resultados obtenidos de estos gráficos sólo serán confiables cuando la diferencia entre los valores de pKa, evaluados a pH ácido y básico (p. ej. pKe1 y pKe2), sea mayor a tres unidades de pH. De acuerdo con la Fig. 5 los valores calculados (pKes1=6.7, pKes2=10.8, pKe1=7.04 y pKe2=10.4) cumplen con dicho criterio.
Se determinó que esta enzima fue una típica proteasa alcalina cuyo pH óptimo es 10. Los valores de las constantes de disociación obtenidos por los tres métodos diferentes correlacionan muy bien entre sí. Los valores de pKa obtenidos a pH alcalino (8-12), es decir para la segunda constante de disociación de la enzima libre y del complejo enzima sustrato (pKe2 y pKes2) son cercanos a los de la tirosina (pKa=9.8-10.5) y/o arginina (pKa=11.6-12.6). Las mayores diferencias entre los métodos lineales y el no lineal se obtuvieron para las constantes de disociación a valores de pH ácido (pKe1 y pKes1). A este respecto, las serina y treonina proteasas así como las aspartil proteasas, utilizan un residuo de ácido aspártico e histidina para llevar a cabo la catálisis. El pKa de un residuo de histidina (pKa=5.5-7) o un ácido aspártico (pKa=3-5) puede oscilar dependiendo del ambiente en el que se encuentre en la enzima. De esta forma los valores de pKe1 y pKes1 obtenidos del análisis lineal concuerdan con los valores de pKa del aspártico y/o histidina (Tabla 3). Por este motivo es importante hacer especial énfasis en realizar un análisis completo y adecuado al estimar los parámetros cinéticos por métodos lineales antes de realizar el ajuste no lineal, para no sobre interpretar los datos experimentales. Sin embargo, para validar la participación de estos aminoácidos en la catálisis enzimática se requiere realizar experimentos adicionales utilizando compuestos que reaccionen de manera específica con estos aminoácidos (tirosina, arginina o histidina) mediante nitración, cloración o iodación de residuos de tirosina u oxidación de grupos sulfuro o realizar mutagénesis sitio dirigida para cambiar estos aminoácidos y con ello determinar si se modifica la actividad enzimática al variar el pH del ensayo, así como la afinidad y la capacidad catalítica de la enzima.
Referencias
1. Renganathan Jayakumar , Shanmugam Jayashree, Balumuri Annapurna, Sundaram Seshadri (2012) Appl Biochem Biotechnol. DOI 10.1007/s12010-012-9902-6.
2. Jyothi Bezawada, S. Yan, Rojan P. John, R. D. Tyagi, R. Y. Surampalli (2011) Biotechnology Research International doi:10.4061/2011/238549.
3. Segel IH (1975) Enzyme Kinetics. John Wiley and Sons, New York.p.957. [ Links ]














