Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
REB. Revista de educación bioquímica
versión impresa ISSN 1665-1995
Rev. educ. bioquím vol.32 no.1 Ciudad de México mar. 2013
Otras comunicaciones
Problema bioquímico. Determinación del ciclo umbral y la eficiencia para la PCR cuantitativa en tiempo real
David R. de Alba Aguayo y Angelica Rueda
Correo E: arueda@cinvestav.mx
Introducción
Desde su implementación en 1985 (1) la reacción en cadena de la polimerasa (PCR del inglés "Polimerase Chain Reaction") ha sido extensamente empleada en la investigación. Esta técnica permite duplicar de manera exponencial una secuencia de ácido desoxirribonucleico (DNA) obteniéndose dos copias en cada ciclo de amplificación a partir de una cadena patrón de DNA. A lo largo del tiempo se han generado numerosas variantes de la PCR que permiten abordar diferentes estrategias experimentales, una de estas es la PCR cuantitativa en tiempo real (qPCR) que, mediante un termociclador acoplado a un espectrofluorómetro y haciendo uso de un agente intercalante fluorescente (como el SYBR Green) cuya fluorescencia aumenta proporcionalmente con el aumento en la cantidad de DNA, nos permite cuantificar simultáneamente (en tiempo real) el número de copias que se están generando en cada ciclo de amplificación.
Existen dos variantes de qPCR, la primera es la qPCR de cuantificación absoluta que se basa en la elaboración de una curva de calibración que se construye mediante la determinación del ciclo umbral en diluciones conocidas de DNA y la segunda es la qPCR de cuantificación relativa que se basa en la relación entre los niveles de expresión de un gen de interés (GOI ó "Gene of Interest") con respecto a los niveles de expresión de un gen de referencia (Ref) que se expresa constitutivamente, ésta ultima estrategia de cuantificación es conveniente para investigar los cambios fisiológicos en los niveles de expresión génica (2).
La Figura 1A corresponde a una curva de amplificación representativa a partir de la cual es posible determinar el punto en el cual la fluorescencia de la reacción sobrepasa la fluorescencia basal (umbral o "threshold"), y se considera como el punto en el cual la reacción de amplificación da comienzo. Este punto es conocido como Cp ("Crossing point") o Ct ("Threshold point" o ciclo umbral). El Ct es un valor definido de forma arbitraria por el usuario aunque es posible definirlo de forma matemática. Obteniendo la segunda derivada de la curva de amplificación, es posible definir el Cp como el ciclo que corresponde al valor máximo de la segunda derivada, adicionalmente, se puede definir el Cp como el punto de despegue ("takeoff point") que es el equivalente al 20% de ese máximo, ambos parámetros permiten encontrar de forma matemática el ciclo en el cual la fluorescencia aumenta significativamente con respecto a la fluorescencia basal.
Si se grafica el Cp obtenido con respecto a la dilución de cDNA correspondiente (en ng) se obtiene una curva de calibración (Fig. 1B) cuya pendiente permite calcular la eficiencia de la reacción de amplificación mediante la Ecuación 1 (3).

Ecuación 1 para el cálculo de la eficiencia en la amplificación de la qPCR (3). Donde E, eficiencia de la amplificación; m, pendiente.
Aunque comúnmente y con la finalidad de simplificar el análisis de la expresión relativa de un gen se asume que las eficiencias para todas las curvas son del 100%, es de vital importancia conocer la eficiencia real de la reacción y tomarla en cuenta al momento de calcular la expresión relativa del gen de interés, ya que al tratarse de un incremento exponencial en el número de copias se puede sobreestimar o subestimar los niveles de expresión relativa. Como ejemplo, una diferencia en la E o ΔE = 0.03 dará una desviación en la relación de expresión del 46% si la EGOI<Eref y de 209% si la EGOI>Eref después de 25 ciclos (3).
Se han desarrollado diversos modelos matemáticos para el cálculo de la expresión relativa de un gen, uno de los más utilizados es el denominado delta delta Ct (ΔΔCt) (4) (Ecuación 2); sin embargo, su principal desventaja es precisamente no tomar en cuenta la corrección por cambios en la eficiencia de la reacción, asumiendo que siempre se tendrá una eficiencia del 100 % (4).
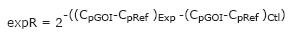
Ecuación 2 para el cálculo de la expresión relativa en la qPC (4). Donde expR, expresión relativa; Ctl, grupo control; Exp, grupo de tratamiento experimental; GOI, gen de interés; Ref, gen de referencia.
Otro método empleado para el cálculo de la expresión relativa del gen de interés es el propuesto por Pfaffl (3) (Ecuación 3). Este método sí toma en cuenta los cambios en las eficiencias de la amplificación y permite calcular de una manera más precisa la expresión relativa del gen de interés con respecto al gen de referencia (3). Este método es de gran utilidad pues también considera las variaciones intrínsecas entre los individuos de un mismo grupo experimental y no sólo las inducidas por las condiciones experimentales -previas y/o posteriores- debidas a algún tratamiento.

Ecuación 3 para determinar la expresión relativa de un gen (3). Donde expR, expresión relativa; EGOI, eficiencia del gen de interés; ERef, eficiencia del gen de referencia; ΔCpGOI, diferencia del Cp del gen de interés del grupo control menos el Cp del gen de interés del grupo experimental (CpGOI Ctl-CpGOI Exp); ΔCpRef, diferencia del Cp del gen de referencia del grupo control menos el Cp del gen de referencia del grupo experimental (CpRef Ctl-CpRef Exp).
Planteamiento experimental
Frecuentemente se define el Ct de forma arbitraria como se muestra en la Figura 1 (línea azul). Otro método rápido y sencillo para determinar el Ct consiste en calcular el promedio (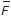 ) y la desviación estándar (DS) de la fluorescencia de los primeros ciclos de amplificación (se pueden usar los primeros 10 ciclos) y posteriormente realizar la suma del valor promedio calculado más 5 veces la DS para obtener un valor de fluorescencia umbral (Ctf) el cual se interpola en la curva de amplificación para determinar el ciclo que corresponderá al Ct (Ecuación 4).
) y la desviación estándar (DS) de la fluorescencia de los primeros ciclos de amplificación (se pueden usar los primeros 10 ciclos) y posteriormente realizar la suma del valor promedio calculado más 5 veces la DS para obtener un valor de fluorescencia umbral (Ctf) el cual se interpola en la curva de amplificación para determinar el ciclo que corresponderá al Ct (Ecuación 4).

Ecuación 4 para determinar valor de fluorescencia umbral (Ctf ) y determinar el Ct de una curva de amplificación de qPCR. Donde Ctf , fluorescencia umbral; 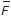 , fluorescencia promedio de los primeros ciclos; DS, desviación estándar.
, fluorescencia promedio de los primeros ciclos; DS, desviación estándar.
Datos
A continuación se presenta una tabla que contiene los resultados experimentales de una corrida de qPCR en un termociclador Rotor-Gene Q Serie 2.0.2. (Qiagen). Para el experimento se utilizaron 300 ng de cDNA en presencia de SybrGreen (Qiagen) más los primers específicos para un gen de referencia (en este caso GAPDH) y un gen de interés a una concentración final de 20 µM, los primers fueron diseñados para amplificar regiones interexónicas en ambos casos. Las condiciones de reacción de la qPCR fueron las siguientes: 1 ciclo a 95ºC por 5 minutos, 55 ciclos de 5 s a 95ºC seguido de 10 s a 60ºC y finalmente 1 ciclo de incremento de temperatura desde 60ºC hasta 95ºC.
Preguntas
1) Utilizando los datos de la Tabla I, grafica las curvas de amplificación tanto para el gen de interés como para el gen de referencia, en ambas condiciones (control y experimental), se . Se recomienda hacer uso de hojas milimétricas.

2) Calcula la Ct utilizando la Ecuación 4 para cada curva de amplificación.
3) Determina la eficiencia utilizando la Ecuación 1 para cada curva de amplificación y usando el valor de la pendiente (m) de la Tabla 1.
4) Calcula el cambio en la expresión relativa del gen de interés (GOI) con la ecuación de ΔΔCt utilizando solamente los valores de Ct obtenidos.
5) Calcula el cambio en la expresión relativa del gen de interés (GOI) con la ecuación de Pfaffl utilizando los valores de Ct y E obtenidos anteriormente.
Referencias
1. Saiki RK, Scharf S, Faloona F, Mullis KB, Horn GT, Erlich HA, Arnheim N (1985) Enzymatic amplification of beta-globin genomic sequences and restriction site analysis for diagnosis of sickle cell anemia. Science 230(4732):1350-4. [ Links ]
2. Pfaffl MW, Horgan GW, Dempfl L (2002) Relative expression software tool (REST) for group-wise comparison and statistical analysis of relative expression results in real-time PCR. Nucleic Acids Res 30(9): p. e36. [ Links ]
3. Pfaffl MW (2001) A new mathematical model for relative quantification in real-time RT-PCR. Nucleic Acids Res 29(9): p. e45. [ Links ]
4. Livak KJ, Schmittgen TD (2001) Analysis of relative gene expression data using real-time quantitative PCR and the 2(-Delta Delta C(T)) Method. Methods 25(4): p. 402-8. [ Links ]














