Introducción
Los organismos poseen múltiples nichos ecológicos en los que se encuentran colonizando diversas comunidades microbianas, presentando una relación de simbiosis entre el huésped y hospedero. Uno de los nichos de gran importancia es el tracto digestivo, donde los microorganismos de la comunidad, facilitan y catalizan gran cantidad de procesos metabólicos, participando en la protección contra patógenos, en el procesamiento de nutrientes y en la regulación de funciones fisiológicas y metabólicas propias del hospedero (Tremaroli y Backhed, 2012). La simbiosis presentada entre los microorganismos con su hospedero, es debido a que obtienen beneficio de su hábitat al disponer de áreas de adhesión, nutrientes para su crecimiento e incluso, modular algunas funciones fisiológicas (Palmer et al., 2007). Son muy diversos los estudios en que se ha destacado el importante rol que los microorganismos del tracto digestivo juegan en beneficio de los animales acuáticos. Por ejemplo, las múltiples interrelaciones huésped-hospedero, han sido reconocidas como un factor influyente en la salud de este último (Merrifield y Ringo, 2014).
Es por lo anterior que el estudio de la composición microbiana del tracto digestivo, resulta fundamental, tanto para conocer a mayor profundidad la biología del hospedero, como para futuras aplicaciones en acuicultura.
La importancia comercial de las especies de camarones de la familia Penaeidae, reside en su rápido crecimiento, ciclo de vida corto y fácil manejo, lo que resulta beneficioso para su utilización en el sector acuícola (Moss et al., 2012; Yuan et al., 2018). La información sobre la microbiota del tracto digestivo de estos crustáceos, se ha enriquecido recientemente debido a nuevas técnicas que permiten realizar la identificación molecular de microorganismos mediante secuenciación masiva del gen 16S ARNr o el metagenoma del tracto intestinal. El fragmento mayormente secuenciado en plataformas de secuenciación masiva, es aquél que contiene las regiones variables V3-V4, principalmente en plataformas Ilumina MiSeq (28%), seguido de Roche y Ion Torrent, aunque estas últimas están por ser descontinuadas (Klindworth et al., 2013; Cornejo-Granados et al., 2018). Se ha documentado que la composición bacteriana intestinal de peneidos como el camarón tigre, Penaeus monodon y el camarón blanco, Litopeaneus vannamei, puede ser muy dinámica durante su cultivo (Rungrassamee et al., 2013; Huang et al., 2016; Zheng et al., 2017), es decir, la predominancia de ciertos grupos se puede ver modificada a través del tiempo, lo que permite inferir que estos cambios obedecen tanto a las necesidades biológicas, como a los diferentes requerimientos nutricionales de la especie durante cada etapa de su desarrollo, además de factores ambientales como la calidad y composición del agua, tipo de alimentación, entre otros. Por ejemplo, la dieta de peneidos cambia drásticamente de la etapa larval a post larval, por lo que el organismo requiere un perfil enzimático distinto entre una etapa y otra para lograr un mejor aprovechamiento de nutrientes (Lemos et al., 2002). A este respecto, podría asumirse que la microbiota aporta funciones enzimáticas adicionales, pero específicas según las necesidades del hospedero.
Los estudios de la composición microbiana en intestino de camarón mediante técnicas moleculares de secuenciación masiva, ofrecen al día de hoy un acercamiento a su distribución y abundancia; sin embargo, hasta hace poco se desconocía casi por completo el papel o función que desempeñan estos microbios en el ciclo de vida del hospedero; de hecho, todavía se desconocen casi en su totalidad las funciones de la microbiota asociada al tracto digestivo del camarón a lo largo de su desarrollo postlarval. Esto se debe principalmente a la necesidad de llevar a cabo un proceso de secuenciación escopeta a partir de muestras de intestinos o heces de camarón, lo que implica extraer también el ADN de un huésped cuyo genoma todavía no se ha ensamblado y, por lo tanto, todavía no es posible filtrar sus secuencias. Sin embargo, programas informáticos basados en predicciones establecidas en base al uso de información de bacterias conocidas y contenidas en bases de datos, han demostrado ser una ruta confiable para llevar a cabo tales aproximaciones. Por ejemplo, en la actualidad es posible llevar a cabo inferencias de la capacidad funcional de un metagenoma bacteriano con relativa certeza, simplemente con conocer las secuencias de genes biomarcadores como rpoB, gyrB o el 16S ARNr, siendo este último el más utilizado (Langille et al., 2013)2013.
Conocer la composición de un determinado microbioma y asociarlo con funciones específicas, puede resultar de gran trascendencia, no solo en el avance del conocimiento sino además en las posibles aplicaciones prácticas, como por ejemplo modificar las condiciones de cultivo para incentivar la dominancia de ciertos grupos cuyas funciones serían deseables, tales como mayores tasas de nitrificación, disminución o aumento del pH, mayor aportación nutricional, aporte de enzimas digestivas, entre otras. La presente revisión se centra en conocer los avances en el conocimiento e importancia de la microbiota del tracto digestivo de camarones peneidos, así como las funciones de ésta y los factores que la modulan.
El tracto digestivo de camarones peneidos
Los camarones peneidos forman parte de la superfamilia Penaeoidea, la cual cuenta con cinco familias en donde se encuentra a la familia Penaeidae, cuyos miembros se caracterizan por poseer un caparazón delgado y liso, así como segmentos abdominales que pueden tener dientes, surcos o muescas dorsales (Fischer et al., 1995).
En términos generales, tracto digestivo de camarones peneidos está compuesto por 1) intestino proximal, donde se encuentra el estómago y ocurre la masticación; 2) el intestino medio o hepatopáncreas que secreta enzimas digestivas y absorción de nutrientes, metabolismo de lípidos y carbohidratos; y 3) el intestino distal que consiste en una estructura tubular en la parte dorsal del organismo y termina en el ano (Fig. 1) (NRC, 2011). El estómago de camarones puede presentar actividad enzimática proveniente del hepatopáncreas o de otras fuentes, ocurriendo el proceso digestivo antes de que el alimento llegue al intestino. Durante este proceso digestivo, los camarones tienen la capacidad de hidrolizar gran variedad de oligo- y polisacáridos (NRC, 2011). La actividad enzimática llevada a cabo por el hepatopáncreas está relacionada con el tamaño de los camarones, la fuente de proteína de la dieta, así como el nivel de proteína; ésta última está relacionada con su crecimiento (Carrillo y González, 2000). Sin embargo, se conoce que las bacterias del tracto digestivo presentan una relación con el desarrollo del hospedero y se podría inferir que, al igual que para el caso de los peces (Nayak, 2010), la microbiota del tracto digestivo determina el adecuado desarrollo y funcionamiento fisiológico del camarón. A pesar de que se intuyen sus funciones, la mayoría de estas siguen sin ser demostradas o ampliamente exploradas.
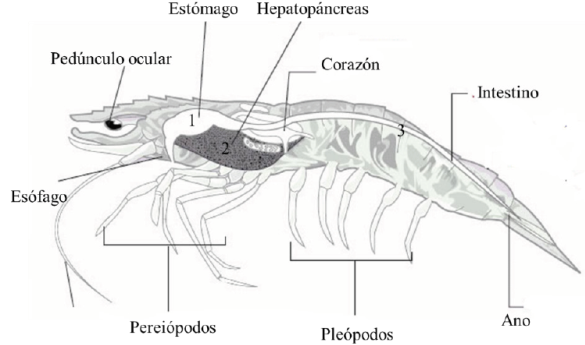
Figura 1 Anatomía de camarones peneidos, resaltando el tracto digestivo: estómago (1),
hepatopáncreas (2) e intestino (3). Imagen modificada de (Bondad-Reantaso et
al., 2001).
Figure 1 Anatomy of penaeid shrimp,
highlighting the digestive tract: stomach (1), hepatopancreas (2)
and intestine (3). Image modifed from (Bondad-Reantaso et al., 2001).
Microbiota del tracto digestivo de camarones peneidos
La microbiota, la cual se compone de los microorganismos asociados a organismos tanto vegetales como animales, son indispensables para su desarrollo (Bordenstein y Theis, 2015; Marchesi y Ravel, 2015). Sin embargo, los estudios basados en la composición y función de la microbiota del tracto digestivo, se han llevado a cabo principalmente en mamíferos, mientras que solo unos cuantos se han enfocado en peces y muy escasos en crustáceos. No obstante, estos han permitido comprobar que las bacterias presentes en el tracto digestivo se asocian con procesos fisiológicos de fundamental importancia para el hospedero (Nayak, 2010; Sommer y Bäckhed, 2013). Por un lado, son importantes en los procesos digestivos, debido a su aporte nutricional directo y a la producción de enzimas que participan en el procesamiento de nutrientes. Por otro lado, juegan un rol importante en la protección del huésped, ya que algunos microorganismos pueden producir compuestos antimicrobianos que ayudan en la defensa contra patógenos. De igual forma, la colonización de bacterias benéficas en el tracto puede desplazar a aquellas patógenas, así como activar el sistema inmune, resultando en una mayor resistencia a enfermedades y finalmente beneficiando el estado de salud del hospedero (Luis-Villaseñor et al., 2015; Zheng et al., 2016).
En el caso de camarones peneidos, estudios recientes han detectado centenares de especies de bacterias pertenecientes a taxones asociados con ambientes marinos y terrestres (Rungrassamee et al., 2014; Vargas-Albores et al., 2017b); lo que lleva a asumir que, al igual que en los mamíferos, la biología de los camarones peneidos está fuertemente ligada a la microbiota asociada a su tracto digestivo.
Breve historia del estudio de la microbiota de camarones peneidos
Las primeras aproximaciones en el estudio de comunidades bacterianas presentes en el intestino de camarones, se llevaron a cabo mediante la identificación por métodos convencionales basados en las características fenotípicas. Una identificación fenotípica bacteriana, se basa en las características de éstas en medios de cultivo; por ejemplo, morfología, desarrollo, así como respuestas bioquímicas y metabólicas (Bou et al., 2011). La desventaja de estos métodos, es que la identificación de bacterias considera solo aquéllas capaces de crecer en las condiciones dadas por el medio de cultivo y el proceso de incubación, dejando de lado aquellas que pudieran incluso ser más representativas, pero incapaces de crecer bajo las condiciones establecidas.
A pesar de sus limitaciones, este tipo de estudios generaron los primeros datos relacionados con la microbiota de peneidos. En primera instancia, se describió la composición bacteriana por aislados de cada región del tracto digestivo, utilizando medios de cultivo de agar TCBS (Tiosulfato citrato bilis sacarosa) y TSA (Agar triptona de soya) y una posterior identificación taxonómica hasta nivel de especie por medio del sistema BIOLOG-GN. Dichos estudios concluyeron que bacterias del género Vibrio forman parte de la microbiota normal en intestino, estómago y hepatopáncreas de camarón (Litopenaeus vannamei); sin embargo, un gran porcentaje no pudo identificarse hasta especie (Gomez-Gil et al., 1998; Oxley et al., 2002).
Oxley et al. (2002) encontraron que en camarones de la especie Penaeus merguiensis cultivada o silvestre, la composición a nivel de género era similar, incluyendo Aeromonas, Plesiomonas, Photobacterium, Pseudoalteromonas, Pseudomonas y Vibrio. Lo anterior fue revelado utilizando agar nutritivo con 4% de agua de mar y monitoreando características bioquímicas y morfológicas, considerando finalmente el uso de claves dicotómicas. Bajo esta aproximación, se detectaron por primera vez algunos miembros de la microbiota; no obstante, no fue posible llevar a cabo comparaciones más profundas utilizando cálculos más sofisticados.
Gracias a estas primeras aproximaciones se comenzó a vislumbrar el rol relevante de la microbiota en el desarrollo del hospedero; aunque más tarde, se demostró que los microorganismos identificados hasta el momento representaban solo una pequeña fracción del total de la población, dando pie al desarrollo de métodos basados en técnicas moleculares, los cuales a partir del año 2000, comenzaron a convertirse en alternativas populares para el análisis de comunidades microbianas, aunque fue hasta finales de la década pasada, cuando comenzó a aplicarse de manera regular en organismos acuáticos (Brunvold et al., 2007; Nayak, 2010).
Primeras Pruebas Moleculares (PCR, DGGE, RFLP)
La implementación de pruebas moleculares aumentó el conocimiento en la diversidad de comunidades microbianas respecto al que se tenía con las técnicas dependientes de cultivo. La clasificación de organismos procariotas (bacterias y arqueas), se basa en el análisis de la amplificación del gen 16S ARN ribosomal (16S ARNr) u otros biomarcadores taxonómicos como la subunidad beta del ADN polimerasa codificada por el gen rpoB (Case et al., 2007) o el gen gyrB que codifica para la ADN girasa (Yanez et al., 2003), entre otros. Sin embargo, el gen 16S ARNr es el más ampliamente utilizado debido a que abarca y clasifica gran parte de los microorganismos procariotas presentes en un ambiente, mientras que los otros biomarcadores complementan haciendo énfasis en grupos taxonómicos específicos; además, las bases de datos del gen 16S ARNr son mucho más robustas en comparación con el resto.
El gen 16S ARNr tiene aproximadamente 1,500 pb con regiones altamente conservadas para el diseño de cebadores y nueve regiones variables que funcionan como códigos para la identificación de diferencias filogenéticas y taxonómicas entre organismos. Además de los cambios nucleotídicos en las regiones variables, se presentan cambios en la longitud de estos fragmentos debido a la susceptibilidad de mutaciones que presentan los bucles o “loops” de las hélices de la estructura del gen (Vargas-Albores et al., 2017a). Los métodos moleculares para el análisis de comunidades microbianas se utilizan en mayor medida debido a la gran información que proporcionan. Por ejemplo, es posible identificar bacterias no cultivables por medio de la amplificación del gen 16S ARNr considerando las secuencias nucleotídicas conservadas para el diseño de cebadores adecuados (Yarza et al., 2014).
Las primeras aproximaciones moleculares de huella genética o fingerprinting, se llevaron a cabo mediante la amplificación del gen 16S ARNr y su posterior análisis por medio de técnicas de separación de productos como la Electroforesis en Gel con Gradiente de Desnaturalización (DGGE) (Liu et al., 2010) y su análogo con gradiente de temperatura (TGGE), así como Polimorfismo de Longitud de Fragmentos de Restricción (RFLP) (Li et al., 2007b), ofreciendo un panorama más amplio para el estudio de la composición microbiana presente en el tracto digestivo de camarones peneidos.
La técnica DGGE permitió conocer la composición y estructura de comunidades bacterianas presentes en intestino de camarones peneidos. La electroforesis de los amplicones del gen 16S en DGGE, demostró ser una herramienta eficiente para el estudio de la diversidad microbiana en varios ecosistemas (You et al., 2002); aunque se observaban los patrones de bandas, era solamente hasta que las bandas eran cortadas y secuenciadas que podía llevarse a cabo una clasificación taxonómica, ofreciendo un panorama más completo de la diversidad de bacterias presentes en cada muestra.
Una de las primeras aproximaciones a una descripción de la microbiota del camarón utilizando la técnica PCR-DGGE, se llevó a cabo en la especie Marsupenaeus japonicus. Liu et al. (2010), evaluaron el efecto del probiótico Bacillus sp. sobre la composición de la microbiota intestinal, extrayendo ADN de intestinos y posteriormente amplificando la región V3 del gen 16S ARNr. De ese modo, los principales hallazgos encontrados fueron referentes a la presencia de los grupos de bacterias en intestino de los camarones control: Gamma proteobacteria no cultivables y secuencias pertenecientes al género Vibrio, mientras que, en el grupo con tratamiento, se encontraron los géneros Pseudomonas y Stappia. En este tipo de ambientes no se tenían registros del género Stappia utilizando los métodos dependientes de cultivo.
En otro estudio, se demostró que el uso de fructooligosacáridos en dietas de camarones, favorecía el crecimiento de las especies de bacterias Alkalibacillus spp. y Micrococcus spp. en el tracto digestivo (Li et al., 2007c). En ese experimento se utilizaron las técnicas PCR-DGGE y secuenciación de las bandas aisladas; aunque algunas de las secuencias no fueron clasificadas hasta nivel de especie. Lo anterior se debió a que las secuencias no fueron encontradas en la base de datos “NCBI” (National Center for Biotechnology Information), la cual no era tan robusta como las actuales. Sin embargo, los resultados demostraron que el uso de fructooligosacáridos modificaba la microbiota intestinal, aunque solo se detectaban unos cuantos taxones, lo que tampoco permitió inferir que taxones contribuyeron al fortalecimiento del sistema inmune. En este sentido, muchos de los estudios resultaban ser básicamente descriptivos; no obstante, un nuevo campo de investigación comenzaba a desarrollarse.
Algunos años después, se realizaron dos análisis comparativos para el conocimiento de la composición bacteriana intestinal de Peanaeus monodon en distintas etapas de desarrollo. Se realizó secuenciación masiva utilizando pirosecuenciación y se encontraron hasta 88 géneros asociados a postlarvas de 15 días, 14 géneros para juveniles de un mes, 27 géneros para juveniles de dos meses y 20 géneros en juveniles de tres meses de edad. El análisis con DGGE permitió conocer los cambios en las comunidades bacterianas presentes con base a las diferencias en el patrón de bandeo y se concluyó que las bacterias de los filos Bacteroides, Firmicutes y Proteobacteria permanecían constantes independientemente de la etapa de desarrollo (Rungrassamee et al., 2013). Dicha técnica ofreció un marco de cobertura más amplio para el análisis de diversidad microbiana.
En 2006 la técnica T-RFLP fue considerada como una de la más utilizadas para el conocimiento de las comunidades microbianas en acuicultura (Balcázar et al., 2006). Ésta se basa en el reconocimiento y corte de secuencias específicas de ADN por enzimas de restricción, para obtener fragmentos de diferente longitud. Los fragmentos obtenidos se separan mediante electroforesis en geles de agarosa y ofrecen un patrón de bandas.
La técnica T-RFLP se ha utilizado para conocer la microbiota asociada a camarón blanco durante la etapa larval. Pangastuti et al. (2009), realizaron una digestión de amplicones del gen 16S ARNr con enzimas de restricción utilizando AluI o RsaI, midiendo el tamaño de los fragmentos generados y comparado con una base de datos, encontrando bacterias clasificadas hasta nivel del filo y subfilo, como Bacteroidetes y alfa-proteobacteria, respectivamente. De igual manera, esta técnica se utilizó para conocer los cambios entre comunidades bacterianas en intestino de camarón blanco frente a diversos tratamientos (Litopeaneus vannamei) (Li et al., 2007c; Thompson et al., 2010). Uno de los principales hallazgos de estas primeras aproximaciones moleculares fue la alta dominancia de filos como Firmicutes y Proteobacteria, concluyéndose además que “la mayoría de las bacterias asociadas con el intestino de camarón son especies no cultivadas y novedosas”.
A pesar del gran aporte brindado al nivel de información taxonómica presente, la utilización de este tipo de métodos no garantizaba la ausencia de errores o sesgo, debido a posibles traslapes de bandas, o a la no identificación de amplicones de baja representatividad, por lo que hoy en día estas técnicas están casi en desuso. Un resumen sobre las técnicas utilizadas para el conocimiento de la composición bacteriana del tracto digestivo de camarones peneidos se muestra en la Tabla 1.
Tabla 1 Estudios del microbioma de camarones peneidos utilizando las primeras técnicas moleculares y secuenciación masiva para análisis de biomarcadores.
| Especie | Órgano | Bacterias | Técnicas | Autores |
|---|---|---|---|---|
| Litopenaeus vannamei | Hepatopáncreas, intestino, estómago | Pseudoalteromonas y Vibrio | Cultivo en placa, PCR y secuenciación | (Tzuc et al., 2014) |
| Litopenaeus vannamei | Intestino | Bacterias del filo Firmicutes y Gamma-proteobacteria | PCR y T-RFLP | (Li et al., 2007b) |
| Penaeus monodon | Intestino | Actinobacteria, Fusobacteria, Proteobacteria, Firmicutes y Bacteroidetes | DGGE y secuenciación vs pirosecuenciación | (Rungrassamee et al., 2014) |
| Marsupenaeus japonicus | Intestino | Vibrio sp. Y Gamma- proteobacteria no cultivables | DGGE y secuenciación | (Liu et al., 2010) |
| Litopenaeus vannamei | Intestino | Cambios en el patrón de fragmentos obtenidos entre tratamientos | T-RFLP | (Thompson et al., 2010) |
El uso de DGGE y la posterior secuenciación de fragmentos, no bastó para conocer la composición total bacteriana intestinal de camarones peneidos (Chaiyapechara et al., 2012), pero gracias al surgimiento de otras técnicas como la secuenciación masiva, se amplió aún más el panorama en los estudios de diversidad microbiana.
La secuenciación de siguiente generación (NGS por sus siglas en inglés) ha resultado ser una opción de bajo costo y alto rendimiento que permite generar gran cantidad de secuencias mediante el uso de diversas plataformas (Kircher y Kelso, 2010). Existen diversos métodos o plataformas de secuenciación que varían en función del tipo de tecnología, la cantidad de lecturas que generan y la longitud de estas. Algunas técnicas son la secuenciación masiva por semiconductores que utiliza Ion Torrent (Life Technologies), pirosecuenciación o secuenciación 454 (Roche Diagnostics, Indianapolis, IN) y la secuenciación por síntesis en fase sólida, como es el caso de Illumina MiSeq (Illumina, San Diego, CA) (Glenn, 2011). Este último tipo de secuenciación presenta menores tasas de error y mayor rendimiento por ejecución (Loman et al., 2012).
La secuenciación masiva ha permitido realizar estudios sobre la microbiota en acuicultura, caracterizando la composición bacteriana presente en el tracto intestinal de camarones peneidos como Litopenaeus vannamei y Penaeus monodon (Cardona et al., 2016; Rungrassamee et al., 2016; Cornejo-Granados et al., 2017; Vargas-Albores et al., 2017b). Estas técnicas han permitido reducir costos y tiempo, a la vez que proveen de datos de manera robusta y altamente confiables.
Estado del arte en el estudio de las comunidades microbianas
En general, la estructura más estudiada de camarones es el intestino, la cual presenta una gran diversidad bacteriana y tiene implicaciones en el desarrollo del crustáceo. Cornejo-Granados et al. (2018), llevaron a cabo un meta-análisis para conocer los factores que influyen en la modulación de las funciones y composición de la microbiota de camarones marinos y de agua dulce; como el tipo de órgano, dieta, hábitat y edad de las diferentes especies. La mayor proporción de estudios reportados corresponden al intestino (80.1%), seguido de la microbiota asociada a todo el crustáceo (11.3%), al hepatopáncreas (6%), a las heces (2%) y en menor cantidad a las branquias (0.7%). Se considera que el intestino, estómago y el hepatopáncreas, e incluso las heces forman parte del tracto digestivo; sin embargo, referente al estómago no se reportan estudios de su microbiota por medio de secuenciación. Mientras que en el caso de peces y algunos crustáceos terrestres, se reportó la presencia de bacterias con actividad quitinolítica y celulolítica y otras bacterias en este órgano (Ringø, 2008; Wang y Wang, 2015). En cuanto a los camarones peneidos, independientemente de la especie, las familias de mayor abundancia en hepatopáncreas son Enterobacteraceae (59.68%), Vibrionaceae (16.23%) y Psedomonadaceae (11.42%). Mientras que en intestino predominan Mollicutes (10.71%), Rhodobacteracea (11.56%) y Vibrionaceae (12.58.3%) (Cornejo-Granados et al., 2018).
La microbiota del tracto digestivo en el desarrollo del camarón blanco, Litopenaeus vannamei
La microbiota del tracto digestivo comienza desde el nacimiento (o eclosión en algunos taxones) del organismo. En animales terrestres, como mamíferos, la microbiota es definida por la que posee la madre, la forma del parto y su alimento inicial; mientras que en animales acuáticos, como camarones peneidos, el tipo de microbiota intestinal se obtiene a partir del contacto directo con el ambiente (agua) (Palmer et al., 2007; Tzuc et al., 2014). El ciclo de vida de un organismo implica cambios en sus necesidades biológicas, ya sea para afrontar retos relacionados al ambiente dónde se desarrolla, o necesidades dictadas por los distintos tipos de sistemas con los que cuenta y lo mantienen vivo y funcional.
El desarrollo de crustáceos es muy dinámico y sufre cambios metamórficos, fisiológicos y metabólicos importantes que van de la mano con los requerimientos nutricionales para su desarrollo. Una vez alcanzada la fase postlarval, el ciclo de muda presenta diversas etapas: Postmuda, intermuda, premuda y muda. La frecuencia del ciclo de muda es mayor durante la primera etapa y va en disminución conforme la edad aumenta (Brusca y Brusca, 2003). El camarón blanco tiene diferentes etapas de desarrollo o estadios: primeramente, se presenta la etapa larval que comprende su forma de nauplio, protozoea y mysis, donde consume alimento primario como microalgas, el cual, en conjunto con la columna de agua, aportan los primeros microbios que comienzan a colonizar el tracto digestivo. Posteriormente, esta microbiota será influenciada por una combinación de factores genéticos propios del hospedero y del medio en el que se desarrolla. Por ejemplo, a diferencia de los camarones que se encuentran en el medio natural, los camarones de granja son expuestos a condiciones nutricionales y fisicoquímicas totalmente distintas. A este respecto, Cornejo-Granados et al. (2018), reportaron que la diversidad bacteriana del tracto intestinal de camarones peneidos es mucho mayor en condiciones de granja registrando índices de diversidad filogenética (IDF) de hasta 10, en comparación con aquellos criados bajo condiciones de laboratorio o colectados del medio natural (IDF ~6).
Los camarones de granja o cultivados, por ejemplo, son alimentados con fórmulas artificiales que contienen altas cantidades de proteína para potenciar su crecimiento; mientras que el camarón en el medio natural se alimenta de zooplancton y fitoplancton. También, se agregan potenciadores de fitoplancton como fertilizantes, y otros productos que, al acumularse, generan condiciones con altas cargas de bacterias y condiciones fisicoquímicas enrarecidas (Martínez-Cordova et al., 2009). En las etapas postlarvales, la diversidad de la microbiota es baja, incrementándose a partir de las etapas juveniles y adultas. Sin embargo, el meta análisis de similaridad (ANOSIM-R) de perfiles taxonómicos de microbiota intestinal de peneidos realizado por Cornejo-Granados et al. (2008), demostró que el estilo de vida, el órgano y la etapa de desarrollo, son factores que influyen en la composición de la microbiota del camarón; sin embargo, la etapa de desarrollo podría ser el factor menos influyente sobre la variación de la microbiota de peneidos.
La etapa postlarval de los peneidos es la más susceptible a enfermedades bacterianas y se desconoce casi por completo el rol que juega su microbiota intestinal en la resistencia ante patógenos. Zheng et al. (2016), reportaron que durante las etapas de desarrollo zoea, mysis y postlarval de camarones sanos, se detectan de manera consistente los filos Proteobacteria y Bacteroidetes, aunque con variaciones en su representatividad. En camarones enfermos, también predominó el filo Proteobacteria, en este caso se aislaron bacterias que pudieron clasificarse hasta nivel de género: Vibrio y Nautella. El anterior estudio se realizó para entender cómo la microbiota intestinal afecta al hospedero según su estado de salud y su etapa de crecimiento. Se conoce que la composición asociada a todo el organismo sufre modificaciones de acuerdo a los cambios durante su desarrollo y dependiendo de su estado de salud. Este estudio partió de un cultivo en placa en agar marino, una posterior amplificación del gen 16S ARNr de las bacterias aisladas y finalmente, la secuenciación de los amplicones obtenidos.
Recientemente, el estudio del perfil taxonómico y su funcionalidad, permitió conocer de manera muy general el comportamiento de los microorganismos durante las etapas de desarrollo del camarón blanco, L. vannamei.Zeng et al. (2017), colectaron organismos cada 15 días durante 60 días de una graja comercial, los cuales fueron previamente expuestos a dosis semanales de Lactobacillus y Bacillus, como probióticos mezclados en el alimento. El perfil taxonómico presentó variaciones significativas estadio-específicas a nivel de Filo. Respecto a la predicción de la funcionalidad del perfil taxonómico, se encontró que los microorganismos participan o presentan funciones como proteínas transportadoras, reparación de ADN, metabolismo de purina y metabolismo de acetato. Así mismo, los géneros Lactobacillus y Bacillus utilizados como probióticos, se han encontrado en proporciones muy bajas (0.04% y 0.9%, respectivamente), por lo que no se presenta su adherencia en el tracto digestivo, aun cuando estos son aplicados de manera desmedida en granjas acuícolas.
Huang et al. (2016) caracterizaron la microbiota asociada al intestino del camarón blanco durante su etapa postlarval y juvenil: al día 14 y en los meses 1, 2 y 3. Para ello, se llevó a cabo la amplificación y secuenciación masiva del gen 16S ARNr considerando las regiones variables 3 y 5, encontrándose que la composición es altamente dinámica, además de influir en el desarrollo postlarval y juvenil del camarón.
En organismos superiores como mamíferos, se ha demostrado la presencia de complejas redes de comunicación celular entre huésped y hospedero, en el que ciertas señales del hospedero dan forma a un cierto tipo de microbiota, pero que, en algunas ocasiones, ese tipo de señales son en realidad promovidas por mensajes primarios de la microbiota, que activan ciertas funciones en el hospedero (Nicholson et al., 2012). A este respecto, existe evidencia de que la microbiota mantiene una señalización con el hospedero, provocando cambios o respuestas metabólicas; sin embargo, la base mecanística de su interacción apenas comienza a revelarse en algunos organismos, aunque por ejemplo, se sabe que una disbiosis intestinal puede afectar el funcionamiento fisiológico de un organismo, haciéndolo susceptible a enfermedades (Holmes et al., 2011). En el caso de organismos menos complejos como los camarones peneidos, los estudios sugieren que el estado de desarrollo tiene influencia sobre la microbiota del tracto digestivo; sin embargo en realidad no se han llevado a cabo estudios sistemáticos para demostrarlo. Para ello se debe considerar el estudio de todos los órganos del tracto digestivo del camarón, además de mantener condiciones casi axénicas, que permitan aislar de mejor manera el factor del estadio de desarrollo. Esta información, revelaría datos desconocidos acerca de la biología del camarón.
Factores determinantes para el desarrollo de la microbiota del tracto digestivo
Muchos son los factores reportados que modifican la microbiota del tracto digestivo en camarones peneidos, incluyendo el uso de probióticos o prebióticos (Li et al., 2007c; Sha et al., 2016), el tipo de ambiente (silvestre o cultivado), las condiciones de estrés provocadas por algún patógeno, así como el desarrollo natural del organismo (Cornejo-Granados et al., 2017), entre otras. Algunas de las especies de crustáceos de importancia acuícola cuya composición microbiana intestinal se ha estudiado, son Fenneropenaeus indicus, Penaeus monodon y principalmente Litopenaeus vannamei (Liu et al., 2011; Chaiyapechara et al., 2012; Tzuc et al., 2014). A este respecto, Li et al. (2018) mencionan que los principales factores que modulan la microbiota intestinal de L. vannamei son el estadio o edad del camarón, el estado de salud, factores ambientales y componentes de la dieta. En relación a la dieta, esta puede ser manipulada para promover el crecimiento de bacterias benéficas, haciendo uso de prebióticos y probióticos.
Los estudios realizados con el fin de comprender mejor la comunidad microbiana en el intestino de camarones peneidos, representan información útil para el uso potencial de estas comunidades. Una posible aplicación es en forma de probióticos, ya que algunas bacterias ayudan a degradar componentes de la dieta debido a su capacidad para producir enzimas extracelulares, dejando biodisponibles nutrientes; además de producir vitaminas útiles para el hospedero. Algunas de las bacterias probióticas utilizadas en camarones son Lactobacillus y Bacillus, capaces de producir péptidos antimicrobianos; así como Streptomyces, Enterococcus faecium y Rhodosporidium paludigenum, las cuales pueden mejorar la respuesta inmune y reducir el estrés oxidativo (Li et al., 2018). Entender como la microbiota interactúa con el hospedero, representa información que puede llevar a comprender tanto la biología de la especie, como la aparición de algunas enfermedades.
Los estudios de la composición bacteriana intestinal de organismos marinos, se han enfocado en evaluar los cambios en su perfil taxonómico ante su estado de desarrollo, la exposición a patógenos, probióticos, tipo de ambiente y uso de bioflóculos o biopelículas (Pérez et al., 2010; Clements et al., 2014). Por ejemplo, al comparar la composición de la microbiota intestinal de camarón blanco de una granja acuícola durante la siembra y la cosecha, mediante la secuenciación masiva de las regiones variables 2 y 3 del gen 16S ARNr, se encontraron diferencias significativas en la alfa y beta diversidad, es decir en la cantidad y tipo de especies encontradas (Gainza et al., 2017); este tipo de modificación en la microbiota puede tener múltiples causas, que van desde el estado de desarrollo del crustáceo, hasta las condiciones del medio en el que se desarrolla.
En cuanto a exposición a patógenos, Rungrassamee et al. (2016) determinaron que la exposición de L. vannamei y P. monodon a la bacteria patógena Vibrio harveyi, aumentó la tasa de mortalidad del camarón cultivado; los autores concluyeron que la estabilidad de la microbiota intestinal es vital para prevenir la colonización de microorganismos patógenos; además, un buen manejo de la misma, podría contribuir a la prevención de enfermedades en el cultivo de camarón.
Una forma de modulación de microorganismos en cultivos de camarón se ha llevado a cabo mediante la introducción de bacterias deseables como los probióticos. Por ejemplo, en camarón blanco, la bacteria Bacillus licheniformis se utilizó como probiótico para L. vannamei, causando una reducción en la cantidad de bacterias del género Vibrio, mejorando la microbiota intestinal y el sistema inmune (Li et al., 2007a; Sha et al., 2016). De la misma forma, se utilizó una mezcla de bacterias del género Bacillus como probiótico para camarones infectados con Vibrio parahaemolyticus, logrando aumentar la supervivencia y resistencia al patógeno, así como una modulación en la microbiota intestinal (Luis-Villasenor et al., 2013; Luis-Villaseñor et al., 2015). Estos estudios demuestran que es posible una alteración de la microbiota intestinal por efecto de la adición de probióticos, ya sea en el alimento o directamente en el estanque.
Otros reportes han mostrado que el uso de alimento adicionado con prebióticos, parece favorecer el crecimiento de cierto grupo de bacterias. Por ejemplo, la adición de fructooligosacáridos de cadena corta en la dieta de L. vannamei, causó un incremento de peso e inmunidad del hospedero, lo que fue asociado a una modificación en la composición de la microbiota intestinal, reflejada en un aumento en la cantidad de bacterias probióticas, por consiguiente mejoró la supervivencia del organismo (Li et al., 2007c; Zhou et al., 2007). También, el uso de probióticos y prebióticos ha mostrado un efecto en la inmuno-modulación, potenciando la sustitución de antibióticos‐ para mejorar la salud del organismo en cultivo (Vargas Albores et al., 2016).
La bioaumentación es una estrategia utilizada en las tecnologías de bioflóculos y biopelículas, utilizando a cierto grupo de bacterias especializadas, mediante una modulación de variables ambientales o sustrato de crecimiento (Salência et al., 2016; Martínez-Córdova et al., 2018). Estas tecnologías ayudan a mejorar la calidad del cultivo de camarones y han mostrado ser un factor influyente sobre la composición de la microbiota intestinal de camarones peneidos como L. vannamei y L. stylirostris (Cardona et al., 2016), ya que éstos consumen masa microbiana como alimento y en consecuencia algunas de las bacterias que conforman este tipo de consorcios, terminan colonizando el intestino y desplazando a otras. A este respecto Garibay-Valdez et al. (2019) demostraron que el consumo de biopelículas no solamente mejoró la respuesta productiva, sino que moduló la microbiota intestinal de L. vannamei, disminuyendo la dominancia característica del filo Proteobacteria, particularmente el géneroVibrio, lo cual es relevante para el cultivo de esta especie, debido a los problemas comunes de vibriosis. Los autores reportaron que, aunque el perfil taxonómico de la microbiota cambiaba con el uso de biopelículas, su funcionalidad aparente permanecía estable, sugiriendo que el camarón posee condiciones que garantizan una redundancia funcional independientemente de cambios en los miembros de su microbiota.
El camarón blanco Litopaneaus vannamei, es la especie más relevante de la acuicultura mexicana y mundial, por lo que es objeto de estudio desde múltiples perspectivas y disciplinas. No obstante, a pesar de los estudios mencionados, todavía se cuenta con poco conocimiento en relación a la microbiota de su tracto digestivo, la cual es una parte importante de su biología. Al igual que para muchos organismos del reino animal, existen factores extrínsecos e intrínsecos que pueden modificar la estructura de la comunidad microbiana de su tracto digestivo; sin embargo, se ha reportado que en el camarón blanco existe un núcleo microbiano (microbial core) que, a pesar de estos factores, se mantiene constante (Huang et al., 2016), lo que hace emerger la hipótesis de que estrategias encaminadas a mantener dicho núcleo serían un aporte para un estado saludable de estos crustáceos.
Específicamente, Huang et al. (2016) mencionan que los filos Proteobacteria, Bacteroidetes y Actinobacteria parecen estar presentes en todo el ciclo de desarrollo de L. vannamei. Además, Firmicutes, Planctomycetes, Chlamydiae y Cyanobacteria también forman parte de la microbiota intestinal natural del camarón, indistintamente de la adición de probióticos y posiblemente del medio en el que se cultivan (Vargas-Albores et al., 2017b). En este sentido, Zoqratt et al. (2018), mencionan que el hospedero ejerce una selección específica de la microbiota del tracto digestivo. Los autores realizaron un análisis en la diversidad de intestino y el ambiente (agua) de dos cultivos de camarón bajo condiciones climáticas distintas. La diferencia en las comunidades microbianas fue más notoria en muestras de agua respecto a los intestinos. Esto puede deberse a la selección ejercida por el hospedero hacia las bacterias que puedan adaptarse en este ambiente.
La diversidad bacteriana intestinal de camarón blanco es reportada por diversos autores a pesar de haber utilizado técnicas muy diferentes. Las bacterias del género Aeromonas, Plesiomonas, Photobacterium, Pseudomonas, Pseudoalteromonas y Vibrio, han sido las más constantes en el intestino de esta especie. El conocimiento en la composición bacteriana, trajo consigo nuevas herramientas para el desarrollo de la acuicultura; por ejemplo, algunos géneros como Vibrio y Pseudoalteromonas se han utilizado como probióticos (Oxley et al., 2002). Otros autores coinciden en proponer al género Pseudoalteromonas como candidato a utilizarse como probiótico por su capacidad para sobrevivir en el tracto digestivo y además producir compuestos antimicrobianos (Tzuc et al., 2014). Por otro lado, este tipo de información puede dar pie a investigaciones relacionadas con la búsqueda de patrones en la abundancia de taxones que podrían tener efectos positivos o negativos sobre el desarrollo y estado fisiológico e inmune del camarón, tal como ocurre en investigaciones relacionadas con mamíferos.
En L. vannamei, la funcionalidad y composición bacteriana presente en intestinos, se relaciona con el estado de salud de este crustáceo. Zhu et al. (2016) realizaron un análisis de la funcionalidad de la composición bacteriana intestinal de dos grupos de camarón blanco, uno enfermo y otro sano; mostrando grandes cambios en las funciones entre los dos grupos. Por ejemplo, en camarones enfermos se registró un aumento de una ruta de factor de virulencia (adhesión focal), la cual es característica de microorganismos patógenos para establecer una infección. Los organismos enfermos resultaron ser más propensos a la invasión de patógenos, lo que estaba asociado no solamente a un sistema inmune deprimido, sino a un cambio en la funcionalidad de su microbiota (Zhu et al., 2016; Xiong et al., 2017). Xiong (2018), describe la relación entre la microbiota intestinal y el estado de salud del camarón; específicamente, organismos con una disbiosis intestinal son más propensos a enfermedades, mientras que aquellos con un balance en la microbiota muestran mayor resistencia a patógenos. Algunas bacterias pueden aumentar o disminuir según el estado de salud. La microbiota del tracto digestivo, participa en múltiples funciones relacionadas con el estado de salud del hospedero y contribuye a su fisiología.
La talla y peso de camarones de edad similar se correlacionan con su composición bacteriana intestinal. Xiong et al. (2017), mantuvieron a los organismos en una granja acuícola bajo las mismas condiciones y los dividieron según presentaran un crecimiento retardado, normal y sobre-crecimiento, con base a su peso y talla. Considerando al crecimiento normal como control, encontraron que la familia Marinicellaceae aumenta su proporción en organismos con sobre -crecimiento, mientras que en los de crecimiento retardado disminuye, respecto al control. La familia Saprospiraceae mostró el mismo comportamiento. Estas familias poseen gran capacidad de hidrólisis y la utilización de fuentes complejas de carbono, lo cual conlleva a un trabajo sinérgico con el hospedero (Sommer y Bäckhed, 2013). Por lo que se correlaciona con un aumento en el metabolismo de carbohidratos en organismos con sobre-crecimiento. Además, incrementó la actividad de enzimas digestivas (amilasa, pepsina, lipasa) en dichos organismos (Xiong et al., 2017). Lo anterior podría sugerir que existe un trabajo facilitado por grupos de microorganismos en el tracto digestivo, los cuales pueden o no, aumentar la eficiencia en la adquisición de nutrientes.
La microbiota del tracto digestivo (estómago, hepatopáncreas e intestino) del camarón blanco, como ya se mencionó, participa en procesos digestivos debido a la aportación enzimática de las propias bacterias. Se ha reportado que bacterias del género Vibrio y Pseudoalteromonas se encuentran formando parte de la microbiota del tracto digestivo del camarón blanco y que además, estos géneros contribuyen a la degradación de algunos componentes dietarios como amilasas, lipasas/esterasas y quitinasas (Tzuc et al., 2014). Por ello se asume que la eficiencia del sistema digestivo sería menor sin el aporte de la microbiota intestinal. En ese estudio se utilizaron cepas con capacidad proteolítica y fueron identificadas mediante la amplificación y secuenciación del gen 16S ARNr.
Las etapas de cultivo de camarón blanco influyen en la composición bacteriana intestinal. En camarones sanos, la microbiota intestinal está regulada por el estado de desarrollo del sistema inmune y la fisiología intestinal sobre la ontogénesis del huésped, lo que da como resultado una microbiota intestinal dependiente de la edad. Gainza et al. (2017) determinaron que los camarones de la etapa de maternización difieren de los de cosecha en su composición bacteriana intestinal bajo condiciones de cultivo intensivo. Se encontró que en la etapa de maternización dominó el filo CKC4 (Bacteria no cultivable), principalmente, seguido de los filos Proteobacteria, Actinobacteria y Firmicutes, mientras que durante la etapa de cosecha los filos dominantes fueron; Proteobacteria, seguido de Chloroflexi y Actinobacteria, siendo esta la etapa con mayor riqueza respecto a la etapa de maternización. Así, con esta información, el conocimiento en la ecología de estos ambientes puede ayudar a optimizar el manejo del cultivo para una mayor producción y calidad como alimento.
Perspectivas
Los estudios del tracto digestivo en camarones peneidos, han comprendido casi exclusivamente a los microorganismos presentes en intestino, dejando de lado los otros dos órganos del sistema digestivo (hepatopáncreas y estómago); además, la capacidad funcional y actividad funcional in situ de estas comunidades microbianas, es un tema muy poco abordado, principalmente debido a la complejidad de discriminar, ordenar e interpretar esta “masa” de datos por medio de herramientas bioinformáticas, las cuales hasta hace poco, no eran lo suficientemente especializadas ni robustas. Es por ello que contemplar estudios de la comunidad microbiana de todo el tracto digestivo, no solamente en términos de caracterización, sino también, sobre la funcionalidad de grupos particulares y de la comunidad en su conjunto, resultan cruciales para el desarrollo y éxito de la producción acuícola, como el cultivo de camarón.
Una de las expectativas a corto plazo es considerar la secuenciación del gen completo, por medio de la tecnología se secuenciación en tiempo real de una sola molécula (SMRT) desarrollada por Pacific Biosciences (PacBio) (Eid et al., 2008; Rhoads y Au, 2015). Wagner et al. (2016), mencionan que la tasa de error obtenida con PacBio es muy similar a la de la plataforma Illumina Miseq y Roche 454; aunque se obtiene una menor cantidad de lecturas, se espera que ésta y otras tecnologías eventualmente desplacen a la actual secuenciación por síntesis de fragmentos pequeños de máximo 2 × 300 pares de bases. Adicionalmente, este tipo de tecnologías cuyas lecturas son más largas, ayudarán eventualmente a obtener el genoma ensamblado de camarones peneidos, con lo que será posible realizar secuenciación escopeta del ADN y ARN del microbioma del tracto digestivo de éstos, filtrando el ADN del hospedero para finalmente producir resultados confiables y conclusiones más acertadas sobre estas poblaciones microbianas y sus respectivas funciones.
Se espera que el uso de metagenómica y metatranscriptómica contribuya de manera significativa al conocimiento de la microbiota del camarón, superando las limitaciones de la inferencia de funciones a partir de biomarcadores. Esto se prevé que ocurra una vez que se hayan logrado ensamblar exitosamente aquellos genomas complejos de camarones peneidos. Esta información podrá ser utilizada desde puntos de vista biológicos y biotecnológicos, la cual además será útil para comprender de mejor manera las interacciones huésped-hospedero y hospedero-hospedero.
Otro asunto trascendental sería la dilucidación de todas las redes de interacción que existen entre los microorganismos que constituyen la microbiota del tracto digestivo, así como con el hospedero. A este respecto, el avance de las ciencias ómicas (Genómica, Transcriptómica, Proteómica y Metabolómica), han producido inmensas cantidades y matrices de datos, los cuales son imposibles de interpretar sin la ayuda de la informática. Una de las técnicas para visualizar redes complejas e integrarlas con cualquier tipo de datos o atributos, es la representación gráfica por nodos y bordes (Kohl et al., 2011). Por ejemplo, los nodos pueden ser representaciones de taxones, funciones, o incluso moléculas biológicas y los bordes conectan los nodos que representan algún tipo de relación. Este tipo de análisis está tomando auge para el estudio de comunidades microbianas y seguramente los camarones peneidos serán incluidos.
Por último, se espera que el estudio del hologenoma del camarón, el cual se considera como la combinación genética y co-evolutiva del huésped y sus microbios, ofrezca información reveladora acerca de los distintos mecanismos a través de los cuales se benefician ambas partes.
Conclusiones
A partir de los estudios consultados se concluye que la microbiota del tracto digestivo de camarones peneidos parece ser fundamental a lo largo de su ciclo de vida. Aunque la información con la que se cuenta al momento es relativamente escasa, se intuye que las funciones de la microbiota son similares a las reportadas en animales superiores, es decir, relaciones de simbiosis y mutualismo, a partir de las cuales el camarón asegura, en cierto modo, su supervivencia y desarrollo. Aunque esta microbiota es altamente variable dependiendo principalmente de factores como la dieta y condiciones ambientales, el filo Proteobacteria es el más común. Por otro lado, se desconoce si a pesar de estos cambios a nivel taxonómico, se mantiene una redundancia funcional de la microbiota. La tendencia hacia los próximos años es que las funciones e interrelaciones huésped-hospedero sean comprendidas a mayor profundidad.











 nova página do texto(beta)
nova página do texto(beta)


