Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Acessos
Acessos
Links relacionados
-
 Similares em
SciELO
Similares em
SciELO
Compartilhar
Boletín médico del Hospital Infantil de México
versão impressa ISSN 1665-1146
Bol. Med. Hosp. Infant. Mex. vol.68 no.2 México Mar./Abr. 2011
Artículo
Serotipos de dengue en México durante 2009 y 2010
Dengue serotypes in México during 2009–2010
Mauricio Vázquez–Pichardo,1 Claudia Rosales–Jiménez,1 Alma Núñez–León,1 Pilar Rivera–Osorio,1 Sergio De La Cruz–Hernández,1 Adriana Ruiz–López,1 Silvia González–Mateos,1 Irma López–Martínez,1 José Cruz Rodríguez–Martínez,2 Hugo López–Gatell,2 Celia Alpuche–Aranda1
1 Instituto de Diagnóstico y Referencia Epidemiológicos (InDRE)
2 Dirección General de Epidemiología; Secretaría de Salud, México, D.F., México
Autor de correspondencia:
Dra. Celia Alpuche Aranda
Correo electrónico: celiam@servidor.unam.mx
Fecha de recepción: 24–01–11
Fecha de aceptación: 27–01–11
Resumen
Introducción. El dengue en México es un problema prioritario de salud pública. Desde el 2008 el Departamento para la Vigilancia Epidemiológica y Virológica del InDRE implementó un nuevo algoritmo de diagnóstico del dengue, que utiliza la Red de Laboratorios Estatales de Salud Pública, para favorecer la representatividad geográfica, la oportunidad, la sensibilidad y la especificidad de la información que se obtiene.
Métodos. La identificación de serotipos se realizó a partir de muestras positivas a la proteína NS1 por ensayo inmunoenzimático (ELISA). Las técnicas que se utilizaron fueron: aislamiento viral, PCR punto final y, desde 2009, RT–PCR en tiempo real (qRT–PCR).
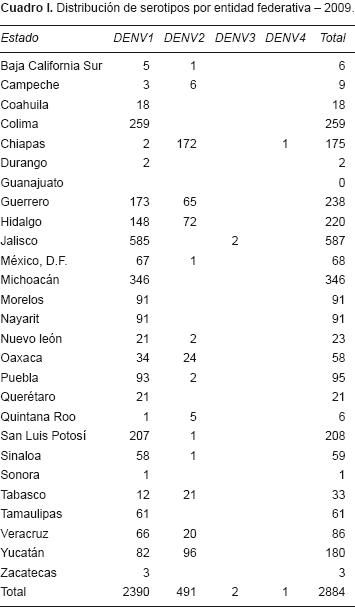
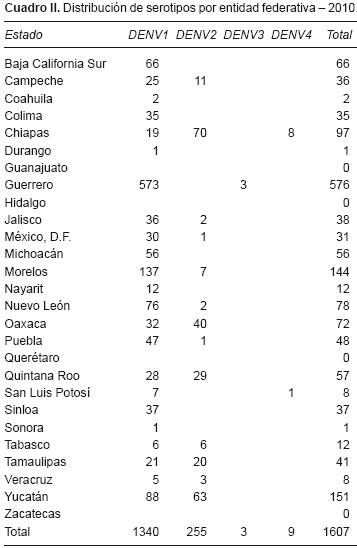
Resultados. En 2009 se analizaron 6,336 muestras; en 2,944 de éstas (46.6%) se identificó el serotipo DENV–1 que predominó sobre el serotipo DENV–2; el serotipo DENV–3 sólo se identificó en dos casos en Guerrero y el serotipo DENV–4 en un caso en Chiapas. En 2010 se analizaron 2,013 muestras. Se identificó algún serotipo en 1,607 muestras (79.88%) y, nuevamente, el serotipo DENV–1 predominó en todo el país. En Chiapas se identificaron los serotipos DENV–1, 2 y 4 y en Jalisco los serotipos DENV–1 y 3. Además, se identificó la circulación del serotipo DENV–3 en Guerrero y apareció el serotipo DENV–4 en San Luis Potosí.
Conclusiones. Por la selección de muestras para vigilancia virológica de dengue mediante la positividad a la proteína NS1 y por la introducción de la técnica de qRT–PCR se optimizó la identificación de serotipos circulantes. La alta endemia, los brotes en nuevas regiones, el predominio del serotipo DENV–1 por varios años y la introducción lenta de otros serotipos, principalmente DENV–3, pueden favorecer la aparición de formas clínicas graves de dengue. La vigilancia epidemiológica inteligente del dengue brindará información para un mejor entendimiento de la enfermedad y promoverá acciones para su control y prevención.
Palabras clave: serotipos de dengue, aislamiento viral, RT–PCR, fiebre por dengue, fiebre hemorrágica por dengue.
Abstract
Background. Dengue is a public health priority in Mexico. Since 2008, the dengue diagnostic algorithm for epidemiological and virological surveillance has been improved at InDRE and the public health laboratory network (RLESP) to optimize geographic representation, opportunity, sensitivity and specificity of the produced information.
Methods. Dengue serotype identification is based on ELISA NS1 positive samples. Methods used are viral isolation, endpoint PCR and, since August 2009, real–time PCR (qRT–PCR).
Results. In 2009, 6,336 serum samples were analyzed and 2,944 (46.6%) were positive for serotype identification. DENV–1 was detected in greater proportion followed by DENV–2, and DENV–3 4 was only identified in two cases in Guerrero and DENV–4 in one case in Chiapas. In 2010, 2,013 serum samples were analyzed and 1,607 (78.8%) were positive for serotype identification. DENV–1 was predominant throughout the country. In Chiapas, DENV–1, 2 and 4 were identified and in Jalisco DENV–1 and 3. DENV–3 was identified in Guerrero again and DENV–4 was detected in San Luis Potosí.
Conclusions. The selection samples through NS1 positive samples and the introduction of qRT–PCR optimized serotype identification. Hyperendemicity, outbreaks in new geographic areas, the predominant circulation of DENV–1 for several years and the slow reintroduction of the other serotypes, mainly DENV–3, could increase clinical cases of severe dengue. An ¡intelligentí epidemiological surveillance program would offer information for a better understanding of the disease and promote action for its control and prevention.
Key words: dengue serotypes, viral isolation, RT–PCR, dengue fever, dengue hemorrhagic fever.
INTRODUCCIÓN
Desde la aparición del hombre las enfermedades virales han sido una de las principales causas del deterioro en la salud del individuo. El dengue es la enfermedad viral transmitida por artrópodos más importante a escala mundial. La etiología de la enfermedad fue descubierta a partir de 1940. En la década de los cincuenta se tenía registro de nueve países con problemas de dengue; actualmente se reportan más de cien. La Organización Mundial de la Salud (OMS) estima que más de 2.5 mil millones de personas se encuentran en riesgo de contraer la enfermedad. Estos datos parecen no ser tan precisos debido al amplio espectro clínico que se ha observado y a la presencia de personas infectadas asintomáticas de las cuales no se tienen datos exactos.1 En México el dengue es considerado, a nivel nacional, como un problema prioritario de salud pública. En 1995 México contaba con 15 estados con circulación viral; actualmente se tienen detectados 29. En 2008 se detectaron entidades con circulación de más de un serotipo al mismo tiempo.2 La hiperendemicidad promueve la presencia de reinfecciones por serotipos diferentes, mismas que están relacionadas con la presencia de cuadros clínicos graves. La infección con el virus de dengue (DENV) puede resultar como una infección subclínica, con fiebre indiferenciada, pasando por un cuadro febril parecido a influenza conocido como fiebre por dengue (FD) hasta una manifestación severa, con presencia de hemorragias, denominada fiebre hemorrágica por dengue (FHD) o el síndrome de choque por dengue (SCD). El cuadro clásico de dengue es: fiebre, cefalea, mialgias, artralgias, dolor ocular, náusea, vómito y, en algunos casos, exantema; en los casos graves se presenta, además, daño hepático dependiente de serotipo y hemorragias a cualquier nivel (epistaxis, gingivitis, melena, etcétera).1,3,4
La FD y la FHD son causadas por cualquiera de los cuatro serotipos del virus perteneciente al complejo dengue; se trata de cuatro serotipos antigénicamente relacionados y conocidos como DENV–1, DENV–2, DENV–3 y DENV–4, pertenecientes a la familia Flaviviridae, del género Flavivirus, que son transmitidos por la picadura del mosquito hembra de las especies Aedes aegypti y Aedes albopictus. La infección con un serotipo confiere inmunidad especifica de por vida para ese serotipo e inmunidad parcial para los otros tres serotipos durante los tres primeros meses después de la infección.5,6
Las técnicas de laboratorio para la detección de infecciones por el virus de dengue se determinan dependiendo del tiempo de iniciada la fiebre (Figura 1). El DENV tiene un periodo de incubación de tres a siete días (antes de que se presente la fiebre) y continúa circulando en la sangre hasta cinco días después de iniciada la fiebre. Durante este periodo (fase aguda) el virus y el antígeno viral pueden ser detectados en el suero. Después del sexto día de iniciada la fiebre se pueden detectar los anticuerpos IgM como diagnóstico de infección actual y, para el día 14, se pueden detectar los anticuerpos IgG. En reinfecciones por serotipos diferentes se observa una respuesta inmune anamnésica, en la cual la viremia es más corta y los anticuerpos IgM se detectan en un periodo más corto y en concentraciones menores; la concentración de los anticuerpos IgG se incrementa en la fase aguda de la enfermedad. Durante esta fase aguda se emplean métodos virológicos para determinar el serotipo viral, como el aislamiento viral en células de mosquitos [C6/36 (A. albopictus), AP61 (A. pseudoscutellaris)] y en células de mamífero [Vero (riñón de mono verde), BHK21 (riñón de hámster)], además de los métodos de biología molecular como la RT–PCR en punto final y en tiempo real.7 Las altas concentraciones de antígeno viral NS1 durante la fase aguda de la enfermedad permiten determinar mediante métodos inmunoenzimáti–cos (ELISA) las infecciones por DENV, más no identificar el serotipo infectante.8,9

Los métodos serológicos para el diagnóstico de infección aguda sólo pueden realizarse con ELISA de captura para la determinación de anticuerpos IgM después de 6 días de fiebre o con muestras pareadas de fase aguda y convalecencia de la primera semana y después de 2 semanas de fiebre, para la determinación del incremento del título de anticuerpos IgG.10,11
Con la necesidad de brindar un diagnóstico oportuno y eficaz el InDRE implementó, en 2008, un nuevo algoritmo de diagnóstico en el que se incluyen todas las técnicas antes mencionadas. Actualmente México cuenta con una Red de Laboratorios Estatales de Salud Pública (RLESP), conformada por 30 laboratorios, que realizan la confirmación del diagnóstico de dengue mediante el uso de técnicas inmunoenzimáticas para detección del antígeno viral NS1, de anticuerpos IgM o de IgG, dependiendo del tiempo de evolución de la enfermedad cuando el paciente busca atención médica. Con el tamizaje de muestras positivas a proteína NS1 se optimiza el muestreo para la identificación de serotipos de dengue en las muestras en las que el virus está presente y se seleccionan muestras de todo el país. Desde agosto del 2009 en el InDRE se implementó la técnica de RT–PCR en tiempo real (qRT–PCR) para el diagnóstico de dengue; previamente se realizaba tipificación con PCR punto final. Durante la pandemia de influenza de 2009 fue posible dotar a la RLESP con equipos para el qRT–PCR y de entrenamiento en esta técnica para detectar influenza. Esta tecnología ha servido de base para el desarrollo y la implementación progresiva de la qRT–PCR para dengue, y no sólo en el InDRE sino también en la RLESP del país.
No existe actualmente una vacuna para la prevención de la enfermedad ni tratamientos antivirales específicos. El tratamiento de los pacientes con sospecha de padecer dengue consiste en la rehidratación, la administración de antipiréticos y de paracetamol; este último debe ser administrado con precaución. El ácido acetilsalicílico está contraindicado.12
Una parte importante de la vigilancia epidemiológica del dengue es la vigilancia virológica que analiza la distribución anual de los diferentes serotipos de DENV. Esto permite identificar serotipos asociados a cuadros de mayor severidad o predecir la posible variación cíclica de estos serotipos y, por tanto, el aumento de la susceptibilidad y el mayor impacto de la enfermedad. En este trabajo describimos la distribución en México de los serotipos de dengue circulantes en los últimos dos años, demostrando que el nuevo algoritmo de diagnóstico optimiza la vigilancia vi–rológica oportuna para generar la información que oriente las acciones de prevención y control de la enfermedad.
MÉTODOS
Para la identificación viral se utilizaron muestras de suero de pacientes con diagnóstico de dengue en fase aguda, confirmado mediante ELISA NS1 (Platelia–Biorad) siguiendo la metodología indicada por la casa comercial. Los sueros provenientes de la RLESP de todo el país se recibieron en el laboratorio de Arbovirus del InDRE y se almacenaron a 4°C hasta su análisis virológico para la identificación de serotipos.
RT–PCR multiplex en tiempo real. El personal del InDRE fue entrenado en el Center For Disease Control and Prevention – Dengue Branch de San Juan, Puerto Rico, en la metodología de qRT–PCR para dengue; esta metodología ya ha sido evaluada y validada para la identificación de serotipos de dengue. Implementada y validada en el InDRE la técnica se comenzó a utilizar como parte del algoritmo de dengue a partir de agosto del 2009. La extracción de ARN se realizó siguiendo las instrucciones del estuche comercial (QIAamp Viral RNA Mini Kit – QIAGEN) y se utilizaron 5 µL del ARN extraído para la reacción de RT–PCR en tiempo real, en un volumen total de 25 µL; se utilizaron iniciadores universales y sondas específicas para cada uno de los cuatro serotipos. La mezcla de reacción se llevó a cabo en un tubo comercial (iScript Platinum III, INVITROGEN) que contenía 100 pmol de cada iniciador y la sonda. La reacción de amplificación se realizó en un termociclador CFX96 (Biorad).13
Aislamiento viral. Esta metodología fue realizada únicamente durante los primeros siete meses del 2009 y fue reemplazada por la RT–PCR en tiempo real. La metodología del aislamiento viral aplicada es una modificación del método de Gubler.14 Se inocularon 50 µL de suero en tubos con una monocapa confluente de células de la línea celular C6/36 mantenida en MEM (GIBCO–BRL) a 5% de suero fetal de ternera (GIBCO–BRL). Los tubos fueron incubados a 28°C durante 8 días. La identificación del serotipo se realizó mediante inmunofluorescencia indirecta, utilizando anticuerpos monoclonales (MAb 2H2, MAb 3H5, MAb 5D4 y MAb 14H10, específicos para DENV–1, 2, 3 y 4, respectivamente). Las células fueron teñidas con un anticuerpo antidengue, producido en ratón y conjugado con FITC, y observadas en el microscopio de inmunofluorescencia para la detección de células infectadas. El aislamiento viral se continúa realizando en una subpoblación por región de los serotipos predominantes DENV–1 y DENV–2 y en todos los aislamientos de DENV–3 y DENV–4.
RESULTADOS
En 2009 se analizaron un total de 6,336 muestras de suero; se identificó el serotipo viral en 2,944 de ellas. El DENV–1 se identificó en mayor proporción seguido del DENV–2 y, finalmente, en la misma proporción DENV–3 y DENV–4 (Figura 2). En 2010 se analizaron un total de 2,013 muestras de suero; se identificó el serotipo responsable de la infección en 1,607 muestras. La distribución de serotipos 1, 2 y 3 se presentó en la misma proporción que en 2009. La identificación de DENV–4 se incrementó, detectándose en 9 muestras (Figura 3).


La distribución de serotipos por entidad federativa en 2009 demostró que los estados de Jalisco, que tuvo un grupo significativo, Michoacán, Colima y San Luis Potosí presentaron un mayor número de identificaciones para DENV–1. El estado en el que no se logró identificar el serotipo circulante fue Guanajuato. El estado en el que se logró identificar la circulación de tres serotipos fue Chiapas, con DENV–1, DENV–2 y DENV–4. En Jalisco se identificaron DENV–1 y DENV–3. En 15 estados se identificó DENV–1 y DENV–2 y en 10 estados únicamente se identificó la circulación de DENV–1 (Cuadro I). El estado de Colima presentó la incidencia más alta: 727.38 por cada 100 000 habitantes (Figura 4).

La distribución por entidad federativa en 2010 demostró que los estados de Guerrero, Morelos, Yucatán y Nuevo León presentaron un mayor número de detecciones para DENV–1. En Querétaro, Zacatecas, Guanajuato y Durango no se logró identificar el o los serotipos circulantes. Chiapas sigue siendo el estado en el que se identificaron tres serotipos circulantes, los mismos que en 2009. Se logró identificar también la circulación de DENV–3 en Guerrero y, en San Luis Potosí, se identificaron DENV–1 y DENV–4. La circulación de DENV–1 y DENV–2 se identificó en 14 estados y en nueve estados únicamente se identificó DENV–1 (Cuadro II). Los estados de Colima y Guerrero presentaron la incidencia más alta observada (Figura 5).

DISCUSIÓN
El dengue es una prioridad de salud pública en México; en 2009 se reportaron al sistema de vigilancia epidemiológica de dengue casi un cuarto de millón de casos probables y en 2010 se tuvieron 127,840 casos probables: una disminución con respecto al año anterior (Figura 6). En los 2 años analizados se observa que el aumento de los casos es a partir de la semana epidemiológica 31 y disminuyendo en la semana 45. Los casos probables cumplen con la sintomatología compatible para dengue pero no se les tomó una muestra para la confirmación en el laboratorio debido a que está establecido, en los lineamientos de vigilancia epidemiológica, el porcentaje de toma de muestras en zonas con alta o baja transmisión. En los estados con presencia de brotes está establecida la toma a tres de cada diez casos probables; en ausencia de brotes o de baja transmisión está establecido el 100% en la toma de muestras.15
Con base en el muestreo de los casos probables la RLESP, en 2009 y 2010 confirmó, con pruebas de laboratorio, un total de 55,961 y 28,688 casos, respectivamente. En 2009, utilizando el aislamiento viral durante el primer semestre del año y la RT–PCR en tiempo real a partir del segundo semestre, se logró 46.46% de identificación de serotipos circulantes. En 2010 se logró identificar los serotipos en 79.88% de las muestras analizadas (positivas a antígeno viral NS1), porcentaje que es similar al obtenido en el Center For Disease Control and Prevention – Dengue Branch de San Juan, Puerto Rico (comunicación personal del Dr. Jorge Muñoz Jordán). Este aumento representa 33% más identificación de serotipos circulantes que en 2009. Esto se puede explicar debido a que la identificación del serotipo por RT–PCR en tiempo real es más sensible que mediante el aislamiento viral.16 Además, para identificar el serotipo por métodos moleculares y por aislamiento viral es necesario contar con una muestra que haya sido manejada y almacenada adecuadamente a 4°C. El éxito del aislamiento viral depende en mayor grado de la red fría para conservar la integridad de las proteínas de superficie, que son necesarias para el reconocimiento y unión al receptor de las células de la línea C6/36. En ensayos de comparación entre aislamiento viral y RT–PCR en tiempo real, realizados en el laboratorio de Arbovirus del InDRE, se han identificado serotipos por aislamiento viral en 80% de las muestras ya tipificadas por RT–PCR en tiempo real (datos no publicados). Esto posiblemente se debió a que se analizaron muestras con un rango de toma de 5 días, seleccionadas en campo por la prueba de confirmación de detección del antígeno NS1. La importancia de la toma de la muestra durante los primeros 3 días después de iniciada la fiebre es que permite que se detecte una mayor carga viral, promoviendo la detección de serotipos por qRT–PCR. La toma de la muestra en este periodo favorece también el aislamiento viral, ya que se sabe que la elevación de la concentración de los anticuerpos IgM o IgG después del cuarto día de iniciada la fiebre puede disminuir la carga viral. Es importante que en los laboratorios estatales la toma, el manejo y el envío de las muestras en fase aguda se realice conservando la red fría hasta la llegada al laboratorio de referencia, lo cual ha mejorado con las nuevas guías para el diagnóstico del dengue, el sistema de calidad y las referencias vigentes desde 2008.12 El nuevo algoritmo de diagnóstico brinda resultados oportunamente, en menos de 24 horas, haciendo que la detección de antígeno viral NS1 y la RT–PCR en tiempo real sean de gran ayuda en la vigilancia virológica, en comparación con el aislamiento viral que requiere 10 días para ofrecer el resultado. Debido a esto, actualmente se está implementando la técnica de qRT–PCR en tiempo real en la RLESP, lo que reforzará la vigilancia virológica en cada estado. El aislamiento viral será utilizado únicamente para la propagación de virus autóctonos que formen parte del cepario del InDRE, lo que permitirá realizar la genotipificación y otros análisis virales.
La identificación de la circulación de más de un serotipo en al menos 14 estados representa un signo de alarma ya que se puede favorecer el incremento de las formas clínicas graves del dengue debido a la presencia de reinfecciones por serotipos diferentes, promovida por el fenómeno de aumento de la infección dependiente de anticuerpos.17 Los serotipos DENV–2 y DENV–3 están más relacionados con la presencia de cuadros clínicos graves. En Tailandia se ha observado este fenómeno desde 1970 y la población infantil es la más afectada.18,19 La presencia de estos serotipos en México puede tener el mismo efecto observado en el Pacifico asiático.
La circulación de DENV–4 en Honduras y Guatemala durante el 200820 y la migración de personas pueden ser la causa de que este serotipo se reintroduzca a México por el sureste del país, en muy baja frecuencia, y de que se haya detectado en el centro del país en 2010.
El aumento de casos observados a partir de la semana epidemiológica 31 está relacionado con el aumento en la temperatura y con la cantidad de lluvia que durante esa época se observa. Es necesario que la vigilancia entomológica sea oportuna para identificar las zonas de mayor riesgo y para tomar las medidas necesarias para el control del vector, promoviendo la prevención antes que la atención médica de los casos, que siempre resulta más costosa.
Es necesario y fundamental que la RLESP cuente con los reactivos e insumos necesarios para realizar el diagnóstico oportunamente y que se permita la aplicación de una vigilancia epidemiológica y virológica inteligente, capaz de brindar información para un mejor entendimiento de la enfermedad y para promover las acciones para su control y prevención.
Agradecimientos
A la Red Nacional de Laboratorios de Salud Pública de México y a la Red Nacional de Epidemiólogos por la detección, notificación y diagnóstico de los casos de dengue. A la doctora Elizabeth Hunsperger y a Manuela Beltrán, de la División de Diagnóstico Serológico e Investigación de Patogénesis Viral, por la donación de anticuerpos monoclonales. Al doctor Jorge Muñoz Jordán, de la División de Diagnóstico e Investigación Molecular, por la donación de células de la línea C6/36 y por la capacitación en qRT–PCR del CDC–Dengue Branch de San Juan, Puerto Rico.
REFERENCIAS
1. Guha–Sapir D, Shimmer B. Dengue fever: new paradigms for a changing epidemiology. Emerg Themes Epidemiol 2005;2:1. [ Links ]
2. SecretarÃa de Salud. Panorama epidemiológico de fiebre y fiebre hemorrágica por dengue en entidades federativas 2009–2010. Disponible en: http://www.dgepi.salud.gob.mx/denguepano/PANORAMAS_2010/PANORAMA%20DENGUE_SEMANA%2007_2010.pdf [ Links ]
3. World Health Organization. Dengue hemorrhagic fever: diagnosis, treatment, prevention and control. Geneva: World Health Organization; 1997. [ Links ]
4. Vázquez–Pichardo M, Rosales–Jiménez C, Rojas–Espinosa O, López–Martínez I, Moreno–Altamirano MMB. Is liver damage dependent on the serotype of dengue virus? – A study in Mexico. Dengue Bulletin WHO 2006;30:114–120. [ Links ]
5. Henchal EA, Putnak JR. The dengue viruses. Clin Microbiol Rev 1990;3:376–396. [ Links ]
6. Weaver S, Barrett A. Transmission cycles, host range, evolution and emergence of arboviral disease. Nat Rev Microbiol 2004;2:789–801. [ Links ]
7. Iniciativa para una Vacuna Pediátrica contra el Dengue (PDVI). Diagnóstico del dengue: recomendaciones de los Consejos de Asia–Pacífico y las Américas para la prevención del Dengue. International Vaccine Institute; 2009. Disponible en: http://www.pdvi.org/email_news/pdf/Recomendaciones_Diagnostico_Del_Dengue_en_Espanol.pdf. [ Links ]
8. Kao CL, King CC, Chao DY, Wu HL, Chang GJ. Laboratory diagnosis of dengue virus infection: current and future perspectives in clinical diagnosis and public health. J Microbiol Immunol Infect 2005; 38:5–16. [ Links ]
9. Bessoff K, Delorey M, Sun W, Hunsperger E. Comparison of two commercially available dengue virus (DENV) NS1 capture enzyme–linked immunosorbent assays using a single clinical sample for diagnosis of acute DENV infection. Clin Vaccine Immunol 2008;15:1513–1518. [ Links ]
10. Hunsperger EA, Yoksan S, Buchy P, Nguyen VC, Sekaran SD, Enria DA, et al. Evaluation of commercially available anti–dengue virus immunoglobulin M tests. Emerg Infect Dis 2009;15:436–440. [ Links ]
11. Innis BL, Nisalak A, Nimmannitya S, Kusalerdchariya S, Chongswasdi V, Suntayakorn S, et al. An enzyme–linked immunosorbent assay to characterize dengue infections where dengue and Japanese encephalitis co–circulate. Am J Trop Med Hyg 1989;40:418–427. [ Links ]
12. Instituto de Diagnóstico y Referencia Epidemiológica. Procedimiento para la aplicación del nuevo algoritmo para diagnóstico por laboratorio de fiebre por dengue y fiebre hemorrágica por dengue. México: InDRE, RNLSP; 2008. Disponible en: http://www.dgepi.salud.gob.mx/denguepano/Procedimientos.pdf. [ Links ]
13. Chien LJ, Liao TL, Shu PY, Huang JH, Gubler DJ, Chang GJ. Development of real time reverse transcriptase PCR assays to detect and serotype dengue viruses. J Clin Microbiol 2006; 44:1295–1304. [ Links ]
14. Gubler DJ, Kuno G, Sather GE, Velez M, Oliver A. Mosquito cell cultures and specific monoclonal antibodies in surveillance for dengue viruses. Am J Trop Med Hyg 1984;33:158–165. [ Links ]
15. Secretaría de Salud. Lineamientos para la vigilancia epidemiológica de fiebre por dengue y fiebre hemorrágica por dengue. México: Secretaria de Salud; 2008. Disponible en: http://www.dgepi.salud.gob.mx/denguepano/Lineamientos.pdf. [ Links ]
16. Callahan JD, Wu SJ, Dion–Shultz A, Mangold BE, Peruski LF, Watts DM, et al. Development and evaluation of serotype– and group–specific fluorogenic reverse transcriptase PCR (TaqMan) assays for dengue virus. J Clin Microbiol 2001;39:4119–4124. [ Links ]
17. Halstead SB, Udomsakdi S, Simasthien P, Singharaj P, Sukhavachana P, Nisalak A. Observations related to pathogenesis of dengue hemorrhagic fever. Experience with classification of dengue viruses. Yale J BioI Med 1970;42:261–275. [ Links ]
18. Halstead SB, Nimmannitya S, Yamarat C, Russell PK. Hemorrhagic fever in Thailand: newer knowledge regarding etiology. Jap J Med Sci Bioi 1967:20(suppl):96–102. [ Links ]
19. Potts JA, Gibbons RV, Rothman AL, Srikiatkhachorn A, Thomas SJ, Supradish PO, et al. Prediction of dengue disease severity among pediatric Thai patients using early clinical laboratory indicators. PLoS Negl Trop Dis 2010;4:e769. [ Links ]
20. Panamerican World Health Organization. 2007: number of reported cases of dengue and dengue hemorrhagic fever, region of the Americas (by country and subregion). Epidemiological Week 52. Disponible en: http://www.paho.org/english/ad/dpc/cd/dengue–cases–2007.htm. [ Links ]














