Introducción
La insuficiencia cardiaca (IC) es una patología en la cual el corazón es incapaz de bombear suficiente sangre oxigenada al cuerpo, interfiriendo con el normal funcionamiento sistémico. Esta afecta a más de 23 millones personas en todo el mundo, cifra que parece ir en aumento cada año y que se ve impulsada por factores como el envejecimiento de la población, la presencia de cardiopatías isquémicas, hipertensión, diabetes, enfermedades reumáticas, valvulares, cardiopulmonares y otras cardiomiopatías1,2. Tiene un impacto tan grande en la salud pública que es considerada la primera causa de muerte en países desarrollados como Estados Unidos3. Sin embargo, no solo países industrializados sufren de esta patología; en países africanos, como Nigeria, también es un grave problema y las personas que la padecen son más jóvenes (laboralmente activos) que las de países desarrollados4. Por su parte, en países en vía de desarrollo como los latinoamericanos, las patologías cardiovasculares como la IC tampoco dan tregua, siendo la primera causa de muerte, debido a cambios demográficos y a hábitos de vida poco saludables5.
Además, aunque esta es una enfermedad que afecta principalmente a adultos, lamentablemente también se presentan múltiples casos pediátricos, deteriorando la calidad de vida de los niños que la padecen por los problemas asociados a esta y a su tratamiento, tales como largos periodos de hospitalización y las dificultades físicas que conlleva esta patología6.
Teniendo en cuenta la alta carga que implica esta enfermedad mundialmente, es importante considerar nuevas estrategias para manejar esta patología que parece no discriminar, deteriorando la salud de las personas globalmente y que, debido a los distintos cambios demográficos y la adopción de estilos de vida menos saludables, amenaza con aumentar su incidencia.
Durante la aparición y la progresión de esta enfermedad se propicia la activación de una serie de mecanismos compensatorios que en una primera etapa pueden ser favorables pero terminan siendo contraproducentes para la función cardiaca, incluyendo una remodelación morfológica, estructural y funcional de los tejidos cardiacos, dada por procesos de hipertrofia (aumento en la masa ventricular, debido al incremento en el volumen celular), pérdida de células musculares cardiacas (cardiomiocitos) y reemplazo de estas por material fibroso7. Igualmente los problemas cardiacos y el envejecimiento pueden llevar a una remodelación subcelular que causa deficiencias en el manejo del calcio, por defectos en organelas como el retículo sarcoplásmico y las distintas proteínas que lo conforman, desencadenando problemas en la función de bombeo del corazón al alterar la capacidad contráctil de este8.
Estas modificaciones tanto a nivel tisular como subcelular llevan a múltiples cambios, que incluyen la alteración de niveles de expresión y liberación de múltiples moléculas. Esto incluye una alteración en los perfiles de micro RNA (miRNA), los cuales han despertado el interés científico en las áreas de la cardiología y la biomedicina en general por su potencial en la práctica clínica. Por esto en el presente artículo se busca mostrar los avances en la investigación sobre el uso de miRNA en el manejo de la IC, exponiendo algunas de sus características biológicas, metodologías usadas en su estudio, así como investigaciones recientes en su uso como biomarcadores y prospectivas como herramientas terapéuticas.
Introducción a la biología de los miRNA y sus métodos de estudio
Existen diversos mecanismos epigenéticos mediante los cuales se logra inhibir o sobreexpresar diferentes genes, al intervenir en los diferentes estadios del proceso de expresión génica sin alterar la secuencia del ADN. Entre los mecanismos de silenciamiento genético a nivel transcripcional encontramos procesos que llevan a la condensación de la cromatina mediante la modificación de histonas, tales como la trimetilación de la histona tres en la lisina 9 o 27, así como la metilación de las islas CpG en la región promotora del ADN, la cual impide la unión de proteínas involucradas en la trascripción y promueve la unión de complejos de remodelación de la cromatina (que pueden llevar a la modificación de histonas)9. También se puede dar una regulación a nivel postranscripcional, en la que intervienen especialmente los miRNA, pequeños RNA de aproximadamente entre 21-25 nucleótidos) que, mediante la asociación con otras proteínas, pueden regular la expresión genética al unirse a la región 3’ UTR del mRNA diana, inhibiendo su traducción a proteína.
Estos miRNA fueron descubiertos en 1993 en el nematodo Caenorhabditis elegans por Lee et al.10, quienes describieron la regulación de la expresión del gen LIN-4 por parte de un RNA no codificante, mediante una interacción RNA-RNA en la región 3’ UTR del mRNA de este gen. Este descubrimiento cambió en gran medida nuestro conocimiento de la regulación genética, llevando a múltiples investigaciones en este campo y a la descripción de miles de miRNA nuevos y procesos celulares en los que intervienen, al punto que actualmente en la base de datos «miRBase» versión 21 se tiene un registro de 1,881 diferentes miRNA en el genoma humano, número que, como indican Londin et al.11 (2015), no representa ni la mitad de todos los existentes, pues en su estudio reportaron 3,707 nuevos miRNA humanos, número que se espera aumente a medida que se llevan a cabo más estudios en diferentes tejidos.
La biogénesis del miRNA comienza con su transcripción por la RNA polimerasa II o en algunos casos III, formando un pri-miRNA que luego es procesado por la RNAsa Drosha, dando lugar a un pre-miRNA de 60-70 nucleótidos12. Posteriormente es llevado al citoplasma con la ayuda de las proteínas Exportin5 y Ran, donde es procesado por la RNAasa Dicer formando un miRNA duplex intermediario de entre 21-25 nucleótidos13. Por último la proteína argonauta II (Argo2) incorpora el miRNA maduro de cadena sencilla para formar el complejo Ago-RNA12, activando de este modo el RNA induced silencing complex (RISC), en el cual el miRNA guía a la proteína Argo2 al mRNA objetivo por un emparejamiento inicial de 2 a 5 nucleótidos y un posterior reconocimiento más avanzado14, lo que lleva a la inhibición de la traducción de este mRNA a proteína y, por tanto, al silenciamiento o reducción de la expresión genética.
Los miRNA pueden ser secretados o liberados (por rompimiento y daño celular) desde su célula de origen para ser transportados en fluidos corporales de forma estable, como un mecanismo de comunicación intercelular15; el cual parece ser un proceso activo y selectivo, pero del que se desconocen muchos aspectos y se requieren más estudios para dilucidarlo16. Este transporte estable se da gracias a que los miRNA son encapsulados o anclados a microvesículas, exosomas, lipoproteínas de alta densidad o la misma proteína Argo2, que los llevan a sus respectivas células receptoras, previniendo su degradación17-20.
Este transporte vía plasmática permite tomar muestras de forma poco invasiva que posteriormente, mediante técnicas de laboratorio y distintos kits comerciales, permiten aislar el miRNA de interés. Además, debido al potencial de estas moléculas se han desarrollado nuevos protocolos a fin de facilitar su obtención y purificación con fines médicos, con la visión de que se conviertan en parte de los test clínicos de rutina21-23.
Después del aislamiento, es posible evaluar la expresión de los miRNA, mediante técnicas como la reacción en cadena de la polimerasa (PCR) cuantitativa con transcripción inversa (qRT-PCR) (la cual consiste en amplificar y cuantificar de forma simultáneamente el ADN complementario obtenido por retrotranscripción del miRNA de interés), en la cual se han desarrollado nuevos protocolos usando primers stem-loop RT (primers con una estructura tallo-bucle que son más estables y facilitan la amplificación de secuencias de nucleótidos cortas) y Oligo(dT) (usados en secuencias con cola de poly adenina), que buscan obtener una alta sensibilidad y especificidad en la cuantificación, evaluar múltiples miRNA en paralelo y aumentar la eficiencia de su análisis24-26. Igualmente otras tecnologías son normalmente usadas en el estudio y cuantificación de miRNA, entre las que resaltan los microarrays, que permiten evaluar la expresión de múltiples miRNA de forma paralela al cuantificar por niveles de fluorescencia la hibridación entre la sonda y el miRNA de interés27. Igualmente, la secuenciación de nueva generación ha cobrado gran importancia en la identificación de miRNA y en la medición de sus niveles de expresión (al cuantificar el número de lecturas durante la secuenciación como medida del nivel de expresión), dada la alta sensibilidad, precisión y eficiencia que llega a tener27.
Posteriormente, mediante diferentes softwares —como NCodeTM, miRDeep2, miRExpress y miRNAKey, entre otros— se obtiene la información sobre el nivel de estos miRNA en medio electrónico28, lo que permite establecer diferencias en los perfiles de expresión entre diferentes grupos (ejemplo pacientes con IC frente a controles sin la enfermedad), a fin de establecer posibles biomarcadores y dianas terapéuticas para distintas enfermedades, incluyendo la IC.
Además de la cuantificación de miRNA, el establecimiento de sus genes diana y la manipulación de sus niveles son aspectos importantes en la investigación biomédica, a fin de tener un mayor entendimiento de las funciones que estos cumplen, las implicaciones de su desregulación y ser usados como una herramienta terapéutica. Los miRNA pueden afectar la expresión de múltiples genes de manera directa (por interacción con la región 3’ UTR del mRNA) e indirecta (por ejemplo, al inhibir un factor de transcripción que afecta a varios genes); algunas técnicas, como la evaluación de niveles de proteína o mRNA, pueden mostrar una relación entre los niveles de miRNA y la expresión génica, pero no discriminan entre una interacción directa o indirecta29. Para establecer que un gen es diana directa de un determinado miRNA, el proceso más usado es una predicción computarizada mediante la evaluación de características como complementariedad de bases, conservación entre especies, energía libre y accesibilidad al sitio de unión, las cuales pueden ser evaluadas mediante diferentes herramientas electrónicas, como miRanda, miRanda-mirSVR, TargetScan y MirTarget2, entre otras30. Esta predicción es luego validada mediante métodos experimentales, siendo el ensayo de lucíferas el más usado, pues mediante la inserción de la región 3′ UTR del gen diana en un vector con el gen para la luciferasa y transfectarlo a determinadas células junto al miRNA de interés, permite establecer si hay una inhibición directa, ya que si el gen es objetivo directo del miRNA la proteína de la luciferasa no será sintetizada y, por ende no se producirá luminiscencia (o se verá reducida)29. También existen otras herramientas menos usadas en la validación de objetivos de miRNA, como las basadas en la imunoprecipitación de los componentes del complejo RISC y posterior análisis del mRNA ligado a este; al igual que la amplificación de los extremos de ADNc mediado por RNA ligasa (RLM-RACE) o su metodología modificada PARE, que se basa en la amplificación e identificación de los productos del clivaje del mRNA por parte de un determinado miRNA31.
En cuanto a la modulación de los niveles de miRNA, las estrategias más utilizadas son el uso de miRNA sintéticos de doble cadena, con modificaciones químicas que favorecen su estabilidad y la captación celular para fines de sobreexpresión, así como el uso de antagomiRs, consistentes en oligonucleótidos (secuencias cortas de nucleótidos) modificados que se unen al miRNA específico inhibiéndolo32. Estos miRNA sintéticos e inhibidores posteriormente son transportados a las células usando diferentes vectores, especialmente de tipo vírico o liposómico32, a fin de manipular la expresión genética y las características celulares de interés.
Gracias a las características biológicas de los miRNA, así como los avances en técnicas y protocolos, se han propuesto nuevas estrategias para abordar enfermedades como la IC, pudiéndose asociar los niveles de miRNA con características patológicas de la enfermedad y manipular su expresión para revertirlas (Fig. 1).

Figura 1 Los miRNA se procesan para formar un miRNA maduro que puede inhibir la expresión génica. Los miRNA se secretan o liberan en fluidos corporales como la sangre, donde son transportados en microvesículas (MV), exosomas (EXS), lipoproteínas de alta densidad (LPD) y la proteína Ago2. En el laboratorio podemos utilizar muestras de tejidos sólidos o plasma para aislar, amplificar, cuantificar y secuenciar miRNA específicos con el fin de establecer posibles biomarcadores y dianas terapéuticas. La información obtenida en medios electrónicos de los miRNA puede ser usada para predecir los posibles genes diana (que luego pueden ser validados por el ensayo de luciferasa) y la síntesis de miRNA o antagomiRs.
Uso de miRNA como biomarcadores de la insuficiencia cardiaca
¿Por qué usar miRNA como biomarcadores?
Dada la alta afectación mundial de la IC, se ha incrementado el número de artículos publicados sobre el uso de biomarcadores, en los cuales se demuestra la viabilidad y la importancia de estos como herramientas para el diagnóstico, estratificación de riesgo, biomonitoreo y guía en el manejo de la enfermedad33.
Actualmente los biomarcadores más usados son los basados en la detección y cuantificación de proteínas; sin embargo, estos presentan algunos problemas, como la complejidad de la composición proteica en la sangre, la diversidad de modificaciones postranscripcionales, la relativa poca abundancia que pueden tener algunas proteínas de interés, al igual que el alto esfuerzo y tiempo necesarios para el desarrollo de agentes de alta afinidad para su detección (por ejemplo, el desarrollo de anticuerpos)34,35. Estos inconvenientes han dificultado el descubrimiento y la implementación de nuevos marcadores basados en proteínas y han puesto en evidencia la necesidad de investigar el uso de biomarcadores basados en otros tipos de moléculas35.
Entre las novedosas opciones para el diagnóstico y pronóstico de enfermedades humanas se ha planteado el perfil de niveles de miRNA, pues presentan características de un buen biomarcador, como estabilidad en fluidos corporales, secuencia conservada entre múltiples especies y expresión de algunos miRNA específica del tejido o condición biológica35. Además también tienen algunas ventajas frente a los marcadores basados en proteínas, tales como una medición rápida y precisa de sus niveles, prácticamente no se requiere el desarrollo de nuevos agentes de detección gracias al desarrollo de técnicas robustas basadas en PCR y secuenciación de última generación, y que gracias a las técnicas de amplificación por PCR es posible realizar análisis de miRNA en volúmenes pequeños de fluidos corporales34.
Perfil de miRNA en el diagnóstico de la insuficiencia cardiaca
Como se mencionó previamente, entre las ventajas de los miRNA figuran su estabilidad en fluidos corporales, su asociación con eventos patológicos y en algunos casos la alta especificad tisular, por lo que diferentes investigaciones han buscado establecer miRNA que sean liberados a fluidos corporales por el tejido cardiaco en respuesta al daño o pérdida celular. Esto ha llevado a la identificación de miRNA como miR-208b, miR-499 y miR-133a, que son liberados casi exclusivamente en condiciones de daño cardiaco, mostrando que el estudio de perfiles de miRNA puede ser útil en la identificación de biomarcadores séricos que reflejen el estado de los órganos y tejidos en enfermedades que afecten la función cardiaca36-38.
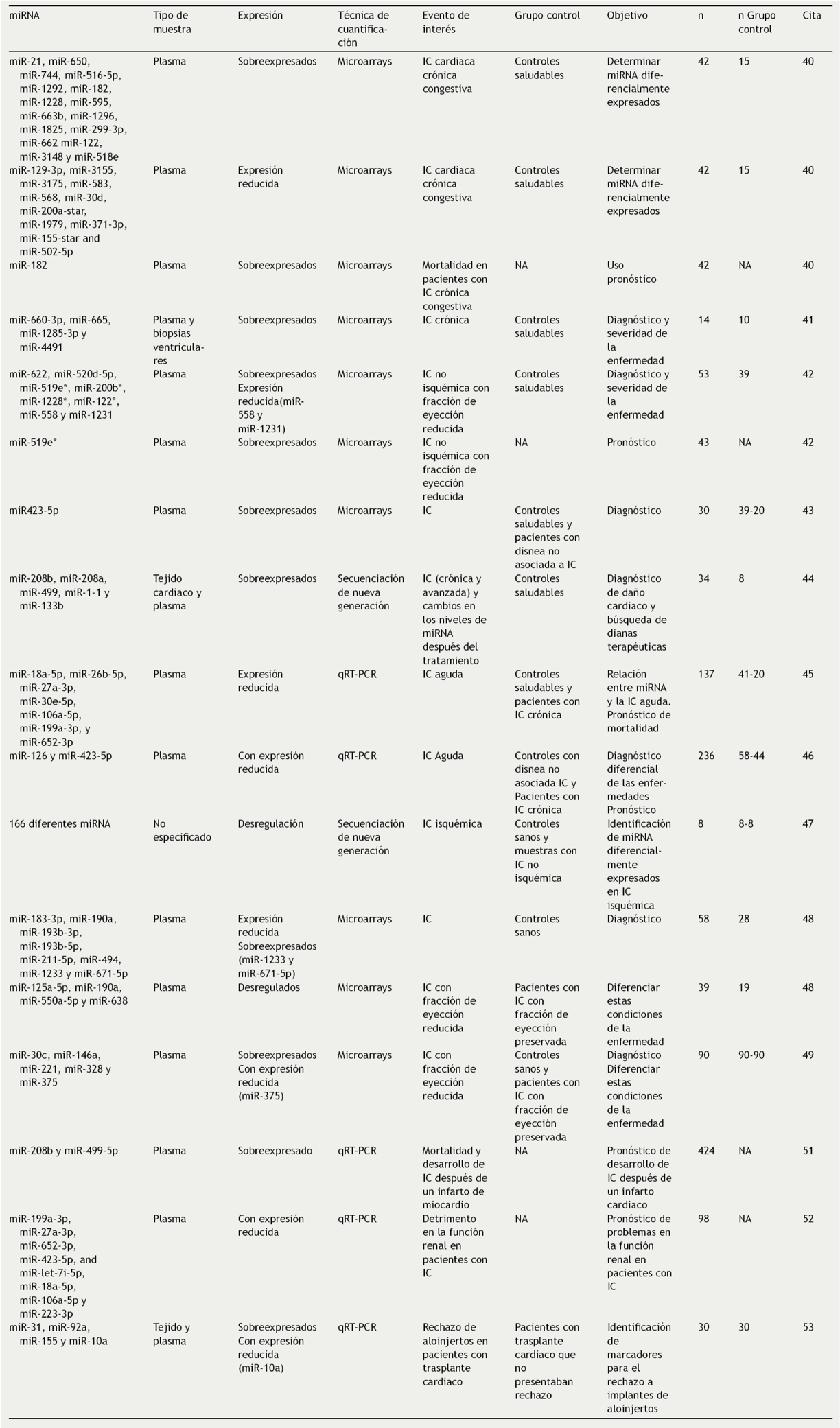
Esta afectación o modificación de tejidos cardiacos, especialmente los asociados a IC, como apoptosis, hipertrofia y fibrosis, están relacionados con alteraciones en la expresión de miRNA en el miocardio, los cuales son potenciales marcadores séricos de interés en el diagnóstico de la enfermedad, especialmente en etapas tempranas39. En miras de lograr esto, estudios como los de Cakmak et al.40 (2015) muestran el alto valor diagnóstico de los miRNA circulantes (extraídos de fluidos corporales), ya que encontraron 17 miRNA sobreexpresados y 11 con expresión reducida en pacientes con IC frente a controles. Igualmente, Vogel et al.41 (2015) y Li et al.42 (2016) encontraron que los perfiles de múltiples miRNA circulantes pueden ser biomarcadores útiles para la IC sistólica y crónica, ya que se relacionan con la presencia y la severidad de la enfermedad (evaluadas por la disminución de la fracción de eyección ventricular), mostrando una importante capacidad en el diagnóstico patológico y evaluación funcional cardiaca.
Adicionalmente Vogel et al.41 reportaron que miR-622, miR-520d-5p, miR-519e*, miR-558 y miR-200b* tienen una capacidad diagnóstica igual e incluso algo superior al marcador proteico propéptido natriurético cerebral N-terminal (NT-proBNP), mientras que Tijsen et al. (2010)43 y Seronde et al.44 (2015) mostraron que la capacidad de estas moléculas no solo se limita a la distinción de casos de IC frente a controles sanos, sino que miR-423-5p y miR-126 pueden distinguir estos de casos de disnea no relacionados a IC, indicando un buen desempeño discriminatorio por parte de algunos miRNA comparado con otro tipo de biomarcadores ya establecidos y ventajas en el uso clínico.
Estos estudios son un ejemplo del alto número de miRNA que se pueden encontrar desregulados en casos de IC, los cuales tienen potencial como herramienta diagnóstica a fin de identificar casos de IC en etapas tempranas y mejorar su manejo clínico.
Uso de miRNA en el manejo de subtipos de la enfermedad
Se estudia el uso de miRNA para diferenciar condiciones específicas, estadios o subtipos de la IC, a fin dar una mejor guía en el abordaje de la misma. En este campo. Akat et al.45 (2014) encontraron diferencias en los niveles de algunos miRNA circulantes entre pacientes con IC vs controles sin la enfermedad y entre pacientes con IC estable vs IC avanzada; incluso evidenciando que los niveles de miRNA de interés cambian después del tratamiento con dispositivo de asistencia ventricular. Por su parte, Ovchinnikova et al.46 (2016) encontraron que 7 miRNA presentaban niveles de expresión significativamente reducidos en pacientes con IC aguda frente a IC crónica y controles sanos, mientras que Seronde et al.44 (2015) reportan que los niveles de miR-126 y miR-423-5p son significativamente mayores en casos de IC aguda que en pacientes con IC avanzada. Estos estudios evidencian que los miRNA pueden ser una herramienta útil, ya que se relacionan con el daño cardiaco y progresión de la enfermedad, permitiendo de forma poco invasiva un seguimiento de los pacientes e identificación de casos avanzados de la misma.
En cuanto a subtipos de la enfermedad, Li et al.47 (2016) encontraron numerosas diferencias en los patrones de expresión de miRNA entre casos de IC isquémica frente a IC no isquémica y recalcan la importancia de estos en la patogénesis de ambas condiciones, aumentando la compresión de las similitudes y diferencias a nivel molecular entre estos subtipos y mostrando que la evaluación de miRNA puede ser de utilidad en la distinción de estas patologías. Otros estudios han identificado diferencias en perfiles de miRNA entre muestras de IC con fracción de eyección reducida vs fracción de eyección preservada, llegando incluso a tener mayor poder discriminatorio que otros marcadores típicos para la enfermedad, como el péptido cerebral natriurético (BNP) y el NT-proBNP, aunque lo que más se recomienda es el uso en conjunto de estos tipos de biomarcadores48,49. Ello que sugiere que estos pueden ser útiles tanto en el diagnóstico de IC como en la estratificación entre estas dos formas de la enfermedad, aspecto importante en el manejo de los pacientes, pues se están implementando nuevas estrategias y regímenes terapéuticos específicos para cada subtipo48.
Uso de miRNA en el pronóstico y la estratificación de riesgos
Otro aspecto importante es la utilidad de los miRNA en la estratificación de riesgo, tanto de desarrollar la enfermedad como de presentar complicaciones derivadas de esta, ya que podría mejorar el abordaje terapéutico y de cuidados tanto hospitalarios como posthospitalarios. En estos aspectos se ha señalado que una baja expresión de múltiples miRNA circulantes, como miR-423-5p, miR-18a-5p y miR-652-3p, es un indicador de un peor pronóstico a largo plazo en pacientes con IC aguda, relacionándose con un mayor riesgo de rehospitalización y mortalidad44,46. Incluso se ha reportado que miR-182 pueden llegar a tener mayor valor en el pronóstico de mortalidad que otros biomarcadores ya establecidos para la enfermedad, como el NT-proBNP y la proteína C reactiva40, lo que propone que la evaluación de miRNA tiene potencial en la identificación temprana de pacientes con un mal pronóstico, buscando mejorar el cuidado y seguimiento después de darles de alta, así como como ofrecer mejores indicaciones a estos y a sus familiares.
También se ha encontrado un uso potencial de los miRNA para evaluar el riesgo de desarrollo de IC isquémica después de un infarto agudo al miocardio, basado en resultados de estudios como el de Matsumoto et al.50 (2013), donde se encontró que los niveles de miR-192, miR-194 y miR-34a se ven elevados en pacientes que desarrollan IC después del infarto en comparación con los que no, así como lo descrito por Gidlöf et al.51 (2013), quienes encontraron que un incremento en los niveles de miR-499-5p y miR-208b se relacionaba con un incremento en el riesgo de muerte y desarrollo de IC después del infarto de miocardio, resultados que indican que el análisis de miRNA ofrece una opción de interés clínico que permitiría dar un mejor abordaje y direccionamiento del tratamiento a los pacientes que después de un ataque cardiaco presentan un alto riesgo de desarrollar IC.
Igualmente se estudia el uso de miRNA para establecer el riesgo de problemas en otros órganos asociados a esta enfermedad, como la disminución de la función renal, encontrándose que la reducción en niveles de algunos miRNA, especialmente miR-199a-3p, puede ser un útil marcador en la predicción de estos eventos, puesto que se asocia con un detrimento temprano en la función renal52, indicando que podría usarse como biomarcador de la progresión de la enfermedad y mejorar el manejo no solo a nivel cardiaco sino del daño a otros sistemas.
Uso de miRNA en la evaluación terapéutica
Otro enfoque en el que se estudia el uso de miRNA es como marcadores en la eficacia de tratamientos contra la IC, pues como lo evidencia un estudio reciente en modelos murinos algunos miRNA, como miR-146a y miR-342-3p, podrían ser usados como marcadores para evaluar la eficacia del tratamiento con el medicamento azilsartan medoxomil (un antagonista de los receptores de angiotensina II), observándose que una restauración de los niveles de estos miRNA inducida por este medicamento se relaciona con una mayor tasa de supervivencia y menor congestión cardiaca53.
Similarmente se ha propuesto el uso de miRNA para evaluar el riesgo de rechazo al trasplante de aloinjertos cardiacos, pues se ha encontrado que la medición de niveles de miR-10a, miR-31, miR-92a y miR-155 en plasma permite distinguir con alta precisión entre pacientes que presentan rechazo y los que no, lo cual es muy importante clínicamente, puesto que el trasplante de corazón es en muchas ocasiones la única oportunidad de supervivencia en etapas avanzadas de la IC54.
Los resultados de estos diferentes estudios muestran que múltiples miRNA tienen potencial como biomarcadores en el diagnóstico temprano, distinción de condiciones o subtipos de la enfermedad, pronóstico, estratificación de riesgo, monitoreo y evaluación de tratamientos (Tabla 1), por lo que se espera que en un futuro puedan ser usados en la práctica clínica rutinaria y entrar en los lineamientos de guías clínicas para el manejo de la IC. Sin embargo una limitación de muchos de estos estudios es el bajo número de muestra, por lo que se requieren estudios donde se evalúe un mayor número de pacientes a fin de dar un soporte basado en la evidencia más firme sobre el uso de miRNA como marcadores de la enfermedad. Además es necesario realizar más análisis comparativos entre el uso de marcadores basados en miRNA, frente a marcadores de uso rutinario de base proteica, con miras a evaluar si los miRNA tienen una capacidad superior significativa o si pueden aportar ms información importante en el manejo clínico de los pacientes.
Uso de miRNA como herramienta terapéutica
El enfoque de la manipulación de la expresión genética con fines terapéuticos ha propiciado importantes avances en la investigación biomédica, mostrando un creciente interés en estrategias como la terapia génica, que mediante la transferencia de genes busca reducir las características clinicopatológicas al modificar la secuencia de nucleótidos.
Sin embargo, la expresión de genes de interés también puede ser modulada por mecanismos que no alteran la secuencia del ADN, tales como los RNA largos no codificantes (lncRNA), que pueden promover procesos como la modificación de histonas, interferencia de miRNA y control de la actividad o estabilidad de algunas proteínas. Lo mismo que la administración exógena de RNA pequeños de interferencia (siRNA), que por complementariedad perfecta inhiben un determinado gen, dando la opción de regular una vía de señalización específica. En esta categoría también encontramos a los miRNA, que son promisorios en la terapéutica, pues uno solo puede inhibir múltiples genes ya que actúan por complementariedad imperfecta, permitiendo modular múltiples vías de señalización y características de la IC.
Prevención de la pérdida de cardiomiocitos
Es notorio desde el punto de vista clínico el uso de miRNA para prevenir la muerte de células cardiacas provocada por eventos como el daño isquémico (deterioro celular por privación de oxígeno), ya sea usando este tipo de moléculas por sí solas o en conjunto con otros tratamientos (Fig. 2A y B). Lo anterior se evidencia en estudios como el de Hu et al.55 (2010), donde se encontró que al administrar miR-210 como terapia en modelos murinos con infarto de miocardio se inducía una mayor angiogénesis (formación de vasos sanguíneos a partir de vasos preexistentes), se reducía la apoptosis y se mejoraba la función cardiaca, puesto que los genes diana de miR-210, especialmente la Ephrina A3 (EFNA3) y la proteína tirosina fosfatasa (PTP1B), se ven involucrados en la inhibición de la angiogénesis y promoción de la muerte celular.

Figura 2 Abordaje terapéutico mediante sobreexpresión o represión de miRNA en la prevención y reparación del daño cardiaco. A) Prevención de la pérdida de cardiomiocitos. B) Mejora de la terapia celular. C) Promoción de la regeneración cardiaca.
Igualmente ensayos en modelos murinos han encontrado que la pérdida o reducción de la expresión de algunos miRNA puede contribuir a la descompensación cardiaca y desarrollo de IC, por lo que su modulación positiva sería de interés en el manejo de la enfermedad. Es el caso de miR-150, el cual juega un papel vital en la sobrevivencia de los cardiomiocitos durante el daño cardiaco isquémico, por su acción represora sobre genes proapoptóticos, como EGR2 y P2RX756; al igual que miR-1, -29c, -30c, -30d, -149, -486 y -499, los cuales tienen efectos represores sobre múltiples factores proapoptóticos, por lo que su reducción contribuye a la pérdida de células cardiacas57.
Además, los miRNA pueden regular otros procesos que contribuyen a la pérdida de células cardiacas, como lo muestran Luo y Zhang58 (2016), quienes encontraron que miR-423-5p media la apoptosis inducida por H2O2 (una especie de oxígeno reactivo que causa estrés oxidativo) y su represión protege a los cardiomiocitos de la muerte, siendo de importancia en la prevención del daño por episodios de isquemia/reperfusión. Estas moléculas también son importantes en la necrosis, otro proceso de muerte celular de importancia que influye en la perdía de función del corazón, campo en el cual se ha reportado que miR-223-5p/-3p pueden regular directamente a los genes TNFR1, DR6, IKK y NLRP3, suprimiendo la necrosis en células cardiacas y mejorando la función ventricular, lo que sugiere su uso potencial en el tratamiento del daño miocárdico59.
Por otro lado, el uso de miRNA puede incrementar los beneficios de la terapia celular; como se puede apreciar en el estudio de Dakhlallah et al.60 (2015), quienes reportan que al usar células madre mesenquimales transfectadas con miR-133a para tratar ratones con infarto al corazón se incrementaba la eficacia del trasplante celular, se mejoraba la función cardiaca, mientras que se evidenciaba una reducción en la fibrosis y apoptosis. Esto debido a que la sobreexpresión de miR-133 reducía la de factores proapoptóticos como Apaf-1, caspasa 9 y caspasa 3, protegiendo a las células implantadas de la muerte celular60. Otro estudio en ratones con infarto de miocardio mostró que la trasferencia de exosomas con miR-126 y miR-210 desde células endoteliales cardiacas hacia células madre progenitoras cardiacas trasplantadas incrementaba la sobrevivencia de estas al activar quinasas que promueven la supervivencia celular y promover cambios metabólicos beneficiosos61, indicando que la transferencia previa al trasplante de miR-126 y miR-210, como en el estudio anterior, podría ser beneficiosa para el tratamiento. Esta reducción en la muerte de células madre es de gran importancia, pues el principal problema de estas terapias celulares es su baja viabilidad en el trasplante60, convirtiendo el mejoramiento de este tratamiento mediante miRNA en una buena oportunidad para aumentar la eficacia de esta herramienta terapéutica.
Recuperación de cardiomiocitos
Uno de los campos en los que se ve una mayor oportunidad terapéutica con los miRNA es la regeneración cardiaca, pues el corazón de los mamíferos adultos tiene una capacidad mínima de renovación de sus células. Aun así, se ha logrado incrementar esta habilidad proliferativa en experimentos con ratones mediante la inducción exógena de algunos miRNA, como miR-590, miR-199a y el clúster miR-302-367, promoviendo la entrada de cardiomiocitos al ciclo celular y teniendo marcados efectos positivos en la reparación cardiaca62,63. Esto debido al rol de miR-590 y miR-199a en múltiples vías que controlan la proliferación celular y la estructuración muscular cardiaca, así como la represión por parte del clúster miR-302-367 sobre de la vía de señalización Hippo, la cual regula aspectos como el tamaño de los órganos y la proliferación celular62,63.
Por otra parte, miembros de la familia de miR-15 impiden la proliferación de cardiomiocitos, por lo que al inhibir estos miRNA en ratones adultos se aumenta la proliferación celular y se mejora la función sistólica ventricular derecha después de eventos de cardiopatía isquémica64. Igualmente se ha visto que miR-34a inhibe la capacidad regenerativa cardiaca al controlar la expresión de genes relacionados con la división celular y la apoptosis, como BCL2, Cíclica D1 y SIRT1, e incluso el uso de antagomiRs para este miRNA mejora la remodelación cardiaca después del infarto de miocardio65. Esto muestra como la modificación de los niveles de determinados miRNA, ya sea por inhibición o por sobreexpresión, es una estrategia promisoria para incrementar la poca capacidad regenerativa del corazón, dando nuevas esperanzas para las personas con daño cardiaco e IC (Fig. 2C).
Reducción de la hipertrofia
Otros miRNA han llamado la atención por su implicación en el proceso de hipertrofia, pues el incremento patológico de la masa ventricular tiene un importante rol en el desarrollo y la progresión de la enfermedad. A fin de proponer estrategias terapéuticas se han establecido miRNA que promueven este proceso y cuya modulación negativa sería prometedora en el tratamiento de la IC, como es el caso de miR-214 y miR-30, puesto que inhiben la expresión del factor de trascripción XBP1 y contribuyen al paso de la hipertrofia cardiaca compensatoria hacia la IC, ya que este gen es importante en la promoción de angiogénesis y en el mantenimiento de las funciones cardiacas normales en los estados tempranos de la hipertrofia66. Igualmente, en estudios con modelos murinos se ha visto que la inyección intravenosa de antagomiRs específicos para miR-132, miR-212 y miR-208a bloquea la hipertrofia cardiaca y el desarrollo de remodelación patológica, mientras que mejora la función cardiaca y aumenta la supervivencia de estos animales, mostrando potencial como una estrategia terapéutica para abordar la enfermedad67,68.
Igualmente se han establecido miRNA con función antihipertrófica y cuya sobreexpresión parece tener efectos positivos en la regulación de procesos de remodelación patológica y progresión de la IC. Es el caso de miR-185 y miR-218, pues ensayos en ratones han mostrado que estos miRNA disminuyen sus niveles al inducir procesos hipertróficos en estos modelos, mientras que su modulación positiva mediante la transfección de miRNA sintéticos a cardiomiocitos de ratones tuvo efectos positivos en la reducción de la hipertrofia, gracias a la regulación que ejerce miR-185 sobre varios genes involucrados en la vía de señalización del Ca+ y miR-218 sobre el factor de transcripción REST69,70. De manera similar se ha encontrado que la sobreexpresión de miR-99a y miR-26a en ratones con IC y cardiomiocitos cultivados reduce el fenómeno de hipertrofia, al menos en parte porque estos miRNA inhiben la traducción de proteínas que promueven este proceso, como la diana de rapamicina (mTOR), el factor de transcripción GATA4 y el factor de transcripción de silenciamiento de RE1 (REST)71,72.
Reducción de la fibrosis
Dado que el corazón humano tiene una capacidad regenerativa casi nula, la formación de una cicatriz fibrosa es el único mecanismo disponible para su reparación; sin embargo, una fibrosis mantenida afecta negativamente la función cardiaca contribuyendo al desarrollo de la IC7. Uno de los factores que promueve este proceso maladaptativo es la angiotensina II (Ang2), por lo cual se ha estudiado el papel de los miRNA en este proceso, encontrándose que fibroblastos cultivados con Ang2 presentan mayores niveles de miR-503 y menores de miR-29, lo que lleva a una mayor proliferación de estas células, al incremento en la producción de colágeno y a la sobrexpresión del factor de crecimiento transformante beta (TGF-β)73,74. Incluso se observó que al restaurar los niveles de miR-29b en cultivos de fibroblastos y modelos murinos expuestos a Ang2 se bloqueaban la fibrosis y la disfunción cardiaca74, indicando que la manipulación exógena de estas moléculas puede tener efectos favorables en este componente del remodelación patológica, previniendo una respuesta maladaptativa y progresión de la IC (Fig. 3).

Figura 3 Uso terapéutico de los miRNA para prevenir procesos de remodelación patológica como la fibrosis e hipertrofia, a fin de impedir y tratar la progresión de la insuficiencia cardiaca.
Similarmente se ha encontrado que la sobreexpresión de miR-328 y miR-208a incrementa significativamente la fibrosis miocárdica, mientras que su represión por medio de antagomiRs o medicamentos reducía este fenómeno en modelos murinos; debido a que miR-328 reprime a TGF RIII, mientras que miR-208a incrementa la expresión de la endoglina, promoviendo así la activación de la vía profibrolítica TGF- 75,76. De forma contraria, miR-24 inhibe la vía TGF-β, por lo que su sobrexpresión mejora la función cardiaca y reduce la fibrosis en modelos murinos, mientras que en experimentos in vitro reduce la diferenciación, la proliferación y la migración de fibroblastos77, mostrando que mediante la modulación de estos miRNA se logra controlar vías promotoras de la fibrosis y remodelación patológica del tejido cardiaco, teniendo un efecto positivo a nivel funcional.
Mejora en la función contráctil
Otra característica de la IC es la alteración de la contractilidad miocárdica, derivada en gran medida por la alteración de la estructura y componentes subcelulares encargados del manejo del calcio, proceso que puede darse al menos en parte por una desregulación de miRNA (Fig. 4). Esto incluye un aumento en los niveles de miR-25, ya que este promueve la pérdida de la función contráctil al inhibir la expresión de la bomba de absorción de calcio del retículo sarcoplásmico (SERCA2a), alterando la capacidad de bombeo del corazón, razón por la cual tratamientos en modelos murinos de inhibición de este miRNA muestran efectos favorables al incrementar la sobrevivencia de los animales, controlar la IC y mejorar la contractilidad y la función cardiacas78.

Figura 4 Mejora en la función contráctil mediante la administración de miRNA sintéticos o antagomiRs, a fin de normalizar la arquitectura subcelular, mejorar el manejo del calcio y reducir el riesgo de arritmias.
No solo suelen darse problemas de arritmias por la reducción de la entrada de calcio al retículo sarcoplásmico, sino por el exceso en liberación de este, fenómeno que puede atribuirse a un incremento en la expresión de los receptores de rianodina (RyR2) y mayor actividad de estos; procesos que se han encontrado en parte regulados por miRNA, pues experimentos en ratones han mostrado que una disminución en los niveles de miR-106b, miR-93 y miR-25 se relaciona con una mayor expresión de RyR2, llevando a una mayor susceptibilidad de arritmias79. Igualmente, otros estudios en corazones de caninos con IC crónica evidenciaron que la sobreexpresión de miR-1 y miR-33 incrementa la arritmogénesis, en parte debido a que miR-33 inhibe la expresión de la subunidad catalítica de la fosfatasa 2A, lo que implica una mayor fosforilación y actividad de los receptores RyR2 l, un incremento en la fuga de calcio e indicadores de proarritmias80.
Otro componente de interés en las células del músculo cardiaco es la Junctophilina 2 (Jp2), una proteína que sirve de anclaje del retículo sarcoplásmico a los túbulos T y la cual es suprimida por mir-24, por lo que la sobreexpresión de este miRNA altera de forma negativa la estructura subcelular y deriva en un acoplamiento contracción-excitación deficiente81,82. Incluso en experimentos in vivo, la supresión de miR-24 previene la transición de una hipertrofia compensada a una hipertrofia descompensada y mejora el acoplamiento excitación-contracción, presentándose como una estrategia de interés en la IC81. Finalmente, cabe mencionar que la sobreexpresión de miR-22 en modelos murinos incrementa la hipertrofia y la disfunción contráctil al alterar la entrada de calcio y su carga en el retículo sarcoplásmico, dado que este miRNA afecta la expresión de múltiples factores de transcripción importantes83, por lo que su inhibición puede ser de importancia en la prevención y la regresión de la IC.
Estos resultados en modelos animales y cultivos celulares dan un indicativo de la utilidad de la regulación terapéutica de los miRNA no solo en la normalización de la estructura cardiaca a nivel tisular, sino a nivel de la organización intracelular y los beneficios que esto trae en la prevención de la progresión de la IC.
Por último, como se mencionó al inicio de este artículo, aunque esta es una enfermedad que afecta principalmente a personas adultas también afecta a la población infantil, por lo que es importante mencionar que se ha encontrado que los perfiles de expresión de miRNA en niños con IC difieren de los que se ven en adultos que presentan la enfermedad, y que el tratamiento con inhibidor de la fosfodiesterasa (PDEi), que es común y muy efectivo en el manejo de la IC pediátrica (pero no en el tratamiento en adultos), altera los niveles de algunos miRNA que parecen prevenir la transición de la hipertrofia a la IC84, lo cual plantea que lo establecido para la enfermedad en personas adultas podría no ser perfectamente extrapolable o equiparable a los casos pediátricos84, incluyendo la investigación en miRNA.
Los casos anteriores dejan ver que los miRNA cobran cada vez más importancia como una de las herramientas disponibles para la regulación genética dirigida a tratar las características patológicas de la IC (Tabla 2). Sin embargo, a fin de que estas nuevas estrategias tengan un impacto en el manejo de la enfermedad en humanos es necesario continuar con la investigación de la acción de los miRNA en la IC, estableciendo qué miRNA específicos tienen el mayor potencial como dianas terapéuticas, para facilitar la creación de protocolos que puedan ser ampliamente usados. También es importante determinar qué dosis de miRNA sintéticos o antagomiRS es la más óptima para revertir los procesos patogénicos cardiacos, así como determinar posibles efectos secundarios de estos tratamientos y umbrales o puntos de corte en cuanto a dosis o tiempo de exposición para prevenirlos. Ya que, como muestran Tian et al.63 (2015), aunque una sobreexpresión controlada de determinados miRNA es beneficiosa en la regeneración cardiaca en ratones, cuando la exposición es por tiempos prolongados puede llevar a la disfunción y desdiferenciación de cardiomiocitos. También hay que tener en cuenta que es necesario establecer vectores no inmunogénicos que eviten la degradación del miRNA, con una buena tasa de entrada a las células receptoras y que permitan selectividad de las células cardiacas a transfectar. Otra importante limitación del uso de miRNA en la terapéutica es el hecho de ser una herramienta relativamente nueva que ha sido probada en su gran mayoría en estudios en cultivos celulares o modelos murinos, por lo que su efecto real en la salud humana aún es desconocido y se requieren ensayos clínicos para elucidarlo y para comparar el desempeño de terapias basadas en modulación de miRNA frente a tratamientos estándares.
Perspectivas futuras
Importantes avances se han dado en los últimos años en el estudio del potencial médico de la evaluación de perfiles de miRNA, así como en la modulación de estos, llevando actualmente a la realización de múltiples ensayos observacionales para establecer miRNA biomarcadores en diversas enfermedades. También se están llevando a cabo ensayos clínicos en fases I y II (MRX34, MRG-201, MRG-106 y SPC3649) de medicamentos basados en miRNA sintéticos y antagomiRs para tratar varios tipos de cáncer, prevenir la fibrosis y combatir la hepatitis C. Esto muestra el interés de la comunidad biomédica en llevar el uso de miRNA a la práctica clínica, como una nueva estrategia para la evaluación y el tratamiento de patologías humanas.
Entre los diferentes miRNA que se han descrito y propuesto son especialmente promisorios aquellos que han demostrado una mayor capacidad que otros biomarcadores ya usados en la práctica clínica o que brindan nueva información sobre la enfermedad y su impacto en la salud a corto, mediano y largo plazo, aunque estudios con mayor número de muestra aún son necesarios. En cuanto a la terapéutica, probablemente los miRNA más novedosos son los aplicados al campo de la regeneración cardiaca, pues existen estrategias usadas en la práctica clínica para reducir procesos como la hipertrofia y la fibrosis y para mejorar la función contráctil; sin embargo, la mayoría de tratamientos rutinarios no tienen un mayor efecto en la recuperación de cardiomiocitos perdidos. Lo anterior sin dejar de resaltar la importancia de otros miRNA que pueden llegar a mejorar el diagnóstico, el pronóstico y el tratamiento de la enfermedad.
Conclusiones
El uso de estrategias basadas en miRNA para el manejo de la IC ha cobrado gran interés en el campo de la biomedicina debido a que pueden ser usados como biomarcadores que brindan información valiosa sobre el estado cardiaco y su impacto sobre la salud. También aparecen como promisorias herramientas en la terapia de la enfermedad por su capacidad de inducir cambios celulares sin alterar la secuencia genética (como otros tipos de terapia génica) y los resultados positivos en ensayos celulares y modelos murinos, que muestran como la modulación positiva o negativa de miRNA clave puede prevenir o revertir la remodelación cardiaca tisular y subcelular. Además, con los avances en las tecnologías genómicas de secuenciación, cuantificación y síntesis de ácidos nucleicos, así como el creciente conocimiento de la biología de los miRNA y su implicación en la IC, se espera que lleguen a tener un impacto positivo en los pacientes con esta enfermedad.











 nueva página del texto (beta)
nueva página del texto (beta)