Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Archivos de cardiología de México
versión On-line ISSN 1665-1731versión impresa ISSN 1405-9940
Arch. Cardiol. Méx. vol.75 no.4 Ciudad de México oct./dic. 2005
Revisión de temas cardiológicos
Genética y biología molecular de las cardiopatías congénitas y adquiridas
Genetics and molecular biology of the congenital, and acquired heart disease
David Cruz Robles,*,****** Aurora de la Peña Díaz,**,****** Minerva Arce Fonseca,***,***** José de Jesús García Trejo,****,****** Óscar A Pérez Méndez,*****,****** Gilberto Vargas Alarcón*****,******
* Departamento de Patología.
** Departamento de Farmacología.
*** Laboratorio de Inmunoparasitología.
**** Departamento de Bioquímica.
***** Departamento de Fisiología.
****** Grupo de Estudio en Genómica y Proteómica de Enfermedades Cardiovasculares. Instituto Nacional de Cardiología, "Ignacio Chávez".
Correspondencia:
Dr. David Cruz Robles.
Departamento de Patología, Instituto Nacional de Cardiología, "Ignacio Chávez"
(INCICH Juan Badiano Núm. 1 Col. Sección XVI, Delegación Tlalpan,
14080. México, D.F.).
Tel. 55732911 Ext. 1217 Fax: 55730926.
E–mail: crumis@hotmail.com
Recibido: 8 de noviembre de 2005
Aceptado: 14 de noviembre de 2005
Resumen
El corazón es el primer órgano que se forma y funciona en el embrión, de tal suerte que todos los eventos subsecuentes en la vida del organismo dependen de la habilidad de este órgano para atender las demandas de oxígeno y nutrientes que éste requiere. Las anormalidades en la formación del corazón, la forma más común de defectos humanos al nacimiento, afecta al 1 % de los nacidos vivos, y su frecuencia en abortos espontáneos se eleva diez veces más. La patofisiología de este tipo de malformaciones congénitas se ha venido enriqueciendo en los últimos años con el conocimiento del Proyecto Genoma Humano; debido al gran avance que se ha producido en el conocimiento genético y molecular de los diferentes genes y cromosomas que suelen ser afectados y muchas veces heredados para producir una enfermedad congénita en general. Esta revisión trata de enfocar su atención sobre la información extraída de los análisis genéticos y moleculares en el diagnóstico, tratamiento y entendimiento de la patogénesis de las enfermedades cardiovasculares pediátricas, dirigidas tanto por los más comunes defectos cardíacos congénitos o heredados, como por los desórdenes esporádicos o adquiridos.
Palabras clave: Defectos cardíacos congénitos. Genética. Biología molecular. Cardiología pediátrica.
Summary
The heart is the first organ to form and function in the embryo, and all subsequent events in the life of the organism depend on the heart's ability to match its output with the organism's demands for oxygen and nutrients. Abnormalities in heart formation, the most common form of human birth defects, afflict nearly 1% of newborns, and their frequency in spontaneously aborted pregnancies is estimated to be tenfold higher. With the completion of the sequencing of the human genome, molecular genetic efforts directed at finding genes for monogenic traits have accelerated dramatically. Breakthroughs in molecular genetic technology have just begun to be applied in pediatric cardiology stemming from the use of chromosomal mapping and the identification of genes involved in both the primary etiology and as significant risk factors in the development of cardiac and vascular abnormalities. This review will focus on information provided by molecular and genetic analysis in the diagnosis, treatment and overall heart disorders.
Key words: Congenital heart disease. Genetics. Molecular biology. Pediatric cardiology.
El corazón es el primer órgano que se forma y funciona en el embrión como un tubo primitivo, de tal forma que todos los eventos subsecuentes en la vida de un organismo dependen de su funcionalidad. Las mutaciones heredadas en los genes que intervienen en el desarrollo cardioembrionario, pueden provocar una enfermedad cardíaca congénita, que es la forma más común de defectos humanos del nacimiento (1% de todos los nacimientos), o anormalidades en el corazón adulto que son la causa más prevalente de morbi–mortalidad en el mundo industrializado.12 En los últimos diez años, se ha observado una marcada transición en los estudios fisiológicos del corazón, de tal suerte que se ha avanzado mucho en el conocimiento de la disfunción cardíaca a nivel genético y molecular. Estos avances científicos y tecnológicos han dado la oportunidad de crear nuevas aproximaciones terapéuticas para la prevención y el tratamiento de enfermedades cardíacas y, además, han generado nuevas incógnitas a resolver en este tema, provocando cambios y nuevas oportunidades en el desarrollo de terapias para las enfermedades cardíacas congénitas y adquiridas.
El entendimiento de los mecanismos del desarrollo cardíaco provee de nuevos conocimientos en los fenotipos cardíacos anormales. El desarrollo de los organismos transgénicos ha permitido reproducir defectos cardíacos gobernados por un solo gen (monogénicos) el cual se depleta o inhabilita en forma homocigota, aunque la mayoría de las mutaciones que causan enfermedad cardíaca en el humano se presentan en forma heterocigota. Por otro lado, los defectos cardíacos congénitos en el humano presentan una gran variabilidad en cuanto a su pene–trancia y expresividad, lo cual indica que, además del efecto que causa el o los genes mutados, las influencias medioambientales también participan en el fenotipo de la enfermedad.3
Los avances recientes en genética molecular han revelado que existen factores genéticos y moleculares ligados a enfermedades cardíacas congénitas y arritmias cardíacas, y han permitido su identificación en el mapa genómico (Fig. 1), con lo cual se ha creado una invaluable oportunidad para innovar diagnósticos genéticos y, en un futuro, alcanzar la terapia génica.

I. Cardiopatía congénita:
Existe una gran variedad de defectos congénitos asociados a mutaciones simples y dichas mutaciones se presentan en un amplio espectro de genes involucrados en la estructura y funcionamiento cardíaco. El nivel para la especificidad cardíaca en estas mutaciones es altamente variable. Algunos genes mutados se asocian a síndromes con una presentación neuromuscular sistémica que también involucra al corazón (por ejemplo: Ataxia de Friedreich y Distrofia muscular de Duchenne). Un amplio rango de defectos cardíacos resulta de estas mutaciones genéticas, incluyendo anormalidades en la función electrofisiológica (defectos de la conducción y arritmias), proteínas de la matriz extracelular, enzimas y transportadores de membrana involucradas en la biosíntesis de ácidos grasos y función mitocondrial, metabolismo de la fosforilación oxidativa, estructura sarcomérica, proteínas contráctiles y factores de transcripción nuclear que regulan la expresión genética miocárdica y su desarrollo programado (Tabla I).
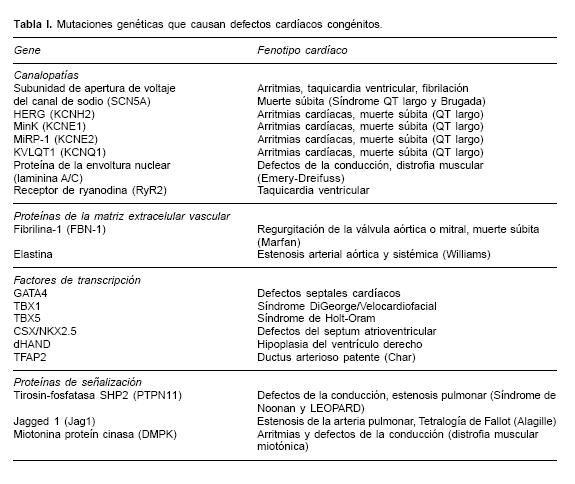
Las malformaciones cardíacas pleiotropicas pueden ser el resultado de mutaciones discretas en factores de transcripción específicos y proteínas clave en el desarrollo embrionario y la morfogénesis cardíaca.4–6 Factores como GATA4, NKX2.5, dHAND, TFAP2 y TBX5, son genes que se expresan tempranamente durante el desarrollo embrionario cardíaco y su expresión es crucial para la activación de otros genes cardíacos. Mutaciones específicas en cada uno de estos genes representan severas anormalidades cardíacas como: defectos septales (GATA4), defectos de la conducción (NKX2.5), hipoplasia ventricular derecha (dHAND), ductus arteriosus patente en el síndrome de Char (TFAP2B) y síndrome de Holt–Oram (TBX5), entre otros, cuya acción es crítica en la disrupción del desarrollo cardíaco temprano y la morfogénesis cardíaca dentro de la génesis de los defectos congénitos cardíacos.7–11
Así mismo, existen defectos genéticos en proteínas involucradas en múltiples rutas de señalización que modulan la proliferación, migración y diferenciación celular en la embriogénesis cardíaca. Se han detectado mutaciones en el gen Jag1en estudios de asociación con el síndrome de Alagille, un desorden autosómico dominante que se presenta con defectos congénitos cardíacos que incluyen estenosis de la arteria pulmonar y tetralogía de Fallot.12
El gene PTPN11 codifica para la tirosin fosfatasa (SHP–2), de tal suerte que mutaciones en este gen pueden causar síndrome de Noonan, el cual se caracteriza por defectos de la conducción, estenosis pulmonar, cardiomiopatía hipertrófica13 y recientemente se le ha involucrado en la patogénesis del síndrome de LEOPARD (lentígines múltiples, electrocardiograma anormal, hipertelorismo, estenosis de la arteria pulmonar, anormalidades genitales, retraso en el crecimiento y sordera) el cual puede ser un desorden alélico.14
Además de las mutaciones puntuales, en regiones codificantes (exones) de genes específicos, una gran cantidad de desórdenes neuromusculares heredados se han referido como síndromes de triple repetición, en los cuales está incluida la ataxia de Friedreich y la distrofia muscular miotónica, que son causadas por secuencias trinucleotídicas repetidas en genes específicos, por ejemplo en el gen de la frataxi–na (FRDA) y el de la miotonin proteína cinasa (DMPK), respectivamente.15,16 Los individuos afectados presentan cardiomiopatía, arritmias cardíacas y defectos en la conducción. En ambos defectos, la severidad de la enfermedad correlaciona con el número de tripletes repetidos, por ejemplo: individuos con ataxia de Friedreich presentan > 200 repetidos GAA, mientras que en la distrofia muscular miotónica, los individuos afectados presentan más de 50 copias del triplete CTG.
Otro tipo de defectos genéticos involucrados en malformaciones del desarrollo estructural cardíaco, son las grandes deleciones o microdeleciones cromosomales que provocan anormalidades conotroncales, defectos del canal atrioventricular y defectos del septum atrial y ventricular.17,18 Defectos del tracto de salida cardíaco son manifestaciones de un desorden genético complejo denominado Síndrome DiGeorge/velocardiofacial o CATCH22 (defectos cardíacos, facies anormales, hipoplasia tímica, paladar hendido, hipocalcemia). La mayoría de los pacientes son hemicigotos para una microdeleción que abarca una región de 1.5 a 3 megabases en el cromosoma 22 (22q11.2), la cual es esencial para el desarrollo normal del arco faríngeo. La deleción del 22q11, es un evento relativamente común que se da en 1 de cada 4,000 nacidos vivos. Se piensa que el gene TBX1, ubicado en la parte central de la región deletada, es un factor crítico en el desarrollo de este defecto congénito.19 Dicho gen codifica para un factor de transcripción involucrado en la regulación del desarrollo cardíaco y es miembro de una familia de genes altamente conservados que comparten un dominio de unión a DNA (el T–box). La reducción de la expresión de TBX1 (la cual ocurre en estado hemicigoto), representa una haploinsuficiencia que repercute ampliamente en la expresión de varios genes de expresión temprana en la morfogénesis cardíaca. Se han reportado otras microdeleciones cromosómicas asociadas a defectos cardíacos congenitos (8p) y es posible que se localicen otras más.20 Las técnicas citogenéticas moleculares actuales presentan una alta resolución, como el caso de la hibridación fluorescente in situ (FISH), que se utiliza rutinariamente para confirmar el diagnóstico clínico de daño cromosómico como en el caso de microdeleciones y pequeñas translocaciones.
Es importante hacer notar que las grandes deleciones genéticas se asocian comúnmente con un amplio espectro de características clínicas adicionales al daño cardíaco. Las malformaciones extracardíacas se asocian frecuentemente con defectos cardíacos congénitos y su incidencia se ha calculado en un 30% de los casos. Dichas anomalías cromosómicas son más prevalentes en individuos con defectos cardíacos que en la población general. Estas malformaciones cardíacas son el resultado de aberraciones cromosómicas como lo es el caso de las trisomías 13, 18 y 21 (Síndrome de Down), así como de la monosomía X0 (Síndrome de Turner), cuyas bases moleculares precisas aún no han sido del todo elucidadas.
II. Arritmias y muerte súbita:
Las arritmias cardíacas son complicaciones frecuentes de enfermedad cardíaca pediátrica y pueden ser la principal causa de muerte súbita. Se han identificado mutaciones en genes que codifican para canales iónicos cardíacos, como factores de riesgo en la patogénesis de arritmias letales y no letales.
El gen SCN5A codifica para un canal de sodio responsable del inicio del potencial de acción. Mutaciones en este gen se asocian a la prolongación del intervalo QT o Síndrome de QT largo (LQTS) el cual predispone al síncope y muerte súbita.21,22 Su fenotipo es una repolarización ventricular anormal que puede resultar en fibrila–ción ventricular idiopática, taquicardia ventricular, defectos de la conducción cardíaca y síndrome de Bragada.23,24 Existen 4 genes más involucrados en la formación de canales de potasio que también se han asociado al LQTS, ellos son el HERG, KCNE1, KCNE2 y KVLQT1 y en ellos se han descrito más de 30 diferentes mutaciones estudiadas en 40 familias con gran heterogeneidad genética.25
Se han identificado mutaciones de sentido erróneo (missense) en el gen del receptor rianodin (RyR2), que es un canal liberador de calcio involucrado en la contracción–relajación sarcomérica, y que causa estrés por sobrecarga de calcio en miocitos, lo cual provoca taquicardia ventricular.26 Algunas mutaciones en el gen de lamini–na A/C, que codifican para proteínas de la envoltura nuclear, se presentan en individuos afectados por la forma autosómica dominante de la distrofia muscular de Emery Dreifuss, en la que se observa lipodistrofia parcial familiar, car–diomiopatía dilatada (DCM), defectos de la conducción atrioventricular y fibrilación atrial.27 Por otro lado, la acumulación de metabolitos intermediarios de los ácidos grasos, como la cadena larga de acilcarnitina, pueden provocar arritmias cardíacas y defectos en la conducción en neonatos. Se han reportado errores del nacimiento en la oxidación de ácidos grasos (por ejemplo, deficiencias en la carnitin:palmitoil–transferasa II, proteína trifuncional mitocondrial o en la carnitil–acil–carnitin translocasa), en la muerte súbita infantil inexplicable y en infantes con defectos de la conducción o taquicardia ventricular.28
II. Vasculopatías:
Dentro de las vasculopatías, consideradas como defectos autosómicos dominantes, se encuentra el síndrome de Marfán, la estenosis aórtica supravalvular y el síndrome de Williams, en los cuales se evidencia el papel crítico de las microfibrillas, y defectos de la matriz extracelular en la patofisiología de estos defectos génicos.29,30 El síndrome de Marfán se caracteriza por anormalidad de los sistemas músculo–esquelético y cardiovascular, además de los ojos, que llevan a una muerte prematura principalmente por una dilatación progresiva del tronco aórtico con una disección aórtica fatal o por insuficiencia valvular aórtica y se asocia con una gran mortalidad neonatal con participación polivalvular y falla cardíaca congestiva severa. La mayoría de los casos con síndrome de Marfán y enfermedad cardiovascular, tienen mutaciones en el gen de la fibrilina y la mayoría de sus familiares presentan diferentes mutaciones ahí. La fibrilina es un constituyente de un complejo multiproteico (que incluye a la elastina) presente en el componente microfibrilar de la pared de los grandes vasos. Mutaciones en los genes que codifican para los componentes de la matriz extracelular (por ejemplo: elastina), son responsables de la estenosis supravalvular aórtica cuyas características obstructivas afectan a la aorta ascendente. El síndrome de Williams se caracteriza por presentar estenosis de las arterias pulmonares o sistémicas.
IV. Cardiomiopatía:
Las mutaciones que causan cardiomiopatías en el humano se han identificado en un amplio espectro de genes nucleares que codifican para proteínas contráctiles y estructurales, enzimas involucradas en el almacenamiento del glucógeno (enfermedades de Cori y Pompe) y degradación de mucopolisacáridos (enfermedad de Fabry), metabolismo de lípidos (β–oxidación de ácidos grasos y deficiencia de la carnitina) y en los genes esenciales para la producción de energía cardíaca (Tabla II), ubicados en el DNA nuclear y mitocondrial (mtDNA). Se ha observado que tanto la cardiomiopatía dilatada (DCM) como la cardiomiopatía hipertrófica (HCM) se presentan en jóvenes y pueden tener componentes genético–familiares. La HCM se ha estudiado más ampliamente debido a que representa la causa más frecuente de muerte súbita en niños y adolescentes. La mayoría de casos familiares de HCM presentan una forma de transmisión autosómica dominante, a excepción, claro, de aquellos casos con mutaciones en el mtDNA, las cuales son heredadas por vía materna (Fig. 2). Se han observado más de 10 genes involucrados en la HCM que codifican para proteínas de la sarcómera como la cadena pesada de α y β–miosina (α y β–MHC), proteína C de unión a miosina, troponina I y troponina T, α–tropomiosina, cadenas ligeras de miosina esencial y regulatoria, titina y α–actina.31–41 También se han detectado mutaciones en pacientes con HCM en genes involucrados en el metabolismo del grupo hemo y Fe++ (Frataxina y COX15)15,37 y en la bioenergética mitocondrial (genes mitocondriales para tRNAs y ATPasa6).38 Un subgrupo de casos conHCM mostró mutaciones en la subunidad reguladora de la proteína cinasa de AMP (AMPK), la cual es un sensor clave y mediador del metabolismo energético.39 En resumen, todos estos resultados sugieren que la interrupción del metabolismo energético mitocondrial cardíaco es la causa de HCM en aquellos pacientes con problemas de contracción sarcomérica y nos pueden ayudar a entender numerosas observaciones clínicas como su heterogeneidad, variabilidad de presentación clínica y su asimetría hipertrófica.


Actualmente, se estima que aproximadamente el 30% de los casos con DCM son heredados, y el 70% son casos esporádicos. Se han identificado genes relacionados con la DCM familiar ligada al cromosomaX (Distrofina, G4.5)40,41 y con la forma autosómica dominante de DCM (acuna, desmina, laminina A/C, δ–sarcoglicano).42–44 En el caso de la DCM ligada al cromosoma X, el gen de la distrofina (enorme proteína del citoesqueleto asociada a la sarcómera) se ve afectado sólo en los miocitos, de tal suerte que las mutaciones que lo afectan se ubican en su promotor tejido específico.45 Mutaciones directas sobre el gen de la distrofina provocan distrofia muscular de Duchenne o de tipo Becker (DMD, DMB), en las cuales se afectan tanto el músculo cardíaco como el esquelético en general.40 La forma más grave de la DMD es provocada por mutaciones o grandes deleciones que afectan el marco de lectura traduccional o aquellas mutaciones puntuales que crean codones de paro, que afectan la producción completa de la proteína. Los varones afectados en la DMD son asintomá–ticos al principio de su adolescencia, pero desarrollan síncope y falla cardíaca congestiva rápidamente progresiva en el final de su adolescencia; las mujeres generalmente presentan la sintomatología más tardíamente. En este tipo de distrofia se debilitan los músculos esqueléticos a edad temprana (3 a 6 años) y posteriormente más del 30% de los casos presentan disfunción cardíaca alrededor de los 14 años; virtualmente, todos los pacientes con DMD desarrollan DCM a los 18 años.
El síndrome de Barth, una miopatía cardioesquelética ligada al X, con neutropenia y cardio–miopatía dilatada que se presenta en la infancia, es producido por mutaciones del gen G4.5 que codifica para la tafacina, una proteína de la familia de aciltransferasas involucrada en la síntesis de fosfolípidos.41–46 Los pacientes con este síndrome presentan niveles elevados de ácidos grasos saturados y muy bajos de cardiolipina, lo cual afecta la fluidez de la membrana de los cardiomiocitos y su función. La displasia arritmogénica del ventrículo derecho (ARVD) se presenta en forma autosómica dominante caracterizada por degeneración progresiva del miocardio, arritmias y gran riesgo de muerte súbita. El análisis de ligamiento ha mostrado que este desorden involucra varios loci de los cromosomas 2, 10, 14 y 17, pero el sitio exacto del defecto genético no se ha determinado.47
Se han descrito varias cardiopatías mitocondriales asociadas a desórdenes neurológicos como el MELAS (miopatía mitocondrial, encefalopatía, acidosis láctica y episodios convulsivos), el MERRF (epilepsia mioclónicay fibras rojas rasgadas) y el síndrome de Leigh38,48 en genes del mtDNA y también en genes nucleares involucrados en el ensamble de la cadena respiratoria mitocondrial (Fig. 2). Estos desórdenes pueden presentarse de forma temprana o tardía y causar cardiomiopatía infantil fatal.49–51 Los estudios moleculares de pacientes con HCM o DCM han aportado gran información para la identificación de nuevas mutaciones en el mtDNA que prevalecen en el tejido cardíaco.52,53 Estas mutaciones también se han encontrado recientemente en genes que codifican para los complejos respiratorios mitocondriales, I, III, y IV, además del gen ATP6 de la F1F2–ATP sintasa (Tabla II). Otros genes nucleares involucrados en la biogénesis mitocondrial o en el ensamblaje de los complejos respiratorios, también son blancos de mutaciones que provocan cardiomiopatías; tal es el caso de SCO2 que controla el ensamblaje de la citocromo oxidasa (Complejo IV), o los genes NDUFS2 y NDUSF4 que son parte estructural de la NADH deshidrogenasa o complejo I mitocondrial.54 Las cardiomiopatías mitocondriales pueden ser causadas por eventos esporádicos como lo son los agentes que causan daño al mtDNA como el alcohol y la adriamicina.55,56 Las mutaciones somáticas así generadas pueden incrementarse durante la isquemia miocárdica y generar daño por estrés oxidativo.55-59 El síndrome de Kearns–Sayre, un desorden neuromuscular con defectos de la conducción atrioventricular y cardiomiopatía, se asocia con deleciones abundantes del mtDNA que se cree que son provocadas de forma espontánea porque no se detectan en la madre o hermanos del afectado.60
En contraste, la DCM se ha asociado a múltiples deleciones en el mtDNA y se pueden presentar de manera dominante o recesiva.61 Las familias que presentan una herencia dominante presentan mutaciones en proteínas necesarias para la replicación del mtDNA, como el gene que codifica para la mtDNApol γ y el gene Twinkle que produce una helicasa, o el metabolismo nucleotídico mitocondrial, como el translocador nucleotídico de adenina.62 Las mutaciones autosómicas recesivas se presentan en factores que juegan un papel importante en el metabolismo nucleotídico mitocondrial, como la timidincinasa 2, timidin–fosforilasa y deoxy–guanosin–cinasa.62 Además, las mutaciones mitocondriales pueden ser inducidas también por zidovudine (AZT), el cual inhibe la acción de las DNApol virales y mitocondriales, con lo cual afecta a la replicación,63 aunque aún no se conoce que este compuesto afecte en el desarrollo de cardiomiopatía en niños tratados con AZT.64
Técnicas de diagnóstico molecular, limitaciones y avances:
Muchos de los genes nucleares implicados en las cardiopatías fueron mapeados por análisis de ligamento en las familias afectadas y esto permitió su posterior identificación como genes candidatos por clonación posicional y secuencia–ción. Una gran variedad de técnicas moleculares, incluyendo la reacción en cadena de la polimerasa (PCR), restricción de fragmentos polimórficos (RFLP's) y polimorfismos conformacionales de cadena sencilla (SSCP), se han utilizado para rastrear alelos defectuosos del caso índice y sus familiares para establecer los patrones de herencia. En la mayoría de los casos, la detección de nuevas mutaciones se realizó sobre las secuencias codificantes (exones) de los genes candidatos y los casos relativamente bien caracterizados de HCM familiar han mostrado que las mutaciones que provocan la enfermedad son casos raros o mutaciones "de novo" en estas familias. Así pues, una correlación genotipo–fenotipo como en mutaciones específicas de β–MHC en la HCM se pueden asociar con una alta incidencia de muerte súbita, mientras que otras mutaciones se asocian con un mejor pronóstico. Recientes avances en la velocidad y sensibilidad de la detección de mutaciones aplicando técnicas como la cromatografía líquida altamente desnaturalizante (DHPLC) o la electroforesis capilar de alta resolución, pueden potenciar el uso de los análisis genéticos–moleculares para establecer un diagnóstico preclínico o clínico y proveer tratamientos en blancos específicos de desordenes cardíacos pediátricos. Así mismo, en un futuro cercano, la disponibilidad de la tecnología a base de chips genéticos permitirá un rastreo rápido y automatizado de mutaciones en el DNA nuclear y mitocondrial.
Desde que las técnicas modernas de imagenología han ayudado a determinar los fenotipos cardíacos en niños afectados, la heterogeneidad genética y la variabilidad intrafamiliar han precisado la elucidación molecular de muchos defectos cardíacos, así como la correlación genotipo–fenotipo que regularmente es difícil de observar. Estas dificultades pueden atribuirse a factores poligénicos o multifactoriales que contribuyen a la expresión de defectos cardíacos específicos, así como también a una gran cantidad de influencias epigenéticas o adquiridas. El progreso es gradual y comienza a definir estos factores epigenéticos y poligénicos, algunos de los cuales son afines al análisis molecular.
Se han realizado estudios de asociación de polimorfismos de bases simples en donde se han detectado genes candidatos para varios padecimientos cardíacos como infarto del miocardio, enfermedad de arterias coronarias y cardiomiopatía hipertrófica (Tabla III). Estos estudios han generado bases de datos que se encuentran disponibles para evaluar los efectos de los polimorfismos en la predisposición a defectos cardíacos específicos y quizá tengan impacto en las opciones de diagnóstico y tratamiento. Los recientes avances en las metodologías han hecho posible la realización de análisis detallados de la expresión genética con el uso de muestras pequeñas de tejido, lo cual es muy importante en el estudio de neonatos y niños. Una de estas metodologías se basa en los microarreglos de DNA (Microarrays). Estos microarreglos son constructos artificiales de DNA arreglados en forma de rejillas (hileras y columnas) en el cual cada elemento ubicado en cada una de las celdas de la rejilla, actúa como una sonda para hibridar a un RNA específico. La expresión génica por análisis de microarreglos ofrece una potente herramienta para establecer las características patofisiológicas de una enfermedad por la evaluación del incremento o decremento de expresión genética, pudiendo aplicarse al diagnóstico y a la evaluación de tratamientos aplicados a los pacientes.

La asociación de genes defectuosos con desórdenes cardíacos no cubiertos por análisis genómicos, necesitan ser seguidos por análisis pro–teómicos para establecer la función y papel patofisiológico de la proteína mutada. Una vez que se ha descrito un gen y su producto, el análisis de su secuencia puede emplearse para identificar secuencias homologas y motivos estructurales y funcionales con proteínas conocidas para un mejor entendimiento de sus actividades. Las interacciones funcionales de las proteínas son potencialmente significativas para estudiar el fenotipo cardíaco. Así, se ha establecido que la titina mutada (derivada de pacientes con HCM) reduce su afinidad de unión con otras proteínas sarcoméricas, como la α–actina, y se pueden caracterizar sus interacciones sinérgicas con factores de transcripción como el NKX2.5 y TBX5 en el desarrollo cardíaco temprano.65
Análisis transgénicos:
Los ratones transgénicos son una herramienta muy utilizada en biología molecular para confirmar un fenotipo específico, que puede ser modificado por la alteración de un gen y su producto proteico, en este tipo de organismos llamados "Knock–out" cuando se suprime el gen, o "knock–in" cuando se altera su expresión de manera positiva (sobre–expresión). Ratones knock–out para la oxidación de ácidos grasos (proteína trifuncional mitocondrial), la transcripción del mtDNA y la bioenergética (factor A de transcripción mitocondrial) y para el gen de la frataxina mitocondrial, desarrollan una rápida progresión de disfunción cardíaca y DCM con datos clínicos de cardiomiopatía asociada con mutaciones específicas en genes involucrados en la función bioenergética mitocondrial.66–68 Estas metodologías han proveído una gran cantidad de información para establecer el papel crítico que juega en gen TBX1 en la etiología del síndrome DiGeorge/Velocardiofacial. Ratones heterocigotos para un alelo TBX1 anulado presentan una gran incidencia de anormalidades en el tracto de salida cardíaco así como otras anormalidades características del síndrome DiGeorge.69
Análisis molecular de anormalidades fetales:
La reconstrucción tridimensional de los defectos cardíacos con la ayuda del ultrasonido, los rayos X y la resonancia magnética nuclear, ha impactado positivamente en el diagnóstico y las estrategias terapéuticas de las enfermedades cardíacas. Actualmente, la mayoría de enfermedades congénitas cardíacas pueden detectarse in útero con estas metodologías, de tal manera que se recomienda hacer evaluaciones subsiguientes de anormalidades extracardíacas y cromosómicas en los pacientes pediátricos, debido a que este tipo de anormalidades se presentan en un 62% y 38% de los casos, respectivamente. El consejo genético, basado en una evaluación prenatal, provee información real acerca de la incidencia, diagnóstico y pronóstico de los defectos cardíacos fetales. El diagnóstico prenatal de malformaciones cardíacas congénitas y sus correlaciones moleculares (por ejemplo: la microdeleción del cromosoma 22q11 en el Síndrome DiGeorge y del 7q en el síndrome de Williams), detectables por técnicas citogenéticas y moleculares subsecuentes a la amniocentesis, ha demostrado ser una potente herramienta en el manejo de las malformaciones del neonato como lo son la transposición de grandes arterias y el síndrome de hipoplasia cardíaca izquierda.
Enfermedades cardíacas adquiridas en niños:
Entre las enfermedades cardíacas adquiridas por neonatos y niños se encuentra la enfermedad de Kawasaki, enfermedad cardíaca reumática aguda y crónica, endocarditis infecciosa y miocarditis. La tecnología genética–molecular se ha utilizado en el diagnóstico temprano de estas enfermedades, aunque de forma limitada; sin embargo, su aplicación puede dar nueva información en el conocimiento general de las mismas. La enfermedad de Kawasaki es una vasculi–tis aguda limitada de la infancia o de la adolescencia temprana y es una de las principales causas de enfermedad cardíaca adquirida en Estados Unidos y Japón.70 Su etiología es desconocida, pues aún con las herramientas moleculares no se ha detectado alguna asociación viral o bacterial con la enfermedad. Si no es tratada, el 25% de los pacientes desarrollan aneurismas en las arterias coronarias. El tratamiento es efectivo si se aplica dentro de los primeros diez días de inicio de la enfermedad (para prevenir el involucramiento coronario), aunque esto es muy complicado para el cardiopediatra, pues debe distinguir la enfermedad de Kawasaki de otras enfermedades en un tiempo relativamente corto. En este sentido, el ultrasonido intravascular es una alternativa para este fin junto con marcadores moleculares que pueden usarse en forma de análisis por microarreglos para confirmar el diagnóstico.
Los análisis moleculares e inmunológicos tienen implícita la presencia de inducción viral (comúnmente se involucra el grupo B de los coxsackie virus o CVB) y respuestas autoinmunes aberrantes en la patogénesis de la miocarditis pediátrica, la cual, en algunos casos, puede evolucionar a DCM. En estudios moleculares recientes, con la ayuda de la PCR, se ha encontrado presencia de adenovirus, además de enterovirus, en el miocardio de niños con miocarditis y DCM.71 Por otro lado, aunque aún no se ha elucidado completamente el mecanismo patológico de la fiebre reumática o de la enfermedad cardíaca reumática inducida por Streptococus, los análisis moleculares han aportado avances importantes en los aspectos críticos autoinmunes de la enfermedad y se espera que futuros análisis de ligamiento y asociación genética generen información importante acerca de los factores genéticos involucrados en la susceptibilidad del paciente. Los datos moleculares también pueden utilizarse en diversas estrategias para el manejo de anormalidades cardiovasculares asociadas con infecciones adquiridas como la hipertensión pulmonar que se puede presentar por infección con HIV.72
Farmacogenética y cardioprotección:
El entendimiento de las enfermedades cardiovasculares a nivel genómico puede permitirnos hacer una mejor estratificación de subclases de pacientes para optimizar y dirigir terapias paciente–específicas.
Los campos relacionados con la farmacogenómica y con la farmacogenética abrazan la promesa de improvisar el desarrollo de drogas y la creación de terapias basadas en la capacidad individual de un organismo para metabolizar dichos compuestos tomando en cuenta la edad, influencia de la enfermedad, factores medioambientales (por ejemplo dieta), medicamentos tomados y factores genéticos individuales como la especificidad de transporte, metabolismo y blanco de la droga. Por ejemplo, un subgrupo de polimorfismos de base simple identificados en los genes humanos como el del receptor beta adrenérgico y la enzima convertidota de angiotensina (ACE), se han asociado con cambios substanciales en el metabolismo o efectos de medicamentos usados en el tratamiento de la enfermedad cardiovascular, y quizá sean informativos para predecir la respuesta clínica (Tabla III).73 La terapia individualizada puede ser crítica para establecer las dosis de las drogas y su eficacia en niños con enfermedades cardiovasculares, una población en la cual la fármaco–cinética de los medicamentos ha sido pobremente definida y muchas veces es impredecible. Los fenotipos inmunológicos y genéticos de los pacientes pediátricos pueden ser de gran ayuda en el establecimiento de estrategias terapéuticas más efectivas, tanto inhibiendo como estimulando respuestas específicas.
Un creciente número de evidencias han establecido que la cardioprotección puede ser evocada tanto por el preacondicionamiento isquémico o por mecanismos farmacológicos (por ejemplo nicorandil y diaxoxide) que pueden ser potencial–mente manejados como una estrategia de protección del órgano y sus tejidos en la enfermedad cardíaca isquémica o el insulto hipóxico, aunque a la fecha exista muy poca información concerniente a las respuestas cardioprotectoras en niños e infantes. Se han realizado modelos experimentales en animales de laboratorio que han establecido las bases moleculares de los mecanismos de cardioprotección, en los cuales se involucra una red de señales de traducción con diversas vías de señalización mediadas por receptores de superficie, activación y translocación subcelular de proteínas cinasas (como la PKC épsilon, P38 MAP cinasa y JUN cinasa) y la apertura de canales sarcolémicos y mitocondriales KATP.74 Se han encontrado niveles elevados de PKC épsilon, P38 MAP cinasa y JUN cinasa en infantes con defectos cardíacos cianóticos e hipoxia, las cuales no están activadas en infantes con defectos acianóticos o sujetos normales; esto indica que la ruta de señales de traducción cardioprotectoras se encuentran parcialmente en operación en infantes con hipoxia.75 En períodos breves de hipotermia, previos a un insulto isquémico prolongado, se ha observado cardioprotección asociada a las proteínas de estrés y a las señales mitocondriales y quizás éstas estén involucradas en el manejo clínico de la taquicardia ectópica por hipotermia.76,77 Futuras investigaciones en esta área podrán revelar moléculas blanco potenciales para una intervención farmacológica altamente específica. Sin embargo, como una nota precautoria, dichas investigaciones pueden llevarse mucho tiempo para incrementar nuestro conocimiento de la red de rutas de señalización que están interactuando. A pesar de los recientes logros en la identificación precisa de los defectos genéticos de señalización que causan arritmias cardíacas, el desarrollo de drogas efectivas (por ejemplo bloqueado–res de canales iónicos específicos), que puedan reducir sustancialmente la mortalidad asociada con severos desórdenes arrítmicos, se ha visto mermado y se incrementará en la medida en que nuestro conocimiento acerca de los complejos circuitos cardíacos, causas múltiples, factores genotípicos y de riesgo involucrados en estas enfermedades, aumente.78
Perspectivas:
Aunque se han realizado avances significativos en el diagnóstico y tratamiento de las enfermedades cardíacas en los niños, existen aún muchas preguntas por resolver a nivel de los mecanismos patofisiológicos básicos de dichos padecimientos. La aplicación de la tecnología genético–molecular ha trazado brechas por las cuales se han abordado las enfermedades cardiovasculares y han permitido el mapeo cromosomal y la identificación de algunos genes involucrados tanto en la etiología primaria como en los factores de riesgo para el desarrollo de dichas anomalías. Las siguientes áreas del conocimiento pueden ser muy promisorias en el futuro:
1) El entendimiento del desarrollo cardíaco y vascular ha sido poco explorado en los infantes, por lo cual la identificación futura de nuevos genes involucrados en la organogénesis y el desarrollo vascular servirá como un importante fundamento para el entendimiento de cómo se generan los defectos genéticos congé–nitos y sus fenotipos cardíacos. Los métodos bioinformáticos pueden emplearse para investigar las bases de datos generadas por medio de técnicas genéticas de rutina. Esto permitirá la subsiguiente clonación de nuevos genes a partir del cDNA de interés, con lo cual se puede lograr la caracterización temporal y espacial de patrones específicos de expresión genética de genes específicos del desarrollo embrionario (usando hibridación in situ).
2) Los mecanismos que gobiernan la especificación temprana de las cámaras cardíacas en el desarrollo del tubo cardíaco no se conocen totalmente, aunque se piensa que pueden estar involucradas nuevas rutas de señalización célula–célula entre las células emigrantes, así como el disparo de programas de expresión genética cámara–específicos, mediados por factores de transcripción especializados como la proteína morfogenética de hueso (MBP). Se pueden crear nuevas áreas para el estudio del papel de las moléculas de señalización (por ejemplo WNT) utilizando animales Knock–out (con gran variedad de trasfondos genéticos) y acceder a su interacción con factores de transcripción críticos como dHAND, NKX2.5, GATA4 y TBX. Algunas aproximaciones metodológicas semejantes pueden ser usadas para elucidar el sistema de conducción cardíaco y para descifrar el papel de los sistemas de señalización que participan en la formación vascular de células endoteliales, enfocándose en la interacción del VEGF, angiopoyetina, TGF y la ruta Notch.
3) Otra área crítica en la investigación cardiológica es la identificación de los reguladores moleculares que controlan la proliferación de los cardiomiocitos. Dichas células son mitóticamente activas durante la embriogénesis y generalmente cesa su proliferación antes del nacimiento. El estudio de las bases moleculares de la proliferación de los cardiomiocitos podría impactar positivamente en los intentos clínicos por reparar el tejido cardíaco dañado. Los mecanismos de regulación del crecimiento celular pueden ser investigados haciendo cuidadosas comparaciones entre los perfiles de expresión génica de miocitos embrionarios y de miocitos postnatales, así como por generación de cultivos de líneas celulares capaces de responder a estímulos inductores de proliferación. Alternativamente, el trasplante celular es un mecanismo utilizado para aumentar el número de miocitos en corazones dañados o isquémicos. Interesantemente, algunos estudios recientes han mostrado que una subpoblación de células troncales cardíacas de adulto inyectadas en un corazón isquémico fueron capaces de reconstituir completamente al miocardio diferenciado, formando tanto miocitos como vasos sanguíneos.79 De tal suerte, se deben realizar grandes esfuerzos en la investigación para definir las condiciones óptimas necesarias para la diferenciación y proliferación de los cardiomiocitos y para su completa integración funcional como células troncales en el miocardio, así como investigar la habilidad de las células troncales para reparar el tejido cardíaco en los niños con cardiomiopatía (por ejemplo DCM), enfermedad de Kawasaki con daño miocardico y ARVD, los cuales pueden ser ayudados por trasplante de células troncales. La visión dentro de las consecuencias cardiovasculares por una función genética anormal y su expresión, puede impactar sobre el desarrollo de estrategias terapéuticas dirigidas y manejo de enfermedades para los niños que presentan desórdenes cardíacos congénitos y adquiridos y quizá reemplacen a tratamientos menos efectivos dirigidos únicamente a rectificar los defectos cardíacos estructurales con mejoras funcionales temporales.
Apéndice:
Enzima convertidora de angiotensina (ACE); cardiomiopatía dilatada autosómica dominante (AD–DCM); fibrilación atrial familiar (AF); enzima desdobladora de glucógeno (AGL); apolipoproteína E (APOE); displasia ventricular derecha arritmogénica (ARVD); defecto septal atrioventricular (AVSD); α–glucosidasa (α–GLU); cadena pesada de la α–miosina (α–MHC); α–tropomiosina (α–TM); receptor β–adrenérgico (β–ADR); cadena pesada de la β–miosina (β–MHC); carnitin–acylcarnitin traslocasa (CACT); carnitil–palmytoil transferasa II (CPTII); δ–sarcoglicano (δ–SGC); cardiomiopatía dilatada (DCM); proteína cinasa de la miotonina (DMPK); ataxia de Friedreich (FRDA); α–galactosidasa (α–GLA); cardiomiopatía hipertrófica (HCM); síndrome humano relacionado al ether–a–go–go (HERG); síndrome de Holt–Oram (HOS); jagged–1 (JAG1); miembro 1 de la subfamilia del canal de potasio dependiente de voltaje, relacionado a Isk (KCNE1); miembro 2 de la subfamilia del canal de potasio dependiente de voltaje, relacionado a Isk (KCNE2); canal 1 de potasio dependiente de voltaje del síndrome de QT largo (KVLQT1); síndrome de QT largo (LQT); distrofia muscular miotónica (MMD); metaloproteinasa de matriz 9 (MMP9); proteína trifuncional mitocondrial (MTP); proteína C de unión a miosina (MYBPC); cadena ligera de la miosina ventricular regulatoria (MYL2); cadena ligera de la miosina ventricular esencial (MYL3); catión orgánico del transportador carnitina 2 (OCTN2); síndrome de Osler–Rendu–Weber (ORW); síndrome de Char–ductus arteriosus patente (PDA); hipertensión pulmonar primaria (PPH); proteína cinasa gamma 2 activadora de AMP (PPKAg2); proteína tiro sin fosfatasa (PTPN11); receptor de rianodina 2 (RYR2); hipoplasia ventricular derecha (RVH); ensamblaje de la citocromo C oxidasa (SCO2); polipéptido α del canal de sodio dependiente de voltaje tipo V (SCN5A); estenosis aórtica supravalvular (SVAS); factor de transcripción T–box 1 (TBXl); factor de transcripción T–box 5 (TBX5); factor de transcripción de la familia AP–2 (TFAP2B); troponina cardíaca T (TNT2); troponina cardíaca I (TNN13); síndrome DiGeorge/velocardiofacial (VCFS/DG); taquicardia ventricular (VT); síndrome de Wolf–Parkinson–White (WPW); cardiomiopatía dilatada ligada al X (X–DCM).
Referencias
1. American Heart Association. Heart disease and stroke statistics. 2004 Update. (American Heart Association, Dallas, Texas, USA, 2003). [ Links ]
2. Joffman JI: Incidence of congenital heart disease: Prenatal incidence. Pediatr Cardiol 1995; 16: 155–165. [ Links ]
3. Hunter D J: Gene–environment interactions inhuman diseases. Nat Rev 2005; 6: 287–298. [ Links ]
4. Benson DW: Advances in cardiovascular genetics and embryology: role of transcription factors in congenital heart disease. Curr Opin Pediatr; 2000; 12: 497–500. [ Links ]
5. Srivastava D: HAND proteins: molecular mediators of cardiac development and congenital heart disease. Trends Cardiovasc Med 1999; 9: 8–11. [ Links ]
6. Benson DW, Silberbach GM, Kavanaugh–McHugh A, Cottrill C, Zhang Y, Riggs S, et al: Mutations in the cardiac transcription factor NKX2.5 affect diverse cardiac development pathways. J Clin Invest 1999; 104: 1567–1573. [ Links ]
7. Garg V, Kathirya IS, Barnes R, Schluterman MK, King IN, Butler CA, et al: GATA4 mutations cause human congenital heart defects and reveal an interaction with TBX5. Nature 2003; 424: 443–447. [ Links ]
8. Schott JJ, Benson DW, Basson CT, Pease W, Silberbach GM, Moak JP, et al. Congenital heart disease caused by mutations in the transcription factor NKX2–5. Science 1998; 281: 108–111. [ Links ]
9. Jay PY, Berul CI, Tanaka M, Ishii M, Kurachi Y, Izumo S: Cardiac conduction and arrhythmia: insights from Nkx2:5 mutations in mouse and humans. Novartis Found Symp 2003; 250: 227–238. [ Links ]
10. Zhao F, Weismann CG, Satoda M, Pierpont ME, Sweeney E, Thompson EM, et al: Novel TFAP2B mutations that cause Char syndrome provide a genotype–phenotype correlation. Am J Hum Genet 2001; 69: 695–703. [ Links ]
11. Bruneau BG, Nemer G, Schmit JP, Charron F, Robitaille L, Caron S, et al: A murine model of Holt–Oram syndrome defines roles of the T–box transcription factor Tbx5 in cardio genesis and disease. Cell 2001; 106: 709–721. [ Links ]
12. KrantzID, Piccoli DA, Spinner NB: Clinical and molecular genetics ofAlagille syndrome. CurrOpin Pediatr 1999; 11: 558–564. [ Links ]
13. Tartaglia M, Mehler EL, Goldberg R, Zampino G, Brunner HG, Kremer H, et al: Mutations in PTPN11, encoding the protein tyrosine phos–phatase SHP–2, cause Noonan syndrome. Nat Genet 2001; 29: 465–468. [ Links ]
14. Legius E, Schrander–Stumpel C, Schollen E, Pulles–Heintzberger C, Gewillig M, Fryns JP: PTPN11 mutations in LEOPARD syndrome. J Med Genet 2002; 39: 571–574. [ Links ]
15. Palau F: Friendreich 's ataxia andfrataxin: molecular genetics, evolution andpathogenesis. Int J Mol Med 2001; 7: 581–589. [ Links ]
16. Korade–Mrnics Z, Tarleton J, Servidei S, Casey RR, Gennarelli M, Pegoraro E, et al: Myotonic dystrophy: tissue–specific effect of somatic CTG expansions on allele–specific DMAHP/SIX5 expression. Hum Mol Genet 1999; 8: 1017–1023. [ Links ]
17. Strauss AW: The molecular basis of congenital cardiac disease. Semin Thorac Cardiovasc Surg Pediatr Card Surg Annu 1998; 1: 179–188. [ Links ]
18. Marino B, Digilio MC: Congenital Heart disease and genetic syndromes: specific correlation between cardiac phenotype and genotype. Cardiovasc Pathol 2000; 9: 303–315. [ Links ]
19. Chieffo C, Garvey N, Gong W, Roe B, Zhang G, Silver L, et al: Isolation and characterization of a gene from the DiGeorge chromosomal region homologous to the mouse Tbxl gene. Genomics 1997; 43: 267–277. [ Links ]
20. Giglio S, Graw SL, Gimelli G, Piróla B, Varone P, Voullaire L, et al: Deletion of a 5–cM region at chromosome 8p23 is associated with a spectrum of congenital heart defects. Circulation 2000; 102: 432–437. [ Links ]
21. Wang DW, Yazawa K, George AL Jr, Bennett PB: Characterization o human cardiac Na+ channel mutations in the congenital long QT syndrome. Proc Nati Acad Sci USA 1996; 93: 13200–13205. [ Links ]
22. Towbin JA, Wang Z, Li H: Genotype and severity of long QT syndrome. Drug Metab Dispos 2001; 29: 574–579. [ Links ]
23. Chen Q, Kirsch GE, Zhang D, Brugada R, Bru–gada J, Brugada P, et al: Genetics basis and molecular mechanism for idiopathic ventricular fibrillation. Nature 1998; 392: 293–296. [ Links ]
24. Bezzina C, Veldkamp MW, van Den Berg MP, Post–ma AV, Rook MB, Viersma JW, et al: A single Na (+) channel mutation causing both long–QT and Brugada syndromes. Circ Res 1999; 85:1206–1213. [ Links ]
25. Splawski I, Shen J, Timothy KW, Lehmann MH, Priori S, Robinson JL, et al: Spectrum of mutations in long–QT syndrome genes. KVLQT1, HERG, SCN5A, KCNE1, and KCNE2. Circulation 2000; 102: 1178–1185. [ Links ]
26. Laitinen PJ, Brown KM, Piippo K, Swan H, Devaney JM, Brahmbhatt B, et al: Mutations of the cardiac ryanodine receptor (RyR2) gene in familial polymorphic ventricular tachycardia. Circulation 2001; 103: 85–90. [ Links ]
27. Bonne G, Di Barletta MR, Varnous S, Becane HM, Hammouda EH, Merlini L, et al. Mutations in the gene encoding laminin A/C cause autosomal dominant Emery–Dreifuss muscular dystrophy. Nat Genet 1999; 21: 285–288. [ Links ]
28. Bonnet D, Martin D, de Lonlay P, Villain E, Jouvet P, Rabier D, et al: Arrhythmias and conduction defects as presenting symptoms of fatty acid oxidation disorders in children. Circulation 1999; 100: 2248–2253. [ Links ]
29. Dietz HC, Cutting GR, Pyeritz RE, Maslen CL, Sakai LY, Corson GM, et al: Marfan syndrome caused by a recurrent de novo missense mutation in the fibrillin gene. Nature 1991; 352: 337–339. [ Links ]
30. Ewart AK, Morris CA, Atkinson D, Jin W, Sternes K, Spallone P, et al: Hemizygosity at the elastin locus in a developmental disorder, Williams syndrome. Nat Genet 1993; 5: 11–16. [ Links ]
31. Bonne G, Carrier L, Bercovici J, Cruaud C, Richard P, Hainque B, et al: Cardiac myosin binding protein–C gene splice acceptor site mutation is associated with familial hypertrophic cardiomyopathy. Nat Genet 1995; 11: 438–440. [ Links ]
32. Thierfelder L, Watkins H, MacRae C, Lamas R, McKenna W, Vosberg HP, et al: Alpha–tropomyosin and cardiac troponin T mutations cause familial hyprtrophic cardiomyopathy: a disease of the sarcomere. Cell 1994; 77: 701–712. [ Links ]
33. Kimura A, Harada H, Parks JE, Nishi H, Satoh M, Takahashi M, et al: Mutations in the cardiac troponin I gene associated with hypertrophic car–diomyophaty. Nat Gene 1997; 16: 379–382. [ Links ]
34. Anan R, Greve G, Thierfelder L, Watkins H, McKenna WJ, Solomon S, et al: Prognostic implications of novel beta cardiac myosin heavy chain gene mutations that cause familial hypertrophic cardiomyophaty J Clin Invest 1994; 93: 280–285. [ Links ]
35. Olson TM, Doan TP, Kishimoto NY, Whitby FG, Ackerman MJ, Fananapazir L: Inherited and de novo mutations in the cardiac actin gene cause hypertrophic cardiomyopathy. J Mol Cell Cardiol 2000; 32: 1687–1694. [ Links ]
36. Satoh M, Takahashi M, Sakamoto T, Hiroe M, Marumo F, Kimura A. Structural analysis of the titin gene in hypertrophic cardiomyopathy: identification of a novel disease gene. Biochem Biophys Res Commun 1999; 262: 411–417. [ Links ]
37. Antonicka H, MattmanA, Carlson CG, Glerum DM, Hoffbuhr KC, Leary SC, et al: Mutations in COX15 produce a defect in the mitochondrial heme biosynthetic pathway, causing early–onset fatal hypertrophic cardiomyopathy. Am J Hum Genet 2003; 72: 102–114. [ Links ]
38. Marín–García J, Goldenthal MJ: La mitocondria y elcorazón. Rev Esp Cardiol 2002; 55:1293–1310. [ Links ]
39. Gollob MH, Green MS, Tang AS, Gollob T, Karibe A, All Hassan AS, et al: Identification of a gene responsible for familial Wolff–Parkinson–White syndrome. N Engl J Med 2001; 344: 1823–1831. [ Links ]
40. Beggs AH: Dystrophinopathy, the expanding phe–notype. Dystrophin abnormalities in X–linked dilated cardiomyopathy. Circulation 1997; 95: 2344–2347. [ Links ]
41. D'Adamo P, Fassone L, Gedeon A, Janssen EA, Bione S, Bolhuis PA, et al. The X–linked gene G4.5 is responsible for different infantile dilated cardiomyopathies. Am J Hum Genet 1997; 61 (4): 862–867. [ Links ]
42. Olson TM, Mchels VV, Thibodeau SN, Tai YS, Keating MT: Actin mutations in dilated cardiomyopathy, a heritable from of heart failure. Science 1998; 280: 750–752. [ Links ]
43. Dalakas MC, Park KY, Semino–Mora C, Lee HS, Sivakumar K, Goldfarb LG: Desmin myopathy, a skeletal myopathy whit cardiomyopathy caused by mutations in the desmin gene. N Engl J Med 2000; 342: 770–780. [ Links ]
44. Tsubata S, Bowles KR, Vatta M, Zintz C, Titus J, Muhonen L, et al: Mutations in the human delta–sarcoglycan gene in familial and sporadic dilated cardiomyopathy. J Clin Invest 2000; 106: 655–662. [ Links ]
45. Towbin JA, Hejtmancik JF, Brink P, Gelb B, Zhu XM, Chamberlain JS, et al : X–linked dilated cardiomyopathy. Molecular genetic evidence of linkage to the Duchenne muscular dystrophy (dystrophin) gene at the Xp21 locus. Circulation 1993; 87: 1854–1865. [ Links ]
46. Neuwald AF: Barth syndrome may be due to an acyltransferase deficiency. Curr Biol 1997; 7: 4665–4666. [ Links ]
47. Rampazzo A, Beffagna G, Nava A, Occhi G, Bauce B, Noiato M, et al. Arrhythmogenic right ventricula cardiomyopathy type 1 (ARVD1): confirmation of locus assignment and mutation screening of four candidate genes. Eur J Hum Genet 2003; 11: 69–76. [ Links ]
48. Wallace DC: Disease of mitochondrial DNA. Ann Rev Biochem l992; 61: 1175–1212. [ Links ]
49. Tanaka M, Ino H, Ohno K: Mitochondrial mutation in fatal infantile cardiomyopathy. Lancet 1990; 336: 1452. [ Links ]
50. Taniike M, Fukushima H, Yanagihara I, Tsukamo–to H, Tanaka J, Fujimura H, et al: Mitochondrial tRNA Ile mutation in fatal cardiomyopathy. Biochem Biophys Res Commun 1992; 186: 47–53. [ Links ]
51. Silvestri G, Santorelli FM, Shanske S, Whitley CB, Schimmenti LA, Smith SA, et al: A new mtD–NA mutation in the tRNA LEU(UUR) gene associated with maternally inherited cardiomyopathy. Hum Mutat l994;3: 37–43. [ Links ]
52. Marín–García J, Goldenthal MJ, Ananthakris–hanan R, Pierpont ME: The complete sequence of mtDNA genes in idiopathic dilated cardiomyopathy shows novel missense and tRNA mutations. J Card Fail 2000; 6: 321–329. [ Links ]
53. Marín–García J, Ananthakrishanan R, Goldenthal MJ, Pierpont ME: Biochemical and molecular basis for mitochondrial cardiomyopathy in neonates and children. J Inhreit Metab Dis 2000; 23: 625–633. [ Links ]
54. Cortes–Hernández P, García–Trejo JJ: Cardiomiopatías mitocondriales En: "Biología Molecular y Corazón". Cap. 3 de "Tratado de Cardiología de la Sociedad Mexicana de Cardiología". Ed. Intersistemas. 2006. pp 107–113. ISBN 970–655–860–8. [ Links ]
55. Serranno J, Palmeira CM, Kuehl DW, Wallace KB: Cardioselective and cumulative oxidation of mitochondrial DNA following subchronic doxo–rubicin administration. Biochim Biophys Acta 1999; 1411: 201–205. [ Links ]
56. Schoppet M, Maisch B: Alcohol and the heart. Herz 2001; 26: 345–352. [ Links ]
57. Corral–Debrinski M, Shoffner JM, Lott MT, Wallace DC: Association ofmitochondrialDNA damage with aging and coronary atherosclerotic heart disease. MutatRes 1992; 275: 169–180. [ Links ]
58. Marín–García J, Goldenthal MJ, Ananthakrishanan R, Pierpont ME, Fricker FJ, Lipshultz SE, et al: Specific mitochondrialDNA deletions in idiopathic dilated cardiomyopathy. Cardiovasc Res 1996; 31: 306–313. [ Links ]
59. Li YY, Hengstenberg C, Maish B: Whole mitochondrial genome amplification reveals basal level multiple deletions in mtDNA of patients with dilated cardiomyopathy. Biochem Biophys Res Commun 1995; 210: 211–218. [ Links ]
60. Holt IJ, Harding AE, Morgan–Hughes JA: Deletions of mtDNA in patients with mitochondrial myopathies. Nature 1988; 331: 717–719. [ Links ]
61. Carrozzo R, Hirano M, Fromenty B, Casali C, Santorelli FM, Bonilla E, et al: Multiple mtDNA deletions features in autosomal dominant and recessive disease suggest distinct pathogenesis. Neurology 1998; 50: 99–106. [ Links ]
62. Zeviani M, Spinazzola A, Carelli V: Nuclear genes in mitochondrial disorders. Curr Opin Genet Dev2003; 13: 262–270. [ Links ]
63. Lewis W, Dalakas MC: Mitochondrial toxicity of antiviral drugs. Nature Med 1995; 1: 1417–1422. [ Links ]
64. Lipshultz SE, Easley KA, Orav EJ, Kaplan S, Stare TJ, Bricker JT, et al: Absence of cardiac toxicity ofzidovudine I infants. Pediatric Pulmonary and Cardiac Complications of Vertically Transmitted HIV Infection Study Group. N Engl J Med 2000; 343: 759–766. [ Links ]
65. Hiroi Y, Kudoh S, Monzen K, IkedaY, Yazaki Y, Nagai R, et al: Tbx5 associates with Nkx2–5 and synergistically promotes cardiomyocyte differentiation. Nat Genet 2001; 28: 276–280. [ Links ]
66. Wang J, Wilhelmsson H, Graff C, Li H, Oldfors A, Rustin P, et al: Dilated cardiomyopathy and atrioventricular conduction blocks induced by heart–specific inactivation of mitochondrial DNA gene expression. Nat Genet 1999; 21: 133–137. [ Links ]
67. Ibdah JA, Paul H, Zhao Y, Binford S, Salleng K, Cline M, et al: Lack of mitochondrial trifunctional protein in mice causes neonatal hypoglycemia and sudden death. J Clin Invest 2001; 107: 1403–1409. [ Links ]
68. Puccio H, Simon D, Cosse M, Criqui–Filipe P, Ti–ziano F, Melki J, et al: Mouse models for Friedreich ataxia exhibit cardiomyopathy, sensory nerve defect and Fe–S enzyme deficiency followed by intramitochondrial iron deposit. Nat Genet 2001; 27: 181–186. [ Links ]
69. Merscher S, Funke B, Epstein JA, Heyer J, Puech A, Lu MM, et al: TBX1 is responsible for cardiovascular defects in velocardio–facial/DiGeorge syndrome. Cell 2001; 104: 619–629. [ Links ]
70. Singh GK: Kawasaki disease: an update. Indian J Pediatr 1998; 65: 231–241. [ Links ]
71. Bowles NE, Ni J, Kearney DL, Pauschinger M, Schultheiss HP, McCarthy R, et al: Detection of virus in myocardial tissues by polymerase chain reaction: evidence of adenovirus as a common cause ofmyocarditis in children and adults. J Am Coll Cardiol 2003; 42: 466–472. [ Links ]
72. Cea–Calvo L, Escribano Subias P, Tello de Menesses R, Lázaro Salvador M, Gómez Sánchez MA, Delgado Jiménez JF, et al: Tratamiento de la hipertensión pulmonar asociada a la infección por VIH con trerpostinil. Rev Esp Cardiol 2003; 56: 421–425. [ Links ]
73. Daley GQ, Cargill M: The heart SNPs a beat:polymorphisms in candidate genes for cardiovascular disease. Trends Cardiovasc Med 2001; 11: 60–66. [ Links ]
74. O'Rourke B: Myocardial KATP channels in preconditioning. Circulation Res 2000; 87: 845–855. [ Links ]
75. Raflee P, Shi Y, Kong X, Pritchard KA Jr, Twweddell JS, Litwin SB, et al: Activation of protein kinases in chronically hypoxic infant human and rabbit hearts: role in cardioprotection. Circulation 2002; 106: 239–245. [ Links ]
76. Ning XH, Xu CS, Song YC, Xiao Y, Hu YJ, Lupinetti FM, et al: Hypothermia preservesfunction and signaling for mitochondrial biogenesis during subsequent ischemia. Am J Physiol 1998; 274: H786–H793. [ Links ]
77. Mosquera Pérez I, Rueda Núñez F, Medrano López C, Pórtela Tórron F, Zavanella Botta C, Castro Beiras A: Tratamiento mediante hipotermia de la taquicardia ectópica de la unión tras cirugía cardíaca infantil. Rev Esp Cardiol 2003; 56: 510–514. [ Links ]
78. Sanguinetti MC, Bennett PB: Anti–arrhythmic drug target choices and screening. Circ Res 2003; 93:491–499. [ Links ]
79. Beltrami AP, Barlucchi L, Torella D, Baker M, Limana F, Climenti S, et al: Adult cardiac item cells are multipotent and support myocardial regeneration. Cell 2003; 114: 763–776. [ Links ]














