Introducción
Cinasas y fosfatasas
La mayor parte de los procesos celulares indis-pensables para la vida, se regulan a través de mecanismos de fosforilación y desfosforilación que se pueden llevar a cabo en los azúcares, las proteínas y los lípidos. A continuación, se describe la acción de las fosfatasas de proteína involucradas en el cáncer.
En las proteínas, el ciclo de fosforilación-desfosforilación depende principalmente de dos tipos de enzimas: las cinasas y las fosfatasas. Las cinasas fosforilan a las proteínas, es decir, son capaces de catalizar la transferencia de grupos fosforilo desde un nucleósido trifosfatado, que comúnmente es el ATP, hacia aminoácidos hidroxilados que forman parte de una proteína (serina, treonina y tirosina); por lo tanto, a las cinasas también se les denomina fosfotransferasas. La adición del grupo fosfato introduce tres cargas negativas en el residuo del aminoácido sobre el que se inserta el fosfato. Cuando esto ocurre, la conformación de las proteínas cambia y quedan al descubierto sitios previamente ocultos, que permiten la interacción con otras proteínas. Esto da lugar a cambios en su comportamiento biológico como: el aumento o disminución de su actividad catalítica, localización subcelular, asociación con otras proteínas, sensibilidad a los ligandos, etc. Estos cambios en el estado de fosforilación de las proteínas a su vez, promueven procesos tan importantes como: la reorganización del citoesqueleto, la activación y regulación de la migración celular, y la diferenciación celular, entre otros (Fernández, 2012; González, 2000; Hanks & Hunter, 1995).
Por otro lado, se encuentran las fosfatasas, estas enzimas son la contraparte de las cinasas (Fernández, 2012). Las fosfatasas catalizan la hidrólisis de ésteres de fosfato, liberan el ion fosfato y en su lugar dejan un grupo hidroxilo. El ciclo de fosforilación de las proteínas, involucra tanto la fosforilación como la desfosforilación, y representa la modificación post-transduccional más común y que mejor se ha estudiado, pues se estima que aproximadamente el 30% de las proteínas se regula por este mecanismo (Khalife, Freville, Vandomme & Pierrot, 2020). Tanto las cinasas como las fosfatasas actúan como interruptores en diversos procesos celulares, ya que se regulan entre sí con un adecuado control y balance que conlleva al correcto funcionamiento de las células.
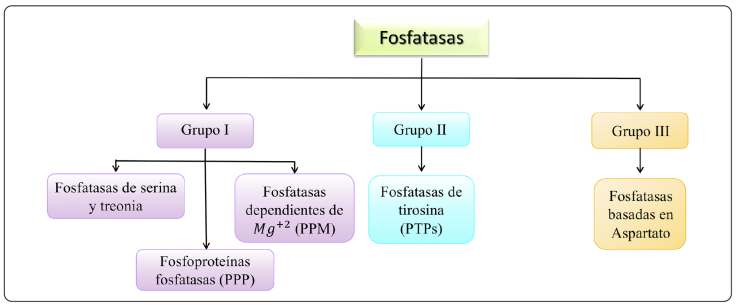
De forma general, las fosfatasas se clasifican en tres grandes grupos de acuerdo a su estructura, secuencia y mecanismo catalítico: en el primero están las fosfatasas que actúan sobre residuos de serina y treonina (Ser/Thr), la gran familia de las fosfoproteína fosfatasas (PPP) y las proteínas fosfatasas dependientes de Mg+2 (PPM); en el segundo la superfamilia de las proteínas fosfatasas de tirosina (PTPs); y en el último las fosfatasas cuya base es el aspartato en la secuencia DXDXT/V en su dominio catalítico (Figura 1). Es importante mencionar que a nivel genético, estos grupos no comparten similitudes ni en su estructura, ni en su secuencia (Larsen, Tremblay & Yamada, 2003; Brautigan & Shenolikar, 2018; Moorhead, Trinkle-Mulcahy & Ulke-Lemée, 2007).
La familia de las fosfatasas de tirosina (PTPs)
La superfamilia de las fosfatasas de tirosina, es un grupo que incluye a la proteína PTP-PEST de interés en esta revisión; se caracteriza por ser el que contiene a más proteínas, presentes en bacterias, levaduras, insectos y vertebrados. Esta familia está constituida por 107 PTPs codificadas en el genoma humano. Se distinguen por el motivo HCR que define su sitio activo, en el que un residuo de cisteína funciona como nucleófilo y es esencial para la actividad catalítica de estas enzimas (Alonso et al., 2004; Kuban-Jankowska, Gorska, Knap, Cappello & Wozniak, 2015; Östman, Hellberg & Böhmer, 2006; Tonks, 2006). Debido a su extensión, esta superfamilia, se ha divido en cuatro subgrupos o clases de acuerdo con la estructura primaria del dominio catalítico (Figura 2): La clase I comprende 38 fosfatasas específicas de residuos de tirosina, también conocidas como clásicas, y 61 duales específicas, es decir, que actúan sobre residuos de tirosina y serina/treonina o sobre residuos de tirosina y treonina (Hendriks, Elson, Harroch & Stoker, 2008; Kuban-Jankowska et al., 2015). La clase II está representada por un único gen que codifica para una fosfatasa de bajo peso molecular (18 kDa) específica de residuos de tirosina (LMWPTP) (Kuban-Jankowska et al., 2015), cuya maquinaria catalítica, aunque es muy similar a las enzimas de la clase I, no está relacionada estructuralmente (Bordo & Bork, 2002; Mustelin, 2007). Por su parte, la clase III está compuesta por tres fosfatasas de treonina/tirosina que participan en la regulación del ciclo celular; y finalmente, la clase IV está formada por cuatro fosfatasas de tirosina y serina/tirosina, que se diferencian de las otras tres por tener un ácido aspártico en el sitio catalítico, en lugar del residuo de cisteína común en las otras clases (Alonso et al., 2004; Kuban-Jankowska et al., 2015; Rayapureddi et al., 2003).

Figura, creatividad personal.
Figura 2 Clasificación general de las fosfatasas de tirosina (PTPs). La marca roja indica la ubicación de la PTP-PEST en la clasificación.
El subgrupo de las PTPs de clase I, se divide a su vez en dos grupos (Paul & Lombroso, 2003; Stoker, 2005): Las que atraviesan la membrana plasmática se llaman PTP-receptoras (RPTP); mientras que las que se ubican de manera intracelular en compartimentos como el citosol o el retículo endoplásmico, se conocen como PTP-no receptoras (nR-PTP) (Stoker, 2005). Las RPTP pueden regular la señalización mediante la desfosforilación de residuos de tirosina de proteínas controladas por ligando (Tonks, 2006). Están formadas por dos dominios PTP citoplásmicos, un dominio proximal de membrana llamado D1 y un dominio distal de membrana llamado D2; también tienen un segmento transmembranal y un dominio extracelular (Andersen et al., 2001). Por su parte, las nR-PTP se caracterizan por tener secuencias reguladoras que flanquean su dominio catalítico y controlan su actividad; ya sea directamente, al interaccionar con este sitio, o indirectamente al influir en la especificidad del sustrato. Estas secuencias no catalíticas también controlan la distribución subcelular y restringen el acceso a sustratos particulares en dichas ubicaciones, con lo que indirectamente las regulan (Pulido, 1998; Tonks, 2006).
Dentro del subgrupo de las PTP-no receptoras se encuentra una familia llamada PEST, recibe este nombre debido a que las proteínas que la integran son fosfatasas ricas en residuos de prolina, ácido glutámico, serina y treonina (PEST). Los miembros de esta familia son tres fosfatasas intracelulares que poseen una organización estructural común, que consta de un dominio de fosfatasa amino terminal, una región central altamente divergente que contiene motivos ricos en prolina y sitios de fosforilación en tirosina que permiten su interacción con otras proteínas, y una cola carboxilo-terminal conocida como dominio de homología carboxilo- terminal (CTH) (Veillette, Rhee, Souza & Davidson, 2009). Los tres miembros de esta familia son: La fosfatasa enriquecida con prolina (PEP), la fosfatasa de tirosina linfoide (LYP), también conocida como PTPN22 o PTPN8, y la protagonista de esta revisión, PTP-PEST, también llamada PTPN12 (Figura 2) (Veillette et al., 2009).
Características estructurales de la proteína PTP-PEST
La proteína PTP- PEST tiene en su extremo C-terminal secuencias ricas en prolina, ácido glutámico, serina y treonina, de ahí la abreviatura PEST. Está codificada por el gen PTPN12 y debido a ello es también conocida por este nombre (Takekawa et al., 1992; Yang, Sommercorn & Tonks, 1993; Zheng & Lu, 2013). En humanos, el gen PTPN12 se ha localizado en el cromosoma 7 en la región q11.23. Esta proteína está compuesta por aproximadamente 780 aminoácidos y tiene un peso molecular de 112 kDa; se expresa de forma ubicua en diferentes tipos de tejidos, aunque se ha visto principalmente acumulada en órganos como el bazo, el hígado y el timo, y se puede encontrar tanto en células hematopoyéticas como en células no hematopoyéticas (Lee & Rhee, 2019; Sirois et al., 2006; Veillette et al., 2009; Zheng & Lu, 2013).
La PTP-PEST contiene entre 50 y 60 aminoácidos en su región N-terminal, seguida de un dominio catalítico PTP de aproximadamente 240 aminoácidos, en donde los residuos Cys231 y Arg237 son particularmente importantes (Davidson, 2001; Lee & Rhee, 2019; Veillette et al., 2009). La cola C-terminal está compuesta por aproximadamente 500 aminoácidos, contiene secuencias PEST y cuatro dominios ricos en prolina nombrados P1, P2, P3 y P4, así como, una secuencia NPLH (asparagina, prolina, leucina, histidina) que es un sitio de anclaje para proteínas involucradas en la transducción de señales (Davidson, 2001; Lee & Rhee, 2019; Veillette et al., 2009). Cabe recalcar que la actividad catalítica de esta fosfatasa, está regulada por un sitio de fosforilación específico en la Ser39 que es esencial para la actividad de esta proteína (Figura 3) (Lee & Rhee, 2019; Nakamura, Palmer, Ozawa & Mashima, 2010; Yaffe & Smerdon, 2001).
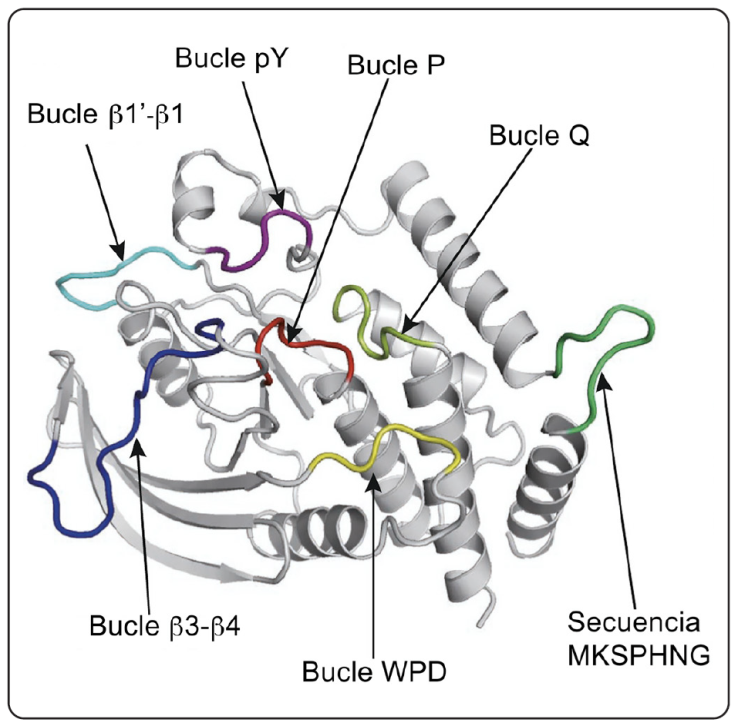
De acuerdo con estudios de cristalografía, la estructura tridimensional de la PTP-PEST (Figura 4) se asemeja a la de las PTP clásicas, pues presenta hojas β centrales flanqueadas por hélices α en cada lado. Dentro de estas estructuras se encuentra el bucle P, que constituye el sitio activo, rodeado de varios bucles que contribuyen con residuos importantes para la formación del sitio catalítico. Los bucles WPD y Q, ubicados a lo largo del bucle P, contienen los residuos catalíticos D19, Q279 y Q282. Flanqueando también el bucle P, pero en sentido contrario a los anteriormente mencionados, se encuentran los bucles pY, β1´-β1 y β3-β4 que contribuyen al reconocimiento de los sustratos (Li et al., 2016). La PTPN12 se diferencia de los otros dos miembros de la familia PEST porque presenta dominios únicos como el bucle WPD, los bucles β1-4 y la hélice α1 (Lee & Rhee, 2019; Li et al., 2016). Dentro del sitio catalítico también se encuentra un enlazador específico con la secuencia MKSPDHNG, que conecta a dos hélices α. Este bucle le permite a la PTP-PEST reconocer a las cinasas dependientes de ciclina (Lee & Rhee, 2019; Li et al., 2016).
PTP-PEST y su interacción con las adhesiones focales
La PTP- PEST está involucrada en diferentes procesos celulares entre los que destaca la migración celular, pues es aquí donde la participación de esta fosfatasa ha sido mejor estudiada, aunque su papel no ha sido completamente elucidado (Davidson, 2001).
Como se sabe, la migración celular es un proceso cíclico, dinámico, altamente coordinado y que involucra múltiples pasos, entre los que se encuentran el ensamble y el desensamble de las estructuras conocidas como adhesiones focales en los bordes delantero y trasero de la célula. En este proceso, la polarización y protrusión del borde delantero, es seguido de la formación y estabilización de la adhesión célula-sustrato, la contracción del citoesqueleto para empujar el cuerpo de la célula hacia adelante, y finalmente, el desensamble de las adhesiones en la región posterior de la célula, da por resultado la retracción del segmento posterior de la célula (Frame, Fincham, Carragher & Wyke, 2002; Larsen et al., 2003; Ridley et al., 2003; Zheng & Lu, 2013).
Las adhesiones focales, son el punto de interacción entre la matriz extracelular (ECM), proteínas de la familia de las integrinas, y el citoesqueleto (Figura 5). Estas estructuras representan los principales sitios de fosforilación y desfosforilación de tirosina, y son una de las estructuras más dinámicas de la célula (Shen, Schneider, Cloutier, Veillette & Schaller, 1998; Zheng & Lu, 2013). La regulación del ensamble y el desensamble de estas estructuras es tan importante, que un desequilibrio inhibe la migración celular (Zheng & Lu, 2013). En la coordinación de la fosforilación y desfosforilación de estas estructuras, están involucradas proteínas como FAK, Src, PTP-PEST y Pyk2, por citar algunos ejemplos (Zheng & Lu, 2013).
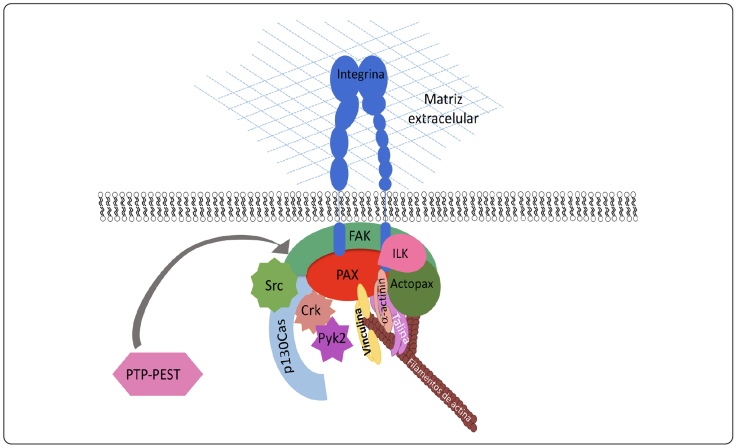
Modificado de (López-Colomé, Lee-Rivera, Benavides-Hidalgo & López, 2017).
Figura 5 Representación esquemática de la organización de las adhesiones focales donde se muestra la participación de diversas proteínas, entre ellas, Fak, Pyk2, Paxilina (PAX), p130Cas, Src, CrkII y PTP-PEST.
El ensamble de las adhesiones focales se inicia con la activación de una integrina por un ligando extracelular (Wehrle-Haller, 2012). Una vez activa, recluta a la proteína adaptadora Talina; que, a su vez, recluta a la proteína adaptadora Vinculina (Humphries et al., 2007; Wehrle-Haller, 2012). La Talina y la Vinculina forman un complejo con FAK, que se autofosforila en el sitio Y397, lo que permite la unión de la proteína Src a su dominio (SH2) (Wehrle-Haller, 2012; Zheng & Lu, 2013). Una vez activadas, FAK y Src fosforilan e incorporan al complejo a las proteínas adaptadoras p130Cas y Paxilina. Estas últimas reclutan a la proteína CrkII y a Pyk2 (Lukic et al., n.d.; Zheng & Lu, 2013). La proteína CrkII activa a la vía de Rac1 para la incorporación de proteínas adicionales a la adhesión focal (Guo & Giancotti, 2004). Es importante hacer notar la importancia de la fosforilación en el reclutamiento y la estabilización de las adhesiones focales. Por otra parte, el desensamble de estas estructuras se da a través de diversos mecanismos (Wehrle-Haller, 2012), en los que la PTP-PEST tiene un papel importante al desfosforilar a proteínas clave en la formación de estas adhesiones, como son FAK, p130Cas y Pyk2 (Angers-Lostau et al., 1999; Côte, Charest, Wagner & Tremblay, 1998; Eleniste, Du, Shivanna & Bruzzaniti, 2012; Garton, Flint & Tonks, 1996; Garton, Burnham, Bouton & Tonks, 1997; Garton & Tonks, 1999; Shen et al., 2000; Zheng & Lu, 2013), Figura 5).
En estudios realizados en fibroblastos deficientes en PTP-PEST, se observó que esta fosfatasa participa en la migración, pues estas células presentan una gran diminución en su movilidad, comparadas con células silvestres, así como niveles elevados de fosforilación en tirosina, en proteínas clave de las adhesiones focales como p130Cas, FAK y Paxilina (Angers-Loustau et al., 1999; Côté et al., 1998; Zheng & Lu, 2013). Adicionalmente, se ha observado que la PTP-PEST, recluta a la dinamina, un motor molecular principalmente involucrado en la endocitosis. La desfosforilación de la dinamina por la PTP-PEST, disminuye su actividad como GTPasa, y promueve su interacción con el dominio FERM de Pyk2, lo que desencadena su desfosforilación. Esto lleva a la inactivación de las cascadas de señalización de Pyk2 y al desacoplamiento de las integrinas, lo que a su vez promueve la reorganización de actina, dentro del podosoma (Eleniste et al., 2012).
PTP-PEST y su participación en la vía de señalización de Rho
La participación de la PTP-PEST ha sido observada en diferentes vías de señalización. Una de las más estudiadas y mejor conocidas hasta el momento es la vía de señalización de Rho.
Rac1 y RhoA forman parte de la familia de las GTPasas pequeñas Rho, y son importantes porque participan en la formación de protrusiones en la membrana y en la retracción del segmento posterior de la célula en el proceso de migración (Nguyen, Kholodenko & von Kriegsheim, 2018). Rac1 estimula la polimerización de actina y la formación de lamelipodios, para impulsar a la célula hacia adelante; mientras que, RhoA promueve el ensamble de adhesiones focales, la contracción del cuerpo celular y la retracción de la parte trasera de la célula (Sastry et al., 2006). Diferentes integrinas y proteínas intracelulares se encargan de la regulación de estas GTPasas facilitando el intercambio del nucleótido GDP, que se encuentra unido a las GTPasas, por nucleótidos GTP. Esto se lleva a cabo por los factores de intercambio de nucleótidos de guanina (GEF), como VAV2 y DOC180. Adicionalmente, su actividad enzimática intrínseca requiere una proteína activadora de GTPasas (GAP) (Sastry et al., 2006; Stiegler & Boggon, 2018). A este grupo pertenece p190RhoGap, que se caracteriza por ser la GAP que presenta mayor interacción con Rac1 y RhoA (Stiegler & Boggon, 2018).
La PTP-PEST regula a las GTPasas RhoA y Rac1 en procesos como el acoplamiento de la protrusión de membrana y la retracción de la parte trasera en la migración celular. En un estudio hecho en fibroblastos de ratón deficientes en esta fosfatasa, se observaron protuberancias de membrana exageradas en el borde delantero de la célula y segmentos largos no retraídos en la parte trasera, así como, una mayor actividad de Rac1 y una disminución en la activación RhoA respectivamente (Sastry et al., 2006; Zheng & Lu, 2013). Estos estudios demostraron que PTP-PEST es fundamental en el control del equilibrio entre la activación de RhoA y la supresión de Rac1, al modular a proteínas como VAV2 y p190RhoGAP, necesarias para la restructuración de la membrana involucrada en el proceso de la migración celular (Sastry et al., 2006). Adicionalmente, PTP-PEST inhibe la actividad de Rac1 al desfosforilar a su factor intercambiador DOC180 (Feller, 2001; Jamieson et al., 2005; Vallés, Beuvin & Boyer, 2004; Zheng & Lu, 2013).
Espejo y colaboradores proponen que la PTP-PEST controla el reclutamiento y/o activación de los complejos de señalización de Rac1 en el borde delantero de la célula. En estos complejos, el estado de fosforilación de la catenina p120, una proteína estructural indispensable para el reclutamiento del andamiaje del citoesqueleto, es esencial. La catenina p120 es un sustrato de la PTP-PEST; esta fosfatasa es capaz de desfosforilar el sitio Y335, en el dominio regulador N-terminal de p120 y controlar su transporte desde la membrana al citosol. El silenciamiento de la PTP-PEST resulta en el aumento de p120 citosólico, particularmente en los lamelipodios, una alteración de la actividad de la GTPasa Rho a través de su interacción con VAV2, y un aumento de la movilidad celular (Espejo et al., 2014).
Finalmente, en las células cancerígenas del colon, se sugiere que la PTP-PEST fortalece las uniones adherentes, al limitar la activación de Rac1 y potenciar la activación de RhoA. En ausencia de la PTP-PEST, la actividad de Rac1 está elevada, lo que se asocia con un aumento de la endocitosis de la E-cadherina, mientras que, RhoA está suprimida, lo que conduce al debilitamiento de las adhesiones, que puede generar una mayor susceptibilidad a las señales quimiotácticas y traducirse en una mayor movilidad celular (Espejo, Rengifo-Cam, Schaller, Evers & Sastry, 2010).
PTP-PEST y su papel en la desfosforilación de EphA3
Otra vía de señalización que involucra a la PTP-PEST, es la vía de EphA3. La interacción efrina-receptor Eph, permite la comunicación entre células a corta distancia, desencadenando modificaciones en el citoesqueleto que se traducen en cambios en la movilidad y la morfología celular (Kania & Klein, 2016). El principal ligando del receptor EphA3 es la efrina 5 (Mansour et al., 2016), y como consecuencia de la activación del receptor se reclutan diversas proteínas efectoras como Src y proteínas intercambiadoras de nucleótidos (GEF) como VAV2 y efexina 1, que influyen en la organización del citoesqueleto directamente o modulando la actividad de las GTPasas RhoA y Rac1 (Kania & Klein, 2016).
Se ha observado que en células de leucemia que sobreexpresan EphA3, se recluta a la PTP-PEST a los complejos de señalización de este receptor, en conjunto con otras proteínas que también están involucradas en la formación de las adhesiones focales como FAK y Paxilina (Day et al., 2013; Vail et al., 2014; Wimmer-Kleikamp et al., 2008). La PTP-PEST controla directamente los niveles de fosforilación de EphA3, así como, sus funciones de señalización (Mansour et al., 2016). La expresión exógena de la fosfatasa reduce significativamente la fosforilación del receptor EphA3 basal inducida por efrina; mientras que, el silenciamiento de la PTP-PEST eleva la fosforilación de EphA3. Asimismo, la PTP-PEST es sustrato de la caspasa 3, que genera dos fragmentos: uno de 75 kDa y otro de 110 kDa. El fragmento N- terminal contiene el sitio catalítico que controla la fosforilación de EphA3 (Mansour et al., 2016). Adicionalmente, este fragmento controla la endocitosis de EphA3 y facilita la desestabilización de la red de actina necesaria para favorecer el movimiento celular. La función de las efrinas en la adhesión celular no ha sido completamente estudiada, por lo que aún falta más investigación para ampliar el conocimiento sobre los mecanismos que controlan la interacción de la PTP-PEST con el receptor EphA3.
PTP-PEST y su participación en la vía PI3K/AKT/mTOR
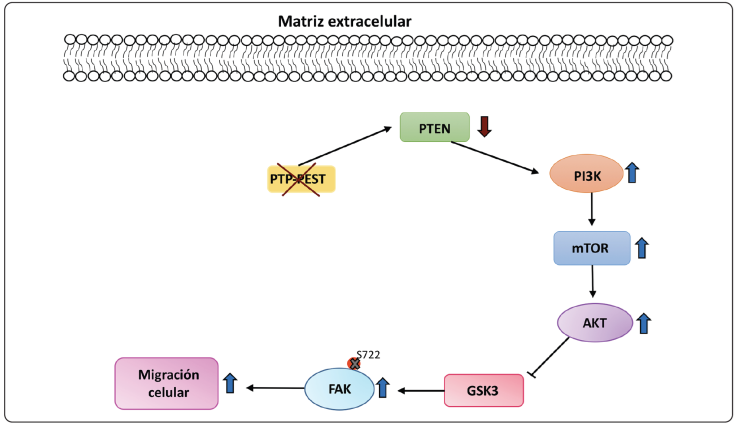
La vía de la cinasa de fosfatidilinositol-3 (PI3K)/AKT/ blanco de rapamicina en los mamíferos (mTOR), está hiperactivada en varios tipos de cáncer, y regula muchos procesos celulares como la proliferación, el metabolismo y la supervivencia (Ersahin, Tuncbag & Cetin-Alay, 2015). La PI3K contiene un sitio de unión para las cinasas dependientes del fosfatidilinositol-3, entre las que se encuentra mTORC2. Al unirse PI3K y mTORC2, fosforilan y activan a la cinasa de serina/treonina (AKT); que, en su estado activo inhibe a la cinasa de la glucógeno sintasa- 3 (GSK3) (Villa-Moruzzi, 2013). Sin embargo, la fosfatasa supresora de los tumores PTEN, es capaz de desfosforilar a la PI3K, con lo que, se inactiva esta vía (Ersahin et al., 2015).
Por su parte, la cinasa de la glucógeno sintasa-3 (GSK3) es un importante regulador de FAK, pues aumenta la fosforilación de su sitio S722 y disminuye la actividad de esta proteína (Villa-Moruzzi, 2013).
Al silenciar a la PTP-PEST en una línea celular de cáncer de ovario, se observó una disminución en la expresión de PTEN. Esto aumenta la activación de la vía PI3K/AKT/mTOR, lo que conlleva a que AKT adquiera la capacidad de inhibir a GSK3. La disminución en la actividad de GSK3 incide en la fosforilación del sitio S722 de FAK, lo que resulta en el aumento de su actividad, y como consecuencia, aumenta la migración celular (Figura 6) (Villa-Moruzzi, 2013).
La disminución de la actividad de la PTP-PEST y su impacto en la vía del HER2
El receptor 2 del factor de crecimiento epidérmico humano (HER2) es un gen que participa en el crecimiento normal de las células y al mutar, actúa como un oncogén y provoca la formación de células cancerígenas asociadas a una alta tasa de mortalidad (de Melo Gagliato, Fontes Jardim, Marchesi & Hortobagyi, 2016; Villa-Moruzzi, 2013). Su alteración está presente en el 20% de los cánceres de mama invasivos, así como, en cáncer de ovario, esófago, gástrico, pancreático, colorectal, y uterino, entre otros tipos (de Melo Gagliato et al., 2016; Gerson, Skariah, Denlinger & Astsaturov, 2017; Villa-Moruzzi, 2013). Se ha visto que la vía del HER2 está modulada por la PTP-PEST, puesto que al abatir la expresión de la fosfatasa, se observa una disminución en la activación de esta vía (Villa-Moruzzi, 2013).
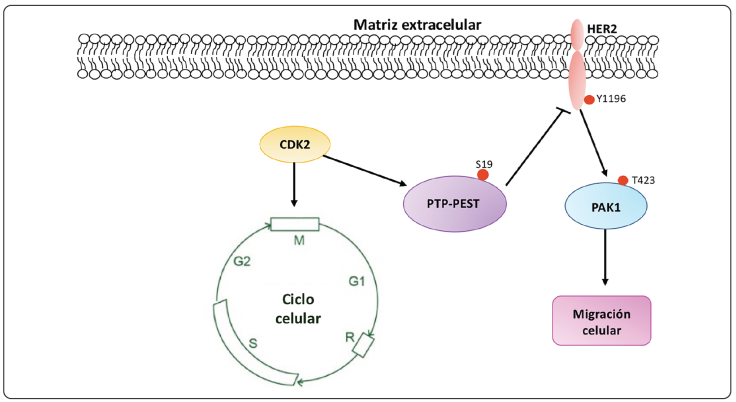
La proliferación celular, particularmente en el cáncer, depende de la desregulación de las proteínas que controlan el ciclo celular. En particular, la proteína cinasa dependiente de la ciclina 2 (CDK2) es un regulador de la transición entre la fase G1 y S del ciclo celular (Kim et al., 2014); y es relevante porque está involucrada en la agresión y recaída del cáncer de mamá y pulmón (Karakas et al., 2016; Kim et al., 2012, 2014; Li et al., 2018; Qu, Liu, Zhong, Li & Zhang, 2015).
De acuerdo con Hui Li y colaboradores, la CDK2 fosforila a la PTP-PEST en su sitio S19, lo que promueve la migración. Cuando este sitio es fosforilado por CDK2, la PTP-PEST no desfosforila el sitio pY1196 de HER2, que es el responsable del reclutamiento y la fosforilación de la proteína cinasa de serina/ treonina-1 (PAK1), la cual, aumenta la migración celular. Lo anterior resulta en una mayor invasión de células cancerígenas (Figura 7) (Li et al., 2018).
PTP-PEST en cáncer
Se han identificado 17 miembros de la familia clásica de las PTPs involucrados en el desarrollo del cáncer, entre los que se cuenta la PTP-PEST. Sun y colaboradores, al estudiar células de cáncer de mama, notaron que había una disminución de la expresión del PTPN12, resultado de la transformación maligna de las células epiteliales mamarias humanas, debido a la activación múltiple de diversas cinasas de tirosina. La diminución de la expresión de la PTP-PEST se debe a deleciones, variantes de secuencias defectuosas o a la pérdida de la expresión del gen de PTPN12. Además, descubrieron que al restaurar la expresión de esta proteína en las células de cáncer de mama se inhibía su proliferación, tumorigenicidad, y su potencial metastásico (Sun et al., 2011). Además, en estudios recientes se ha descubierto que la reducción de la expresión de la PTP-PEST incrementa significativamente la capacidad proliferativa de las células de cáncer de mama, in vitro (Piao et al., 2015). Sin embargo, no es el único tipo de cáncer en el que se ha involucrado a esta fosfatasa: Na Shen y colaboradores observaron que al transfectar células de cáncer colorectal con una mutante de PTPN12 que atenúa su efecto inhibidor, se observa que se inhibe la desfosforilación de la proteína adaptadora SHC, con lo que, aumenta la expresión de CCND1 y se acelera la transición de la fase G1 a la fase S en el ciclo celular aumentando, así, la aparición de células cancerígenas (Shen et al., 2019).
En el cáncer de colon se ha observado que una disminución en la expresión de la PTP-PEST provoca la desregulación de la vía de la GTPasa Rho y la desestabilización de la uniones adherentes de la célula con un incremento de la movilidad celular y una mayor transición epitelio-mesénquima, además de una invasión rápida de las células cancerígenas (Espejo et al., 2010). Se ha reportado también la participación de esta fosfatasa en células invasoras de glioblastoma. En este caso, la PTP-PEST regula el crecimiento invasivo al formar un complejo con las proteínas p130Cas y Vcp (Valosin-containing protein, por sus siglas en inglés), lo que estabiliza las adhesiones focales y equilibra el crecimiento invasivo de estas células. De esta manera, las células de glioblastoma que expresan a la PTP-PEST son menos invasivas debido a que cuentan con adhesiones focales más estables; del mismo modo, las células con bajos niveles de expresión de PTP-PEST son más invasivas, debido a que el desmontaje de las adhesiones focales es más dinámico (Chen, Morales, Guerrero, Sun & McCarty, 2018).
También se ha documentado la participación de la PTP-PEST en la invasión de células cancerígenas papilares renales inducida por especies reactivas de oxígeno (ROS). En estas células, la oxidación inactiva a la fosfatasa, disminuye la desfosforilación de ABL1, e induce un aumento en el crecimiento tumoral y la supervivencia de las células (Lee & Rhee, 2019; Xu et al., 2018). Adicionalmente, se ha observado la participación de micro-RNAs en la disminución de la expresión de la PTP-PEST. En el caso del cáncer de ovario, el micro-RNA, miR-194 disminuye la expresión de la PTP-PEST, lo que aumenta la progresión de los tumores en este tipo de cáncer (Liang, Cheng, Ren & Zhang, 2016).
Con base en lo anterior, es innegable la participación de la PTP-PEST como un regulador en diferentes tipos de cáncer en humanos como el cáncer de mama, el de ovario, y el de colon, entre otros (Zhangyuan et al., 2018). Adicionalmente, se ha documentado su participación en las vías de señalización relacionadas con este padecimiento al restringir la acción de cinasas pro-oncogénicas (Villa-Moruzzi, 2013). Esta proteasa es muy importante en la regulación de la adhesión, la movilidad y la invasión celular, a través de su interacción con, e inhibición de las cinasas anteriormente mencionadas (Piao et al., 2015). Debido a esto, la PTP-PEST se ha considerado candidata para el desarrollo de tratamientos terapéuticos contra algunos tipos de cáncer, sin embargo, los procedimientos a utilizar aún están en investigación.
Conclusiones
Puesto que las fosfatasas son reguladores clave de las vías de señalamiento intracelular involucradas en una gran variedad de patologías, existe un gran interés en utilizarlas como blancos terapéuticos. A partir del descubrimiento de la defosfatina en 1993 (Imoto et al., 1993) se han desarrollado una gran variedad de inhibidores a partir de productos naturales, péptidos, fosfonatos, mimótopos, y RNA de silenciamiento (Zhang, 2017). Sin embargo, ha sido difícil elaborar un inhibidor que sea selectivo, biodisponible y capaz de bloquear con eficacia el sitio activo. El hecho de que las PTPs posean un sitio activo altamente conservado, representa un problema en la elaboración de inhibidores selectivos. Por otra parte, la mayoría de estos sitios poseen una gran cantidad de cargas positivas, lo que implica que cualquier inhibidor cargado negativamente tendría problemas de permeabilidad celular (Zhang, 2017).
En la actualidad, no se han descrito inhibidores específicos para la PTP-PEST. Sin embargo, recientemente se ha descrito que el Auranofin, una sal de oro que se utiliza para el tratamiento de la artritis reumatoide, tiene un efecto inhibidor sobre la actividad de la PTP-PEST en un modelo murino de daño al miocardio (Yang et al., 2020). Sin embargo, aunque el Auranofin está aprobado por la FDA (Food and Drug Administration de Estados Unidos de América), dista mucho de ser un inhibidor específico. No se conoce el mecanismo de acción de este compuesto, pero se sabe que inhibe la liberación de proteínas proinflamatorias, como la ciclooxigenasa 2, la sintasa del óxido nítrico y la reductasa de tioredoxina, además de ser un regulador de la vía de NF-kappaB (Yamashita, 2021). La ausencia de inhibidores específicos para la PTP-PEST representa una oportunidad importante para el desarrollo de fármacos que, al coadyuvar en el estudio de estas fosfatasas, podrían ser terapéuticamente importantes en enfermedades como el cáncer. Es por esto que resulta importante seguir investigándola, así como su papel en la fisiología celular.











 nova página do texto(beta)
nova página do texto(beta)