¿Qué es la plasticidad celular?
Las células eucariontes de los organismos pluricelulares se encuentran en un microambiente en donde convergen diversas señales biofísicas y bioquímicas, que contribuyen al establecimiento de la homeostasis tisular. Las células pueden manifestar una plasticidad al responder o adaptarse a esas señales. La plasticidad celular ha sido definida como la capacidad que tiene el genotipo de una célula para producir células con diferentes fenotipos, en respuesta a variaciones microambientales (Fordyce, 2006; Whitman & Agrawal, 2009; Pigliucci, Murren & Schlichting, 2006). La plasticidad celular es una propiedad inherente a todas las células, es un mecanismo que se presenta tanto en el desarrollo embrionario como en los tejidos adultos, en contextos fisiológicos o patológicos (Jessen Mirsky & Arthur-Farraj, 2015). En condiciones fisiológicas, la plasticidad celular es fundamental para los procesos de reparación tisular y de regeneración de órganos, contribuyendo al mantenimiento de la homeostasis tisular; mientras que en condiciones patológicas, la plasticidad también es una propiedad de las células enfermas, como en el caso de las células cancerosas que pueden adquirir fenotipos que favorecen la metástasis (Varga & Greten, 2017).
Tipos de Plasticidad Celular
Los órganos de vertebrados adultos utilizan diferentes mecanismos moleculares para mantener sus funciones y su masa. Cuando un tejido es dañado, se activan diferentes programas de reparación tisular en las células somáticas adultas que involucran procesos como: la proliferación, la desdiferenciación y la transdiferenciación, entre otros (Stanger, 2015; Merrell & Stanger, 2016). La desdiferenciación implica que una célula diferenciada readquiera marcadores y propiedades de la célula que le dio origen; es decir, características de la célula progenitora o troncal (Merrell & Stanger, 2016). La transdiferenciación, por el contrario, se refiere a la conversión de una célula diferenciada en otra célula igualmente diferenciada, de un origen similar o diferente al de la capa germinal que le dio origen; mientras que la transición epitelio-mesénquima (EMT, del inglés Epithelial-Mesenchymal Transition), es un tipo de transdiferenciación propia de las células epiteliales que puede ser parcial o total y mediante la cual las células epiteliales adquieren un fenotipo de tipo mesenquimal, al perder o disminuir la expresión de algunos de sus rasgos epiteliales como: la morfología, la polaridad, la expresión de factores transcripcionales y de microRNAs; además de exhibir cambios en la expresión y la organización de las proteínas del citoesqueleto, entre otros (Merrell & Stanger, 2016).
Esta revisión, describe principalmente el conocimiento actual sobre la plasticidad del hepatocito y sobre los mecanismos moleculares que la regulan, así como en describir su participación y relevancia en la fisiología y patología del hígado, particularmente en procesos hepáticos como: la homeostasis metabólica y la regeneración, así como en la inflamación crónica, la fibrosis y el desarrollo del hepatocarcinoma (HCC). Cabe señalar, que la plasticidad de las células no parenquimatosas del hígado y su relevancia en la fisiopatología hepática merece también ser sujeta a una futura revisión.
El hígado: estructura y funciones
El hígado es un órgano esencial en la regulación y el mantenimiento de la homeostasis metabólica de los vertebrados (Kang Mars & Michalopoulos, 2012). Las funciones hepáticas son muy amplias y variadas, al llevar a cabo procesos como la absorción, la síntesis, el almacenaje, el metabolismo y la redistribución de nutrientes como: los lípidos, los carbohidratos, las proteínas y las vitaminas. Además, es el principal órgano que metaboliza, detoxifica a los compuestos xenobióticos y exhibe una alta capacidad regenerativa en respuesta al daño (Taub, 2004) (Figura 1). El proceso de la regeneración hepática ha sido descrito desde la antigüedad en la mitología griega, al relacionarlo con la historia del castigo a Prometeo, quien fue condenado a permanecer encadenado a una roca por robar el fuego de los dioses; así, cada día un águila devoraba parte de su hígado, que se restauraba debido a su capacidad regenerativa permitiendo que el ave rapaz se alimentara indefinidamente de su órgano (Taub, 2004).
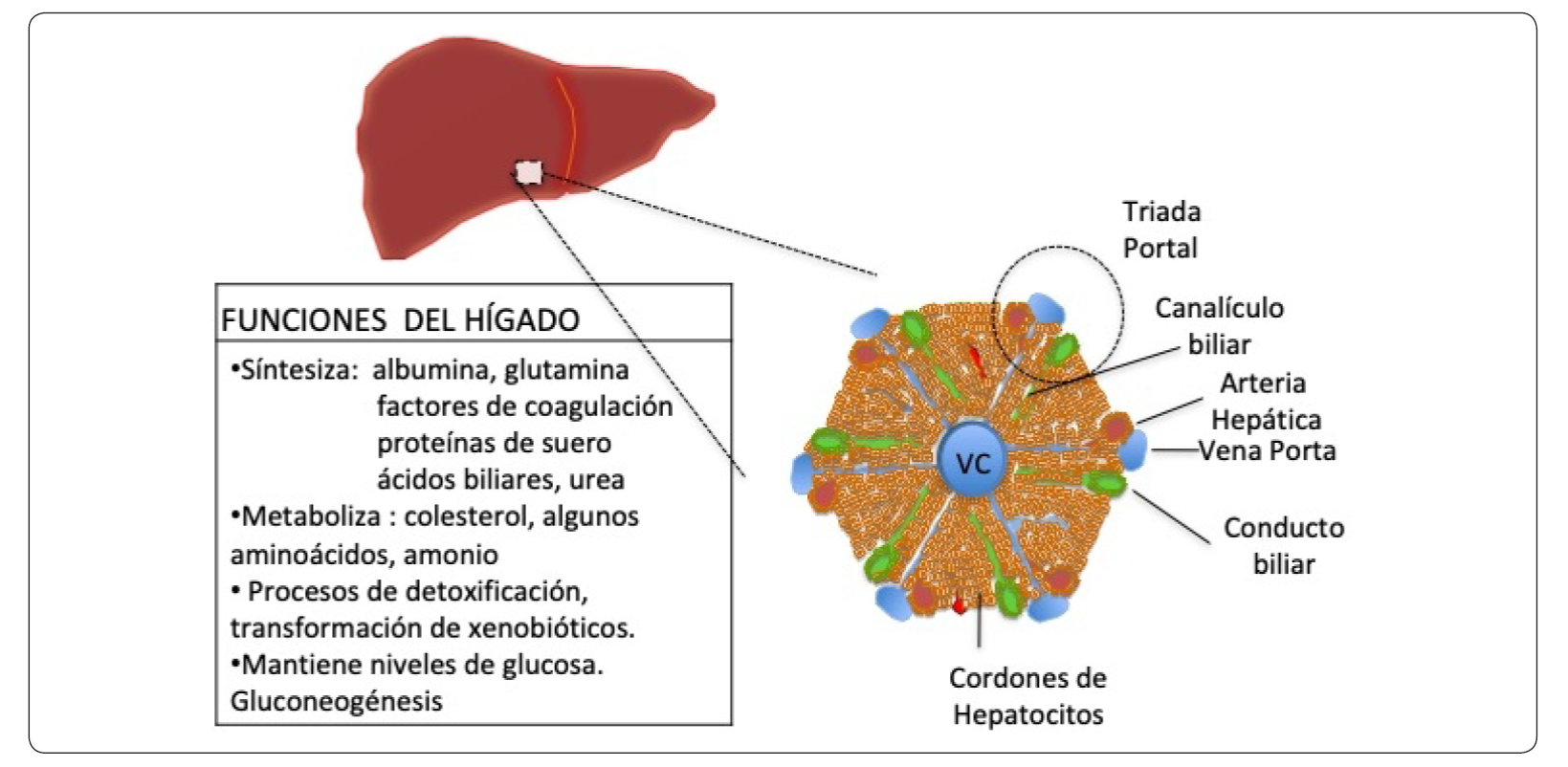
Figura 1 La unidad funcional del hígado: el lóbulo hepático. Representación esquemática del lóbulo hepático indicando la triada portal (arteria hepática, vena porta y canalículo biliar), la vena central (VC), así como la distribución de los hepatocitos. Elaboración personal.
La unidad estructural del hígado es el lobulillo hepático, con forma de hexágono y en cuyo centro se encuentra la vena centrolobulillar y hacia sus vértices se localiza la triada portal; esta última, compuesta por ramas de la arteria hepática, del conducto biliar y de la vena porta (Figura 1). Esta red vascular establece un flujo sanguíneo unidireccional que fluye desde la triada portal hacia la vena central. En el hígado existen diferentes tipos celulares clasificados en dos principales que son: las células parenquimatosas y las células no parenquimatosas. Las células parenquimatosas o hepatocitos, constituyen alrededor del 80% del volumen hepático y representan el 60% de todas las células que componen al hígado. En tanto que las células no parenquimatosas comprenden un 6.5% de su volumen y representan el 40% de las células restantes. Los tipos de células no parenquimatosas son las: epiteliales del conducto biliar o colangiocitos, sinusoidales o endoteliales, estrelladas hepáticas HSC (del inglés Hepatic Stellate Cells), de Kupffer o macrófagos y NK hepáticas (del inglés Natural Killer) (Lecluyse, Witek, Andersen & Powers, 2012; Tsutsumi, Nakamura, Ueno, Torimura & Aguirre-García, 2017).
Los fenotipos del hepatocito
Los hepatocitos representan al grupo celular más abundante del hígado, que está constituido por diferentes poblaciones celulares que se han clasificado de acuerdo con su disposición en el lóbulo hepático y a su ploidía. En la Tabla I se describen los diferentes marcadores morfológicos y moleculares característicos de los hepatocitos, por lo que cabe señalar que cada una de las diferentes sub-poblaciones de ellos tienen funciones metabólicas muy especializadas (Jungermann & Keitzmann, 1996; Tanami et al., 2017; Gentric & Desdouets, 2014). En el lobulillo hepático, los hepatocitos se distribuyen en forma de láminas o cordones, que se extienden desde la vena centrolubilillar hacia la triada portal (Colnot & Perret, 2011). En esta estructura coexisten diferentes microambientes que generan una “zonación metabólica” en el hígado, denominada “zonación hepática”, que se establece en parte por el tránsito diferencial de nutrientes y niveles de oxígeno que provienen de las arterias, venas y conductos biliares (Figura 2). Se han definido 3 grandes zonas hepáticas: 1) la zona 1 o periportal, es donde convergen el conducto biliar, la vena porta y la arteria hepática; en esta zona se localizan los hepatocitos llamados periportales (PP) que tienen un metabolismo gluconeogénico y participan en la biosíntesis del colesterol; 2) La zona 2 o media, se localiza entre la región periportal y la región pericentral; 3) La zona 3 o pericentral, se encuentra adyacente a la vena central y contiene a los hepatocitos denominados pericentrales (PC) que tienen un metabolismo glucolítico y llevan a cabo la biosíntesis de los ácidos biliares y de la glutamina (Katz, Teutsch, Jungermann & Sasse 1977; Kietzman, 2017) (Figura 2).
Tabla I Características morfológicas y marcadores moleculares de los hepatocitos con fenotipo epitelial.
| Características | Marcadores | Referencias |
| Morfología | •Células con forma poligonal •Mono- o bi-nucleados •Microvellosidades en la membrana |
(Kmiec, 2001; Kreutz et al., 2017; Stanger, 2015) |
| Popularidad |
Basolateral: MRP3, MRP4, MPR6, Na+/K+ ATP-asa,
OATP Apical: MDR1, MDR3, MRP2, DPPIV, BRCP Canalículo biliar: MRP2, MDR1, BSEP, NTCP Uniones celulares: E-cadherina, ZO-1, Ocludina, Claudina 1, Conexina 32 |
(Decaens & Cassio, 2001; Deharde et al., 2016; Gissen & Arias, 2015; Moghe et al., 1996; Treyer & Müsch, 2013; Wang & Boyer, 2004) |
| Ploidía | 2N, 4N, 8N | (Celton-Morizur & Desdouets, 2010; Kreutz et al., 2017) |
| Citoesqueleto | •Actina organizada de manera cortical •Citoqueratina 8, Citoqueratina 18 |
(Ballatori et al., 2006; Feldmann, 1989; Snykers, De Kock, Rogiers & Vanhaecke, 2009) |
| Factores transcripcionales | HNF1, HNF4α TBx3 C/EBP CAR, PXR, RXRα |
(Cicchini et al., 2015; Kheolamai & Dickson, 2009; Kuijk et al., 2016; Maurel, 2010; Snykers et al., 2009) |
| miRNAs | miR200c miR34a |
(Lu, Jolly, Levine, Onuchic & Ben-Jacob, 2013) |
| Secreción |
Proteínas en plasma: Albúmina, transferrina,
Lipoproteínas (Apo-A1, Apo-B100), ceruloplasmina, urea.
Ácidos biliares Inhibidores de proteasas: α1-antitripsina, antitrombina, α2- macroglobulina Factores que modulan la coagulación: factores V, VII, IX, X; fibrinógeno, protrombina, plasminógeno Proteínas de respuesta aguda: proteína C-reactiva, proteína del complemento C3 |
(Ferrini, Pichard, Domergue & Maurel, 1997; Geiger et al., 1982; Kaimori et al., 2007; Maurel, 2010; Pösö, Penttila, Suolinna & Lindros, 1986; Runge et al., 2000; Sierra-Santoyo, López, Hernández & Mendoza-Figueroa, 1994) |
| Metabolismo de fármacos | Familia del CYP450: CYP1A1, CYP1A2, CYP2A6, CYP2B6, CYP2C8, CYP2C9, CYP2C19 CYP2D6, CYP2E1, CYP3A4, CYP2E1, CYP2B1, CYP3A2, CYP4A1, CYP3A7, CYP7A1 | (Ferrini et al., 1997; Hewitt et al., 2007; Maurel, 2010; Tsutsui et al., 2006; |

Figura 2 Zonación metabólica. Representación de la zonación metabólica en un sinusoide a lo largo del eje porto-central indicando el flujo de O2, Nutrientes, vías de señalización y rutas metabólicas. Elaboración personal.
Los hepatocitos son células cuyo promedio de vida oscila entre 200-300 días. A la fecha, se han identificado dos sub-poblaciones adicionales de hepatocitos PP y PC, con funciones asociadas al mantenimiento de la masa hepática en condiciones homeostáticas o de daño (Bird & Forbes, 2015). 1) La subpoblación PP incluye a los hepatocitos híbridos periportales (HybHP) que representan entre el 4 y 5% del total (Font-Burgada et al., 2015). Estos hepatocitos son positivos al factor transcripcional Sox9 y a algunos marcadores de células epiteliales del conducto biliar, como la proteína osteopontina. La razón por la que se les conoce como hepatocitos híbridos es porque presentan marcadores tanto de hepatocitos como de colangiocitos; estos hepatocitos HybHP se caracterizan por proliferar en respuesta a señales de daño hepático (Font-Burgada et al., 2015). 2) La subpoblación de los hepatocitos PC se caracteriza por ser diploide y positiva a Axina2 y al factor transcripcional Tbx3 (Wang, Zhao, Fish, Logas & Nusse, 2015). Esta subpoblación parece ser la que contribuye fundamentalmente a la renovación de los hepatocitos en el hígado en ausencia de daño, considerando que esta subpoblación puede renovar hasta el 40% de ellos en un año, en organismos como el ratón (Wang et al, 2015).
La ploidía es una característica peculiar de los hepatocitos y se refiere al número (N) de juegos de cromosomas presentes en una célula; se considera que los hepatocitos en general son células diploides (2N), pero conforme van madurando su ploidía puede aumentar, llegando a ser 4N, 8N e inclusive 32N y mono- o bi-nucleados. Así por ejemplo, en los roedores la ploidía aparece al momento del destete y aumenta conforme avanza la edad del animal; en otros contextos, como la regeneración del hígado de ratas sometidas a un proceso de hepatectomía parcial, se ha observado un incremento en el número de hepatocitos poliploides, principalmente en los tetraploides (4N) y octaploides (8N) (Sigal et al., 1999; Wang et al., 2015). Cabe mencionar que el grado de ploidización tiene una distribución zonal: la mayor cantidad de los que son poliploides se concentran en la Zona 2, aunque existen hepatocitos poliploides también en las Zonas 1 y 3 (Tanami et al., 2017). La ploidía se ha asociado a procesos de senescencia celular, lo que compromete la capacidad proliferativa de los hepatocitos y su función metabólica; así, los metabólicamente más activos tienen más copias de genes asociados a esa actividad. El fenómeno de ploidización ocurre por alteraciones en la división celular, ya sea porque no se completan procesos como la citocinesis (división celular) o la cariocinesis (división nuclear) (Gentric & Desdouets, 2014). Además, se ha sugerido que la ploidía en los hepatocitos podría conferir protección ante el daño al DNA, en condiciones en las cuales hay un aumento de los radicales libres y de las especies reactivas del oxígeno que se generan como producto de la función metabólica de los hepatocitos (Gentric & Desdouets, 2014; Tanami et al., 2017).
Vías de señalización involucradas en el establecimiento del fenotipo del hepatocito
La identidad celular es el resultado de la interacción entre múltiples señales bioquímicas y biofísicas con clara evidencia de la contribución de diferentes vías de señalización para mantener o modificar la identidad del hepatocito, entre las que se encuentran: la vía del TGF-β, la ruta Hippo y la ruta Wnt, así como las vías de Notch y Hedhehog (Hh), descritas a continuación.
La vía del TGF-β y los factores transcripcionales Smad
El TGF-β (del inglés Transforming Growth Factor-Beta) fue identificado en 1980 y es el factor prototipo de la superfamilia del TGF-β, que abarca más de 60 miembros presentes en vertebrados e invertebrados y se subdivide en dos grandes subfamilias. La primera está integrada por los TGF-β1, 2 y 3, las activinas, nodal y las inhibinas; la segunda por las BMP (del inglés Bone Morphogenetic Proteins) y los GDF (del inglés Growth and Differentiation Factors) (Roberts et al., 1980). La vía de señalización canónica del TGF-β inicia con la unión del ligando en forma de dímero a dos receptores transmembranales con actividad de cinasa de residuos de serina y treonina (Ser/Thr). Primero, el TGF-β activo se une al receptor transmembranal tipo II llamado TβRII, que tiene actividad constitutiva de cinasa de residuos de Ser/Thr, para reclutar al receptor tipo I llamado TβRI o ALK5 (del inglés Activin Receptor-Like Kinase 5). En el complejo heterotetramérico formado entre los receptores TβRI y TβRII, este último transfosforila al primero en una secuencia rica en residuos de glicina y serina, denominada dominio GS. La fosforilación en este dominio activa al receptor TβRI o ALK5, quien propaga la señal intracelularmente a través de la fosforilación de las proteínas llamadas Smad; en el caso de las señales del TGF-β y la activina se promueve la fosforilación de las proteínas Smad2 y Smad3 en el motivo SXS, localizado en su extremo C-terminal (Macías-Silva et al., 1996). Las Smad2/3 activas, forman complejos heterotriméricos entre ellas o con la Smad4, para que en conjunto sean translocadas al núcleo y regulen la expresión de sus múltiples genes blanco (Massagué, 1998).
Las proteínas Smad2/3 son vitales para el desarrollo embrionario del hígado; los ratones heterocigotos para Smad2+/− y Smad3+/−, generados por entrecruzamiento entre ratones Smad2+/− y Smad3+/−, mueren a la mitad de su gestación al día 14.5. El análisis histológico muestra que los hígados de estos ratones heterocigotos son de menor tamaño con respecto a los ratones normales (Weinstein et al., 2001). En un hígado adulto, la activina y el TGF-β regulan la homeostasis de los hepatocitos al actuar como factores citostáticos (anti-proliferativos) y pro-apoptóticos (muerte celular programada), manteniendo a los hepatocitos en la etapa G0 del ciclo celular o estado de quiescencia (Rodgarkia-Dara et al., 2006). Esta vía de señalización se activa en los procesos de regeneración hepática (Macías-Silva. Li, Leu, Crissey & Taub, 2002; Vázquez-Victorio et al., 2015). Además, estudios realizados en dos líneas celulares de hepatocitos de ratón (MMH/E14 y WT/3A) reportan que el TGF-β induce la binucleación de los hepatocitos y un incremento en su ploidía, como resultado de una falla en la citocinesis por un mecanismo que involucra la activación de la cinasa Src y la inactivación y deslocalización de la proteína RhoA (De Santis Puzzonia et al., 2016). Resulta interesante que el fenotipo del hepatocito cambie en el proceso de binucleación inducido por el TGF-β y que adquiera marcadores de tipo mesenquimal, como el incremento en los niveles del factor transcripcional Snail (De Santis Puzzonia et al., 2016). Si bien el TGF-β regula la homeostasis hepática al actuar como un supresor de tumores, puede exhibir un papel dual en el desarrollo de algunas patologías, al ser un fuerte promotor de la fibrosis y el HCC. El desarrollo de estas patologías es promovido por un daño crónico que puede resultar de padecimientos como la hepatitis B y C, por el consumo excesivo de alcohol o por el metabolismo de algunos medicamentos, entre otros. En un contexto de daño crónico, el TGF-β actúa como uno de los principales factores pro-fibrogénicos, pro-tumorales y eventualmente pro-metastásicos (Dooley & ten Dijke, 2012; Fabregat et al., 2016).
La ruta Hippo y los cofactores transcripcionales YAP/TAZ
La vía de señalización Hippo se identificó inicialmente en la mosca de la fruta Drosophila melanogaster en los años 90; el nombre de esta vía deriva de la cinasa de residuos de Ser/Thr llamada Hippo (Hpo), que en mamíferos corresponde a las cinasas MST2 y MST1 (del inglés, Mammalian Ste20-like protein kinase 2 o 1) (Patel, Camargo & Yimlamai, 2017). Esta vía de señalización regula procesos relevantes como el control del tamaño de los órganos, el mantenimiento de las poblaciones de células troncales, la diferenciación de distintos tipos celulares y la transición epitelio-mesénquima; además, la ruta Hippo es un importante regulador del sistema inmune y del desarrollo de tumores (Yimlamai, Fowl & Camargo, 2015; Moroishi et al., 2016). En mamíferos, la ruta Hippo ejerce una regulación negativa de sus efectores, los correguladores transcripcionales YAP (del inglés, Yes-Associated Protein 1) y TAZ (del inglés, Transcriptional coactivator with PDZ-binding motif), a través de una cascada de fosforilaciones iniciada por las cinasas Mst1 y Mst2, que en asociación con la proteína de andamiaje WW45 o Sav1 (Salvador 1) fosforilan y activan a las cinasas Lats1 y Lats2 (del inglés, Large Tumor Supressors 1 y 2) (Pan, 2010). Las cinasas Lats1 y 2 activadas se asocian con los activadores de cinasas MOB1A y MOB1B para llevar a cabo la inactivación de los correguladores transcripcionales YAP y TAZ, mediante su fosforilación en residuos de Ser; esta fosforilación promueve la translocación de los correguladores del núcleo hacia el citoplasma y entonces pueden ocurrir dos situaciones: por un lado, las proteínas YAP y TAZ son retenidas en el citoplasma por su asociación con las proteínas 14-3-3 o bien son degradadas vía el Sistema Ubiquitina-Proteosoma (UPS, del inglés Ubiquitin-Proteosome System) (Hao, Chun, Cheung, Rashidi & Yang, 2008; Kanai et al., 2000; Yagi, Chen, Shigesada, Murakami & Ito, 1999; Liu et al., 2010).
En el hígado de un organismo adulto se ha visto que YAP controla el fenotipo de los hepatocitos a lo largo del eje porto-central del lóbulo hepático (Fitamant et al., 2015) (Figura 2). YAP se expresa principalmente en los hepatocitos PP, a diferencia del corregulador transcripcional de la ruta Wnt llamado β-catenina que se expresa principalmente en los hepatocitos PC (Fitamant et al., 2015). La zonación hepática depende de que los niveles de YAP se mantengan bajo un estricto control; por ejemplo, los hepatoblastos son células bipotentes que pueden dar origen a hepatocitos o colangiocitos; cuando los hepatoblastos expresan altos niveles de YAP promueven su mantenimiento y evitan su diferenciación hacia los hepatocitos; mientras que el aumento de los niveles de YAP en los hepatocitos, puede favorecer su proliferación; sin embargo, si los niveles de YAP aumentan arriba de cierto umbral, los hepatocitos se desdiferencian hacia hepatoblastos al inhibir la expresión de HNF4α, un factor transcripcional maestro en el control de la identidad del hepatocito (Lee et al., 2016; Yimlamai et al., 2016).
Se han generado ratones KO (del inglés, knockout) que carecen de la expresión de algunos de los genes que codifican para los diferentes componentes de la ruta de Hippo y en todos lo casos se ha observado la presencia de hígados de mayor tamaño y altos niveles de YAP, aunque se desconocen los mecanismos involucrados. En estos modelos de ratón KO se ha observado, por ejemplo, que en los hígados de ratones KO para la cinasa Mst1 o para la Mst2, aumenta la proliferación de los hepatocitos, mientras que en los ratones KO de Sav1, una proteína andamio o scaffold de Lats1/2, son las células troncales las que proliferan y se diferencian (Lee et al., 2010; Lu, Finegold & Johnson, 2018; Zhang et al., 2010; Zhou et al., 2009). En contraste, durante la progresión del HCC ocurre un incremento en los niveles de las proteínas YAP y TAZ; el aumento de TAZ en pacientes con HCC correlaciona con el incremento de algunas citocinas pro-inflamatorias como CCL2, IL6 y CXCL1 (Hagenbeek et al., 2018).
La ruta Wnt y el cofactor transcripcional β-catenina
La actividad de la ruta Wnt/β-catenina tiene un papel fundamental en el desarrollo embrionario del hígado, desde la etapa de especificación del endodermo anterior hasta el establecimiento de la zonación hepática, así como en la proliferación y la maduración de los hepatocitos (Russell & Monga, 2018; Perugorria et al., 2019). Los niveles de la proteína β-catenina son mayores en los hepatocitos PC en comparación con los hepatocitos PP (Figura 2). Esto se debe en parte, a que en los PP hay una mayor expresión de la proteína APC (del inglés, Adenoma Poliposis Coli), un regulador negativo de la ruta Wnt que forma parte del complejo de destrucción de la β-catenina (Benhamouche et al., 2006; Colnot & Perret, 2011; Burke et al., 2018).
En la vía canónica de la ruta Wnt, los niveles proteicos del corregulador transcripcional β-catenina se mantienen bajos en ausencia del ligando gracias al complejo de destrucción de la β-catenina, el cual es un multicomplejo conformado por GSK3 (del inglés, Glycogen Synthase Kinase 3), APC, CK1α (del inglés, Casein Kinase 1α) y la proteína andamio o scaffold llamada Axina (Russell & Mongaa, 2018). En este complejo, la β-catenina es fosforilada en la Ser45 por la CK1α, lo que promueve su subsecuente fosforilación en las Ser33, la Ser37 y en la Thr41 por la cinasa GSK3, propiciando la asociación de la β-catenina con la proteína β-TRCP (del inglés, β-Transducin Repeat Containing Protein) que es una ligasa E3 de ubiquitina, la cual poli-ubiquitina a la β-catenina y promueve su degradación vía el proteosoma. Por el contrario, cuando un ligando Wnt se une al receptor tipo GPCR llamado Frizzled y a los correceptores LRP5 o LPR6 (del inglés, Low density lipoprotein Receptor-related Protein 5 o 6) se activa la ruta Wnt y se inactiva el complejo de destrucción, lo que permite un incremento en los niveles de la proteína β-catenina que entonces es translocada al núcleo e interacciona con los factores transcripcionales TCF y LEF (del inglés, T-Cell Factor y Limphoid Enhancer Factor, respectivamente) para regular la expresión de sus genes blanco. Entre los genes blanco del complejo β-catenina/TCF/LEF se encuentran los que codifican para: Axina2, la glutamina sintetasa 2 y para las enzimas del citocromo P450, CYP2E1 y CYP1A2, entre otros (Russell & Monga, 2018). En el núcleo de los hepatocitos PC, el complejo β-catenina/TCF/LEF induce la expresión de genes asociados a un fenotipo pericentral en respuesta a una mayor actividad de la ruta Wnt; en tanto que en los hepatocitos PP, se expresan altos niveles de APC por lo que los niveles de β-catenina son bajos (Benhamouche et al., 2006). La eliminación de la expresión del regulador negativo APC en el hígado provoca que los hepatocitos adyacentes a la región periportal adquieran un fenotipo de hepatocito PC, y por el contrario, si se disminuyen los niveles de β-catenina en los hepatocitos PC, estos adquieren un fenotipo PP (Kietzman, 2017).
En pacientes con HCC se han encontrado mutaciones en el gen CTNNB1 que codifica para la β-catenina, muy cerca de los sitios de fosforilación de esta proteína que son importantes en la regulación de su estabilidad y su translocación al núcleo (Monga, 2015). En condiciones patológicas, la β-catenina favorece el desarrollo de tumores al estimular la proliferación y la sobrevivencia de las células cancerosas, así como por promover el metabolismo, la angiogénesis y la tolerancia inmunológica del tumor (Russell & Monga, 2018).
La vía de Notch y el corregulador transcripcional NICD
La vía de señalización canónica de Notch funciona en la comunicación intercelular yuxtacrina, que se activa por la interacción entre el receptor Notch, presente en una célula, con su ligando expresado en la membrana de la célula adyacente. En mamíferos existen 4 tipos de receptores Notch que se caracterizan por presentar un solo pase transmembranal, mientras que existen 5 diferentes ligandos: Jagged1, Jagged2, Delta1, Delta3 y Delta-like 4 (Morell, Fiorotto, Fabris & Strazzabosco, 2013). La interacción ligando-receptor promueve un corte proteolítico del dominio intracelular del receptor Notch denominado NICD (del inglés, Notch Intracellular Domain) por la γ-secretasa. El dominio NICD es translocado al núcleo, en donde actúa como corregulador transcripcional uniéndose al factor transcripcional RBPJ (del inglés, Recombination signal Binding Protein immunoglobulin kappa J) regulando la expresión de sus genes blanco como Hes y Hey, entre otros (Morell et al., 2013).
En el hígado, la vía de Notch participa en la formación de los conductos biliares. La activación sostenida del receptor Notch2 en los hepatocitos promueve que éstos se transdiferencien hacia colangiocitos (Chen et al., 2012; Jeliazkova et al., 2013). Adicionalmente, se ha reportado que la vía de Notch es activada en hígados fibróticos (Ni et al., 2018).
La vía de Hedgehog y los factores Gli
Esta vía se descubrió en Drosophila, en donde la mutación del gen hh que codifica al ligando Hedgehog (Hh del inglés, Hedgehog), provoca la formación de espículas en la epidermis de las larvas de la mosca, dándoles la apariencia de un puercoespín (Briscoe & Thérond, 2013). En mamíferos se han descrito 3 ligandos: Sonic- (Shh), Desert- (Dhh) e Indian- (Ihh) hedgehog. La vía se activa por la unión de un ligando al receptor Patched que consta de 12 pases transmembranales. Esta interacción permite la activación de la proteína transmembranal Smoothened induciendo su función y permitiendo con ello la propagación de la señal, la cual conduce a la translocación de los factores transcripcionales Gli al núcleo (Briscoe & Thérond, 2013).
En el hígado, la vía de Hh contribuye al mantenimiento de los hepatoblastos, células progenitoras del hígado que dan origen a los hepatocitos y a los colangiocitos; sin embargo, conforme los hepatoblastos se diferencian hacia hepatocitos disminuye la expresión de los ligandos Hh (Hirose, Itoh & Miyajima, 2009; Sicklick et al., 2006; Omenetti, Choi, Michelotti & Diehl, 2011). Así mismo, los niveles de expresión del receptor patched también se mantienen bajos en los hepatocitos, en comparación con los progenitores hepáticos (Sicklick et al., 2006).
La citocina IL-6 y la vía JAK/Stat3
La interleucina-6 (IL-6) es una citocina pleiotrópica que es secretada en condiciones de inflamación en respuesta al TNFα o a la activación de la vía TLR4 (del inglés, Toll like Receptor 4). En el hígado, la IL-6 participa en diversos procesos como son la proliferación celular y la inflamación. En el proceso de regeneración hepática, inducido por una hepatectomía parcial (PH, del inglés Partial Hepatectomy), se produce IL-6 en las primeras horas posteriores a la cirugía y actúa como una factor mitogénico, mientras que en procesos inflamatorios promueve la síntesis de las proteínas de la fase aguda hepática, como el fibrinógeno, la proteína C reactiva, la haptoglobina y la proteína amiloide del suero A, entre otras (Schmidt-Arras & Rose-John, 2016).
Para llevar a cabo sus acciones, la IL-6 se une al receptor IL-6R y a la glicoproteína gp130 en la membrana de las células; la formación de este complejo promueve la activación de la cinasa JAK (del inglés, Janus Tyrosine Kinases) que fosforila diversos residuos de tirosina en gp130, generando con ello sitios de acoplamiento que permitirán la unión de diferentes proteínas y la activación de sus efectores como el factor transcripcional Stat3 o las cinasas MAPK y PI3K (Schmidt-Arras & Rose-John, 2016). En particular, la activación de la vía IL-6/JAK/Stat3 regula procesos como la sobrevivencia de las células hepáticas, la regeneración hepática y la inducción de la respuesta aguda en el hígado (Wang, Lafdil, Kong & Gao, 2011). Stat3 es miembro de las proteínas STAT (del inglés, Signal Transducer and Activator of Transcription) y es activado por fosforilación en la Tyr705 por la cinasa JAK, en respuesta a estímulos como el de IL6 o bien de IL5, VEGF, HGF o EGF (Itoh, Saitoh & Miyazawa, 2018). La fosforilación de Stat3 promueve la homo- o hetero-dimerización de esta proteína, que luego es translocada al núcleo para regular la expresión de sus genes blanco (Itoh et al., 2018).
Plasticidad del hepatocito en contextos sanos y patológicos
Una de las propiedades más sorprendentes de los hepatocitos es su plasticidad; es decir, su capacidad de cambiar su estado de quiescencia por un estado proliferativo o cambiar su fenotipo, con el propósito de mantener la homeostasis del hígado (Sell, 2002; Stanger, 2015). Esto ocurre en determinados contextos fisiológicos o patológicos, como en el caso de la regeneración hepática, la fibrosis, el HCC y la colestasis; en estos escenarios, los hepatocitos exhiben una gran plasticidad que les permite desdiferenciarse o transdiferenciarse en respuesta al microambiente y adquirir un fenotipo con características mesenquimales, o bien generar otros tipos celulares como los colangiocitos o inclusive células con características troncales; esto con la finalidad de restaurar la homeostasis del hígado o bien conducir al desarrollo de patologías (Figura 3). Algunos de los contextos fisiológicos y patológicos en donde se manifiesta la plasticidad del hepatocito, se detallan a continuación:
Homeostasis hepática
Los tejidos mantienen su integridad en condiciones homeostáticas a través de la activación de diversos mecanismos como la proliferación o la transdiferenciación de sus células parenquimales, o bien por la diferenciación y proliferación de las células troncales residentes (Stanger, 2015). En el hígado, se ha documentado que existen subpoblaciones de hepatocitos que contribuyen de manera diferencial al mantenimiento de la masa hepática en condiciones fisiológicas o patológicas (Figura 3). Un grupo de investigación propone que los hepatocitos PP son los que generan al resto de los hepatocitos en un modelo que denominan Transmisión o “Streaming” (Zajicek, Oren & Weinreb, 1985), mientras que otro grupo ha sugerido que solamente una subpoblación de hepatocitos PP, denominados híbridos, son capaces de restaurar la masa hepática, pero solamente en contextos como la regeneración hepática inducida por una hepatectomía parcial (Font-Burgada et al., 2015). Por otro lado, Wang y col. en 2015, haciendo estudios de rastreo de linaje realizados por un año en ratones, sugieren que una subpoblación de los hepatocitos PC, positivos a Axina2, mantiene la masa hepática en condiciones de homeostasis (Wang et al., 2015).
Regeneración hepática y la capacidad proliferativa de los hepatocitos
El hígado está expuesto a múltiples tipos de daño, ya sea como resultado de su actividad metabólica, por la exposición a patógenos o por la detoxificación de xenobióticos. Existen diferentes modelos para estudiar la capacidad regenerativa del hígado, ya sea administrando algún compuesto químico o realizando la remoción quirúrgica de una parte del órgano (Kang et al., 2012). Un modelo clásico de daño químico es el tratamiento con tetracloruro de carbono CCl4, en donde la administración aguda o crónica de este compuesto, provoca la muerte de algunos hepatocitos y genera nódulos de regeneración en las áreas de necrosis (Pritchard & Apte, 2015). Cabe resaltar que la restitución de la masa hepática en este modelo, involucra además de la proliferación de los hepatocitos, la proliferación y la diferenciación de las células troncales del hígado (Petersen, Zajac & Michalopoulos, 1998). Otro de los modelos clásicos en el estudio de la regeneración hepática es la PH realizada en roedores. El modelo consiste de la remoción quirúrgica de 2/3 partes del hígado (lóbulos mayores en roedores) y la restauración de la masa hepática entre 2 y 3 semanas después de que se realizó la cirugía (Michalopoulos, 2010; Katoonizadeh, 2017). A diferencia de los modelos químicos, en el modelo de PH la restitución de la masa hepática se lleva a cabo principalmente por la proliferación de los hepatocitos, quienes a pesar de ser considerados como células quiescentes comienzan a proliferar en respuesta al daño y no parece haber una participación de células troncales o progenitoras (Taub, 2004; Kang et al., 2012). Es importante resaltar que en el proceso de regeneración en respuesta a una PH, los hepatocitos atraviesan por un proceso de EMT parcial, que se caracteriza por una disminución en la expresión de marcadores epiteliales como la E-cadherina, así como de marcadores funcionales como la albúmina y el HNF4α, entre otros, así como por el aumento temporal en la expresión de marcadores mesenquimales como la vimentina, la α-sma (del inglés, alpha-smooth muscle actin) y el factor transcripcional Snail, entre otros, (Tabla II y III) (Figura 3). Además, los hepatocitos pueden también producir temporalmente colágena tipo I (Oh, Swiderska-Syn, Jewell, Premont & Diehl, 2018). Este cambio de fenotipo tipo EMT durante la regeneración hepática, parece ser necesario para que los hepatocitos proliferen; sin embargo, se requieren experimentos adicionales para comprender su participación en el proceso de la regeneración hepática (Oh et al., 2018).
Tabla II Marcadores moleculares de hepatocitos con fenotipo mesenquimal en modelos in vitro.
| Contexto | Organismo/Modelo | Marcadores de fenotipo | Referencias | |
| Normal | Rata | Cultivo primario | •Snail | (Franco et al., 2010) |
| Ratón | Línea celular: MMH | •HNF4α | (Cicchini et al., 2006) | |
| Línea celular: AML 12 | •miR30 | (Zhang et al., 2012) | ||
| Fibrosis | Rata | Cultivo primario | •miR21 | (Zhao et al., 2014) |
| Ratón | Cultivo primario | •E-cadherina Albúmina |
(Kaimori et al., 2007; Nitta, Kim, Mohuczy & Behrns, 2008) | |
| Cáncer | •Colágena 1α1 •Actina organizada en fibras de estrés •Vimentina |
|||
| Humano | Muestra de tejido. Líneas celulares: Hep3B, SK-HEP-1, Mahlavu |
•Twist | (Firrincieli, Boissan & Chignard, 2010; Yang et al., 2009) | |
| Humano | Líneas celulares: HuH7, SK-HEP-1, Mahlavu | •miR122 | (Tsai et al., 2009) | |
| Ratón | Línea celular: Hep3B, HepG2, MHCC97 | •miR101 | (Zhao et al., 2015) | |
| Línea celular: AML12 | ||||
| Humano | Muestra de tejido. Líneas celulares: HepG2, SMMC7721, Bel7402, HCCLM3, MHCC97H |
•miR451 | (Huang et al., 2015) | |
Tabla III Marcadores moleculares de hepatocitos con fenotipo mesenquimal en modelos in vivo.
| Contexto | Organismo/modelo | Marcadores de fenotipo | Referencias | |
| Regeneración | Ratón | Hepatectomía parcial | •E-cadherina | (Oh et al., 2018) |
| •Albúmina | ||||
| •α-SMA | ||||
| •Colágena 1α1 | ||||
| •Vimentina | ||||
| •Zeb1 | ||||
| •MMP9 | ||||
| Fibrosis | Ratón | CCl4 (2 veces/semana) /6 semanas | •FSP-1 | (Zeisberg et al., 2007) |
| Ratón | DDC: 4-12 semanas CCl4: (2 veces/semana) /3-9 semanas | •α-SMA | (Gu et al., 2017) | |
CCL4: tetracloruro de carbono; DDC: 3,5-dietoxicarbonil-1,4-dihidrocolidina.
Múltiples señales y vías de señalización orquestan el proceso de regeneración hepática. Se ha demostrado que durante el proceso de regeneración, los niveles de β-catenina aumentan y se promueve la proliferación de los hepatocitos; mientras que si disminuyen los niveles de β-catenina entonces se retrasa el proceso de regeneración (Tan, Behari, Cieply, Michalopoulos & Monga, 2006). La ruta Hippo esta activa en los hepatocitos maduros quiescentes y se inactiva en la regeneración, de manera que los niveles de los correguladores transcripcionales YAP y TAZ se incrementan después de realizada la PH y las proteínas exhiben una localización nuclear. YAP particularmente favorece el proceso de regeneración hepática, al regular positivamente la expresión de genes como Cyr61 y AmotL2 (Grijalva et al., 2014; Lu et al., 2018). La vía de Hh también participa en el proceso de regeneración hepática. Los hepatocitos quiescentes no expresan a los ligandos Hh, pero pueden expresar y secretar a los ligandos Shh e Ihh durante el proceso de regeneración hepática (Ochoa et al., 2010). Además de las vías de señalización antes mencionadas, los factores de crecimiento como HGF (del inglés, Hepatocyte Growth Factor), EGF (del inglés, Epidermal Growth Factor), FGF (del inglés, Fibroblast Growth Factor), VEGF (del inglés, Vascular Endothelial Growth Factor) e IGF (del inglés, Insulin-like Growth Factors) son moléculas que también juegan un papel fundamental en el proceso de regeneración hepática, participando en los procesos de activación, proliferación, diferenciación y sobrevivencia de los diferentes tipos celulares en el hígado (Böhm, Kóhler, Speicher & Werner, 2010).
Desdiferenciación y transdiferenciación de los hepatocitos
La transición Epitelio-Mesénquima (EMT) es un proceso de transdiferenciación reversible que se presenta en las células epiteliales (Nieto, Huang, Jackson & Thiery, 2016). Este proceso se caracteriza por la pérdida o disminución de la expresión de marcadores epiteliales como la E-cadherina, junto con la pérdida de la polaridad y de las uniones celulares; en tanto que las células van adquiriendo marcadores mesenquimales como los factores transcripcionales: Snail1, Snail2, ZEB1, ZEB2, Twist1, Twist2 y Twist3, así como proteínas del citoesqueleto como la vimentina y la α-sma (Moustakas & Heldin, 2016; Gupta & Maitra, 2016) (Tabla II y III). El proceso de EMT ocurre en diferentes contextos fisiológicos o patológicos y se ha clasificado en 3 tipos principales: 1) La EMT tipo 1 ocurre durante el desarrollo embrionario; 2) la EMT tipo 2 se presenta en la reparación de heridas, la regeneración tisular y la fibrosis; 3) la EMT tipo 3 ocurre en los carcinomas y se encuentra asociada a procesos como la invasión y la metástasis (Yoshida, Murata, Yamaguchi, Matsuzaki & Okazaki, 2016).
En tejidos adultos como el hígado, la EMT se presenta en procesos fisiológicos como la cicatrización y la reparación tisular, pero también puede ocurrir en patologías como la fibrosis hepática que al volverse crónica puede conllevar al desarrollo de cirrosis y HCC (Yu, Li, Shi & Li, 2018). La fibrosis hepática puede desencadenarse a partir de daños al hígado de tipo crónico o agudo y durante su evolución se produce un exceso de matriz extracelular en el hígado (ECM, del inglés Extracellular Matrix). Así mismo, la fibrosis puede resultar de enfermedades crónicas como la hepatitis y con el tiempo esta enfermedad puede progresar y derivar en enfermedades como la cirrosis hepática y el HCC (Leask & Abraham, 2004). El principal tipo celular hepático responsable de los grandes depósitos de matriz extracelular son las HSC, quienes una vez que son activadas en respuesta a los cambios en la rigidez de la ECM del órgano se transdiferencian hacia miofibroblastos (MFB, del inglés Myofibroblasts), el cual es un tipo celular que se caracteriza por producir un exceso de proteínas de la ECM (Mannaerts et al., 2015). Las HSC no son la única fuente celular en la generación de MFB y por lo tanto, no son las únicas responsables del aumento en la deposición de la matriz extracelular; de hecho, se ha propuesto que otros tipos celulares del hígado pueden contribuir a la formación de MFB, como por ejemplo las diferentes células epiteliales hepáticas después de pasar por un proceso de EMT, como las células endoteliales, las células del conducto biliar y los hepatocitos (Zeisberg et al., 2007; Taura et al., 2010; Wells, 2010). Cabe mencionar que existe evidencia de que los hepatocitos pasan por un proceso de EMT parcial en un cultivo primario, pero hasta el momento la evidencia de que este proceso ocurra in vivo se mantiene en controversia. El proceso de EMT se ha estudiado en diferentes modelos que van desde el cultivo in vitro hasta modelos in vivo de trazado de linaje. En los modelos in vitro como el reportado por Kaimori et al. 2007, los hepatocitos de organismos murinos mantenidos en cultivo primario, exhiben una morfología alargada tipo fibroblasto y además expresan marcadores de células mesenquimales como la vimentina; sin embargo, el cambio morfológico de los hepatocitos en cultivos in vitro ocurre en condiciones muy diferentes a las del órgano como tal, sugiriendo que el cambio fenotípico en los hepatocitos es resultado del proceso de cultivo de los hepatocitos y no precisamente refleja el comportamiento in vivo. Para evaluar si los hepatocitos presentan una EMT, el grupo de Zeisberg et al., 2007 empleó un modelo de trazado de linaje de los hepatocitos, en el cual las células derivadas de los hepatocitos expresan β-galactosidasa; a estos ratones se les indujo fibrosis con CCL4 y se encontró la colocalización entre el marcador epitelial albúmina y el marcador mesenquimal FSP-1, mediante inmunofluorescencia; sin embargo, aún no hay una demostración contundente de que los hepatocitos adquieren una morfología mesenquimal en un contexto de fibrosis. El grupo de Taura et al., 2010, empleando el modelo de trazado de linaje, generó ratones transgénicos cuyos hepatocitos expresaban a la β-galactosidasa, cuya expresión estaba acoplada a la del promotor de albúmina, en tanto que acopló la expresión de GFP a la del promotor de colágena 1α1, para marcar a las células mesenquimales; sin embargo, no lograron observar colocalizaciones entre los marcadores epitelial (albúmina) y mesenquimal (colágena); por lo que sus estudios no apoyan que los hepatocitos pasen por una EMT en modelos de fibrosis. Sin embargo, se ha reportado que en el contexto de la regeneración hepática, los hepatocitos experimentan un proceso de EMT parcial que es clave para la regeneración (Oh et al., 2018). Por lo tanto queda por investigar si realmente los hepatocitos experimentan un proceso de EMT y en qué contextos fisiopatológicos ocurre.
Transformación de los hepatocitos en el cáncer de hígado o hepatocarcinoma
El daño crónico del hígado relacionado a la cirrosis, la hepatitis B y C, al consumo crónico de alcohol, al tabaquismo, la diabetes, la obesidad, la esteato-hepatitis no alcohólica (NASH) y al consumo de aflatoxinas B1 producidas por hongos del género Aspergilllus, entre otros, puede dar lugar al desarrollo del cáncer de hígado (Balogh et al., 2016; Tunissiolli et al., 2017). Los principales tipos de cáncer hepático son el HCC con un 75-85% de los casos y el colangiocarcinoma con un 10-15% del total de los casos reportados, de acuerdo a las estadísticas globales de cáncer reportadas en 2018 (Bray et al., 2018). Diferentes tipos celulares pueden contribuir al desarrollo del cáncer de hígado, como los hepatocitos, los colangiocitos y las células ovales (consideradas como las células troncales residentes del hígado); sin embargo, los hepatocitos son el principal tipo celular que contribuye a desarrollar HCC (Sell, 2002; Anfuso, El-Khobar, Sukowati & Tiribelli, 2015; Tummala et al., 2017).
El HCC es el tipo de cáncer de hígado más común y el séptimo con más casos en el mundo en 2018 (Balogh et al., 2016; Bray et al., 2018). Se han establecido diferentes modelos de daño químico para el estudio del desarrollo del HCC, empleando compuestos como: el N-2-AAF (del inglés, 2-Acetylaminofluorene), la DEN (del inglés, Diethylnitrosamine), el furano y la AFB1 (del inglés Aflatoxin B1). Cada uno de estos compuestos químicos actúa en zonas específicas del lóbulo hepático y por lo tanto, afectan de manera diferencial a los distintos tipos celulares del hígado (Sell, 2002; Bakiri & Wagner, 2013). De tal manera que, el N-2-AAF y la AFB1 actúan principalmente en la zona periportal, mientras que el DEN actúa en la zona pericentral (Sell, 2002); por otro lado, el furano afecta más al lóbulo caudal del hígado (Elmore & Sirica, 1991). Sin embargo, aún falta esclarecer por qué ciertas zonas o lóbulos del hígado son más susceptibles al daño por compuestos químicos específicos.
Transdiferenciación de los hepatocitos a células biliares en la colestasis
Los hepatocitos también poseen la capacidad para transdiferenciarse directamente en colangiocitos, cuando esta población celular se ve afectada en la colestasis. La colestasis es un padecimiento que se refiere a la obstrucción o disminución del flujo biliar (Cullen, 2010) (Figura 3). Existen diferentes modelos para el estudio de la colestasis, como la ligación de los conductos biliares. La realización de este procedimiento en conjunto con la administración de DAPM (del inglés 4,40-diaminodiphenyl methane) permite tener un modelo en el cual los hepatocitos atraviesan por un proceso de transdiferenciación y dan lugar a nuevos colangiocitos, que formarán nuevos conductos biliares para compensar la obstrucción biliar (Michalopoulos, Barua & Bowen, 2005). Los ratones KO condicionales para la proteína RBPJ son un modelo de colestasis; estos ratones KO mimetizan el síndrome conocido como Alagille (ALG) en humanos, que se caracteriza por una disminución en la cantidad de conductos biliares afectando la función del hígado y se desarrolla una colestasis crónica. Los ratones KO para RBPJ tienen una deficiencia en la formación de los conductos biliares pero el organismo se mantienen vivo debido a que los hepatocitos se transdiferencian hacia colangiocitos y forman conductos biliares funcionales que drenan la bilis y permiten la vida del organismo, aun con un hígado colestático (Schaub et al., 2018). El TGF-β es la principal citocina que regula directamente el proceso de transdiferenciación de los hepatocitos hacia colangiocitos, demostrando así que la plasticidad de un tipo celular es fundamental para aminorar el daño en un órgano como el hígado (Schaub et al., 2018).
Implicaciones fisiopatológicas y terapéuticas de la plasticidad del hepatocito
A partir del conocimiento de que una célula ya diferenciada conserva una plasticidad que le permite convertirse en otro tipo celular, podemos plantear preguntas como: ¿cuáles son los interruptores moleculares para regular el fenotipo de una célula?, ¿cómo estos reguladores podrían contribuir a la homeostasis en condiciones fisiológicas como la regeneración y la reparación de heridas? y ¿cómo es su contribución al desarrollo de patologías como la fibrosis y el cáncer?
Los hepatocitos son células muy versátiles y multifuncionales, que contribuyen al establecimiento de la estructura del hígado y la función hepática, por lo que son sujetos a diversos protocolos de investigación en temas como la regeneración y el desarrollo embrionario del hígado, así como en el estudio del metabolismo de nutrientes y la toxicidad de los medicamentos, entre otros (Shulman & Nahmias, 2013). Los hepatocitos mantenidos en cultivos primarios son uno de los modelos biológicos más usados para estudiar las funciones del hígado; sin embargo, este modelo tiene una gran limitante, ya que los hepatocitos cambian su fenotipo paulatinamente desde que se aíslan y luego se cultivan; es decir, los hepatocitos experimentan un proceso de EMT parcial en cultivo, que los hace adquirir las características de una célula tipo mesenquimal, como se muestran en las Tablas II y III. El periodo de tiempo en el cual los hepatocitos presentan predominantemente sus características epiteliales en cultivo primario es de aproximadamente unas 24-48 h (Cassim, Raymond, Lapierre & Bilodeau, 2017). Ante este problema, diversos grupos de investigación, incluido el nuestro, trabajamos en comprender cómo diversas señales bioquímicas y biomecánicas regulan el fenotipo del hepatocito en cultivo, para poder así diseñar estrategias que permitan mantener su fenotipo por periodos prolongados en un cultivo primario. En este campo se han desarrollado muchas estrategias para conservar el fenotipo epitelial de los hepatocitos, a través de los siguientes tipos de cultivo: en un sandwich de colágena tipo I, con otras células hepáticas, 3D (tridimensional) en organoides, sobre micro-estampado de proteínas específicas y recientemente en microchips, los cuales son microplataformas de cultivo que mimetizan en escalas nanométricas, las condiciones biomecánicas del hígado como la rigidez, el flujo de nutrientes, la presencia de ácidos biliares, la expresión de factores que ayudan al mantenimiento del fenotipo y la función del hepatocito. Además, se están desarrollando plataformas para cultivar y mantener a las diferentes subpoblaciones de hepatocitos (PP y PC), e investigar in vitro cómo regulan diferencialmente el metabolismo de nutrientes (Huh, Hamilton & Ingber, 2011; No, Lee, Lee & Lee, 2015). En general, podemos concluir que una mejor comprensión de la plasticidad del hepatocito beneficiaría el diseño y el desarrollo de estrategias terapéuticas para el tratamiento de enfermedades hepáticas como la fibrosis, la cirrosis y el cáncer hepatocelular.











 nueva página del texto (beta)
nueva página del texto (beta)



