Introducción
as oxigenasas son enzimas oxidoreductasas (EC 1.13 o EC 1.14) que oxidan sus sustratos introduciendo uno (monooxigenasas) o dos (dioxigenasas) átomos de oxígeno a partir del oxígeno molecular. Usualmente en su sitio activo contienen un elemento metálico el cual, a través de cambios en su estado de oxidación, activa al oxígeno para que ocurra la reacción. Fueron descritas por primera vez en un estudio sobre la fenolasa (Mason, Fowlks & Peterson, 1955) y de la pirocatecasa (Hayaishi Katagiri & Rothberg, 1955), enzimas que en microorganismos y animales participan en el metabolismo de ciertos compuestos como el catecol (Kojima et al., 1967) y el L-DOPA (Prabhakaran, Kirchheimer & Harris, 1968). Desde entonces se ha reportado su participación en diversas rutas metabólicas, como la oxidación de compuestos aromáticos en bacterias (Mallick, Chakraborty & Dutta, 2011) y la síntesis y catabolismo de diversos metabolitos en plantas (Mitchell & Weng, 2019).
Este amplio grupo de enzimas se puede clasificar en función del cofactor que utilizan para activar el oxígeno. Están las oxigenasas dependientes de hemo, las dependientes de cobre, las dependientes de FAD-FMN y las dependientes de Fe no hémico, estas últimas se dividen en dos grupos, las enzimas que contienen dos átomos de Fe en su sitio activo y aquellas que solo tienen uno. Las que tienen dos átomos se dividen en cuatro grupos acorde a su estructura (Nordlund & Ekland, 1995; Leahy, Batchelor & Morcomb, 2003); mientras que las enzimas que solo tienen un átomo de Fe varían acorde al donador de electrones de este átomo, este donador puede ser un centro Rieske [2Fe-2S], una molécula de α-cetoglutarato o una molécula de pterina (Bruijnincx, Van Koten & Gebbink, 2008).
Las oxigenasas tipo Rieske/mononuclear, objeto de esta revisión, son enzimas que poseen en su estructura un dominio conteniendo un centro tipo Rieske [2Fe-2S] y un segundo dominio conteniendo un Fe mononuclear no hémico. Estas enzimas dependen de un sistema de transporte de electrones. Fueron descritas por primera vez como parte del sistema de degradación del benzeno en bacterias del género Pseudomonas (Axcell & Geary, 1973 y 1975) y han sido estudiadas porque pueden oxidar diversos compuestos aromáticos xenobióticos, cuya degradación por métodos no enzimáticos es difícil.
Características generales de las oxigenasas Rieske/mononuclear
El centro Rieske [2Fe-2S] fue descrito inicialmente como parte del citocromo bc de una mitocondria (Rieske, MacLennan & Coleman, 1964), en este centro metálico, un átomo de Fe está coordinado por dos residuos de cisteína y el otro átomo de Fe lo está por dos residuos de histidina (Figura 1A), a diferencia de los centros [2Fe-2S] clásicos en los que los cuatro residuos que coordinan a los dos átomos de Fe son cisteínas. Este centro Rieske se encuentra en el dominio que forma el amino terminal de la proteína. Los cuatro residuos de coordinación del Fe, así como la región de unión de este centro, están totalmente conservados en estas enzimas (Van Doren, Gennis, Barquera & Crofts, 1993; Davidson, Ohnishi, Atta-Asafo-Adjei & Daldal, 2005).

Figura 1 Coordinación de los centros metálicos en las oxigenasas Rieske/mononuclear. A) Centro Rieske [2Fe-2S]. B) Fe mononuclear no hémico. Los átomos de azufre y hierro se muestran como esferas de color amarillo y naranja respectivamente. Los residuos de aminoácidos de la proteína que coordinan a los Fe se representan como varillas, con carbonos en color negro, nitrógenos en azul y oxígenos en rojo. La figura se realizó con el software UCSF Chimera (Pettersen et al., 2004). Elaboración personal.
Una característica funcional importante de los centros Rieske es que su potencial de óxido-reducción varía dependiendo de sus interacciones con los aminoácidos que rodean el centro metálico y los residuos que lo coordinan (Colbert, Couture, Eltis & Bolin, 2000). Existen centros Rieske de alto potencial redox como el del citocromo bc y otros de menor potencial como el de las ferredoxinas y el de las oxigenasas Rieske/mononuclear.
El Fe mononuclear no hémico está coordinado por dos residuos de histidina y un residuo de aspártico totalmente conservados (Figura 1B). A este arreglo se le conoce como la tríada 2-His-1-carboxilato, en este dominio es donde tiene lugar la activación del oxígeno y la reacción enzimática no sólo de las oxigenasas Rieske/mononuclear, sino de otros tipos de oxigenasas que tienen esta tríada (Koehntop, Emerson & Que, 2005). El átomo de Fe se coordina de una forma octaédrica con los tres residuos previamente mencionados y tres moléculas de agua, en algunos casos la coordinación con el residuo aspártico es bidentada, uniendo solo dos moléculas de agua, pero conservando la forma octaédrica (Hegg & Que, 1997; Que, 2000).
Las oxigenasas Rieske/mononuclear reciben los electrones del NAD(P)H a través de un sistema de transporte de electrones que en algunos casos consiste de tres componentes: una ferredoxina reductasa, cuyo grupo prostético FAD recibe los electrones del NAD(P)H y los transfiere a una ferredoxina y la oxigenasa, que recibe los electrones de la ferredoxina (Figura 2 A). Mientras que en otros casos consiste en dos componentes: una reductasa dependiente de FMN o FAD y la oxigenasa (Figura 2 B). La estructura de las oxigenasas Rieske/mononuclear consiste en un homotrímero de subunidades α catalíticas (α3) (Figura 3 A) o bien en un heterohexámero formado por tres subunidades α y tres subunidades β (α3β3) (Figura 3 B). La función precisa de la subunidad β es desconocida hasta la fecha, pero se sabe que no es esencial para la actividad catalítica, por lo que se ha propuesto que puede ayudar a la estabilidad estructural del heterohexámero o bien regular la actividad enzimática de las subunidades α (Hurtubise, Barriault & Sylvestre, 1998; Kauppi et al., 1998).

Figura 2 Representación esquemática de los tipos de oxigenasas Rieske/monuclear. (A) de tres componentes y (B) de dos componentes. Los electrones llegan a los centros Fe-S de la oxigenasa de uno en uno, pero se necesitan dos para que ocurra un ciclo catalítico. Elaboración personal.
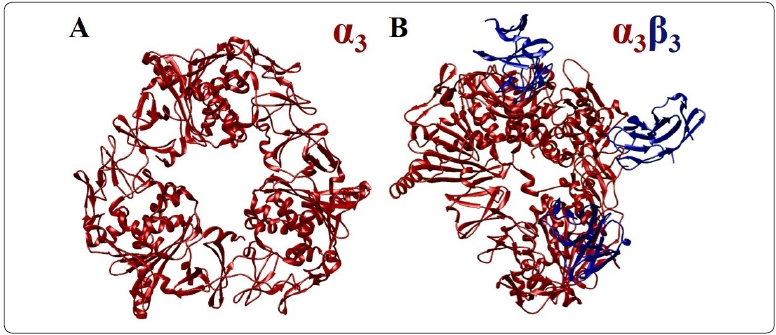
Figura 3 Representación en lazos del heterohexámero de la carbazol dioxigenasa (PDB: 2DE6). (A) Homotrímero formado por subunidades α. (B) Heterohexámero formado por las subunidades α y β. La figura se realizó con el software UCSF Chimera. Elaboración personal.
Como parte del ciclo catalítico, el centro Rieske transfiere electrones al Fe mononuclear para la activación del oxígeno, por lo que dada la gran separación de estos centros metálicos dentro de un mismo monómero (Figura 4 A), la transferencia de electrones tiene lugar desde el centro Rieske de una subunidad al Fe mononuclear de la subunidad vecina que está mucho más cercano (Figura 4 B). Esta transferencia está mediada por un residuo de aspartato o glutamato de la subunidad a la que pertenece el centro Fe mononuclear que recibe el electrón (Parales, Parales & Gibson, 1999). La mutación de cualquiera de los residuos que coordinan a los átomos de Fe del centro Rieske, al Fe mononuclear o del residuo ácido que participa en la transferencia de electrones entre los centros metálicos produce la pérdida de actividad de estas enzimas (Van Doren et al., 1993; Jiang, Parales, Lynch & Gibson, 1996; Parales, Parales & Gibson, 1999).
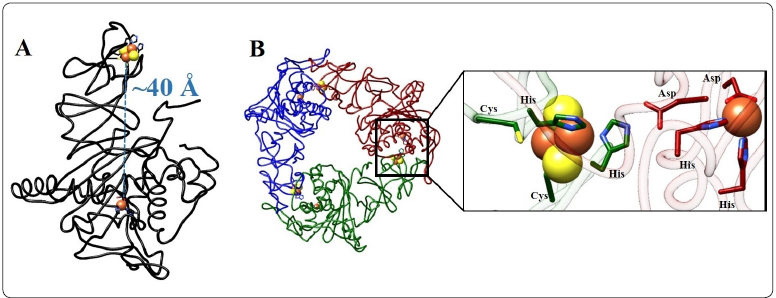
Figura 4 Transferencia de electrones entre los centros metálicos de las oxigenasas Rieske/mononuclear. A) Representación en lazos de un monómero de la oxigenasa de prolina betaína (PDB: 3VCA); con una línea punteada se indica la distancia entre sus dos centros metálicos. B) Representación en lazos del trímero de la oxigenasa de prolina betaína y acercamiento mostrando los residuos de aminoácidos que coordinan a los centros metálicos y del residuo de aspártico que asiste la transferencia de electrones desde el centro Rieske de una subunidad al Fe mononuclear no hémico de su subunidad vecina. Los residuos de aminoácidos se representan como varillas. Los carbonos se muestran en colores verde, azul o rojo cobrizo, dependiendo de la subunidad, los nitrógenos en azul y los oxígenos en rojo. Los átomos de azufre y hierro se muestran como esferas de color amarillo y naranja respectivamente. La figura se realizó con el software UCSF Chimera. Elaboración personal.
Clasificación y distribución filogenética de las oxigenasas Rieske/mononuclear
La base de datos de dominios conservados CDD (Conserved Domain Database, https://www.ncbi.nlm.nih.gov/cdd), hace una clasificación por dominio de las oxigenasas Rieske/mononuclear que permite relacionar la información que se tiene acerca de su estructura, función y evolución. Filogenéticamente, el dominio del centro Rieske pertenece a la familia denominada cd03469_Rieske_RO_alpha_N, que hasta la fecha comprende 16 subfamilias y el domino del Fe mononuclear no hémico pertenece a la familia denominada cd00680_RHO_alpha_C que hasta la fecha comprende 10 subfamilias (Tabla I).
Tabla I Clasificación de las oxigenasas Rieske/mononuclear de acuerdo a la base de datos de dominios conservados. (A) Dominio Rieske, perteneciente a la familia cd03469_Rieske_RO_alpha_N (B) Dominio Fe mononuclear no hémico, perteneciente a la familia cd00680_RHO_alpha_C, En ambos casos se muestran las subfamilias que hasta el momento han sido reportadas.
| Familias | |
|---|---|
| cd03469 Rieske_RO_Alpha_N | cd00680 RHO_alpha_C |
| Subfamilias | |
| cd03472 Rieske_RO_Alpha_BPDO_like | cd08878 RHO_alpha_C_DMO-like |
| cd03479 Rieske_RO_Alpha_PhDO_like | cd08879 RHO_alpha_C_AntDO-like |
| cd03480 Rieske_RO_Alpha_PaO | cd08880 RHO_alpha_C_ahdA1c-like |
| cd03531 Rieske_RO_Alpha_KSH | cd08881 RHO_alpha_C_NDO-like |
| cd03532 Rieske_RO_Alpha_VanA_DdmcC | cd08882 RHO_alpha_C_MupW-like |
| cd03535 Rieske_RO_Alpha__NDO | cd08883 RHO_alpha_C_CMO-like |
| cd03536 Rieske_RO_Alpha_DTDO | cd08884 RHO_alpha_C_GbcA-like |
| cd03537 Rieske_RO_Alpha_PrnD | cd08885 RHO_alpha_C_1 |
| cd03538 Rieske_RO_Alpha_AntDO | cd08886 RHO_alpha_C_2 |
| cd03539 Rieske_RO_Alpha_S5H | cd08887 RHO_alpha_C_3 |
| cd03541 Rieske_RO_Alpha_CMO | |
| cd03542 Rieske_RO_Alpha_HBDO | |
| cd03545 Rieske_RO_Alpha_OHBDO_like | |
| cd04337 Rieske_RO_Alpha_Cao | |
| cd04338 Rieske_RO_Alpha_Tic55 | |
Se ha propuesto otro método de clasificación de estas enzimas que toma como criterio las características estructurales del sistema de transferencia de electrones que alimenta a la oxigenasa y de esta misma. Así, se clasifican según el tipo de reductasa o de ferredoxina con el que trabajan, por ejemplo, si en la reductasa el dominio de unión del FAD/FMN se encuentra en el amino o en el carboxilo terminal o en el caso de la ferredoxina si el centro metálico es de tipo Rieske [2Fe-2S] o del tipo [3Fe-4S] y si la oxigenasa posee o no una subunidad β (Kweon et al., 2008; Chakraborty, Ghosal, Dutta & Dutta, 2012). El componente oxigenasa es el que juega el papel más importante en la diversificación funcional de estas enzimas, por lo que cualquier método de clasificación, debe de considerar de manera primordial a este componente.
Las oxigenasas Rieske/mononuclear están ampliamente distribuidas en bacterias (proteobacterias, actinobacterias, algunas cianobacterias y firmicutes) e incluso en algunas arqueas (Chakraborty et al., 2012) se ha descrito que estas enzimas están presentes también en bacterias termófilas, sin embargo, hasta el momento no se ha reportado la función de alguna de ellas (Chakraborty, Suzuki-Minakuchi, Okada & Nojiri, 2017). La mayoría de las oxigenasas Rieske/mononuclear presentes en bacterias participan en rutas de degradación de compuestos aromáticos policíclicos, como el naftaleno (Ensley, Gibson, & Laborde, 1982; Ensley & Gibson, 1983), carbazol (Sato et al., 1997), bifenil (Haddock & Gibson, 1995), benzoato (Wolfe et al., 2002), cumeno (Dong et al., 2005), oxoquinolina (Rosche, Tshisuaka, Fetzner & Lingens, 1995), ftalato (Batie, Lahaie & Ballous, 1987), benzeno (Mason, Butler, Cammack & Shergill, 1997), tolueno (Jiang, Parales, & Gibson, 1999), entre otros. También se han encontrado oxigenasas Rieske/mononuclear en bacterias que oxidan compuestos no aromáticos como la glicina betaína (Shao, Guo, Zhang, Yu, Zhao & Pang, 2018), el cloruro de benzalconio (Ertekin, Konstantinidis & Tezel, 2017), la prolina betaína (Daughtry et al., 2012) y la carnitina (Zhu et al., 2014).
En eucariontes, estas enzimas no se encuentran tan ampliamente distribuidas. En plantas, hasta la fecha se ha encontrado que participan en la síntesis del osmoprotector glicina betaína (GB) a partir de colina (Rathinasabapathi et al., 1997), en la formación de clorofila b a partir de la clorofila a y en el catabolismo de esta (Tanaka et al., 1998; Pruzinska, Tanner, Anders, Roca & Hortensteiner, 2003). En animales, participan en la degradación del colesterol (Yoshiyama-Yanagawa et al., 2011) y en algunas levaduras en el catabolismo de colina (Linder, 2014).
La historia evolutiva de estas enzimas está relacionada con el de proteínas multidominio, este tipo de proteínas se originan después de que ocurre algún proceso en el genoma que permite la creación de genes nuevos, entre estos procesos está la fusión génica que permite que genes con función distinta se fusionen y den origen a un gen con una nueva función (Long, Betrán, Thornton & Wang, 2003). Aunque encontrar el mecanismo específico por el cual se originó una proteína con dos o más dominios no es sencillo, en el caso particular de las enzimas Rieske/mononuclear la fusión de dos genes que codificaban a las proteínas de función distinta pudiera ser el origen de este tipo de enzimas. El dominio Rieske pertenece a la superfamilia de proteínas que contienen hierro-azufre (ISP por sus siglas en inglés), cuyos miembros tienen un plegamiento similar y por lo tanto este dominio pudo haber surgido de una proteína ancestral cuya función estuviera relacionada con el transporte de electrones (Schmidt & Shaw, 2001; Chakraborty & Dutta, 2011). En el caso del dominio de unión al Fe mononuclear, su plegamiento es parecido al de las proteínas de la superfamilia denominada Betv1, cuyos miembros tienen funciones variadas. En particular se han encontrado similitudes estructurales entre la naftaleno dioxigenasa y la proteína PITP, que no tiene actividad catalítica pero media el intercambio de fosfatidilinositol y fosfatidilcolina entre diferentes membranas (Yoder et al., 2002). Tanto el dominio del Fe mononuclear no hémico de la naftaleno dioxigenasa como la PITP poseen una cavidad central, que en el caso de la PITP es donde se unen los fosfolípidos y en el de la dioxigenasa es donde se une el naftaleno (Bashton & Chothia, 2007). También se ha propuesto que la proteína PA1206 de Pseudomonas es muy parecida estructuralmente al dominio de unión del Fe mononuclear de las oxigenasas Rieske/mononuclear (Chakraborty & Dutta, 2011). Sin embargo, ni PITP ni PA1206 unen iones metálicos y tampoco tienen conservados los residuos de unión del Fe mononuclear, totalmente conservados en las oxigenasas Rieske/mononuclear.
Relaciones estructura-función de las oxigenasas Rieske/mononuclear
Hasta el momento se han reportado estructuras cristalográficas de 13 oxigenasas Rieske/mononuclear: carbazol dioxigenasa (PDB: 2DE6, 1WW9, 3GKQ, 3GCF), dicamba monooxigenasa (PDB: 3GKE), oxoquinolina monooxigenasa (PDB: 1Z01), naftaleno dioxigenasa (PDB: 1NDO, 4HJL, 2HMO, 2B1X), bifenil dioxigenasa (PDB: 1ULI, 2GBW, 3GZX), tolueno dioxigenasa (PDB: 3EN1), cumeno dioxigenasa (PDB: 1WQL), nitrobenceno dioxigenasa (PDB:2BMO), nitrotolueno dioxigenasa (PDB: 5XBP), dioxigenasa de hidrocarburos aromáticos policíclicos (PDB: 2CKF), prolina betaína monooxigenasa (PDB: 3VCA), oxigenasa de 3-cetoesteroide (PDB: 4QDC, 2ZYL) y una oxigenasa Rieske/mononuclear de función desconocida (PDB: 3N0Q). Acorde a la clasificación de la base de datos de dominios conservados del NCBI, los dominios Rieske/mononuclear de la carbazol dioxigenasa y la oxoquinolina monooxigenasa corresponden a los cd03548/cd08878, los de la dicamba monooxigenasa a los cd03532/cd08878, los de la naftaleno dioxigenasa, nitrotolueno dioxigenasa y nitrobenceno dioxigenasa a los cd03535/cd08881, los de la bifenil dioxigenasa, tolueno dioxigenasa, cumeno dioxigenasa y la dioxigenasa de hidrocarburos aromáticos policíclicos a los cd03472/cd08881, los de la prolina betaína monooxigenasa y la oxigenasa de función desconocida a los cd03469/cd08884 y los de la oxigenasa del 3-cetoesteroide al cd03531 sin tener aún un cd asignado para el dominio del Fe mononuclear no hémico. Es importante mencionar que las oxigenasas con los mismos dominios conservados y misma estructura tridimensional cumplen diferentes roles fisiológicos es decir, presentan una plasticidad funcional, por ejemplo la oxoquinolina monooxigenasa y la carbazol dioxigenasa tienen la misma estructura tridimensional (Figura 5 A), pero están codificadas por distintos genes y cumplen roles fisiológicos distintos, la carbazol dioxigenasa tiene una amplia especificidad por el sustrato, sin embargo, no se ha estudiado si es capaz de oxidar la oxoquinolina (Nojiri et al., 1999), hasta el momento se desconoce la especificidad por el sustrato de la oxoquinolina monooxigenasa. La misma situación ocurre en el caso de la bifenil dioxigenasa y la cumeno dioxigenasa (Figura 5 B). La plasticidad funcional ha sido descrita para otras enzimas como las deshidrogenasas/reductasas de cadena media (Riveros-Rosas & Julián-Sánchez, 2006), esta plasticidad funcional en las oxigenasas Rieske/mononuclear con el mismo dominio conservado se pudiera deber al grado de conservación de los residuos que conforman el sitio activo, en el caso de la carbazol dioxigenasa y naftaleno dioxigenasa (Figuras 6 A y 6 B), los residuos que se encuentran a 5 Å del sitio de unión del sustrato tienen un bajo de grado de conservación, pudiendo explicar las diferentes funciones que cumplen las enzimas con los dominios cd03548/cd08878 y cd03535/cd08881. Sin embargo, en el caso de la prolina betaína monooxigenasa (cd03469/cd08884), el grado de conservación de los residuos es más elevado, se conocen dos enzimas con los mismos dominios cuya función es distinta a la de prolina betaína monooxigenasa, la glicina betaína monooxigenasa y cloruro de benzalconio monooxigenasa (Erketin et al., 2017; Shao et al., 2018).
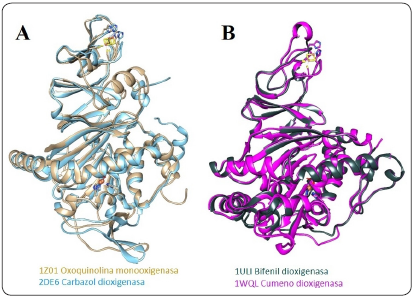
Figura 5 Diferente función, misma estructura. Comparación estructural de diferentes oxigenasas Rieske/mononuclear con los mismos dominios conservados pero diferente función. Oxoquinolina monooxigenasa y Carbazol dioxigenasa tienen los dominios conservados que pertenecen a las subfamilias cd03548/cd08878 mientras que la bifenil dioxigenasa y la cumeno dioxigenasa tienen los dominios conservados que pertenecen a las subfamilias cd03472/cd08881. La figura se realizó con el software UCSF Chimera. Elaboración personal.
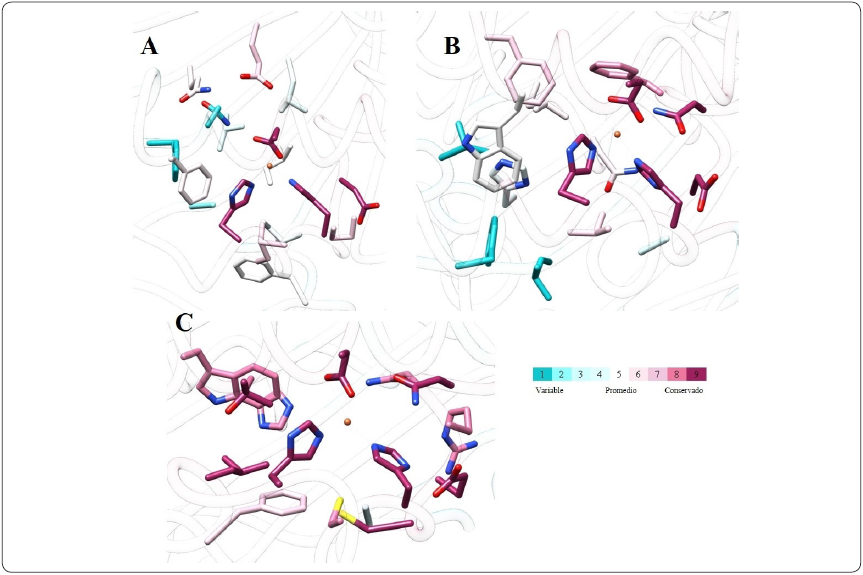
Figura 6 Conservación de aminoácidos en oxigenasas Rieske/mononuclear. Representación esquemática del sitio activo de tres oxigenasas cuya estructura se obtuvo con el sustrato unido al sitio activo, (A) PDB:2DE7 Carbazol dioxigenasa cd03548/cd08878), (B) PDB:1O7G Naftaleno dioxigenasa cd03535/cd0888, (C) PDB:3VCP Prolina betaína monooxigenasa cd03469/cd088841, en donde se muestran en varillas los aminoácidos a 5 Å de distancia del sitio de unión del sustrato. Los nitrógenos se muestran en azul, oxígenos en rojo y azufres en amarillo. El átomo de hierro se muestra como esfera de color naranja. La escala de conservación se muestra en la imagen como barra de colores. La figura se realizó con el software UCSF Chimera. El análisis de conservación de residuos se realizó con el programa Consurf (Ashkenazy et al., 2016). Elaboración personal.
La especificidad por el sustrato de las oxigenasas Rieske/mononuclear es un aspecto que ha sido estudiado en algunas de ellas, por ejemplo, la bifenil dioxigenasa de Pseudomonas sp. LB400 es capaz de oxidar mayor variedad de bifenilos policlorados que la de Pseudomonas pseudoalcaligenes KF707, (Gibson, Cruden, Haddock, Zylstra & Brand, 1993), las enzimas son diferentes entre ellas en sólo 20 residuos, la mutación de un solo residuo en el sitio activo de la enzima de P. pseudoalcaligenes KF707 permite cambiar de manera considerable la especificidad por el sustrato de esta (Suenaga, Goto & Furukawa, 2006). La carbazol dioxigenasa tiene la capacidad de oxidar compuestos que son estructuralmente parecidos al carbazol pero en algunos casos como el fluoranteno no lo son y también los oxida, esta enzima puede incluso oxidar al bifenilo (Nojiri et al., 1999), esta última oxidación también la lleva a cabo la naftaleno dioxigenasa, dependiendo del sustrato, esta enzima puede llevar a cabo reacciones de monooxigenación, desaturación, sulfoxidación y desalquilación confiriéndole una capacidad de oxidación de compuestos mucho más amplia (Resnick, Lee & Gibson, 1996). La especificidad por el sustrato de estas enzimas está determinada principalmente por los residuos que forman el sitio de unión de este, la forma en que potenciales sustratos se unan a este y el cómo la unión de estos afecte el ambiente del Fe mononuclear. Una afectación en este ambiente puede causar cambios en la carga de este átomo y modificar la manera en que el sustrato o potencial sustrato se una al sitio, causando también cambios en la estabilidad y/o forma de unión de los intermediarios de la reacción enzimática (Wang, Li & Liu, 2017), otro factor importante es el tamaño del sitio, por ejemplo, la oxigenasa de compuestos aromáticos policiclicos de Sphingobium sp. PNB tiene un sitio de unión al sustrato bastante más grande que el de otras oxigenasas, lo que le permite oxidar compuestos de diversos tamaños (Khara, Roy, Chakraborty, Dutta & Dutta, 2018).
Retos en el uso y aplicación de las oxigenasas Rieske/mononuclear
Es importante tener en cuenta que la intención de usar en procesos de tipo industrial a cualquier biocatalizador como lo son las enzimas conlleva ciertos retos. Respecto a las oxigenasas, Van Beilen, Duetz, Schmid & Witholt (2003), mencionan que se deben de considerar aspectos como la capacidad de obtener de manera heteróloga a la enzima, la cantidad de enzima recombinante que se puede obtener, la eficiencia catalítica, la eficiencia de unión del sustrato y producto de la reacción enzimática, la transferencia de oxígeno en el medio y su disponibilidad para la enzima, la posibilidad de que los productos de la reacción enzimática sean oxidados de nuevo, la disponibilidad de cofactores y la solubilidad de los sustratos. En el caso de las oxigenasas Rieske/mononuclear, dado que la mayoría de estas enzimas son de origen procarionte los problemas respecto a la obtención de manera heteróloga de estas enzimas y la cantidad de enzima recombinante pudieran ser aspectos fáciles de atender, de hecho, enzimas como la ftalato dioxigenasa de Burkholderia cepacia y la carbazol dioxigenasa de Pseudomonas stutzeri han logrado ser obtenidas de forma recombinante a niveles de gramos por litro en cultivos líquidos (Jaganaman, Pinto, Tarasev & Ballou, 2007; Larentis, Sampaio, Martins, Rodrigues & Alves, 2011). A través de la ingeniería de proteínas se ha logrado que algunas oxigenasas Rieske/mononuclear utilicen un mayor número de moléculas y se ha logrado también aumentar la eficiencia catalítica (Özgen & Schmidt, 2019). Un factor que pudiera ser importante a considerar es la disponibilidad de cofactores, dada la naturaleza del sistema de transferencia de electrones de estas enzimas, es necesario contar con cofactores como el NAD(P)H y el FMN/FAD, estas moléculas son costosas y por lo tanto supondrían una limitante en el uso y aplicación de las oxigenasas Rieske/mononuclear, una alternativa sería el uso de cofactores sintéticos menos costosos que mimetizan la función de los cofactores biológicos (Zachos, Nowak & Sieber, 2019), en ambos casos es importante considerar que es necesario reducir estas moléculas, existen diversos métodos probados mediante los cuales se puede lograr ésto (Wu et al., 2013), un escenario ideal sería aquel en donde los centros metálicos de la oxigenasa pudieran ser reducidos directamente sin la necesidad de los otros componentes del sistema de transferencia de electrones, en este sentido, compuestos químicos como la ditionita de sodio pueden lograr este fin, sin embargo, el costo de su uso de nueva cuenta impone una limitante.
Potenciales aplicaciones biotecnológicas de las oxigenasas Rieske/mononuclear
Las oxigenasas Rieske/mononuclear de bacterias han sido utilizadas para sintetizar dos compuestos con alto valor comercial: el colorante índigo, muy usado en la industria textil, y el fármaco indinavir, un inhibidor de una proteasa del VIH con aplicaciones terapéuticas. La síntesis química del colorante índigo, además de ser laboriosa, utiliza y produce compuestos químicos altamente contaminantes (Bialy, 1997). La biosíntesis de este compuesto se basa en la oxidación del indol (un intermediario de la ruta de biosíntesis del triptófano, Ensley et al., 1983) y no solo produce menos compuestos contaminantes, sino que económicamente es más conveniente que la síntesis química. Un importante intermediario en la síntesis del indinavir se puede obtener mediante la enzima tolueno dioxigenasa (Wackett, Kwart & Gibson, 1988).
Probablemente el área en la que la participación de las oxigenasas Rieske/mononuclear podría destacar más es en la de biorremediación, en particular las oxigenasas de bacterias. A pesar de la capacidad de estas enzimas de oxidar compuestos policíclicos aromáticos, hay poca información acerca de su aplicación o uso directo para remediar áreas contaminadas con dichos compuestos. Sin embargo, en lodos activados de plantas de tratamiento de aguas se ha encontrado que la comunidad de microorganismos presente posee una diversidad de oxigenasas importante que permite a esta comunidad degradar compuestos aromáticos (Jadeja, More, Purohit & Kapley, 2014), incluyendo bacterias previamente desconocidas con capacidad para degradar compuestos aromáticos (Li et al., 2017). Se han estudiado comunidades de microorganismos en áreas contaminadas con diferentes compuestos, encontrándose que hay una importante diversidad de microorganismos cultivables y no cultivables que forman parte de dichas comunidades y que poseen una gran cantidad de genes de oxigenasas Rieske/mononuclear, muchos de los cuales codifican para oxigenasas de las que no se han descrito homólogos cercanos (Ni Chadhain, Norman, Pesce, Kukor & Zylstra, 2006). Estas comunidades pueden ser estimuladas añadiendo algún compuesto en particular como el naftaleno, lo que permite la selección de microorganismos con una mayor capacidad para degradar este compuesto (Ni Chadhain et al., 2006). Además, estas comunidades son muy estables durante períodos prolongados de tiempo, lo cual es ideal para la biorremediación de áreas altamente contaminadas (Yagi & Madsen, 2009) y pueden tolerar cambios en el ambiente causantes de estrés abiótico, tales como, el aumento en la salinidad de los suelos (Li et al., 2012). Dada la gran cantidad de secuencias de oxigenasas Rieske/mononuclear recientemente obtenidas existe la oportunidad de encontrar nuevas funciones, enzimas con una mayor eficiencia catalítica o una especificidad por el sustrato más amplia (Martin, Malagnoux, Violet, Jakoncic & Jouanneau, 2012).
Otra área de desarrollo potencial de aplicación de las oxigenasas Rieske/mononuclear tiene como base la enzima que participa en el catabolismo de la cafeína en Pseudomonas putida CBB5, una bacteria que puede incluso utilizar la cafeína como única fuente de carbono y nitrógeno (Summers et al., 2012). A pesar de que la capacidad de ciertos microorganismos de metabolizar cafeína ha sido estudiada por bastante tiempo, la participación de las oxigenasas Rieske/mononuclear no había sido establecida hasta hace poco tiempo y ello plantea la posibilidad de utilizar estas enzimas o incluso los microorganismos que poseen los genes para las mismas, para llevar a cabo procesos como el bio-descafeínado, biorremediación de suelos contaminados con cafeína o síntesis de metabolitos con valor comercial que forman parte del catabolismo de la cafeína (Summers, Mohanty, Gopishetty, & Subramanian, 2015).
En conclusión, las oxigenasas Rieske/mononuclear son enzimas con una amplia diversidad funcional y dado que su aparentemente más extendido rol fisiológico es la oxidación de compuestos aromáticos policíclicos tóxicos o de interés industrial, constituyen una potencial herramienta biotecnológica que hasta el momento no se ha explotado. Otra área de potencial desarrollo es el uso de estas enzimas para la síntesis de compuestos importantes para diferentes industrias o en el área de la salud. El número de genes y secuencias de proteínas de enzimas Rieske/mononuclear son mucho más grandes que el número de enzimas bioquímica o estructuralmente caracterizadas o con función comprobada hasta el momento, lo cual es una gran ventana de oportunidad para seguir estudiando este importante grupo de enzimas.











 nueva página del texto (beta)
nueva página del texto (beta)


