Introducción
Rotavirus de la especie A
Los rotavirus de la especie A (RVA) son la causa más importante de las gastroenteritis virales en mamíferos alrededor del mundo, y en el caso de los RVA en humanos su relevancia en la prevalencia de esta enfermedad es igual en países desarrollados y en vías de desarrollo (Fields, Knipe & Howley, 2013). Durante el año 2000, se estima que los RVA causaron 528,00 muertes en niños menores de 5 años, y tras la aplicación de vacunas (RotaTeq y Rotarix) hubo una reducción a 215,00 muertes estimadas durante el año 2013 (Tate, Burton, Boschi-Pinto & Parashar, 2016). Los RVA pertenecen al género Rotavirus dentro de la familia Reoviridae, y comparten sus características morfológicas (Matthijnssens, et al., 2011). El genoma viral se divide en 11 segmentos del RNA de doble cadena y codifica para 12 proteínas; seis estructurales (VP 1-4, 6 y 7) y seis no estructurales (NSP 1-6) (Desselberger, 2014). La partícula viral icosaédrica carece de envoltura lipídica y mide 100 nm incluyendo las espículas, está compuesta de tres capas concéntricas de proteínas (TLP, por sus siglas en inglés Triple-Layered Particle), donde la capa externa se compone por VP4 y VP7, la intermedia por VP6 y la interna por VP2 (Jayaram, Estes & Prasad, 2004). El genoma viral se encuentra asociado a VP1 (RNA polimerasa dependiente del RNA) y a VP3 (la guanilil-metil-transferasa) dentro del virión (Desselberger, 2014).
Las TLPs reconocen a los receptores celulares mediante VP4 que previamente es procesada por cortes proteolíticos con tripsina para producir a VP5 y VP8 (Dormitzer et al., 2002a; Dormitzer, Sun, Wagner & Harrison, 2002b). El virus es internalizado por endocitosis o por penetración directa, y la capa más externa se disocia por baja concentración de Ca2+ en los endosomas, y se libera la partícula de doble capa (DLP) (Ludert, Michelangeli, Gil, Liprandi & Esparza,1987). Las DLP libres en el citoplasma son transcripcionalmente activas mediante VP1 y VP3. Los transcritos virales sintetizados emergen de la DLP, y poseen estructura tipo cap en el extremo 5’, similar a los RNA mensajeros celulares, y en el extremo 3’ tienen la secuencia conservada UGACC presente en todos los genes de los RVA (Desselberger, 2014). La proteína NSP3 recluta a los RNA mensajeros virales y se une al factor de inicio de la traducción 4G (eIF4G), lo que resulta en una inhibición de la traducción celular y la facilitación de la síntesis de proteínas virales; esto probablemente por limitar la circularización de los RNAm celulares con cap y poliadenilados y favorecer la circularización de los RNAm virales con cap y la secuencia UGACC-3’ (Piron, Vende, Cohen & Poncet, 1998; Poncet, Aponte & Cohen,1993; Vende, Piron, Castagné & Poncet, 2000). Además, en el citoplasma de la célula infectada se forman cuerpos de inclusión llamados viroplasmas compuestos por NSP2 y NSP5, en los que se lleva a cabo la replicación del genoma y la morfogénesis viral (Silvestri,Taraporewala & Patton, 2004). Las partículas virales inmaduras de los viroplasmas migran hacia el retículo endoplásmico, a través de interaccionar con NSP4 (Taylor, O’Brien & Yeager, 1996). Las partículas virales en el retículo endoplásmico adquieren una envoltura lipídica transitoria y se integran a las proteínas externas del virión, VP4 y VP7 (Estes & Greenberg, 2013). La partícula viral completa es liberada por lisis celular o asociada a vesículas procedentes del retículo endoplásmico (Crawford et al., 2017).
Virus y traducción celular
Los virus se comportan como parásitos intracelulares obligados debido a que utilizan gran variedad de funciones celulares durante la replicación viral. Los virus producen proteínas multifuncionales, pero no han desarrollado una maquinaria de síntesis de proteínas similar a los ribosomas. Durante el ciclo de replicación viral se modula la actividad de los ribosomas para favorecer la traducción de los RNA mensajeros virales y sintetizar las proteínas involucradas en la replicación del genoma y el ensamble de la partícula viral (Walsh et al., 2012). Fallas durante la traducción afectan la replicación viral por lo que los virus han desarrollado estrategias para controlar la actividad de los complejos de la traducción. Además, el control de la traducción celular por los virus ha aportado información muy relevante sobre los mecanismos de regulación de la maquinaria de síntesis de proteínas (Walsh et al., 2012).
La traducción celular es un mecanismo de regulación post-transcripcional de la expresión génica, y altera rápidamente la abundancia espacio-temporal de las proteínas celulares. El proceso de traducción se divide en tres fases: iniciación, elongación y terminación, donde la iniciación es el paso con mayor regulación e involucra el reclutamiento del RNA mensajero a los ribosomas (Hinnebusch & Lorsch, 2012; Iwasaki et al., 2016).
Previo al reclutamiento de los RNA mensajeros en los ribosomas celulares, se forma el complejo de preiniciación 43S compuesto por la subunidad ribosomal menor 40S, factores eucariontes de inicio de la traducción (eIF) eIF1, eIF1A, eIF5, el complejo multiproteíco eIF3 y el complejo ternario eIF2-GTP-RNA de transferencia iniciador cargado con metionina (Met-tRNAi). El RNA mensajero se une a eIF4E mediante el cap en el extremo 5’, y a PABP mediante el extremo 3’ poliadenilado, lo que da como resultado la posible circularización de los RNAm celulares con cap y poliadenilados (Wells, Hillner, Vale & Sachs, 1998). El factor eIF4G funciona como andamio entre eIF4E y PABP además de asociarse a una helicasa del RNA (eIF4A) (Jackson, Hellen & Pestova, 2010). Estas interacciones promueven la traducción del RNA mensajero, que por el efecto protector de PABP es menos susceptible a la degradación en el citoplasma celular (Vicens & Westhof, 2014; Wells, Hillner, Vale & Sachs,1998). También el factor 4G interacciona con eIF3 unido a la subunidad ribosomal menor 40S y se previene la formación del ribosoma completo 80S, hasta que el codón de inicio de la traducción en el RNA mensajero sea ubicado (Figura 1A) (Hinnebusch, 2014). El reconocimiento del codón inicio da lugar a la hidrólisis de GTP del complejo ternario, promovida por eIF5, liberando así a los factores de inicio de la traducción y permitiendo la unión entre las subunidades ribosomales (40 y 60S) lo que permite el inicio de la síntesis del péptido que luego se regula por factores de la elongación (EF-Elongation Factors). La formación del enlace peptídico es catalizada por el RNA ribosomal (RNAr). La síntesis del péptido concluye cuando algún codón de término (UAA, UAG o UGA) es detectado por el factor eRF1 (Eukaryotic release factor 1), y la unión entre el RNA de transferencia y el péptido sintetizado es hidrolizada (Walsh & Mohr, 2011).
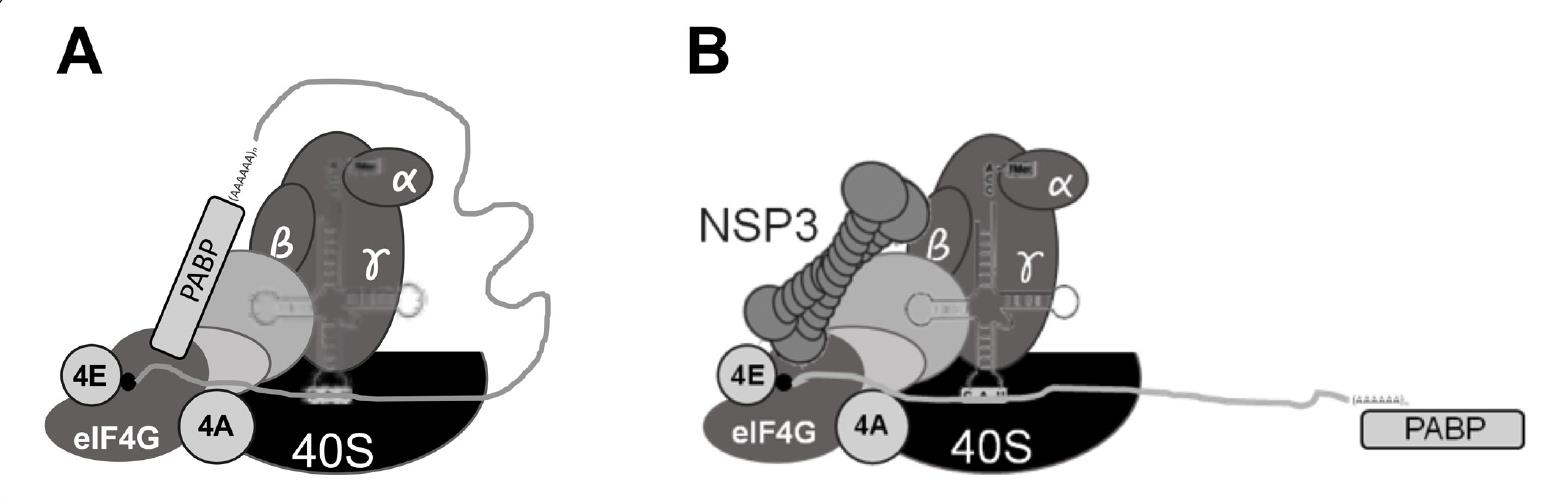
Figura 1: Modelo de la iniciación de la traducción y del efecto de NSP3 para desplazar a PABP. (A) La circularización de los RNA mensajeros celulares se favorece por la unión de eIF4E con el extremo 5’ y PABP con el extremo 3’, mientras que eIF4G funciona como andamio al unirse a eIF4E y PABP, y además recluta a una helicasa del RNA (eIF4A). Finalmente, eIF4G se une al complejo de preiniciación 43S (subunidad ribosomal 40S, eIF2, eIF3 y eIF5) para la búsqueda del codón de inicio óptimo para la traducción celular. (B) NSP3 se une a eIF4G en el mismo sitio que PABP, lo que lineariza los RNAm mensajeros reduciendo su eficiencia para ser traducidos.
Diversos virus previenen el reclutamiento de los ribosomas hacia los RNA mensajeros celulares, por secuestro y modificación de algunos factores involucrados en la traducción celular. El RVA reduce notablemente la síntesis de proteínas celulares desde tiempos tempranos de la infección, favoreciendo la traducción de los mensajeros virales. NSP3 es el regulador de la expresión génica de los RVA a nivel traduccional debido a que reconoce específicamente a los mensajeros virales, y se une a eIF4G en el mismo sitio y con mayor afinidad que lo hace PABP (Figura 1B) (Fields, Knipe & Howley, 2013; Piron, Vende, Cohen & Poncet,1998; Poncet, Laurent & Cohen, 1994).
Gen y estructura de NSP3 (dominios)
La proteína no estructural 3 (NSP3, Non Structural Protein 3) es producto del gen 7 de los RVA (cepa SA11), y muestra identidad entre 46 y 85 % para los 12 genotipos propuestos para NSP3 (Ito, Sugiyama, Masubuchi, Mori & Minamoto, 2001; Matthijnssens et al., 2011; Matthijnssens et al., 2008). En la cepa de RVA SA-11, la longitud del gen es 1,104 pb con regiones no traducidas de 25 y 131 nucleótidos en 5’ y 3’, respectivamente; y codifica para una proteína de 315 aminoácidos (Ito, Sugiyama, Masubuchi, Mori & Minamoto, 2001). Variantes genéticas de NSP3 presentan mutaciones no conservadas y duplicación de dominios generados por recombinación, posicionados dentro o fuera del marco abierto de lectura (ORF, del inglés Open Reading Frame) de NSP3. La mayoría de estas variaciones se producen in vitro por cultivo del virus a elevadas multiplicidades de infección (Arnold, Brownback,Taraporewala & Patton, 2012). La proteína de 315 aminoácidos se divide en tres dominios principales: en el extremo amino terminal (aminoácidos 4-149) se une el RNAm viral, en el extremo carboxilo terminal se une el factor eIF4G (aminoácidos 206-314), y en los aminoácidos 150 al 205 se localiza la región mínima para la dimerización (Piron, Delaunay, Grosclaude & Poncet, 1999). La parte central de la proteína se conforma de una región larga del tipo coiled coil (aminoácidos 159-245), y se caracteriza por un patrón hepta-repetido (HPPHPPP)n con residuos hidrofóbicos (H) y polares (P) (Piron, Delaunay, Grosclaude & Poncet,1999). Además, en el coiled coil se unen dos proteínas celulares: HSP90 y RoXaN (aminoácidos 225-258 y 167-237, respectivamente) (Dutta et al., 2011; Poncet, Laurent & Cohen, 1994). En la Figura 2 se muestran los dominios y regiones de NSP3.
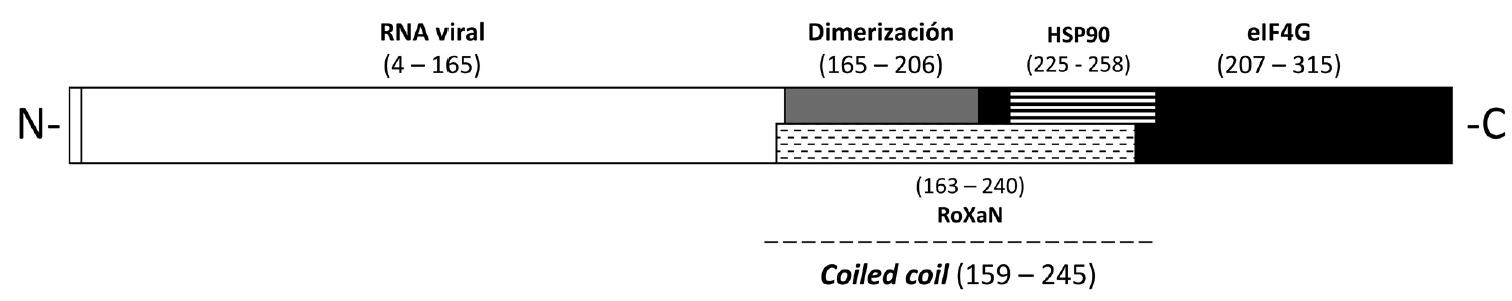
Figura 2: Representación esquemática de NSP3. NSP3 posee 315 residuos y 34 kDa de masa molecular. El extremo amino terminal (4-164) une específicamente al RNAm viral a través de la secuencia conservada en 3’ (GACC) y el extremo carboxilo terminal (207-315) se une al factor de inicio de la traducción 4G. La parte central (165-206) corresponde a la región mínima para dimerizar. Los dominios de unión a las proteínas celulares HSP90 (225-258) y RoXaN (163-240) se encuentran inmersas en una región extensa del tipo coiled coil (159-245).
El dominio de unión al RNA viral (aminoácidos 4-164) se cristalizó en conjunto con un hexanucleótido (5-GUACC-3’) y se resolvió su estructura a una resolución de 2.45 Å. Así se determinó que el dominio de unión al RNA es un heterodímero asimétrico con una interfaz extensa y características polares que forman un túnel donde se une una sola molécula del RNA viral (Figura 3) (Deo, Groft, Rajashankar & Burley, 2002). Por otro lado, el dominio de unión a eIF4G (aminoácidos 206-315) se cristalizó fusionado a GST con un péptido del factor 4G (aminoácidos 132-160) y se observa como un homodímero simétrico con dos sitios disponibles para unir a eIF4G. El resto de NSP3 no se ha cristalizado, no obstante, en la cristalografía del extremo carboxilo se observa una región larga conformada por alfa hélices que confirman la existencia de una parte de la región coiled coil predicha por herramientas bioinformáticas (Figura 4) (Groft & Burley, 2002).
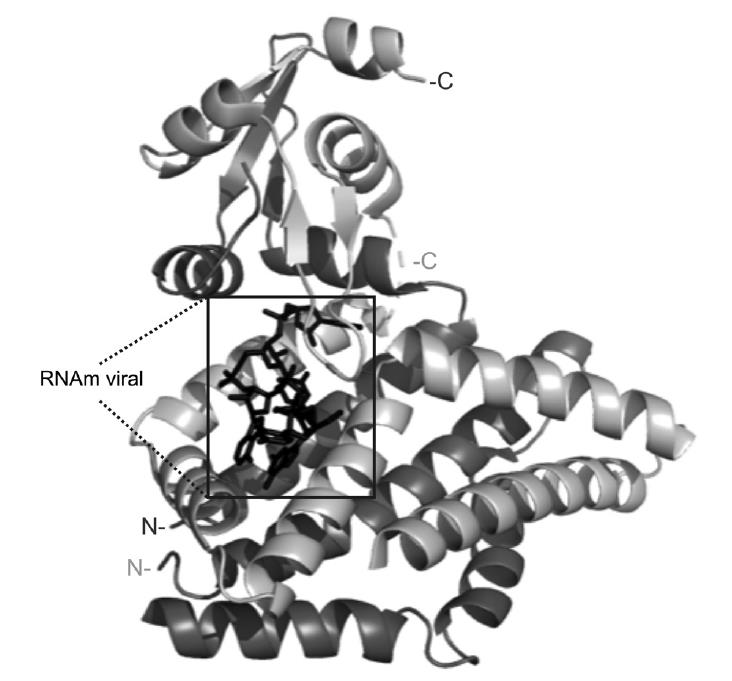
Figura 3: Estructura cristalográfica del extremo amino terminal de NSP3. El dominio de unión al RNA viral (4-164) se muestra co-cistralizado con el hexaribonucleótido consenso (5’-GUGACC-3’) presente en el extremo 3’ de los mensajeros del RVA. Una sola molécula de RNAm viral estabiliza al homodímero asimétrico. Monómeros de NSP3 (gris). Hexaribonucleótido (negro). PDB 1KNZ.
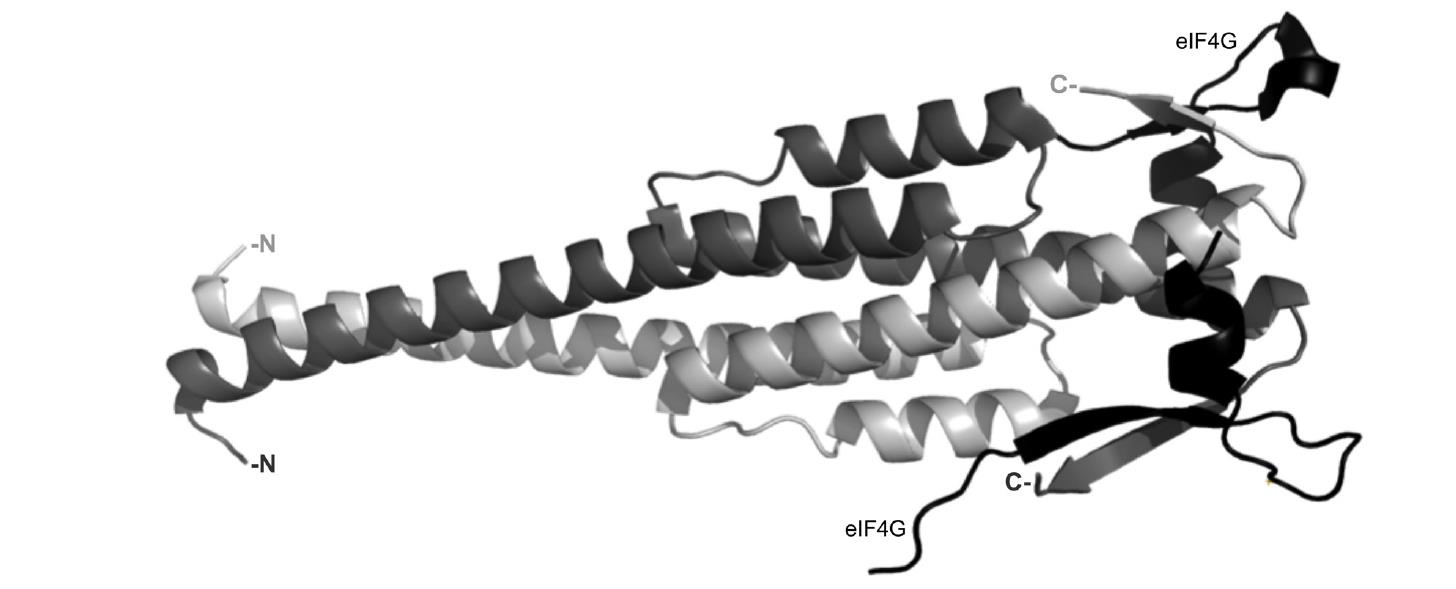
Figura 4: Estructura cristalográfica del extremo carboxilo terminal de NSP3. El dominio de unión al factor eIF4G (206-315) se muestra como homodímero simétrico unido a un fragmento de eIF4G (132-160). El dímero de NSP3 muestra dos posibles sitios de unión a eIF4G. Monómeros de NSP3 (gris). Fragmento de eIF4G (negro). PDB 1CZQ.
Interacciones de NSP3
El dominio de unión al RNA en el extremo amino terminal de NSP3 (NSP3-N) reconoce la secuencia conservada UGACC presente en la región 3’ terminal no traducida (UTR, por sus siglas en inglés Unstraslated Region) de los RNAm de RVA (Poncet, Laurent & Cohen,1994). Las interacciones entre NSP3-N/RNAm viral son electrostáticas con los residuos Asp100 y Leu104 en un monómero, así como Ala122’ y Leu142’ en el otro monómero, y es necesario que el extremo 3’ del RNA esté libre. La interacción con los últimos cuatro nucleótidos es indispensable, dado que variaciones de tres nucleótidos pierden afinidad por NSP3. Experimentos de dicroísmo circular (DC) muestran que NSP3 unido al RNA viral es estable por un aumento del contenido de estructuras alfa hélice (Poncet, Laurent & Cohen,1994). Por ensayos de entrecruzamiento con luz ultravioleta (UV cross-linking, por sus siglas en inglés), se demostró que los extremos del dominio de unión al RNA cooperan con la unión de las moléculas del RNA viral (Piron, Delaunay, Grosclaude & Poncet,1999). Se piensa que la interacción entre NSP3 y los RNA mensajeros virales es indispensable para la traducción eficiente así como el codón de inicio (AUG), y longitudes mínimas en los extremos UTR 5’ y 3’ (5 y 11 nucleótidos, respectivamente) (Gratia et al., 2016).
El extremo carboxilo terminal de NSP3 (NSP3-C) interactúa con eIF4G, y evita que PABP sea reclutado en los complejos traduccionales, y esta interacción es 10 veces más afín que PABP-eIF4G, por lo que in vitro NSP3 desplaza a PABP de su unión a eIF4G (Groft & Burley, 2002; Piron, Vende, Cohen & Poncet, 1998). La interfaz entre NSP3 y eIF4G se lleva a cabo mediante interacciones hidrofóbicas y polares (Figura 4) (Groft & Burley, 2002). Además, un complejo ternario se forma entre NSP3 con eIF4G y RoXaN, y la proteína PABP es relocalizada en el núcleo celular. Mutaciones que afectan la interacción entre NSP3-eIF4G son incapaces de inhibir la traducción celular y para relocalizar a PABP-C en el núcleo (Harb et al., 2008; Padilla-Noriega, Paniagua & Guzmán-León, 2002). Además, la unión de eIF4G hacia NSP3 favorece la traducción de RNA mensajeros celulares y virales (Keryer-Bibens et al., 2009; Vende, Piron, Castagné & Poncet, 2000).
Adicionalmente, la chaperona HSP90 y la proteína celular RoXaN (Rotavirus X Protein Associated to NSP3) interaccionan con la región coiled coil en la parte central de NSP3, una región de la proteína que también contiene al dominio mínimo de dimerización (Figura 2). HSP90 se une a los monómeros recién sintetizados y asiste a la dimerización de la proteína tras la hidrólisis de ATP. La interacción de HSP90 con NSP3 produce intermediarios de la dimerización y evita la degradación por el proteasoma. El bloqueo de la actividad ATPasa de HSP90 por 17DMAG inhibe la dimerización y facilita la degradación de NSP3, y se impide la localización nuclear de PABP-C así como la interacción con eIF4G (Dutta et al., 2011) RoXaN es una proteína de unión al RNA, y fue descrita por la interacción con NSP3 mediante dominios LD similar a los presentes en la proteína paxilina. Interacciones entre residuos con carga e hidrofóbicos participan en la interacción entre NSP3 y RoXaN, por lo que cambios en la naturaleza de estos residuos afectan la formación del complejo ternario eIF4G-NSP3-RoXaN, así como la relocalización de PABP-C al núcleo (Harb et al., 2008; Leppek & Stoecklin, 2014; Vitour et al., 2004).
Función inhibitoria de la traducción celular de NSP3
El control de la maquinaria de traducción es indispensable durante el ciclo de replicación de los RVA; esto disminuye la síntesis de proteínas celulares y promueve la traducción de proteínas virales. NSP3 inhibe la traducción celular al interactuar con eIF4G, lo que se piensa que inhibe la circularización de los extremos 5’-3’ de los RNAm celulares mediada por eIF4E-eIF4G-PABP (Figura 1) (Piron, Vende, Cohen & Poncet, 1998; Walsh & Mohr, 2011). La función inhibitoria de NSP3 ha sido evaluada ampliamente con diversas estrategías experimentales. Las proteínas virales se acumulan mientras la infección por RVA progresa, y existe una evidente disminución de la síntesis de proteínas celulares. Ensayos de co-inmunoprecipitación con anticuerpos anti-eIF4G muestran que conforme aumenta la cantidad de NSP3 acumulada, la asociación entre PABP-C y eIF4G disminuye gradualmente, por lo que la inhibición traduccional correlaciona con dicha interacción. Algunas proteasas virales (de picornavirus, poliovirus, HIV-1, etc.) participan en la inhibición de la traducción celular por cortar al factor 4G en los extremos amino y carboxilo terminal, lo que afecta la circularización del RNAm. En infecciones con RVA no se han detectado productos de corte proteolítico de eIF4G mientras que su asociación con eIF4E y eIF4A no se modifica (Castello, Álvarez & Carrasco, 2011; Novoa & Carrasco, 1999; Piron, Vende, Cohen & Poncet, 1998; Walsh et al., 2012). Adicionalmente, los RVA inducen la fosforilación del factor eIF2α mediante la cinasa PKR, lo que típicamente se relaciona con un bloqueo general de la síntesis de proteínas, no obstante, en estas condiciones existe traducción preferencial para algunos genes de respuesta a estrés (Montero, Rojas, Arias & López, 2008). Además, se demostró que la expresión de NSP3 es suficiente para inhibir la traducción de proteínas celulares. Ensayos de incorporación de (35S)Metionina (35S)Cisteina en células BSC-1 infectadas con virus vaccinia recombiantes para expresar a NSP3 silvestre producen un abatimiento mayor al 90% de la traducción celular y de proteínas del virus vaccinia. Asimismo, los dominios de unión a eIF4G y de dimerización resultan ser indispensables para la inhibición traduccional, mientras que el dominio de unión al RNA viral no afecta la función inhibitoria de NSP3 (Padilla-Noriega, Paniagua & Guzmán-León, 2002). Recientemente se reportó que mutaciones puntuales en la región coiled coil afectan la inhibición traduccional, y se relacionan con la acumulación y dimerización de la proteína (Contreras-Treviño et al., 2017).
La relocalización nuclear de PABP-C se relaciona con la función inhibitoria de la traducción de NSP3, y resulta de la formación del complejo ternario (eIF4G-NSP3-RoXaN). La proteína PABP tiene una localización dinámica que se regula por interacciones con los RNA mensajeros y/o otras proteínas celulares (paxilina y eEF1), en particular PABP-C libre del RNA reconoce a la importina-alfa mediante los dominios de reconocimiento del RNA (RRM, por sus siglas en inglés RNA recognition motifs), y causa que PABP-C retorne al núcleo para unirse a los transcritos celulares recién sintetizados (Khacho et al., 2008; Kumar, Shum & Glaunsinger, 2011; Woods, Kantidakis, Sabe, Critchley & Norman, 2005). Una deleción del extremo carboxilo terminal (NSP3(4G) y mutaciones puntuales que afectan la interacción con RoXaN (R187E y K191E) son incapaces de relocalizar a PABP-C en el núcleo. Además, se observa que en ambos casos NSP3 es completamente incapaz de co-inmunoprecipitar con eIF4G y RoXaN, por lo que la formación del complejo ternario es esencial para la relocalización al núcleo de PABP-C (Harb et al., 2008). El reciente hallazgo de que una doble mutante de NSP3, que combina las dos mutaciones que afectan la interacción con RoXaN (R187E:K191E), tiene un nivel de acumulación menor en comparación con la proteína silvestre revela que la dinámica de síntesis y degradación de NSP3 es potencialmente relevante para relocalizar PABP-C al núcleo (Contreras-Treviño et al., 2017). Por otro lado, la relocalización nuclear de PABP-C no se presenta cuando NSP3 se expresa en presencia de 17DMAG, un inhibidor de la chaperona HSP90, lo que indica que los dímeros de NSP3 son necesarios para la función inhibitoria (Dutta et al., 2011). No obstante, una mutación en el residuo triptófano 170 por alanina es incapaz de dimerizar y conserva un 50% de la función inhibitoria de la traducción celular con respecto a la proteína silvestre (Contreras-Treviño et al., 2017). En conjunto los datos anteriores sugieren que dos de las funciones de NSP3, relocalización de PABP-C e inhibición de la traducción celular, no responden de manera idéntica a mutaciones puntuales en la proteína.
La naturaleza del genoma de los RVA y sus estrategias de replicación han permitido obtener rearreglantes genéticas por infecciones consecutivas en cultivos celulares, en particular un virus rearreglante del RVA de la cepa SA11 posee secuencias nucleotídicas duplicadas en el gen que codifica para NSP3 (SA11-4Fg7re) y mantiene la capacidad infecciosa. Esta variante de NSP3 con 533 aminoácidos presenta dominios duplicados (RNA, dimerización, unión a RoXaN), y un peso molecular de 60 kDa, es incapaz de relocalizar a PABP-C mientras que la unión con RoXaN, con eIF4G y la capacidad para dimerizar se mantienen (Arnold, Brownback, Taraporewala & Patton, 2012). La inhibición de la traducción celular durante la infección por los RVA se atribuye a NSP3, debido al efecto que tiene NSP3 recombinante sobre la traducción celular (Padilla-Noriega, Paniagua & Guzmán-León, 2002), y con base en que el uso de interferentes de RNA específicos para NSP3 afectan la inhibición traduccional por RVA, independientemente del estado de fosforilación del factor eIF2( (Montero, Rojas, Arias & López, 2008). Durante el ciclo de replicación de los RVA existe un secuestro nuclear de los transcritos celulares y es atribuido a NSP3, y se revierte cuando se bloquea la expresión de NSP3 por RNA de interferencia, por lo que se ha propuesto que el bloqueo del transporte nuclear-citoplásmicos del RNA y proteínas coopera con la función inhibitoria traduccional de NSP3 (Rubio, Mora, Romero, Arias & López, 2013). El exporte nuclear del RNAm depende de modificaciones que suceden en el núcleo y es asistido por la exportina 1 (CMR1), adicionalmente se une a factores de inicio de la traducción celular como eIF4E y PABP-C resguardándolo de la degradación en el citoplasma. La proteína PABP-C emerge del núcleo unida al RNAm, y a la proteína paxilina. Se ha reportado interacción entre PABP-C y RoXaN a través de un intermediario del RNA, además, RoXaN reconoce secuencias del tipo ARE (AU-rich elements, por sus siglas en inglés) que regulan la estabilidad de los RNA mensajeros celulares (Harb et al., 2008; Leppek & Stoecklin, 2014; Vitour et al., 2004). La proteína RoXaN es mayormente nuclear; mientras que PABP-C es citoplásmica, y durante la infección con RVA existe un intercambio del compartimento celular de estas dos proteínas (Harb et al., 2008). En conjunto, estos datos sugieren que NSP3 secuestra en el citoplasma a un factor necesario para el exporte nuclear de los mensajeros celulares, probablemente favoreciendo su localización y degradación.
Recientemente se reportó que la función inhibitoria de la traducción de NSP3 se adquiere previamente a la formación de los dímeros estables, y se propone que esto ocurre por un secuestro de eIF4G desde la síntesis de NSP3 en los ribosomas, y a esta forma funcional se le denomina intermediario de dimerización (Figura 5) (Contreras-Treviño et al., 2017). Este hallazgo implica que la chaperona HSP90, que forma parte de los intermediarios de dimerización, juega un papel central en la adquisición de la función inhibitoria de la traducción de NSP3. Además, la distinción que se hace entre intermediarios de dimerización y dímeros estables de NSP3 abre una nueva avenida de investigación sobre la relevancia de estas formas de la proteína en relación a las distintas funciones que realiza.
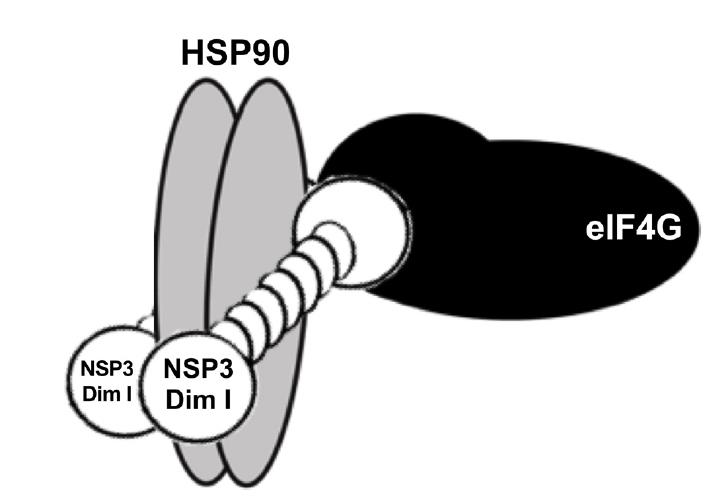
Figura 5: Intermediarios de la dimerización de NSP3. Los intermediarios de la dimerización de NSP3 se definen por su interacción con la chaperona HSP90. El hallazgo de mutantes de NSP3 que no forman dímeros estables, pero contienen el dominio de unión a HSP90 intacto, y la alta afinidad de NSP3 por eIF4G (10 veces mayor que la de PABP), sugieren fuertemente que los intermediarios de dimerización secuestran a eIF4G recién sintetizado.
Adicionalmente, se han encontrado diferencias temporales para cepas distintas de RVA, el efecto inhibitorio requiere 3.5 horas para la cepa RF con un efecto máximo del 90%, mientras que RRV alcanza un máximo del 50% en 6 horas (Gratia et al., 2015).
Estimulación de la traducción viral por NSP3
El ciclo replicativo de los RVA ocurre en el citoplasma celular y particularmente los RNAm virales son sintetizados dentro de la partícula viral por VP1 (polimerasa del RNA). Los transcritos de los RVA poseen en el extremo 5’ una estructura tipo cap similar a los mensajeros celulares, mientras que el extremo 3’ no tiene poliadenilación y en su lugar posee una secuencia conservada en todos los genes de los RVA (UGACC) que es reconocida por NSP3 (Poncet, Laurent & Cohen, 1994). NSP3 estimula la traducción de los mensajeros virales y ha sido debatida por sus funciones aparentemente contrastantes. El contexto de la infección viral enfrenta las funciones opuestas de NSP3 (inhibición y estimulación de la traducción), de las cuales sólo la inhibición de la traducción celular, pero no la estimulación de la traducción viral, es susceptible al nivel de expresión de NSP3 en experimentos de transfección con RNA interferente. No obstante, en estas condiciones aumenta la liberación de partículas virales infecciosas que se relaciona con una mayor síntesis de RNA viral y disminución del efecto citopático (Montero, Arias & López, 2006). Por otro lado, la estimulación traduccional por NSP3 se ha estudiado a través de genes reporteros (luciferasa) con UTR de los RVA y se encontró que aumenta la actividad de la luciferasa para los mensajeros virales poliadenilados con magnitudes distintas. El reconocimiento del RNAm viral es indispensable para la estimulación traduccional, debido a que deleciones o mutaciones puntuales (W87/A) en el extremo amino terminal de NSP3 afectan la afinidad y por consecuencia disminuye la señal de la luciferasa. La cantidad del RNA inicial introducido por electroporación disminuye conforme el efecto inhibitorio traduccional aumenta, ya que cuando la línea celular usada en los experimentos (Cb20) expresa constitutivamente a NSP3 favorece la traducción 325 veces de los RNA mensajeros similares al RVA a las 6 horas, a pesar de disminuir siete veces la cantidad del RNA inicial. Es notable que la cantidad de NSP3 acumulada es 6.8 veces menor comparada con la infección del RVA en células MA104 (8 hpi), además en tiempos tempranos de la infección (1 ó 2 hpi) existe una traducción de los RNA mensajeros virales, aunque NSP3 no sea detectable (Gratia et al., 2015; Vende, Piron, Castagné & Poncet, 2000). Los datos anteriores en conjunto sugieren que las funciones antagónicas de NSP3 para inhibir o estimular la traducción son críticamente sensibles al nivel de expresión y tiempo en el que esto ocurre, además de estar ligados de alguna manera a la replicación viral.
Oligomerización de NSP3
El dímero de NSP3 se ha relacionado con la función inhibitoria traduccional, y la chaperona HSP90 asiste en su formación. La parte central de NSP3 tiene una región mínima para dimerizar (residuos 150-206), y fue identificada a través del sistema del doble híbrido en levadura (Piron, Delaunay, Grosclaude & Poncet,1999). Esta región se encuentra dentro del coiled coil, donde además incluye un fragmento del dominio de unión a eIF4G, así como los dominios de unión a RoXaN y HSP90. NSP3 tiende a homo-oligomerizar cuando se expresa en células infectadas por el RVA, células de insecto y en sistemas in vitro de traducción (Mattion, Cohen, Aponte & Estes,1992). HSP90 asiste a NSP3 en la dimerización a través de los residuos 225 al 258 de los monómeros recién sintetizados, y en un sistema de traducción in vitro con lisados de reticulocitos los dímeros tardan 15 minutos en completar el proceso. Estas interacciones tempranas entre HSP90 con los monómeros de NSP3 se les denomina intermediarios de la dimerización, debido a que los inmunoprecipitados con anti-HSP90 producidos por traducción in vitro se disocian de NSP3 conforme avanza el tiempo (< 15 minutos), y en lisados inmunoprecipitados de células infectadas con los RVA que fueron sometidos a electroforesis en ausencia de un agente reductor (beta-mercaptoetanol), no se detectan dímeros unidos a HSP90. La formación de los dímeros de NSP3 correlaciona con su capacidad para interaccionar con eIF4G y relocalizar a PABP-C en el núcleo (Dutta et al., 2011). Los fragmentos amino terminal (residuos 4-164) y carboxilo terminal (residuos 206-315) se han cristalizado por separado, y se observan como un homodímero asimétrico y simétrico, respectivamente (Deo, Groft, Rajashankar & Burley, 2002; Groft & Burley, 2002). La dimerización de NSP3 disminuye con mutaciones en la región coiled coil, en particular la mutación en el triptófano 170 por una alanina, modifica una posición importante para la oligomerización y estabilidad de NSP3, y por contraparte al cambiar la tirosina 192 por alanina aumenta la cantidad de dímeros estables de NSP3 (Contreras-Treviño et al., 2017). Además, la variante duplicada de NSP3 producida por el virus SA11-7Fg4re mantiene la capacidad para oligomerizar aunque es incapaz para relocalizar a PABP-C (Arnold, Brownback, Taraporewala & Patton, 2012).
Intermediarios de la dimerización con función inhibitoria de la traducción celular
Las interacciones tempranas de NSP3 con HSP90 dan como resultado a los intermediarios de dimerización, que fueron detectados en ensayos de traducción in vitro de lisados de reticulocitos; estas interacciones son inestables frente a bajas concentraciones de SDS (0.02%), y muestran un patrón electroforético similar al monómero (34 kDa) (Contreras-Treviño et al., 2017; Dutta et al., 2011). Además, mutaciones puntuales en el coild coil de NSP3 afectan la formación de dímeros y presentan una función inhibitoria parcial (aprox. 50%), estos cambios están fuera del dominio de unión a HSP90, por lo que estas mutantes de NSP3 deben producir mayoritariamente intermediarios de dimerización, pero no dímeros maduros libres de interacciones con la chaperona HSP90 (Figura 5) (Contreras-Treviño et al., 2017).
Estabilidad de NSP3
La proteostasis a nivel celular funciona como un mecanismo de regulación para la función de diversas proteínas y comprende desde la síntesis, el plegamiento y la degradación (Hartl, Bracher & Hayer-Hartl, 2011). El tiempo de vida media de NSP3 determinado por ensayos pulse-chase es de 200 minutos en el sistema de expresión del virus vaccinia (Piña-Vázquez, De Nova-Ocampo, Guzmán-León & Padilla-Noriega, 2007), y es susceptible a la degradación por la vía del proteasoma cuando se inhibe a HSP90 (Contreras-Treviño et al., 2017; Dutta et al., 2011). Recientemente, se encontró que los dímeros de NSP3 se degradan con mayor rapidez con respecto a los intermediarios de dimerización, con un tiempo de vida media de 60 y 134 minutos, respectivamente (Contreras-Treviño et al., 2017).
Conclusiones
La traducción celular es uno de los procesos génicos con mayor nivel de regulación, por lo que los virus desarrollaron diversas estrategias para utilizarla en su beneficio. NSP3 se ha propuesto como el regulador a nivel traduccional de la expresión génica de los RVA, por su capacidad para interactuar con los mensajeros virales y con proteínas celulares que participan en la traducción celular. Específicamente NSP3 posee la capacidad de estimular la traducción viral e inhibir la síntesis de proteínas celulares, y sus RNA mensajeros carecen de una etiqueta fundamental de los RNAm celulares, la cola de poli-A, por lo que estudiar los mecanismos que regulan ambas funciones es un modelo muy atractivo para conocer funciones celulares de regulación traduccional a detalle.
El mecanismo de inhibición de la traducción celular de NSP3 era atribuido totalmente a la formación de dímeros estables, así como a la interacción con eIF4G y RoXaN. No obstante, en estudios recientes se ha demostrado que la función se adquiere previo a la aparición del dímero y las interacciones del RNA mensajero celular con eIF4G, esto sugiere que la adquisición de la función es un mecanismo más complejo de lo que se pensaba y que las propiedades intrínsecas de NSP3 y de la chaperona HSP90 que asiste a su plegamiento y dimerización son partícipes de ella.











 nueva página del texto (beta)
nueva página del texto (beta)


