Introducción
El arsénico es un elemento químico presente en ambientes acuáticos, con efectos mutagénicos, citotóxicos y genotóxicos y ampliamente reconocido como carcinogénico (Rosen, 1971; Liu, Zhang, Chen & Sun, 2011). Se ha propuesto que alrededor de 150 millones de personas pueden estar en riesgo al consumir agua contaminada con arsénico y en algunos países representa un grave problema de salud, ya que la población consume agua con concentraciones de arsénico mayores a 10 μg/L, siendo el máximo permitido por la Organización Mundial de la Salud (Nordstrom, 2002; Khan & Ho, 2011).
Ciertas actividades humanas son responsables de la contaminación de suelos y aguas, como la minería, desperdicios industriales y el uso de pesticidas en la agricultura, entre otros (Nordstrom, 2002; Paez-Espino, Tamames, de Lorenzo & Cánovas, 2009). La dificultad para remover o biorremediar sitios contaminados con arsénico, así como su toxicidad, depende en parte del estado de oxidación o valencia del metaloide. El As puede presentar una variedad de estados oxidativos, siendo los estados pentavalente (V) y trivalente (III) las formas más comunes de encontrarlo y el As(III) como la forma más tóxica. El arsenato (AsO4 3-) formado por arsénico pentavalente puede ser asimilado a través de las membranas celulares de las bacterias por los transportadores de fosfato (PO4 3-), mientras que el arsenito (AsO2 -) compuesto por arsénico trivalente puede entrar a través de las aguagliceroporinas (Paez-Espino, Tamames, de Lorenzo & Cánovas, 2009). Una vez dentro de la célula, el arsenato es reducido a arsenito por arsenato reductasas (ArsC) y es expulsado por bombas de expulsión de arsenito (ArsB). Se ha observado que el arsenito puede estimular la generación de especies reactivas de oxígeno (ROS), las cuales dañan proteínas, lípidos y al DNA (Alí, Khan & Sajad, 2013). Además de la participación de proteínas como ArsC y ArsB, existen otros mecanismos de resistencia al arsénico; por ejemplo, la oxidación del arsenito, siendo el gen aoxB el que codifica para la subunidad catalítica de la arsenito (III) oxidasa (AoxAB), (Quéméneur et al., 2008). Éstos y otros mecanismos de resistencia al arsénico han sido revisados en diversos trabajos (Ver Paez-Espino, Tamames, de Lorenzo & Cánovas, 2009; Yang & Rosen, 2016).
El arsénico es abundante en la corteza terrestre y puede originarse de diversas fuentes naturales (Mandal & Suzuki, 2002). En particular, altas concentraciones de As pueden encontrarse en aguas subterráneas y manantiales termales. Estas fuentes de agua pueden ser empleadas para consumo humano y otras actividades recreativas; como es el caso de los manantiales termales de Araró, Michoacán, México. En los manantiales termales de Araró se han reportado elevadas concentraciones de As, las cuales pueden alcanzar los 6.9 mg/L (Vázquez-Vázquez, Cortés-Martínez & Alfaro-Cuevas-Villanueva, 2015). En el trabajo realizado por Prieto-Barajas, Alfaro-Cuevas, Valencia-Cantero & Santoyo, (2017), se confirmaron las altas cantidades de As detectando concentraciones que van desde 2.7 a 6.6 mg/L durante las cuatro estaciones del año. El As resultó ser un factor relevante para determinar la biodiversidad bacteriana de los tapetes microbianos de las pozas, durante cuatro muestreos en diferentes épocas del año. Uno de los principales grupos reportados en dicho trabajo fueron las bacterias del género Bacillus, división Firmicutes. Los resultados mostraron que el As, junto con otros factores fisicoquímicos como la temperatura (( 500C), es uno de los elementos más importantes que seleccionan las comunidades bacterianas en los manantiales termales analizados (Prieto-Barajas, Alfaro-Cuevas, Valencia-Cantero & Santoyo, 2017). En este contexto, el objetivo de este trabajo fue identificar genes (arsB, arsC, aoxB, ACR3), como un potencial mecanismo de resistencia al arsénico en cepas aisladas de los tapetes microbianos de las pozas termales de Araró, Mich. Por lo tanto, se identificó la presencia de genes ars en 21 de las 37 cepas de Bacillus analizadas y mediante la amplificación por PCR, secuenciación y análisis filogenéticos, se identificaron los genes arsB y arsC. Adicionalmente, se analizó la correlación entre la tolerancia al arsenato (V) y arsenito (III) de las cepas bacterianas con la presencia de los genes ars. Los resultados demuestran que las cepas de Bacillus que muestran ser hipertolerantes al As (≥16mM), contienen genes ars que podrían contribuir a la resistencia al arsénico.
Materiales y Métodos
Cepas microbianas
En el presente estudio se trabajó con 37 cepas del género Bacillus aisladas de dos pozas termales de Araró, Michoacán, México (Prieto-Barajas, Alfaro-Cuevas, Valencia-Cantero & Santoyo, 2017). Los 37 aislados pertenecen a las especies Bacillus licheniformis (18 cepas); Bacillus cereus (6 cepas); Bacillus pumilus (5 cepas); Bacillus subtilis (4 cepas); Bacillus vietnamensis (2 cepas) y Bacillus megaterium (2 cepas). Todos los aislados han sido identificados a través de la secuenciación del gen ribosomal 16S (Prieto-Barajas, Alfaro-Cuevas, Valencia-Cantero & Santoyo, 2017). Los aislados fueron cultivados en medio de cultivo Agar Nutritivo (AN), a 37°C, para su uso rutinario en el Laboratorio y conservadas a -70°C en glicerol al 50% más medio nutritivo.
Amplificación y secuenciación de genes
El DNA genómico de cada cepa fue aislado por medio del protocolo de (Mahuku, 2004) y la extracción se comprobó mediante electroforesis en gel. Se diseñaron “primers” específicos para la amplificación de los genes arsB, arsC, aoxB y ACR3 (Tabla I). Para la amplificación por la reacción en cadena de la polimerasa (PCR, por sus siglas en inglés) se emplearon las siguientes condiciones: Un ciclo de desnaturalización inicial a 95°C por 3 min, treinta ciclos de desnaturalización (95°C/30s), alineamiento (59°C/1min), extensión (72°C/1min) y un ciclo de extensión final a 72°C por 5min. Los productos de PCR fueron purificados con del kit Wizard® SV Gel and PCR Clean-Up System (Promega), y se mandaron secuenciar a MR.DNA sequencing services (Texas, USA).
Tabla I Oligonucleótido diseñados para la amplificación de genes de resistencia a arsénico: arsC, arsB, ACR3 y aoxB. (f: Oligonucleótido directo, r: Oligonucleótido reverso).
| Gen | Secuencia de los oligonucleótidos | Longitud (pb) | Tamaño del amplicón |
|---|---|---|---|
| arsC | f 5´- TGGTACTTTTTCCACACACTTTCA-3´ r 5´-TGTGGCACATGTCAAAAAGCA-3´ | 24 21 | 335pb |
| arsB | f 5´-TGGAATCGCAACTCGGACAA-3´ r 5´-CCACCACTAGCAAAGGTTCG-3´ | 20 20 | 700pb |
| ACR3 | f 5´-AGTGATTAGCGCTAGCAATGAAA-3´ r 5´-TGGTGAGGATCCGATGTTGC-3´ | 23 20 | 638pb |
| aoxB | f 5´-GCACTGGGGCTCGACTTC-3´ r 5´-CCAGGAAAACGCTGCTTACG-3´ | 18 20 | 650pb |
Análisis filogenéticos
La identificación de los genes de resistencia a arsénico fue llevada a cabo con búsquedas de homología tipo Blastx en la base de datos del GenBank. En el Blastx, las secuencias de nucleótidos son transformadas a secuencias de aminoácidos y con éstas se calcula el grado de identidad de las secuencias. Los análisis filogenéticos de las secuencias de nucleótidos de los genes arsB y arsC se llevaron a cabo utilizando el programa MEGA 5. Las filogenias se construyeron utilizando el método del “vecino más cercano”. Otros métodos mostraron topologías similares: máxima verosimilitud y mínima evolución. (Tamura et al., 2011). Las secuencias de genes arsB y arsC caracterizados se obtuvieron del NCBI y se realizaron los correspondientes alineamientos. Un valor de confianza para el conjunto de datos de secuencias alineadas se obtuvo mediante la realización de análisis de Bootstrap de 1000 repeticiones. Para enraizar los árboles se emplearon las siguientes secuencias para el árbol de arsB Paenibacillus polymyxa M1 (HE577054.1) y para el árbol de arsC, Aspergillus parasiticus SU-1 696 (JZEE01000681.1).
Concentraciones máximas de tolerancia al As
Las concentraciones máximas de tolerancia se realizaron mediante el crecimiento de los aislados en cajas de Petri con medio agar nutritivo (AN) y medio mínimo (M9) con concentraciones crecientes de arsenato de sodio dibásico heptahidratado Na2HAsO4-7 H2O (4, 8, 16, 32, 64 y 128 mM) y (meta) arsenito de sodio AsNaO2 (4, 8, 16, 32, 64 y 128 mM). Los cultivos se incubaron a 37°C durante 72 hrs. El crecimiento se determinó a través de la observación de las colonias en el medio.
Resultados
Identificación de los genes arsB y arsC.
Los oligonucleótidos fueron diseñados con base en secuencias conservadas de los genes que codifican las enzimas ArsB, ArsC, AoxB y ACR3 (Tabla I). De las 37 cepas bacterianas analizadas se obtuvieron amplificados del tamaño esperado en 21 cepas para los genes arsB y arsC; sin embargo, ninguna cepa dio positivo para la amplificación de los genes aoxB y ACR3.
Los productos de PCR amplificados fueron secuenciados y por búsquedas de tipo Blastx en la base de datos del GenBank, se identificaron como proteínas ArsB y ArsC con altos porcentajes de identidad (Tabla suplementaria). Cuatro de las veintiún cepas dieron positivo a arsB, con una alta identidad (84-99%) a genes arsB de especies como: Psychrobacter, Bacillus y Escherichia coli. Diecisiete cepas mostraron identidad del 91 al 100% con genes arsC de Paenibacillus polymyxa, Bacillus paralicheniformis, Planococcus antarcticus y al grupo de Bacillus subtilis.
Tabla suplementaria: Cepas bacterianas que dieron positivo a la amplificación por PCR y búsqueda de homología por Blastx en el NCBI.
| Especie y cepa bacteriana | Tamaño del amplicón (pb) | Relación más cercana por Blastx | Identidad % | Número de acceso |
|---|---|---|---|---|
| B. megaterium ZAP20 | 529 | Bomba de flujo de arsénico/ Psychrobacter sp. | 97% | WP_058368437.1 |
| B. licheniformis ZAP31 | 346 | Bomba de flujo de arsénico / Psychrobacter sp. | 99% | WP_058368437.1 |
| B. licheniformis ZAP10 | 358 | Proteína de Resistencia al arsénico arsB/ Bacillus | 84% | WP_004398718.1 |
| B. cereus ZAP43 | 474 | Bomba de flujo de arsénico / Escherichia coli | 99% | ORT41606.1 |
| B. cereus ZAP40 | 348 | Arsenato reductasa / Planococcus antarcticus | 98% | WP_006829557.1 |
| B. cereus ZAP15 | 257 | arsC / Grupo Bacillus subtilis | 100% | WP_020451674.1 |
| B. cereus ZAP64 | 421 | arsC / Bacillus sp. | 100% | WP_003182412.1 |
| B. licheniformis ZAP98 | 399 | Arsenato reductasa/ Bacillus paralicheniformis | 91% | OMI10446.1 |
| B. licheniformis ZAP73 | 401 | arsC / Bacillus | 99% | WP_003182412.1 |
| B. licheniformis ZAP14-1 | 278 | Arsenato reductasa arsC/ Bacillus licheniformis | 100% | WP_069500528.1 |
| B. subtilis ZAP33 | 404 | Arsenato reductasa / Grupo Bacillus subtilis | 100% | WP_025810781.1 |
| B. licheniformis ZAP12 | 404 | Arsenato reductasa / Paenibacillus polymyxa | 100% | WP_014600010.1 |
| B. subtilis ZAP13 | 407 | Arsenato reductasa / Grupo Bacillus subtilis | 99% | WP_025810781.1 |
| B. licheniformis ZAP14-2 | 390 | Arsenato reductasa / Paenibacillus polymyxa | 100% | WP_014600010.1 |
| B. subtilis ZAP100 | 273 | Arsenato reductasa / Paenibacillus polymyxa | 100% | WP_014600010.1 |
| B. licheniformis ZAP79 | 408 | Arsenato reductasa / Paenibacillus polymyxa | 93% | WP_040103021.1 |
| B. pumilus ZAP072 | 403 | Arsenato reductasa / Paenibacillus polymyxa | 93% | WP_040103021.1 |
| B. pumilus ZAP16 | 399 | Arsenato reductasa / Paenibacillus polymyxa | 93% | WP_040103021.1 |
| B. licheniformis ZAP17 | 399 | Arsenato reductasa / Bacillus paralicheniformis | 91% | OMI10446.1 |
| B. licheniformis ZAP14-1 | 408 | Arsenato reductasa / Bacillus paralicheniformis | 91% | OMI10446.1 |
| B. cereus ZAP09 | 362 | arsC / Bacillus | 99% | WP_023857526.1 |
Filogenias de los genes arsB y arsC
Para la asignación filogenética de las secuencias arsB y arsC se utilizaron secuencias del GenBank que mostraron identidad con las secuencias problema como grupos de referencia en la realización del árbol filogenético. Para el árbol de las secuencias arsB se utilizó a Paenibacillus polymyxa M1 (arsB) como grupo externo y para el árbol de arsC se utilizó a Aspergillus parasiticus US-1 696 (arsC). El análisis filogenético colocó las secuencias arsB en dos grupos. En particular, la secuencia arsB de Bacillus licheniformis ZAP10 se agrupó con genes arsB de B. subtilis, Bacillus cereus y Bacillus gibsonii (Figura 1a). Las otras secuencias arsB de B. licheniformis ZAP31, B. megaterium ZAP20 y B. cereus ZAP43, formaron un grupo con genes arsB de E. coli, Staphylococcus arlettae y cepas de Bacillus sp.
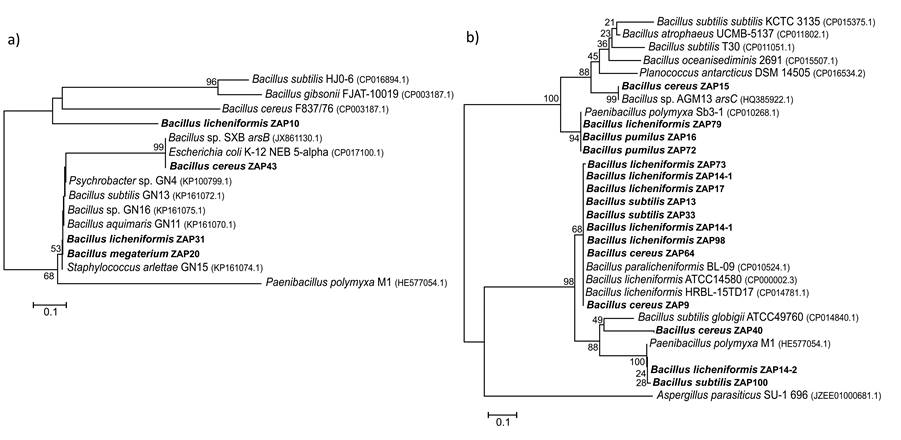
Figura 1: Filogenias de las secuencias arsB (a) y arsC (b). Para la reconstrucción filogenética se utilizó el método del “vecino más cercano” y como medida de soporte Bootstrap después de 1000 repeticiones, utilizando el programa MEGA 5.
En el caso de las secuencias arsC, el análisis filogenético distinguió entre dos grupos distantes de secuencias (Figura 1b). Uno conformado por cepas ZAP aisladas de las pozas de Araró pertenecientes a B. licheniformis, B. subtilis y B. cereus con cepas ATCC de referencia de B. licheniformis, B. subtilis y B. paralicheniformis. El otro grupo incluyó tres secuencias de las cepas B. cereus ZAP15, B. licheniformis ZAP79, B. pumilus ZAP16 y ZAP72 con especies de Bacillus sp.
Hipertolerancia al arsenato (V) y arsenito (III)
El arsénico se presenta principalmente en dos formas químicas: arsenato y arsenito, por lo que los microorganismos han desarrollado mecanismos de resistencia a ambos estados de oxidación. Para determinar la tolerancia a estos compuestos se crecieron las cepas en medios AN y M9 con cantidades crecientes de arsenato y arsenito (4, 8, 16, 32, 64 y 128 mM). Cuando fueron cultivadas en los medios M9 y AN sin el metaloide (condiciones control), todas las bacterias crecieron de manera óptima, igualmente, todos los aislados fueron tolerantes a concentraciones de As(V) y As(III) de 8 mM, sin embargo, el porcentaje de aislados bacterianos capaces de crecer en presencia del As disminuyó de forma estadísticamente significativa a partir de la concentración 16 mM para el caso del As(III) y de 32 mM para el caso del As(V), hasta llegar a una proporción 0.0 y de 0.13 en la concentración de 128 respectivamente (Tabla II).
Tabla II: Proporción de aislados bacterianos tolerantes a As(III) y As(V) a distintas concentraciones.
| Concentración (mM) | As | Medio M9 | Medio AN | ||||
|---|---|---|---|---|---|---|---|
| Tolerantes | Proporción | Valor P a | Tolerantes | Proporción | Valor P | ||
| 0 (Control) | (V) | 37 | 1 | - | 37 | 1 | - |
| (III) | 37 | 1 | - | 37 | 1 | - | |
| 8 | (V) | 37 | 1 | 0.5 | 37 | 1 | 0.5 |
| (III) | 37 | 1 | 0.5 | 37 | 1 | 0.5 | |
| 16 | (V) | 37 | 1 | 0.5 | 26 | 0.7 | 0.0002 |
| (III) | 11 | 0.29 | <0.0001 | 24 | 0.64 | <0.0001 | |
| 32 | (V) | 33 | 0.89 | 0.0190 | 26 | 0.7 | 0.0002 |
| (III) | 6 | 0.16 | <0.0001 | 4 | 0.1 | <0.0001 | |
| 64 | (V) | 20 | 0.54 | <0.0001 | 15 | 0.4 | <0.0001 |
| (III) | 0 | 0 | <0.0001 | 0 | 0 | <0.0001 | |
| 128 | (V) | 5 | 0.13 | <0.0001 | 7 | 0.18 | <0.0001 |
| (III) | 0 | 0 | <0.0001 | 0 | 0 | <0.0001 | |
aValor de p indica la significancia estadística de la diferencia de las proporciones entre el control y el tratamiento señalado según un análisis de frecuencias de X 2 seguido de un análisis de proporciones.
La Figura 2 muestra un listado de las 37 cepas de Bacillus analizadas en este trabajo, así como la presencia/ausencia de genes ars y la tolerancia a las diferentes concentraciones de As(V) y As(III). En general, se puede observar que las cepas hipertolerantes [(128 mM de As (V) y 32 mM As (III)], B. megaterium ZAP20, B. licheniformis ZAP31, B. licheniformis ZAP10 y B. cereus ZAP43, dieron positivo a la presencia de los genes arsB y arsC, siendo estas cepas las que mostraron mayor resistencia al As. Otras 17 cepas con niveles altos de resistencia al arsénico (V y III), pero inferiores a las del primer grupo, dieron también positivo a la amplificación del gen arsC, que codifica para la arsenato reductasa ArsC pero no a la amplificación del gen arsB que codifica para la bomba de expulsión de arsenito. No obstante, no se descarta que otros mecanismos de resistencia estén presentes en las demás cepas de Bacillus, ya que algunas de ellas también fueron tolerantes a altas concentraciones de arsénico [(64 mM As (V) y 32 mM As (III)], sin haber dado positivo para los genes arsB y arsC. Sin embargo, se puede observar una correlación clara entre la presencia de los genes arsB y arsC como posibles mecanismos de resistencia en las cepas hipertolerantes al As.

Figura 2: Comparación de los niveles de tolerancia al arsenato y arsenito de las 37 cepas de Bacillus analizadas en este trabajo y su asociación con la presencia/ausencia de los genes arsC y arsB. La intensidad de colores de blanco a negro de los cuadros indican la concentración de arsenato o arsenito resistido por las cepas. Los cuadros de color verde claro indican la detección del gen arsC, mientras que los cuadros color verde oscuro indican la presencia de arsB, detectados por medio de la PCR.
Discusión
En un trabajo realizado recientemente por Prieto-Barajas, Alfaro-Cuevas, Valencia-Cantero & Santoyo, (2017), se observó un efecto de la estacionalidad y ciertos parámetros fisicoquímicos sobre la diversidad cultivable de comunidades bacterianas de dos pozas termales en Araró (Prieto-Barajas, Alfaro-Cuevas, Valencia-Cantero & Santoyo, 2017), localizado en el centro del Eje Volcánico Transmexicano y relativamente cercano (20 Km) a la zona de los Azufres, en Michoacán (Israde-Alcántara & Garduño-Monroy, 1999). Al analizar diversos parámetros fisicoquímicos, las altas concentraciones de arsénico resultaron ser un factor importante que influyó de forma significativa sobre la diversidad bacteriana en una de las pozas analizadas. Otros reportes también habían encontrado niveles altos de As en algunas otras fuentes termales recreativas en Araró, con valores que oscilan entre 0,01 mg / L y 6,26 mg / L, que superan las normas de la Organización Mundial de la Salud (OMS), siendo un riesgo potencial para la salud (Vázquez-Vázquez, Cortés-Martínez & Alfaro-Cuevas-Villanueva, 2015). Por lo tanto, al descubrir los altos niveles de As en el agua de las pozas termales, surgió la hipótesis de que dichas cepas poseen elementos genéticos que les brindan resistencia al As para poder sobrevivir. Cabe destacar que el 76% de los filotipos analizados correspondieron al Phylum Firmicutes y principalmente al género Bacillus. Así, de las 37 cepas analizadas el 56.7% dieron positivo al ser amplificadas por PCR los genes arsB y arsC. Los genes arsB y arsC son dos de los genes más ampliamente distribuidos en bacterias resistentes al As, ya que la reducción del arsenato a arsenito, llevada a cabo por la arsenato reductasa ArsC; así como su posterior expulsión por la bomba de expulsión de arsenito ArsB, es uno de los mecanismos más eficientes de resistencia a la toxicidad del As (Bachate, Cavalca & Andreoni, 2009; Cervantes & Gamiño, 2017; Oremland & Stolz, 2003). Los genes arsB y arsC forman parte del operón arsRDABC, que contiene cinco genes o tres, como el arsRBC, siendo ambos localizados en diversos replicones, de tipo cromosomal o plasmídico (Oremland & Stolz, 2003). Sin embargo, el que no se hayan amplificado los genes ACR3 y aoxB, no permite descartar su presencia en este trabajo. Las razones podrían ser diversas, incluyendo un esfuerzo mayor para modificar las condiciones de amplificación o diseño de nuevos oligos. Así mismo, se podría realizar otro tipo de estudio más amplio, como un análisis metagenómico de dichos ambientes que podría revelar la presencia de esos genes o incluso, de otros elementos que brinden un mecanismo diferente de resistencia al As (Yang & Zhang, 2017).
Al realizar los análisis tipo Blastx se encontró una alta identidad con las proteínas ArsB y ArsC de diversas especies, como Bacillus, Paenibacillus, Psychrobacter y Planococcus. Algunas de estas proteínas han sido ya caracterizadas, por lo que su función ha sido comprobada como un mecanismo de resistencia al As. Por ejemplo, en un trabajo realizado por Bachate, Cavalca & Andreoni (2009), se aislaron diversas cepas bacterianas tolerantes al As de un suelo agrícola contaminado con este metaloide en Bangladesh, logrando reducir 2 mmol l−1 de As(V) a As(III), en condiciones aerobias. Algunas cepas del género Bacillus contenían arsenato reductasas y bombas de expulsión de arsenito. Otro trabajo realizado por Tripti, Sayantan, Shardendu, Singh & Tripathi (2014), también analizaron la capacidad de toma y remoción de arsenato (V); así como la reducción de arsenato (V) a arsenito (III), en una cepa de Bacillus licheniformis (DAS1). De forma interesante las cepas mostraron que concentraciones inhibitorias mínimas (CMI) para As (V) y As (III) eran 10 y 7 mM, respectivamente. Aunque el trabajo anterior reporta buenas capacidades de la cepa DAS1 para tomar, remover o reducir el arsenato y arsenito, la cepa no muestra altos niveles de resistencia al As, lo que limitaría su uso en ambientes contaminados con altas concentraciones de dicho elemento tóxico (Tripti, Sayantan, Shardendu, Singh & Tripathi, 2014). De acuerdo con Cai, Liu, Rensing & Wang, (2009), se consideran cepas altamente resistentes (hiper) al As cuando reportan concentraciones mínimas inhibitorias (MIC), igual o mayor a 14 mM de As. Los autores reportan el aislamiento de 58 cepas hiperresistentes al As de suelos altamente contaminados con dicho metal, incluyendo géneros como Acinetobacter, Agrobacterium, Arthrobacter, Comamonas, Rhodococcus, Stenotrophomonas y Pseudomonas, pero no de Bacillus. Un dato interesante reportado por los autores fue que se encontró una alta frecuencia de transferencia horizontal de los genes arsB/ACR3 entre las cepas aisladas. Lo anterior coincide con nuestros análisis filogenéticos, donde genes arsB de B. licheniformis cepas ZAP10 y ZAP31 no se agrupan en un mismo clado, sino que están ubicados en grupos distintos. El mismo caso se observa para las secuencias arsC de las cepas B. licheniformis ZAP79 y ZAP73 igualmente separadas en el análisis filogenético.
En otro trabajo realizado por Cavalca, et al., (2010), se reporta el aislamiento de bacterias resistentes al arsénico asociadas con las raíces de la planta silvestre Cirsium arvense de un suelo contaminado con arsénico y la búsqueda de posibles actividades promotoras del crecimiento vegetal. Comparando con nuestros resultados, cinco cepas que aislamos de las pozas termales de Araró lograron tener una tolerancia de hasta 32mM de arsenito (III) y 128 mM de arsenato (V) (Figura 2). Cabe destacar que la mayor concentración a la cual lograron sobrevivir fue cuando las cepas fueron cultivadas en medios nutritivos (AN), comparado cuando se cultivaron en medios mínimos. Y es que un medio nutritivo contiene mayores cantidades de fosfatos, los cuales pueden competir con el As (V), para ser internalizados a través de los transportadores de fosfato (Cervantes & Gamiño 2017; Rosen & Liu, 2009).
Conclusiones
Los genes arsB y arsC identificados en cepas de Bacillus y en particular en aquellos aislados hipertolerantes al As, podrían constituir uno de los principales mecanismos genéticos de sobrevivencia en un ambiente acuático con altas concentraciones del metaloide, como son las pozas termales de Araró, Michoacán.











 text new page (beta)
text new page (beta)


