Introducción
En la actualidad, la supercomputación tiene una vital importancia a la hora de lograr avances significativos en ciencias tan diversas como la biología (Smith, 2014), la medicina en la simulación de respuesta a medicamentos (Kim et al., 2015) y la matemática, logrando conseguir en horas los resultados que en computadores personales puede tardar meses. Debido a esto, es común encontrar en distintas partes del mundo máquinas robustas que tienen como objetivo procesar grandes cantidades de datos en tiempos cortos, para que los investigadores puedan continuar con su labor y obtener avances en la ciencia mucho más rápido que en tiempos pasados.
La supercomputación se integra de distintas unidades de procesamiento; CPUs, GPUs1 y Xeon PHI son las más empleadas en la actualidad. Gracias a las arquitecturas multi-core y many-core, las capacidades de los supercomputadores, que en la actualidad alcanzan mil billones de operaciones de punto flotante por segundo (FLOPS), conocido así por sus siglas en inglés, aumentan de gran manera, pero para aprovechar estas tecnologías se debe contar con software realmente paralelizable.
Computación de alto rendimiento
La computación de alto rendimiento o High Perfomance Computing (HPC) es un área que abarca varios paradigmas de programación paralela, incluyendo lenguajes e interfaces de programación, software especializado, conferencias internacionales, entre otras, se define desde el ámbito técnico y científico (Nielsen, 2016).
El uso de HPC en distintos ámbitos de la bioinformática es más frecuente, como en el análisis de big data, procesamiento de señales biomédicas, deep learning2, machine learning, entre otras (Min et al., 2016) esto se debe a las ventajas que esta tecnología ofrece en cuanto a tiempo de ejecución y herramientas para que investigadores y científicos puedan optimizar el tiempo en sus investigaciones.
En la actualidad la supercomputación es un campo ampliamente estudiado, de hecho se considera uno de los tres pilares de la investigación, gracias a que muchos de los avances en la ciencia y la ingeniería están basados en ella (González et al., 2015) en la literatura se encuentran implementaciones realizadas por investigadores en donde buscan solucionar problemas u optimizar recursos, incluso usando máquinas virtuales (Huang et al., 2006) y para dar solución a problemáticas en campos tan variados como las matemáticas (Behrends et al., 2016), diseño de turbinas de gas (Alhatim, 2016), secuenciación (Al-Ali et al., 2016), entre otras.
Bioinformática
La bioinformática es la aplicación de la estadística y las ciencias computacionales en el campo de la biología, donde el principal objetivo es mejorar el entendimiento de los procesos biológicos (Gobalan y John, 2016). La bioinformática ha aportado en muchos roles de la ciencia, por ejemplo, en el cambio climático y el problema del crecimiento de la población (Batley y Edwards, 2016), en la agricultura (Esposito et al., 2016), en la medicina, en el entendimiento de enfermedades congénitas, entre otras.
Gracias a los avances en las tecnologías genéticas como Next Generation Sequencing (NGS) y en el campo de la biología molecular, la comunidad científica ha generado grandes cantidades de información (Fatumo et al., 2014), por esta razón la bioinformática ha tomado mucha importancia en los últimos años, ya que gracias a ella, a los algoritmos y técnicas que aplica, se pueden analizar ágilmente los volúmenes de datos demasiado grandes y así obtener información relevante para las investigaciones.
Software bioinformático altamente paralelizable
Como se mencionó anteriormente, para aprovechar las ventajas de la supercomputación al máximo, se requiere hacer uso de todos los procesadores y esto se logra con software altamente paralelizable. En la bioinformática existen herramientas diseñadas para correr en máquinas de alto rendimiento como ABySS (Simpson et al., 2009) que es un ensamblador en paralelo, MAFFT (Katoh y Standley 2013) que puede hacer alineaciones múltiples sobre diferentes CPUs y BIOS-ParallelBlast. Este último se empleó en el siguiente estudio para optimizar la ejecución de NCBI-Blast [https://blast.ncbi.nlm.nih.gov/Blast.cgi] sobre Xeon Phi. NCBI-Blast es el software bioinformático más popular para hacer alineamientos locales sobre secuencias de nucleótidos o proteínas contra bases de datos previamente formateadas.
BIOS-ParallelBlast (PB) es un wrapper desarrollado en Python por el Centro de Bioinformática y Biología Computacional de Colombia BIOS [https://www.bios.co] el cual permite la ejecución en paralelo sobre un conjunto de recursos computacionales usando dos esquemas de paralelización - Trivial Parallelization TP y by-sequence Parallelization bSP -. En términos generales, un archivo de secuencias se dividió en pequeñas partes dependiendo de los esquemas de paralelización y de los algoritmos de división que se apliquen (BIOS-FastaSplitter, LongerSplitter, SorterSplitter o PhiSplitter). Una vez realizada la división se procesó cada archivo con un NCBI-Blast independiente. Al final se unen los resultados de los alineamientos en un archivo de salida único. En su versión actual soporta diferentes tipos de comparación (blastx, blastp y blastn). Esta herramienta se explica con más detalle en la sección de la metodología.
Importancia de Xeon PHI en la actualidad
En la actualidad, gracias a la gran eficiencia energética y al poder computacional que aportan los Xeon PHI a las arquitecturas heterogéneas, se emplea en diferentes ámbitos de la ciencia y la tecnología, por ejemplo, en la tecnología cloud y en especial en big data (Paranjape et al., 2012).
Cómo optimizar el alineamiento local en Xeon PHI
Actualmente se han planteado diferentes formas de mejorar el rendimiento del NCBI-Blast y de diferentes alineamientos locales en las arquitecturas multi-core. Una de estas alternativas es dividir la secuencia query y la base de datos en partes mucho más pequeñas, distribuir estas partes mediante MPI e hilos OpenMP y combinar los resultados de todos los procesos en una sola salida (Sawyer et al., 2015).
Metodología
Operación de BIOS-ParallelBlast (PB)
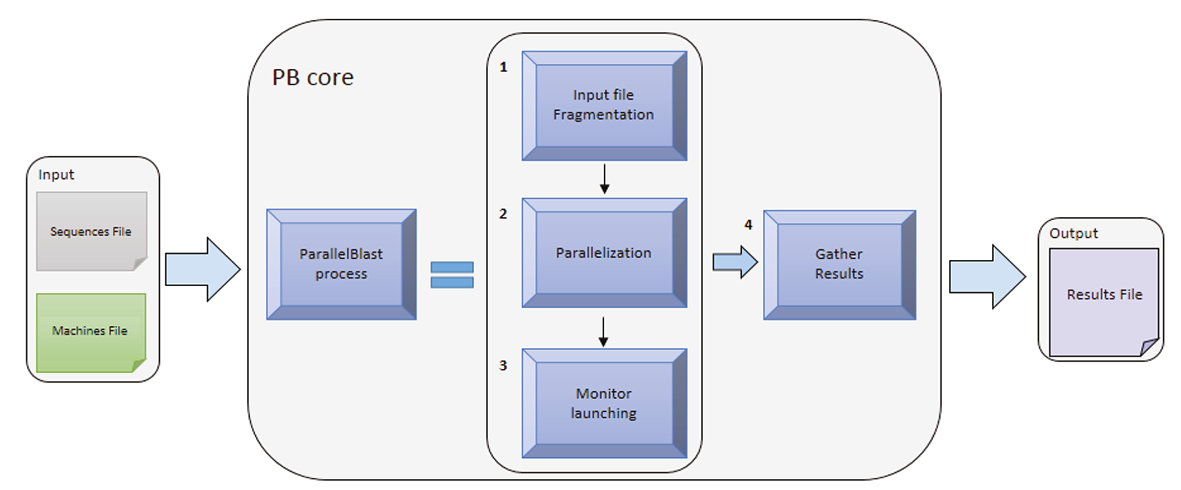
El flujo de trabajo general se muestra en la Figura 1 de BIOS-ParallelBlast.
Input. PB recibe dos archivos como entrada:
Sequences File: Archivo en formato FASTA (secuencias identificadas por el caracter “>”) [http://www.ncbi.nlm.nih.gov/BLAST/blastcgihelp.shtml]
Machines file: Una línea por cada nodo (hostname) que se usará por PB
PB core. El proceso descrito puede variar dependiendo del esquema de paralelización seleccionado (TP o bSP). Información sobre estos esquemas se presenta en las secciones siguientes. PB puede soportar blastx, blastn y blastp.
Input file fragmentation. Teniendo en cuenta el esquema de paralelización escogido, la entrada se divide en pequeños fragmentos, usando uno de los cuatro algoritmos de división de secuencias (BIOS-FastaSplitter, LongerSplitter, SorterSplitter o PhiSplitter), los cuales se presentan en las siguientes secciones.
Parallelization. Puede ser TP o bSP.
Monitor launching. Una vez que el segmento del archivo se envía al procesado por una máquina seleccionada, un proceso de monitoreo se lanza por PB. Esta fase de monitoreo permite a PB enviar un nuevo segmento solo una vez, donde el proceso se termina en una máquina (bSP) y une las salidas de las alineaciones en un solo archivo (TP y bSP).
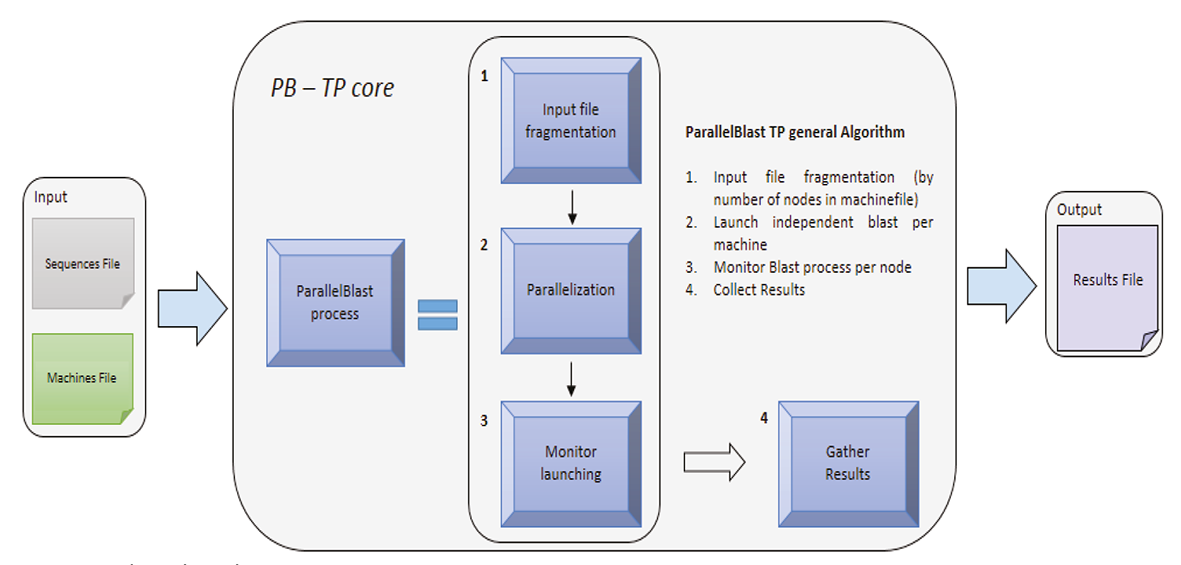
Trivial parallelization
El primer esquema de paralización disponible en PB es Trivial Parallelization - TP, sobre el cual pueden ejecutarse cuatro algoritmos de división.
BIOS-fastaSplitter
Este algoritmo divide las secuencias de entrada en pequeñas piezas con el número de secuencias calculadas basado en el número de máquinas incluidas en el archivo de hosts.
donde
TotalNumSeqArchivoEntrada = |
Número total de secuencias a analizar |
NumeroMaquinas = |
Número de nodos incluidos en el archivo de hosts |
Este algoritmo no tiene en cuenta las longitudes de las secuencias, por lo que es ideal cuando se ejecuta sobre Reads, que tienen la misma longitud. Se recomienda el uso de este algoritmo cuando se lanza PB únicamente sobre nodos de CPU (no sobre Xeon Phi).
LongerSplitter
En primer lugar, calcula si la cantidad de secuencias y su longitud es apropiada, si cumple con las condiciones, calcula la longitud total de todas las secuencias contenidas dentro del archivo, excluyendo su encabezado y divide este número entre el número de máquinas donde se ejecutará PB, con el fin de tener una repartición equivalente cuando se cuenta con secuencias de diferentes longitudes dentro del archivo.
donde
TotalLongitudSeqArchivoEntrada = |
Longitud total de las secuencias a analizar, excluyendo su encabezado |
NúmeroMáquinas = |
Número de nodos incluidos en el archivo de hosts |
Si no cumple con los requisitos ordena el archivo con secuencias por longitud, en orden descendente, y reparte las secuencias en el número de archivos solicitados por iteraciones. Si el número de máquinas es n, las primeras n-secuencias se copian en los n-archivos, una por archivo, continuando hasta terminar la totalidad de las secuencias. El resultado son n-archivos con la misma cantidad de secuencias ordenadas por longitud. El propósito de esta división es tener una longitud total de las secuencias de cada archivo lo más similar posible.
Se recomienda el uso cuando se cuenta con archivos de secuencias de diferentes longitudes y cuando se desea ejecutar PB únicamente sobre nodos de CPUs (no sobre Xeon Phi).
SorterSplitter
En primer lugar, ordena las secuencias dentro del archivo por longitud en orden descendente y repite el proceso del algoritmo BIOS-fastaSplitter (dividir en archivos más pequeños, todos con la misma cantidad de secuencias). El propósito de este algoritmo es generar archivos de diferentes tamaños (en orden decreciente, el primer archivo será el más grande y el último el más pequeño) para que los nodos con CPUs analicen los archivos más grandes y los nodos con Xeon Phi los archivos más pequeños. Se requiere adicionalmente que el archivo de hosts diferencie de forma notoria los nodos con CPUs y los nodos con Xeon Phi (se recomienda usar el sufijo -mic para Xeon Phi).
PhiSplitter
En primer lugar, calcula dentro del archivo de hosts, cuántas máquinas son nodos de CPUs y cuántas son nodos de Xeon Phi, debido a que estos factores son necesarios para calcular la cantidad de secuencias que contendrán los archivos. Cabe aclarar que este algoritmo crea archivos 3 veces más grandes para los nodos de CPUs que los archivos para los nodos de Xeon Phi.
TotalLongitudSeqArchivoEntrada = |
Longitud total de las secuencias a analizar, excluyendo su encabezado |
TotalNodosCPU = |
Número de nodos de CPUs incluidos en el archivo de hosts |
TotalNodosPhi = |
Número de nodos de Xeon Phi incluidos en el archivo de hosts |
El factor diferenciado de tamaño (tres) se encontró luego de pruebas de comparación de tiempos de ejecución de alineamientos con NCBI-Blast, realizados con los mismos archivos de entrada y la misma base de datos sobre Nodos de CPUs y Nodos de Xeon Phi.
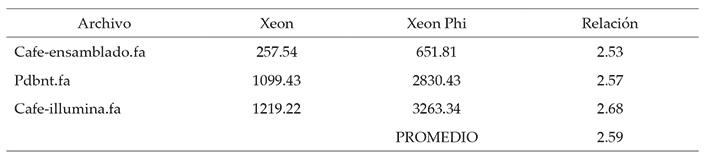
El uso de este algoritmo se recomienda cuando se ejecuta PB sobre nodos con CPUs y nodos de Xeon Phi (Tabla 4, Comparación Xeon y Xeon Phi).
Tabla 1 Archivos de secuencias

* Estas secuencias se pueden descargar en el siguiente enlace: (https://github.com/simonorozcoarias/ncbi-blast_sobre_xeon_phi)
El flujo de trabajo de PB-TP, en general, se presenta en la Figura 2, de flujo de trabajo PB-TP.
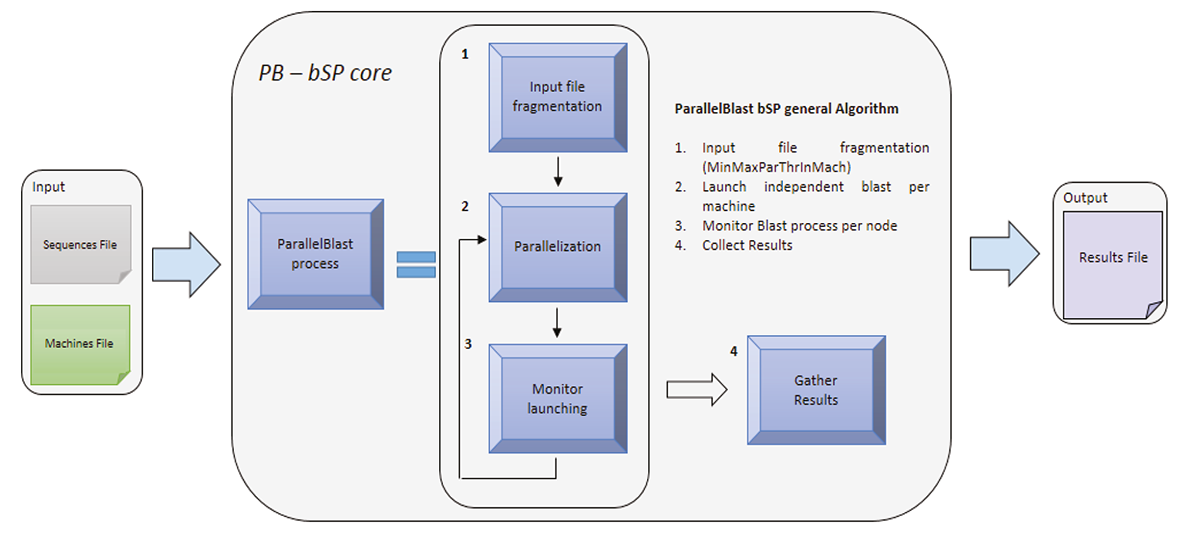
By-Sequence Parallelization
El segundo algoritmo de paralelización disponible en PB es by-Sequence Parallelization-bSP que consiste en dividir el archivo de secuencias de entrada en partes más pequeñas con el número de secuencias calculadas basado en el mínimo número de hilos disponibles en el grupo de máquinas incluidas en el archivo de hosts. Por ejemplo, si el archivo contiene dos máquinas con 32 hilos disponibles en la primera y 64 hilos disponibles en el segundo, el archivo de entrada se divide en 32 partes. A continuación, estas piezas se procesan en los nodos en un orden creciente acorde con los nodos que están disponibles o que ya terminaron de procesar su archivo de entrada. El número de secuencias a ser procesadas en un ciclo de PB-bSP se obtiene por la siguiente ecuación
donde
MinMaxParThrInMach = |
Mínimo número de hilos paralelos a ser ejecutados por máquina en el archivo de hosts |
El número total de ciclos se calcula (ciclo = procesamiento de una pieza más monitoreo de esa ejecución) de la siguiente forma
El flujo de trabajo de PB-bSP se muestra en la Figura 3.
Resultados
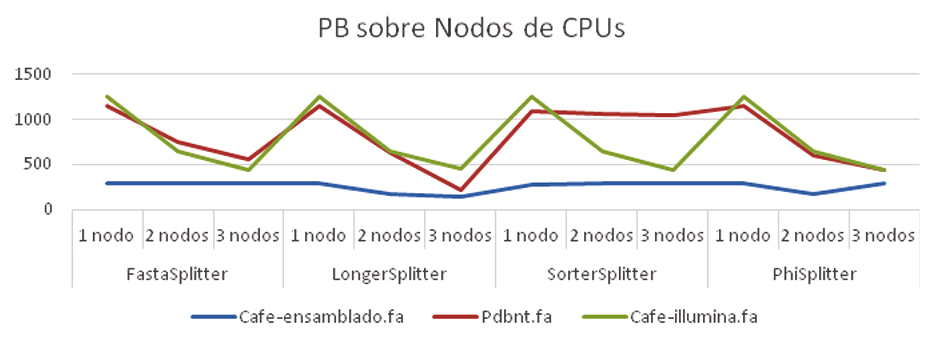
Las pruebas se realizaron sobre nodos de CPUs con Xeon E5-2670, cada uno con 32 cores (HT) a 2.60GHz y 256 GB de memoria RAM y con tarjetas Xeon Phi 5110p con 60 cores a 1.053 GHz, cada una con 8 GB de RAM y número total de 240 hilos. Es importante mencionar, que solo se incluyen los resultados del paradigma de paralelización TP, debido a que es el más efectivo. La versión de NCBI-Blast sobre la cual se ejecutaron las pruebas fue 2.2.31.
Los archivos se comparten bajo el protocolo NFS. En la Tabla 1 se presentan los archivos utilizados en las ejecuciones. Todos los archivos contienen bases de nucleótidos, además se usó la opción blastx de PB. El archivo de referencia fue env_nr [ftp://ftp.ncbi.nlm.nih.gov/blast/db/FASTA/env_nr.gz], que contiene 6.907.428 secuencias de aminoácidos, previamente formateado para NCBI-Blast.
Resultados algoritmos de división
A continuación, se muestran los resultados correspondientes a las salidas de los algoritmos de división utilizados por PB. Cabe aclarar que para el algoritmo de división PhiSplitter se emplearon 1 nodo de CPUs + 1 tarjeta Xeon Phi y 1 nodo de CPUs + 2 tarjetas Xeon Phi, respectivamente.
Los valores corresponden al porcentaje de peso en cada una de las partes en que se divide el archivo.
Comparación entre ejecución de NCBI-Blast en un nodo con CPUs y con una tarjeta Xeon Phi
A continuación, se muestran los resultados de la ejecución de PB sobre 1 nodo de CPUs y una tarjeta Xeo Phi. El tiempo se muestra en segundos. La base de datos de referencia fue la misma que para los demás análisis (env_nr).
Conclusiones
Se logró demostrar que por medio de la división de los archivos de entrada fue posible optimizar el tiempo de ejecución del programa NCBI-BLAST para el análisis de secuencias de nucleótidos usando CPUs en conjunto con Xeon Phi.
Un nodo con CPUs (Xeon E5-2670) con 32 cores en HT y 256 GB de memoria RAM, ejecutan en promedio 2.6 veces más rápido un alineamiento con NCBI-Blast que un nodo de Xeon Phi (5110p) accedido por SSH, con 60 cores, 240 hilos y 8 GB de memoria RAM. Sin embargo, implementado el esquema de paralelización TP combinado con el algoritmo de divisón PhiSplitter se logra usar de forma óptima PB sobre nodos de CPU y Xeon Phi al mismo tiempo. Esto se debe a que el algoritmo tiene en cuenta el factor de ejecución de los nodos con CPUs, eliminando así el tiempo de diferencia entre las múltiples ejecuciones y reduciendo considerablemente los tiempos muertos de los recursos.
Cuando el archivo de secuencias de entradas tiene muy pocas secuencias y estas tienen longitudes variantes, su paralelización se complejiza, afectado el tiempo de ejecución. No se logra una disminución considerable al usar las tarjetas Xeon Phi en comparación con su ejecución en una sola máquina.
Si el conjunto de nodos donde será ejecutado PB está compuesto únicamente por nodos con CPUs y el archivo de entrada consiste en secuencias de diferentes longitudes, es más óptimo usar el esquema de paralelización TP combinado con el algoritmo de división LongerSplitter, ya que tiene en cuenta las longitudes de las secuencias y crea archivos más uniformes.
En caso de que los nodos donde se ejecutará PB sean exclusivamente nodos de CPUs y el archivo de secuencias comprenda solo secuencias de la misma longitud, es recomendable el uso del esquema de paralelización TP y el algoritmo de división BIOS-fastaSplitter.











 nueva página del texto (beta)
nueva página del texto (beta)