1. Introducción
Desde el origen de la inteligencia artificial en los años cuarenta con algunos trabajos incipientes se buscó utilizar a las computadoras como herramienta de ayuda para resolver problemas de interés para los humanos [1].
Es a partir del influyente trabajo de Alan Turing en 1950 para determinar si una máquina era inteligente, realizando la prueba que lleva su nombre, que de manera formal inicial la inteligencia artificial y el número de contribuciones científicas relacionadas con esta área se incrementó significativamente, surgiendo así el Aprendizaje Automático, cuyo objetivo es desarrollar técnicas que permitan que las computadoras aprendan [1,2].
Una técnica usada para el aprendizaje automático son las redes neuronales artificiales. Las cuales consisten en un conjunto de unidades, llamadas neuronas artificiales, conectadas entre sí para transmitirse señales. Las evoluciones de los métodos de aprendizaje en conjunto con las redes neuronales dieron origen al aprendizaje profundo (Deep Learning) el cual está formado por un conjunto de algoritmos de aprendizaje automático que intenta modelar abstracciones de alto nivel en datos usando arquitecturas computacionales que admiten transformaciones no lineales múltiples e iterativas de datos expresados en forma matricial o tensorial.
Estas técnicas son usadas en gran cantidad de proyectos entre los cuales podemos encontrar el procesamiento digital de imágenes [1].
El procesamiento digital de imágenes ha adquirido, en años recientes, un papel importante en las tecnologías de la información y el cómputo. Actualmente, es la base de una creciente variedad de aplicaciones que incluyen diagnóstico médico, percepción remota, exploración espacial, visión por computadora, entre muchas otras. El procesamiento digital de imágenes (PDI) es el conjunto de técnicas que se aplica a las imágenes digitales con el objetivo de mejorar la calidad o facilitar la búsqueda de información, usando como herramienta principal una computadora. Hoy en día, el PDI es un área de investigación muy específica en computación [2].
Durante los últimos 15 años, un número creciente de técnicas referentes a imágenes digitales y su procesamiento en formato digital, ha sido introducido en la práctica médica [3]. Como en el caso del presente trabajo que se usa el procesamiento digital de imágenes para la detección de tuberculosis.
La tuberculosis (TB), también conocida como tisis, es una enfermedad infecciosa crónica, causada por un germen llamado Mycobacterium tuberculosis. La bacteria suele atacar los pulmones principalmente, pero puede también dañar otros órganos del cuerpo humano. La TB se disemina a través del aire, cuando una persona provocando, de esta manera, la propagación de la enfermedad. Puede ser prevenible y curable si es detectada a tiempo de otra forma podría causar la muerte del paciente. Para saber si una persona tiene la enfermedad de tuberculosis se pueden realizar pruebas como una radiografía de tórax o un cultivo de una muestra de esputo [5]. En México, una de las principales causas de mortalidad, es la tuberculosis con una tasa de 9.24 % por cada 100 mil habitantes, según datos del Sistema Estadístico y de Defunciones de la Dirección General de Epidemiología.
De acuerdo con la Organización Mundial de la Salud, la tuberculosis es una de las 10 principales con TB pulmonar tose, estornuda o habla [4] causas de mortalidad en el mundo. En 2015 cerca de 10.4 millones de personas enfermaron de tuberculosis y 1.8 millones murieron por esta enfermedad.
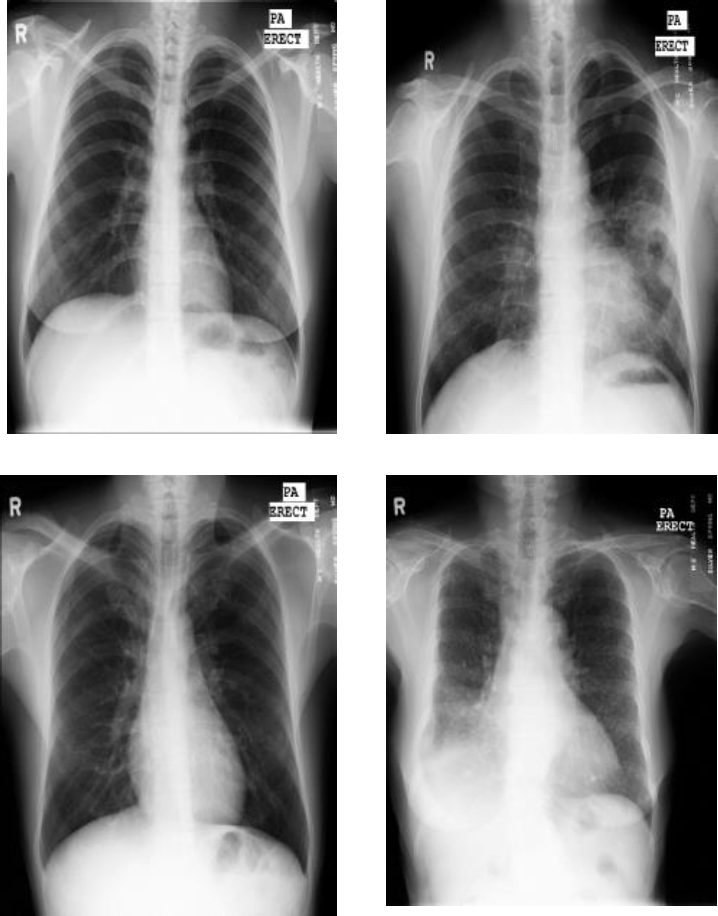
Más del 95% de las muertes por tuberculosis se producen en países del tercer mundo [6]. En la Figura 1 podemos apreciar 4 imágenes de Rayos X, las dos imágenes de la izquierda muestran a pacientes saludables y las dos de la derecha muestran a pacientes con la enfermedad detectada.
Los métodos de diagnóstico y tratamiento de la enfermedad han mejorado, sin embargo, no se alcanzará a cumplir la meta que se tenía para el 2020 de la Estrategia Fin a la TB [7]. Sin duda este problema de salud pública sigue siendo un gran reto para el sistema de salud de los países, principalmente en vías de desarrollo.
Un repaso muy interesante de la evolución de las técnicas de análisis y procesamiento de imagen médica desde los años 80 puede encontrarse en [8].
La clasificación de imágenes hace referencia a la tarea de extraer clases de información de una imagen, existen dos tipos de clasificación: supervisada y no supervisada. La clasificación supervisada parte de un conjunto de clases conocido, estas clases deben caracterizarse en función del conjunto de variables mediante la medición de las mismas en individuos cuya pertenencia a una de las clases no presente dudas, mientras que la clasificación no supervisada no establece ninguna clase, aunque es necesario determinar el número de clases que queremos establecer, y dejar que las defina un procedimiento estadístico [9].
En el presente trabajo se aplica la inteligencia artificial en la clasificación automática de imágenes radiografías de tórax de pacientes con tuberculosis y sin tuberculosis. Estas imágenes son clasificadas en dos posibles categorías: enfermo o sano.
El Resto del artículo se encuentra organizado de la siguiente manera: en la sección 2 se presenta una breve descripción del trabajo relacionado. En la sección 3 se presenta el método propuesto, así como una descripción de las diferentes etapas que lo componen. Por último, se presentan los resultados de la evaluación del método propuesto y las conclusiones del presente trabajo.
2. Trabajo relacionado
El procesamiento de imágenes tiene un amplio uso, por ejemplo, en [10] el procesamiento de imágenes es usado para extraer regiones de interés con propiedades que pueden ser potencialmente relacionadas con el diagnóstico médico de Parkinson, usa la tecnología de diagnóstico asistido por computadora para procesar las imágenes, extraer las texturas, hacer una segmentación de la imagen y encontrar el área de interés.
Dentro de [11] encontramos el uso del procesamiento de imágenes, reconocimiento de patrones e inteligencia artificial para ayudar a detectar clúster de micro calcificaciones en imágenes digitalizadas de mamografías. Existen otras técnicas para realizar el procesamiento de imágenes como es el caso de [12] utiliza una técnica de espectroscopia Raman para obtener mapas espectrales con resolución espacial especifica (1 a 5 micrómetros) sobre una región seleccionada de la muestra para acceder y visualizar información relevante sobre la distribución espacial en cualquier muestra acerca de su composición bioquímica.
Algunos trabajos de interés donde se puede encontrar un compendio de técnicas para mejorar imágenes médicas son [13-15], y para eliminación de ruido de la imagen usando técnicas que van desde la erosión, extracción y otras comúnmente utilizadas en el estado del arte, pueden ser consultadas en [16-18].
Otra investigación encontrada sobre el procesamiento de imágenes se encuentra en [19] la que al igual que [11] ayuda al pronto diagnóstico de cáncer de mamá utilizando procesamiento de imágenes, dentro de esta investigación podemos encontrar que usa la técnica de segmentación por textura, las imágenes utilizadas como prueba son de una base de datos, la cual cuenta con imágenes de masas cancerígenas y micro-calcificaciones manualmente etiquetadas por expertos. En [19] llevan a cabo la identificación de cáncer de mama utilizando imágenes térmicas, realiza un procesamiento digital de las imágenes, utilizando un análisis de textura de las imágenes para identificar y extraer todas las regiones de interés.
3. Método propuesto
El método propuesto fue desarrollado en Python. Las características de las imágenes que son utilizadas como atributos de clasificación son extraídas con KERAS. KERAS es una biblioteca de Redes Neuronales de código abierto creada en Python que contiene la arquitectura de RESNET50, esta arquitectura ayudará a extraer las características de las imágenes mediante arreglos.
En el presente trabajo se utilizaron tres métodos de clasificación: El primero método usado basado en máquinas de vectores de soporte (SVM, Support Vector Machine), el cual es un modelo supervisado de aprendizaje con algoritmos asociados que analizan los datos y reconocen patrones, se utiliza para la clasificación y el análisis de regresión [17].
El segundo método usado es basado en regresión logística (Logistic Regression), el cual es un algoritmo de aprendizaje automático de clasificación que se utiliza para predecir la probabilidad de una variable dependiente categórica que sea dicotómica, es decir, que contenga datos que puedan ser clasificados en una de dos posibles categorías (vivo o muerto, enfermo o sano, sí o no, etc.).
Una regresión logística, por lo tanto, requiere que la variable dependiente sea binaria. Además, el factor de nivel 1 debería representar el valor "deseado". Solamente las variables significativas deben incluirse como variables independientes que, a su vez, deberían ser independientes entre sí [16].
El tercer método usado es basado en los vecinos más cercanos (KNN, K-Neighbors Classifier), el cual es un algoritmo basado en instancias de tipo supervisado de Aprendizaje Automático. Este método es particularmente útil para clasificar nuevas muestras (valores discretos) o para predecir o estimar valores futuros (regresión, valores continuos). Sirve esencialmente para clasificar valores buscando los puntos de datos más similares (por cercanía) aprendidos en la etapa de entrenamiento y haciendo conjeturas de nuevos puntos basado en esa clasificación [2].
Para el presente trabajo se usó la base de datos Montgomery, las imágenes de rayos X de esta base de datos se adquirieron del programa de control de la tuberculosis del Departamento de Salud y Servicios Humanos del Condado de Montgomery, MD, EE. UU. Este conjunto contiene 138 radiografías, de las cuales 80 radiografías corresponden a pacientes sanos (normales) y 58 radiografías presentan manifestaciones de tuberculosis (anormales). Esta base de datos se encuentra disponible. Todas las imágenes están des-identificadas y disponibles en formato DICOM. El conjunto cubre una amplia gama de anomalías, incluyendo derrames y patrones miliares. El conjunto de datos incluye lecturas de radiología disponibles como un archivo de texto. Cada una de las imágenes tiene una etiqueta que ayuda a identificar las imágenes. Las etiquetas pueden ser: con TB (etiqueta con el número “1”, éxito), y normales o sin TB (Etiqueta con el número “0”, fracaso).
Se realizó un pre-procesamiento a las imágenes utilizadas. Existen dos partes principales en el pre procesamiento: (1) Relleno (Padding) (2) Redimensionamiento (Resizing). Ambas etapas se realizan después de que las imágenes fueron extraídas, estas etapas dan como resultado una matriz para cada imagen de entrada con dimensiones de 224x224 y con números del 0 al 255, esto corresponde a una imagen de 224x224 en 3 canales (RGB), la finalidad de este proceso es proporcionar a la red una matriz de estas dimensiones. Una vez que estas etapas fueron realizadas entran en la red ResNet50. En la figura 2 se muestra el proceso que realiza la Red.
La última etapa del pre procesamiento es cuando entran a la Red para extraer las características de las imágenes que serán utilizadas como atributos de clasificación. La Red toma como entrada la matriz generada de [x224x3] y en cada capa va realizando convoluciones a la matriz y de esta forma genera mapas de extracción.
En la penúltima capa de la Red se obtiene un vector de dimensión 2048 el cual contiene las características generales de la imagen tales como saturación, luminosidad, intensidad, entre otras.
3.1. Procesamiento para validación cruzada
Una vez que se obtienen los arreglos con las características de las imágenes para TB y Normal, se crean las etiquetas en un documento de texto para nombrar cada una de las imágenes que se utilizarán para el entrenamiento del programa. Dentro del programa de procesamiento se llaman las etiquetas y las características y se van creando las relaciones entre etiquetas y características para después ser convertidas en arreglos.
Cuando el sistema termina de ordenar los datos para su mejor interpretación convierte las relaciones en 0 y 1, para de esta forma poder interpretarlas, esta información en la entrada a los métodos de aprendizaje automático para llevar a cabo la clasificación de las imágenes.
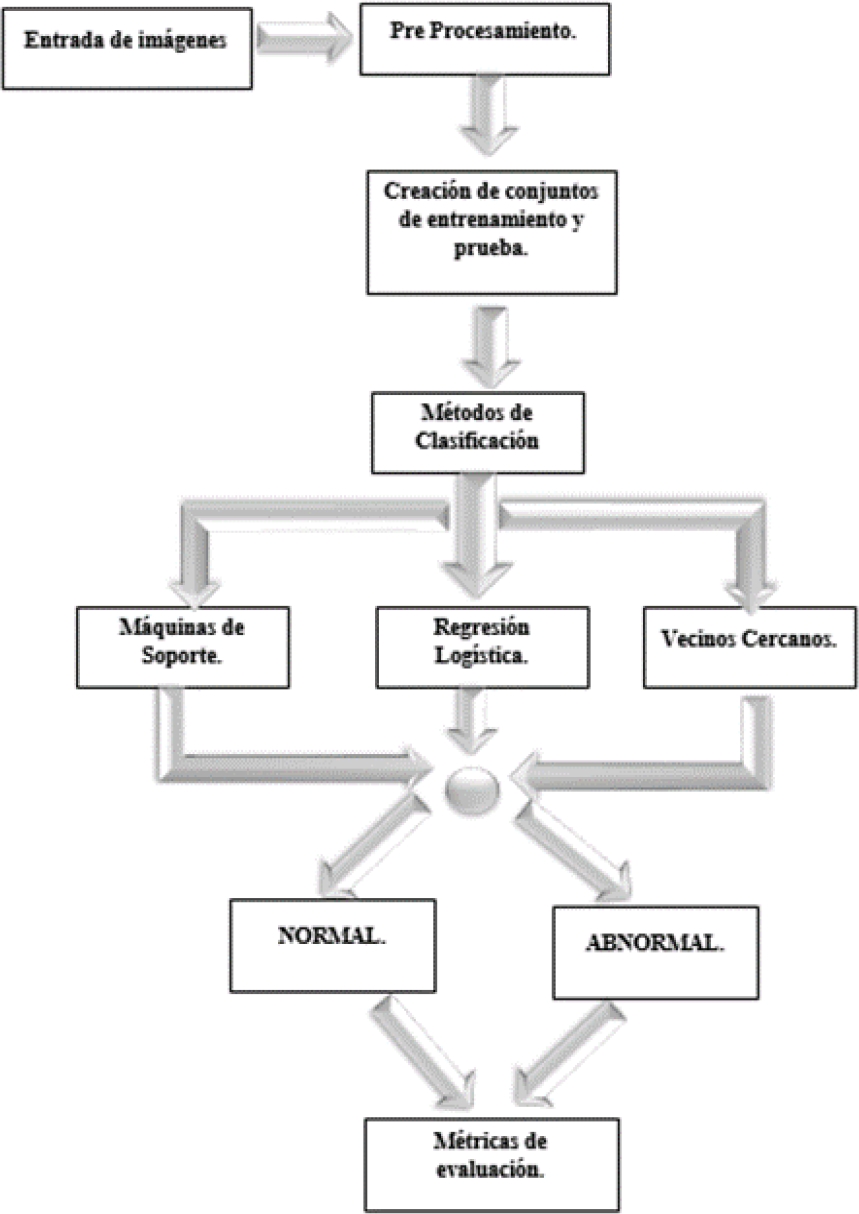
En la Figura 3 se muestra el diagrama del procesamiento realizado para el escenario de validación cruzada, con cada uno de los métodos de clasificación automática.
3.2 Procesamiento para conjunto de entrenamiento y prueba
Para el segundo escenario se crearon conjuntos de entrenamiento y prueba. 80% de las imágenes se usaron para el entrenamiento y el 20% restante se usó para prueba, esto con el fin de que las imágenes de prueba no sean vistas nuca por el conjunto de entrenamiento. En la Figura 4 se muestra el diagrama de la Metodología usada para los conjuntos de entrenamiento formados.
Para cada clasificación realizada, registramos las siguientes métricas de evaluación: Accuracy (exactitud), Precision (Precisión), Recall (Recuerdo) y F-Measure (medida estadística F). En el estado del arte es común nombrar a estas métricas por sus nombres en inglés.
En la siguiente sección se muestran los resultados obtenidos.
4. Resultados
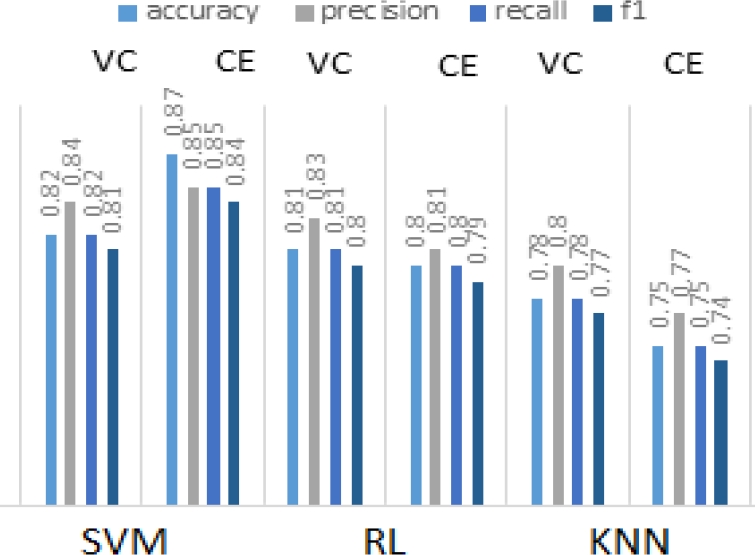
En la gráfica de la figura 5 se muestra una comparativa de resultados entre ambos escenarios, Validación cruzada (VC) y Conjuntos de entrenamiento (CE).
Para cada uno de ellos se muestran las cuatro métricas de evaluación registradas.
En la gráfica se puede observar que el mejor resultado de ambos escenarios se obtiene al utilizar SVM como método de aprendizaje, y los peores valores arrojados fueron lo de Vecinos cercanos para ambos escenarios de clasificación en prácticamente todas las métricas. En la tabla 1 se muestran los resultados obtenidos en el escenario de validación cruzada para las cuatro métricas de evaluación. Mientras que en la tabla 2 encontramos los resultados obtenidos con los conjuntos de entrenamiento y prueba formados.
Tabla 1 Resultados Validación Cruzada
| Clasificación | Accuracy | Precision | Recall | F1 |
| SVM | 0.82 | 0.84 | 0.82 | 0.81 |
| Logistic Regression | 0.81 | 0.83 | 0.81 | 0.80 |
| K- Neighbors Classifier | 0.78 | 0.80 | 0.78 | 0.77 |
Tabla 2 Resultados conjuntos de entrenamiento y prueba
| Clasificación | Accuracy | Precision | Recall | F1 |
| SVM | 0.87 | 0.85 | 0.85 | 0.84 |
| Logistic Regression | 0.80 | 0.81 | 0.80 | 0.79 |
| K- Neighbors Classifier | 0.75 | 0.77 | 0.75 | 0.74 |
El escenario que muestra un mejor desempeño es de los conjuntos de entrenamiento y prueba, el cual es el escenario de clasificación más deseable, cuando se cuenta con suficientes instancias para formar ambos conjuntos, debido a que el conjunto de entrenamiento nunca ve al conjunto de prueba, evitando de esta manera el poder tener algún tipo de influencia o tendencia a la hora de llevar a cabo la asignación de la categoría a la imagen bajo estudio. Se puede observar, además, que en ambos escenarios el clasificador que muestra un mejor desempeño es SVM.
5. Conclusiones
En el presente trabajo se presentan resultados de clasificación automática de imágenes médicas en dos categorías: con y sin tuberculosis.
Para llevar a cabo la clasificación, se extraen características utilizando aprendizaje profundo y la red neuronal RESNET-50. Se implementaron dos escenarios de clasificación: validación cruzada y formando conjuntos de entrenamiento y prueba. El escenario con mejores resultados fue en el que se formaron conjunto de entrenamiento y prueba con una exactitud superior al 85%.
El método de clasificación que muestra el mejor desempeño en los dos escenarios implementados en el presente trabajo es SVM. Como se puede apreciar en los resultados obtenidos en el presente trabajo, estos superan con mucho al azar, y permiten llevar a cabo la clasificación de imágenes de una manera eficiente. Los resultados obtenidos permiten ver la viabilidad de la metodología utilizada.
También nos permite identificar el mejor escenario de clasificación y método de aprendizaje automático para llevar a cabo la clasificación de radiografías con y sin tuberculosis.











 nova página do texto(beta)
nova página do texto(beta)