1. Introducción
En el área médica la inspección visual de imágenes microscópicas1 no solo proporciona información importante tanto cualitativamente como cuantitativamente ante la presencia de diversas patologías, también es útil para complementar exámenes de rutina aumentando la confiabilidad en el diagnóstico [1]-[3]. Particularmente, durante el análisis cualitativo de una imagen microscópica de células sanguíneas intervienen factores que pueden afectar su estudio e interpretación, tales como la calidad en la preparación de la muestra, o bien el cansancio y la falta de experiencia del especialista clínico. Respecto al análisis cuantitativo, aunque actualmente se dispone de tecnología especializada como los contadores hematológicos, su mayor desventaja es que no realizan un análisis visual de la imagen y su mecanismo de interpretación implica un tratamiento físico-químico de la muestra. Como una consecuencia, se destruye la muestra original. Además, la disponibilidad de dichos equipos y el costo de adquisición y mantenimiento, eleva los costos del examen de la muestra. Aunque existen métodos con alto grado de precisión para la identificación de los elementos de estudio [4], estos son poco accesibles por causas como disponibilidad geográfica o la falta de personal capacitado para usarla. Entre estos métodos se encuentran la citogenética y la biología molecular [5] así como la determinación del inmunofenotipo [6]. En los primeros se determinan alteraciones cromosómicas numéricas o estructurales, en tanto que en el último se experimentan reacciones con antígenos específicos para la identificación del tipo de patología.
Con estos precedentes, el procesamiento digital de imágenes se distingue como una alternativa adecuada para el análisis de imágenes microscópicas en el área médica, lo cual se observa claramente en la basta cantidad de técnicas propuestas hasta el momento para la detección de patologías como la leucemia [10]-[14] o el análisis microscópico de tejidos [15]; en los cuales la interpretación visual es imprescindible para lograr la identificación de los objetos de estudio. En relación a las fases que componen el procesamiento digital de imágenes, una de las más importantes es la segmentación cuyo propósito es obtener las regiones de interés que permitan describir mejor la información en la imagen. Su importancia radica en que los resultados obtenidos durante esta fase determinan el éxito en etapas posteriores de procesamiento. Es importante mencionar que en algunas aplicaciones de visión artificial o tratamiento de imágenes, el proceso de segmentación es usado como una etapa de preprocesamiento; en otras no se utiliza y en otras se considera como una etapa independiente. Particularmente este trabajo, esta enfocado al último caso.
Dentro de los enfoques de segmentación más comúnmente utilizados se encuentran: la umbralización [1], [10], [11], [13]-[16], [18], [21]-[23], [25], [26], [29], [31], [37], el agrupamiento [1], [14], [16], [19], [24], [27]-[31], los bordes [10]-[13], [15]-[17], [20], [22]-[24], la textura [16], el crecimiento de regiones [15], [16] y los contornos activos [10], entre otros. En la Tabla 1 se muestra una revisión breve al respecto, en la cual se puede apreciar el uso de combinaciones de técnicas utilizadas en el proceso de segmentación de muestras biológicas. No obstante, en la mayoría de los casos expuestos, la segmentación es el producto de la aplicación de una secuencia de variantes pertenecientes a enfoques específicos. Esta manera de tratar a la imagen provoca que ciertos criterios de homogeneidad, tales como: nivel de gris, color o textura, sean favorecidas en ciertas regiones de la imagen dejando en desventaja otros criterios que también deben ser cubiertos para lograr una segmentación adecuada. Idealmente, las regiones obtenidas de una segmentación deben ser uniformes y homogéneas con respecto a uno o varios criterios de homogeneidad; además, el interior de dichas regiones debe ser simple y sin muchos huecos pequeños. De igual manera, regiones adyacentes deben tener valores significativamente distintos con respecto al criterio de homogeneidad en el cual ellos son uniformes. Asimismo, los límites de cada segmento deben ser simples, espacialmente precisos y no irregulares [7].
Tabla 1 Enfoques comunes de segmentación utilizados en imágenes microscópicas
| Enfoque | Técnica Utilizada | Tipo de Imagen | # Imágenes | % Eficiencia | Referencia | ||||
| Agrupamiento | Umbralización | Bordes | Textura | Regiones | |||||
| X | X | X | ME, S, G, CxC, SR, SM | Tejido de carcinoma | 160 | 83 | [15] | ||
| X | X | X | X | X | AW, Th, S, SRG, Gr, THu, FC, RC | Histológicas y citológicas | - | - | [16] |
| X | C, OM, D, LL | Leucocitos normales | 113 | - | [17] | ||||
| X | UG, MC | ALL, AML | - | - | [18] | ||||
| X | KRC | ALL | 165 | - | [19] | ||||
| X | CC, So, C, D, HF | ALL (BD de ASH) | 80 | 98 | [20] | ||||
| X | M, D | ALL-IDB2 | 75b, 65n | 89.72 | [21] | ||||
| X | X | O, S | Frotis sanguíneo | 128 | - | [22] | |||
| X | X | propio | Frotis sanguíneo | 20 | 85 | [23] | |||
| X | C, D, E | ALL-IDB1 | 120 | 96.67 | [12] | ||||
| X | X | O, S | ALL, AML, CLL, CML | 90 | - | [13] | |||
| X | X | KMC, UG | Leucocitos, eritrocitos plaquetas | 78 | 95.5 | [1] | |||
| X | X | AZ, O, OM, AW, CA | Leucocitos normales y anormales | - | - | [10] | |||
| X | X | O, FCM, Km, TW | Leucocitos anormales | - | - | [11] | |||
| X | X | KNN, THu | Leucocitos | - | 92 | [24] | |||
| X | AZ | ALL-IDB1 | 108 | 92 | [25] | ||||
| X | O | ALL-IDB1, ALL-IDB2 | 121 | 32 | [26] | ||||
| X | O | Células de leucemia | - | - | [27] | ||||
| X | SDM, Di, WC | ALL-IDB2 | 180 | 36.72 | [28] | ||||
| X | X | FC | Eritrocitos leucocitos inmaduros | - | 80 | [29] | |||
| X | KMC, MSA | AML | 6 frotis | - | [30] | ||||
| X | X | TW, Km, EH, AC, G | ALL | - | 73.7 | [14] | |||
| X | X | O, TW, FCM | ALL | - | - | [31] | |||
CxC- Cruce por cero; AW- Algoritmos watershed; Th- Thresholding; SRG- SR growing; Gr- Grafos; THu-Transformada Hu; RC- Redes de convolución; OM- Operadores Morfológicos; D- Dilatación; LL- Llenado; MC- Mejora de contraste; KRC- Kernel induced rough C-means; CC- Clustering en color; So- Sobel; HF-Hole-filling; M- Manual; E- Erosión; AZ- Algoritmo Zack; CA- Contornos activos; FCM Fuzzy C-means; Km-K-means; TW- Transformada watershed; SDM- Estimulación de medidas discriminantes de vars; Di-Dispersión intra-entre clusters; WC- Watershed controlado; MSA- Mean-shift algorithm; AC- Ajuste de constraste; Ge- Geometría.
Las técnicas de segmentación mostradas en la Tabla 1 difieren en la forma en la que enfatizan uno o más de los criterios de homogeneidad deseados en la imagen. De ésta manera, si se considera el nivel de gris como un criterio de homogeneidad, entonces, técnicas como Otsu (O) [37], umbralización global (UG) [18], mínimo error (ME) [15], gradiente (Gr) [16], canny (C) [20] o snake (S) [22], brindan resultados adecuados en imágenes con histogramas preferentemente bimodales, debido a que se basan en el análisis del histograma de la imagen o bien, en cambios bruscos en el nivel de gris de pixeles contiguos. Por lo tanto, al estar claramente separados en clases (i.e., modas del histograma), se facilita su agrupamiento. Por otra parte, técnicas basadas en agrupamiento o crecimiento de regiones como K-means clustering (KMC) [30], fuzzy clustering (FC) [29], split merge (SM) o seeded region (SR) [15] tienden a producir regiones irregulares causadas por el cálculo inadecuado del número de grupos (i.e; número de regiones) requerido al inicio del algoritmo respectivo. En este caso, otros criterios de homogeneidad como el color o la textura se encuentran en desventaja al no ser evaluados. Por otra parte, considerando que las técnicas pertenecientes a un mismo enfoque de segmentación utilizan procesos distintos para determinar las regiones de interés, la imagen producida es diferente en cada caso.
En este sentido, los algoritmos evolutivos mul-tiobjetivo (Multiobjective Evolutionary Algorithms (MOEAs)) brindan una alternativa factible en el proceso de segmentación de imágenes al permitir evaluar más de una técnica al mismo tiempo, evitando el uso secuencial o semi-secuencial de múltiples técnicas de segmentación. En el presente trabajo se hace uso de un MOEA llamado NSGA-II (Non Sort Genetic Algorithm) propuesto en [32], el cual ha utilizado en la segmentación de imágenes. Por ejemplo, en [44] se propone un método de segmentación por umbralización que utiliza como funciones objetivo la varianza intra-clase ajustada, la entropía de Shannon y la entropía bidimensional.
El método se probó con 3 categorías de imágenes. Sin embargo, aunque los resultados de segmentación obtenidos son relativamente buenos para imágenes con un bajo nivel de ruido, su mayor desventaja radica en que este método utiliza la varianza intra-clase ajustada y por lo tanto, el uso de recursos computacionales aumenta durante el proceso de optimización. Por otro lado, Mukhopadhyay et. al. describen en [8] un esquema de agrupamiento genético difuso multiobjetivo para la segmentación de imágenes de resonancia magnética. Este esquema utiliza NSGA-II para obtener los parámetros para un agrupamiento óptimo, demostrando finalmente la eficiencia de su método con respecto a otras técnicas existentes. Finalmente, Faceli et. al. [9] presentan un método para determinar el número de grupos óptimo para una segmentación por agrupamiento utilizando NSGA-II. El método es probado con distintos tipos de imágenes mostrando un desempeño eficiente.
A continuación se describen algunos puntos relevantes respecto al problema de la segmentación de imágenes, mientras que en la Sección 3 se presentan los fundamentos teóricos. Las Secciones 4 y 5 contienen los detalles del desarrollo y las pruebas realizadas. Finalmente, en la Sección 6 se muestran las conclusiones.
2. Antecedentes
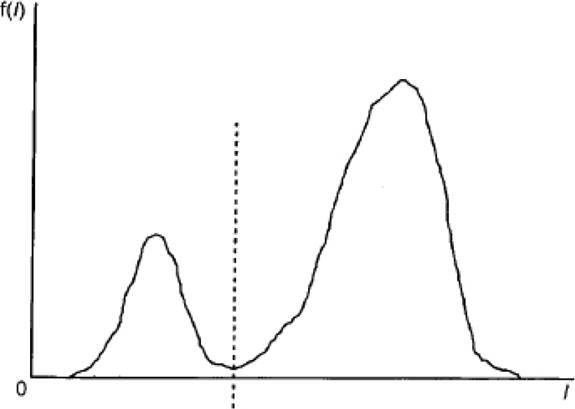
El enfoque de umbralización en la segmentación de imágenes es una de las técnicas utilizadas con mayor frecuencia en el procesamiento de imágenes. En general, en una imagen que contiene objetos que sobresalen del fondo, las modas estan bien definidas en sus histogramas. En la Figura 1 se muestra el caso de un histograma ideal, donde el valor mínimo de la distribución proporciona un nivel de intensidad de gris adecuado como valor de umbral (línea punteada). Además, la moda situada en la parte derecha de la Figura 1 representa el conjunto de pixeles más claros, mientras que la moda situada en la parte izquierda de la figura corresponde a los pixeles más obscuros.
Las técnicas de umbralización se basan en suponer que existe un solo objeto sobre un fondo uniforme, constituyendo una alternativa útil en esquemas de ajuste de iluminación o en el proceso de eliminación de fondos, lo cual puede simplificar las fases subsecuentes en el análisis de la imagen [33]. En la umbralización se considera la intensidad del nivel de gris en la imagen u otro atributo de los pixeles, de tal forma que al no tomar en cuenta la coherencia espacial de la región, es común que dos pixeles separados en la imagen puedan pertenecer a la misma región si sus atributos son similares. La Figura 2 presenta la segmentación de una imagen con distintos valores de umbral, haciendo evidente la importancia en el proceso de la determinación del valor adecuado (umbral) en el histograma para la separación de regiones.

Fig. 2 Caso de segmentación por umbralización de un eritrocito anormal. Sup.-Izq. Imagen original en escala de grises. Sup.-Der. Histograma de la imagen. Inf.-Izq. Segmentación con un valor de umbral de 55, Inf.-Der. Segmentación con un valor de umbral de 95
Un aspecto importante en la segmentación de imágenes es el criterio de uniformidad. En el caso del enfoque de umbralización, generalmente corresponde al nivel de intensidad de gris en la imagen. No obstante otros criterios que pueden regir el proceso de segmentación son: la brillantez, el color, la textura e incluso el movimiento. En el caso específico de imágenes con objetos grandes que presentan pocos detalles en su superficie, la segmentación implica dividir la imagen en regiones, donde cada región tiene un alto grado de uniformidad previamente establecido. Desafortunadamente, las necesidades existentes en el mundo real requieren satisfacer diversos criterios de uniformidad a un mismo tiempo para lograr resultados satisfactorios en el proceso de segmentación haciendo difícil encontrar un punto de equilibrio en el cumplimiento de los criterios requeridos. En el caso de las imágenes microscópicas que contienen objetos de interés tales como células, parásitos o bacterias, el enfoque de umbralización es útil debido a que en la mayoría de los casos los objetos a identificar resaltan con respecto al fondo, como se puede observar en la Figura 3. De ésta manera, obtener una sola región resulta útil para seleccionar el objeto de interés, o bien el fondo.
Como se ha mencionado anteriormente, si se considera la distribución de los niveles de gris como el criterio de uniformidad para llevar a cabo la segmentación, entonces el problema consiste en encontrar el valor de nivel gris o umbral que permita separar adecuadamente las modas en el histograma de la imagen, como se ilustra en la Figura 1. Por lo tanto, el conjunto de pixeles dentro del rango determinado por el umbral obtenido tiene niveles de gris similares y de esta manera, estos pixeles conforman una región de interés. Existen diversos métodos de segmentación por umbralización, en [34]-[36] se puede encontrar una amplia revisión al respecto. Algunas técnicas clásicas de este enfoque son el método de Otsu [37], la entropía entre clases [38], [39] y la umbralización del mínimo error [40]. Cada una de ellas trata la información del histograma de una forma distinta para obtener los valores de umbral, favoreciendo algunos criterios para ciertas categorías de imágenes y disminuyendo otros.
Por otra parte, el desempeño de los métodos se ve afectado cuando el histograma de la imagen no tiene modas claramente definidas o bien la cantidad de éstas aumenta, haciendo mas difícil determinar la posición de picos y valles. Estas características típicamente corresponden a imágenes con ruido o con leves cambios en sus niveles de gris (ver Figura 4). Por lo tanto, la variación en el proceso del cálculo de los umbrales y la presencia de varios picos en el histograma hace difícil encontrar los valores óptimos de umbral.
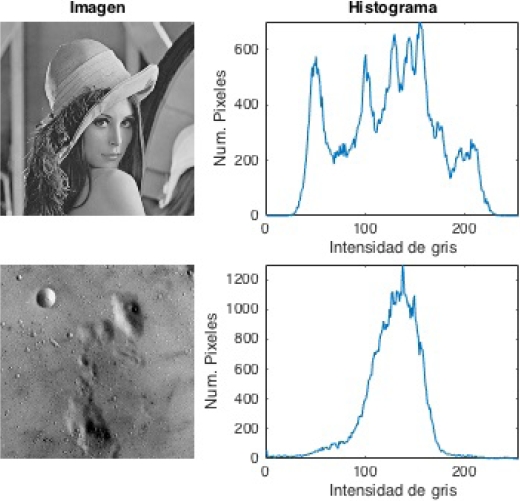
Fig. 4 Ejemplo de imágenes con histogramas que presentan varios picos y modas no bien definidas [41]
Una alternativa útil para abordar la problemática antes descrita es el uso de MOEAs. Los enfoques de cómputo evolutivo, inspirados por la evolución natural, hacen uso de operadores evolutivos comunes como: la selección, cruza y mutación; esto significa que a partir de una población de soluciones candidatas (individuos), se sigue un proceso de reproducción recombinando las soluciones existentes para generar nuevas soluciones. Por lo tanto, los procesos de selección de padres y los mecanismos de supervivencia inspirados en la evolución natural, permiten obtener un subconjunto de soluciones óptimas del problema en una sola ejecución [42]. En síntesis, las técnicas evolutivas son técnicas estocásticas que generan rápidamente una partición razonablemente óptima y garantizan la convergencia a una partición óptima asintóticamente [43].
La complejidad que caracteriza al proceso de segmentación de imágenes, hace que la consideración y evaluación de varios criterios simultáneamente denoten una mayor percepción de los compromisos existentes entre ellos. De tal forma que, obtener un conjunto de soluciones no dominadas implica un proceso de optimización multiobjetivo. Las técnicas de optimización mul-tiobjetivo, algoritmos evolutivos y bio-inspirados han demostrado resultados satisfactorios en la segmentación de imágenes [43]-[45]. Entre los criterios de umbralización reportados en la literatura y con más frecuencia utilizados estan: la varianza entre clases, la entropía, la probabilidad global de error y el método de ajuste de curvas Gaussianas [46]-[49]. Además, como es de esperarse, el consumo de recursos para el cálculo de cada criterio es distinto. En [50] se puede encontrar un caso aplicado para la segmentación de imágenes de eritrocitos, donde se evalúan los criterios de varianza intra-clase versus entropía y varianza entre-clase versus entropía, demostrando que la segunda combinación presenta un mejor desempeño respecto al uso de recursos computacionales en la evaluación de las funciones objetivo.
3. Fundamentos
3.1. Optimización multiobjetivo evolutiva
Los enfoques más comunes para resolver problemas de optimización multiobjetivo con algoritmos evolutivos son: métodos de agregación y las técnicas de población basadas en Pareto y no-Pareto [51]. En el presente trabajo se hace uso de una técnica de Pareto, cuyo mecanismo para la selección de la solución se basa en el concepto de dominancia de Pareto [52]. No obstante, independientemente del enfoque utilizado, un problema de optimización multiobjetivo generalmente tiene la forma [53]:
donde
El valor de cada función objetivo depende del vector de variables de decisión. De esta forma, el vector de funciones objetivo es denotado como:
Un vector de decisión
3.2. Algoritmo genético de ordenamiento no dominado (NSGA-II)
NSGA-II es uno de los métodos de optimización multiobjetivo evolutiva más utilizado [53]. Se caracteriza por encontrar múltiples soluciones óptimas de Pareto, seguir un principio elitista, utilizar un mecanismo explícito de preservación de la diversidad y hacer énfasis en las soluciones no dominadas. La Figura 5 muestra el esquema general de NSGA-II y su funcionamiento detallado se describe a continuación. En cualquier generación
3.3. Enfoques de segmentación
Los métodos de segmentación se pueden clasificar en enfoques paramétricos y no paramétricos. En el primer caso, se trata de ajustar el histograma de la imagen a una suma de distribuciones Gaussianas y posteriormente encontrar los umbrales óptimos en las intersecciones de estas Gaussianas. La desventaja de este enfoque es evidente para el caso de histogramas unimodales, donde al no existir valles resulta imposible localizar el umbral. En los enfoques de segmentación no paramétricos el cálculo de umbrales no requiere algún tipo de ajuste de parámetros, siendo el análisis de discriminantes [39] o la entropía [37], algunos de los métodos más utilizados y cuyo fin es maximizar la separabilidad de las clases. La segmentación por umbralización consiste en encontrar un valor de umbral que permita separar las regiones de interés en la imagen. En el caso de un solo valor de umbral se denomina segmentación binivel y la imagen de salida consiste en 2 regiones: objetos de interés y fondo. Cuando se obtienen 2 o más valores de umbral
Por otra parte, satisfacer todas las propiedades de una segmentación adecuada al mismo tiempo contrapone algunas de ellas, por ejemplo: regiones homogéneas y uniformes no solo tienen muchos agujeros pequeños sino también límites irregulares. Así, regiones adyacentes pueden ser mezcladas y los límites entre esas regiones se pierden [56]. Estos factores propician que las técnicas existentes sean útiles en dominios limitados y condiciones de adquisición de imágenes preferentemente controladas. En nuestro caso particular se hace uso del enfoque no paramétrico utilizando como funciones objetivo: la varianza entre-clase de Otsu y la entropía.
3.3.1. Varianza entre-clase de Otsu
El cálculo de la varianza entra-clase de Otsu tiene su fundamento en el análisis de discriminantes (o medidas de separabilidad de clase) [59]. El método sugiere maximizar la suma ponderada de las varianzas entre-clase de los pixeles del fondo y del objeto para establecer un umbral óptimo. Cabe decir que el mismo efecto se obtiene al minimizar la dispersión intra clase y como es de suponerse, el método proporciona resultados satisfactorios cuando la cantidad de pixeles en cada clase es similar [34].
Para definir las varianzas intra-clase y entre-clase, permítanos considerar que los niveles de gris
donde:
Las medidas de separabilidad de las clases (o medidas de criterio discriminante) se obtienen de la siguiente forma a partir del análisis de discriminantes:
La ecuación (6) es la varianza intra-clase mientras que (7) es la varianza entre-clase. A partir de esto, se observa que (6) requiere mayores recursos computacionales. La media total de la imagen original se define como:
De esta manera, el umbral óptimo
En nuestro caso se utiliza (7), el cual finalmente se define como:
3.3.2. Entropía
Similarmente como la varianza entre-clase de Otsu, otra técnica para obtener el umbral de una imagen, es el uso de la entropía de la distribucion de los niveles de gris; es decir, del grado de incertidumbre de los datos del histograma. De esta manera, si la entropía es la cantidad de información promedio que contienen los datos, entonces los niveles de gris con menor probabilidad son los que aportan mayor información. Cuando los datos son igualmente probables, todos aportan información relevante y la entropía es máxima [60]. Algunos autores sugieren que la maximización de la entropía de la imagen umbralizada es un indicativo de la máxima transferencia de información [38], [61]-[64]. En otros casos, la perspectiva consiste en tratar de minimizar la entropía cruzada entre la imagen de entrada en nivel de gris y la imagen de salida binaria como indicativo de preservación de la información [65]-[68], o bien como una medida de entropía difusa [69], [70].
Dentro de las primeras aplicaciones de la entropía a la umbralización de imágenes se encuentran [71], [72], donde se hace uso de la entropía de Shannon [60] para determinar los umbrales. Para definir el concepto de entropía de una imagen
donde
Así la entropía total de una imagen
Con estas consideraciones, el valor óptimo de umbral es aquel que maximiza la entropía total definida en (13). Por lo tanto, el umbral queda definido como un problema de optimización por:
4. Formulación del problema
Como se ha mencionado, el propósito de segmentar una imagen no es mas que agrupar los pixels que la componen en función de algún criterio de homogeneidad, de tal forma que como producto de la segmentación se obtengan regiones homogéneas distintas de acuerdo a los grupos de pixels que cumplan con dicho criterio. Este proceso de asignar los pixels que representan patrones de una cierta región requiere de un proceso de selección que generalmente contempla varias alternativas.
La formulación de la segmentación como un problema multiobjetivo permite evaluar de manera simultánea las alternativas consideradas para el agrupamiento de los pixeles. Comúnmente las opciones a evaluar estan en conflicto, siendo algunos ejemplos: maximizar la conectividad o minimizar el error del clasificador utilizado para la evaluación de la segmentación [43]. La consideración de esta combinación de alternativas es compleja, así el enfoque de optimización multiobjetivo es una técnica apropiada para resolver este problema.
Por otra parte, para segmentar una imagen usando el enfoque de umbralización existen diferentes técnicas, tales como la varianza entre-clase de Otsu, la entropía o las mezclas Gaussianas. Sin embargo, usualmente cada técnica proporciona un valor de umbral distinto, provocando diferencias en la cantidad de regiones obtenidas en la imagen de salida. En este trabajo se propone realizar una segmentación binivel con un enfoque de umbralización no paramétrico, utilizando como funciones objetivo la varianza entre-clase de Otsu y la entropía, de tal forma que el propósito es encontrar el valor de umbral adecuado que permita optimizar ambos objetivos. Por lo tanto, una vez optimizados los objetivos, se genera el frente de Pareto, el cual representa las soluciones no dominadas de las funciones objetivo. Cabe resaltar que el frente de Pareto también representa una manera simple de observar el compromiso entre las funciones objetivo, facilitando la toma de decisión en caso de requerir el uso de una única solución. Para comenzar con la formulación del problema de segmentación, a partir de (1) tenemos que:
donde
5. Resultados experimentales
Las imágenes utilizadas para el desarrollo del presente trabajo corresponden a glóbulos rojos anormales (dracriocitos). Los frotis sanguíneos preparados con una tinción de Giemsa fueron proporcionados por la Benemérita Universidad Autónoma de Puebla, México. Las imágenes fueron adquiridas con ayuda de una cámara digital de 1.3 megapixeles, la cual fue montada en un microscopio óptico y vistas con una precisión de 100X. La resolución de las imágenes fue ajustada a 128x128 pixeles y en ellas se puede apreciar una o más células del mismo o de distinto tipo, como se muestra en la Figura 6.

Fig. 6 Imágenes adquiridas. Las flechas señalan a las células de interés (dracriocitos), caracterizados por una ligera protuberancia en un extremo
La selección de los ejemplares de interés (dracriocitos) se realizó manualmente validándose por un experto del área clínica. De ésta manera se obtuvo un total de 71 imágenes. Es importante subrayar que NSGA-II esta codificado en C [32], y una vez obtenido el conjunto de umbrales sub-óptimos, el proceso de umbralización se realizó en Matlab V.7. La ejecución fue en un CPU core i7 con 8Mb de memoria.
Dado que la umbralización realizada es binivel, atendiendo la sugerencia de [39], se optó por utilizar solo el canal G de la imagen debido a que este presenta un mejor constraste, como se vislumbra en la Figura 7.
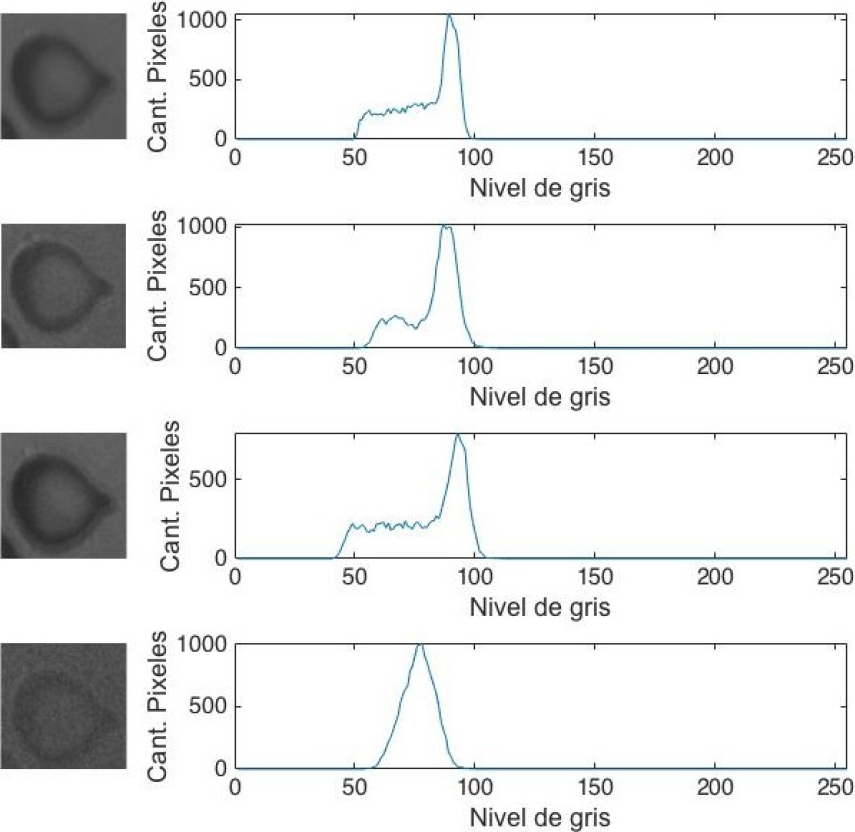
Fig. 7 Imágenes: escala de gris (fila 1), canal R, G y B, respectivamente en filas 2 a 4. A la derecha, histograma de cada imagen
El diagrama mostrado en la Figura 8 muestra las fases de la propuesta. El proceso de umbralización se llevó a cabo con tres conjuntos de imágenes. El primero está formado por las imágenes capturadas sin aplicar algún tipo de pre-procesamiento. El segundo conjunto corresponde a las imágenes obtenidas después de aplicar un filtro Gaussiano para eliminar ruido Gaussiano, dado que generalmente las imágenes adquiridas adquieren cierta cantidad de ruido Gaussiano caracterizado por pequeñas variaciones en la imagen y su origen radica en las diferentes ganancias del sensor, el ruido en la digitalización, etc [73]. El preprocesamiento consistió en aplicar un filtro Gaussiano de paso bajo de tamaño [3 3] y desviación estándar (sigma) 0.5. En el tercer conjunto se utiliza un grupo de imágenes a las cuales se agregó ruido Gaussiano con parámetros media 0 y varianza 0.01. La Figura 9 presenta imágenes correspondientes a cada una de estas categorías, tres ejemplos por columna.
En otro punto, para distinguir el compromiso existente entre las funciones objetivo definidas en (9) y (14), la Figura 10 muestra la representación gráfica de la varianza entre-clase de Otsu así como de la entropía obtenidas para la primera imagen de la Figura 9.
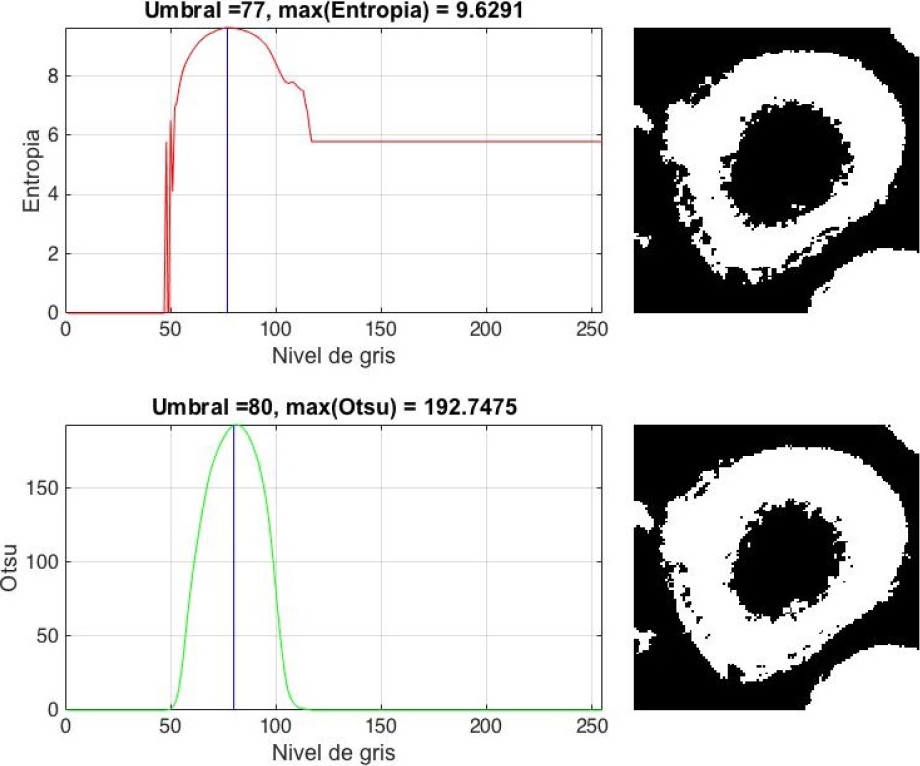
Fig. 10 Compromisos entre las funciones objetivo entropía y varianza entre-clase de Otsu. La línea azul representa la ubicación del valor de umbral. A la derecha se observan los efectos de la umbralización para entropía y varianza entre-clase de Otsu, respectivamente
En el caso de la varianza entre-clase de Otsu, el valor máximo es alcanzado en la posición 77, mientras que para la entropía este se obtiene en la posición 80. Esto provoca salidas distintas en la segmentación generando efectos que se hacen más evidentes en las imágenes con ruido, como se observa en la Figura 11, en la cual la diferencia en el valor del umbral obtenido por cada método difiere en más de 20 unidades. Considerando que el espacio de búsqueda es de 256 elementos, el efecto en la segmentación final es significativamente distinto.
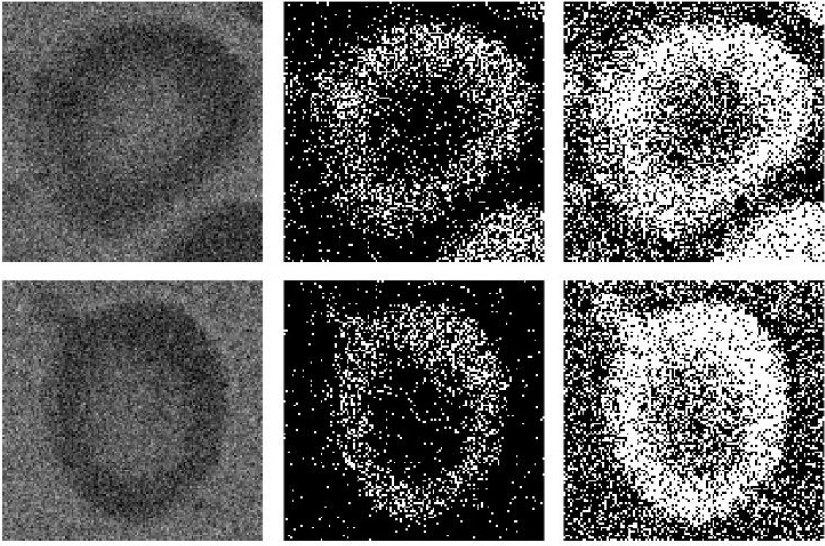
Fig. 11 Segmentación de imágenes con ruido. Columna 2.- con entropía, umbrales: img1= 54, img2= 62. Columna 3: con varianza entre-clase de Otsu, umbrales: img1= 80, img2= 86
Por otra parte, para generar los frentes de Pareto que proporcionan los umbrales óptimos al evaluar las funciones objetivo: varianza entre-clase de Otsu y entropía, se utiliza NSGA-II con cada conjunto de imágenes por separado, es decir; las imágenes sin preprocesamiento, las que incluyen preprocesamiento y las que tienen ruido Gaussiano.
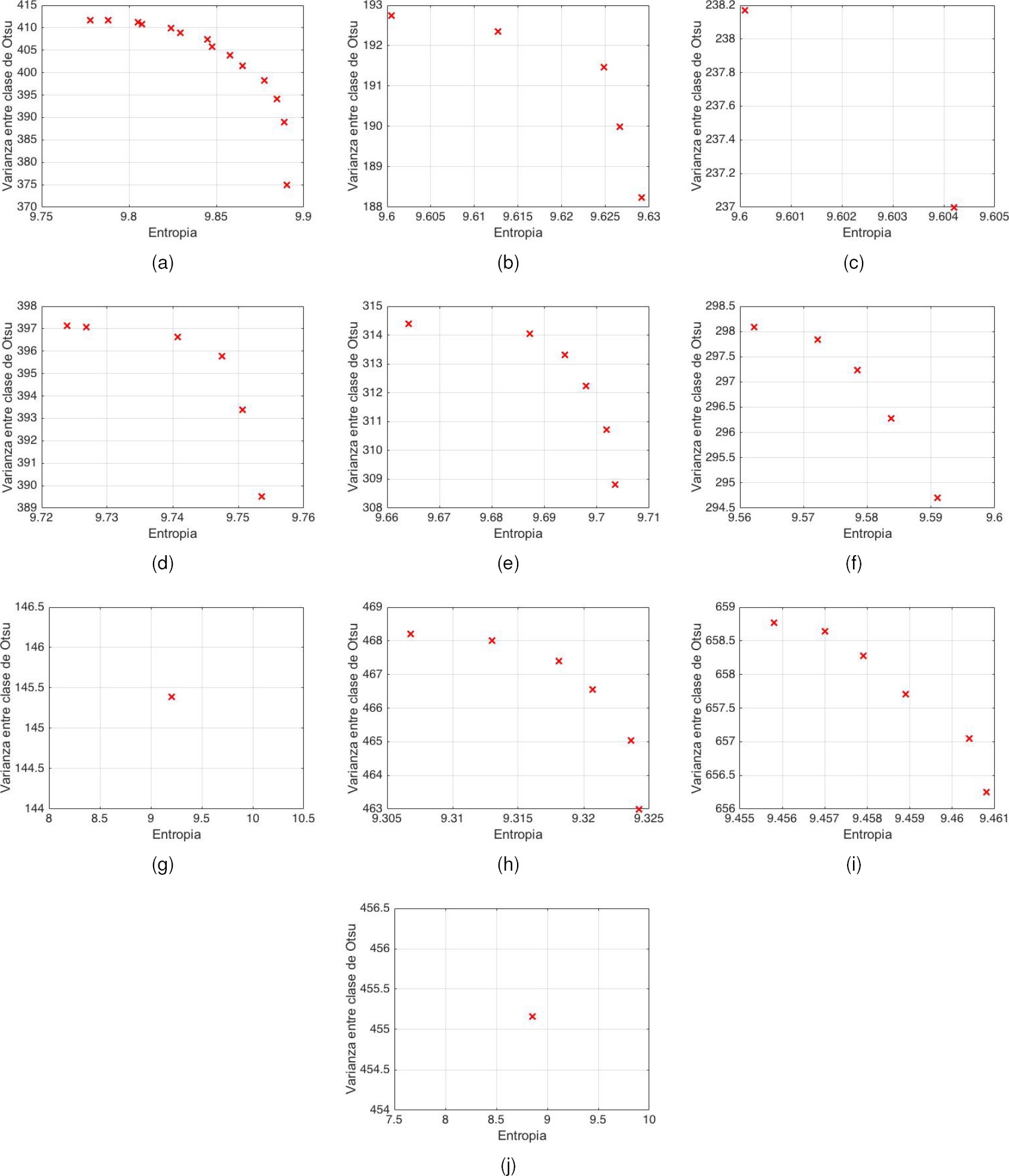
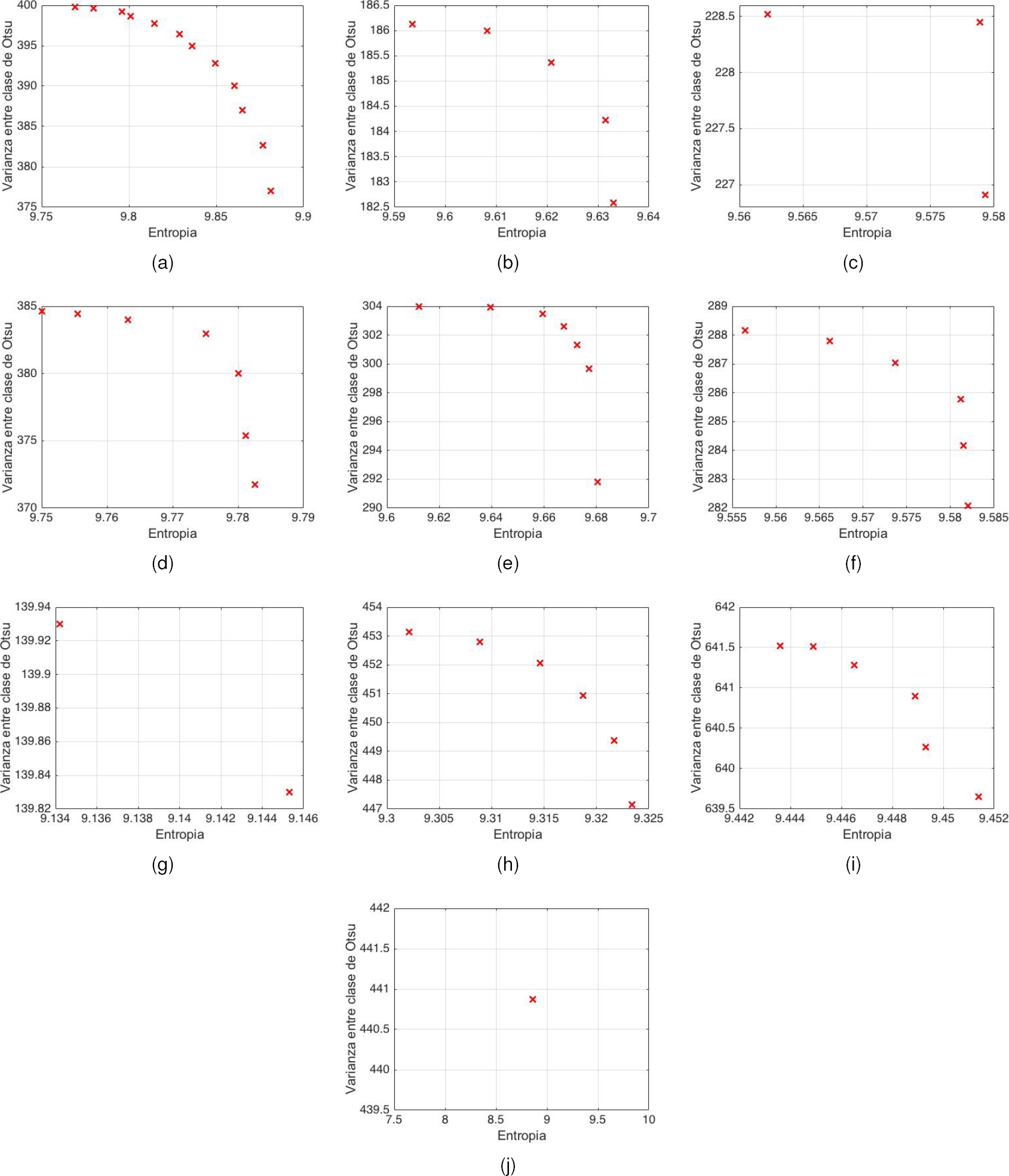
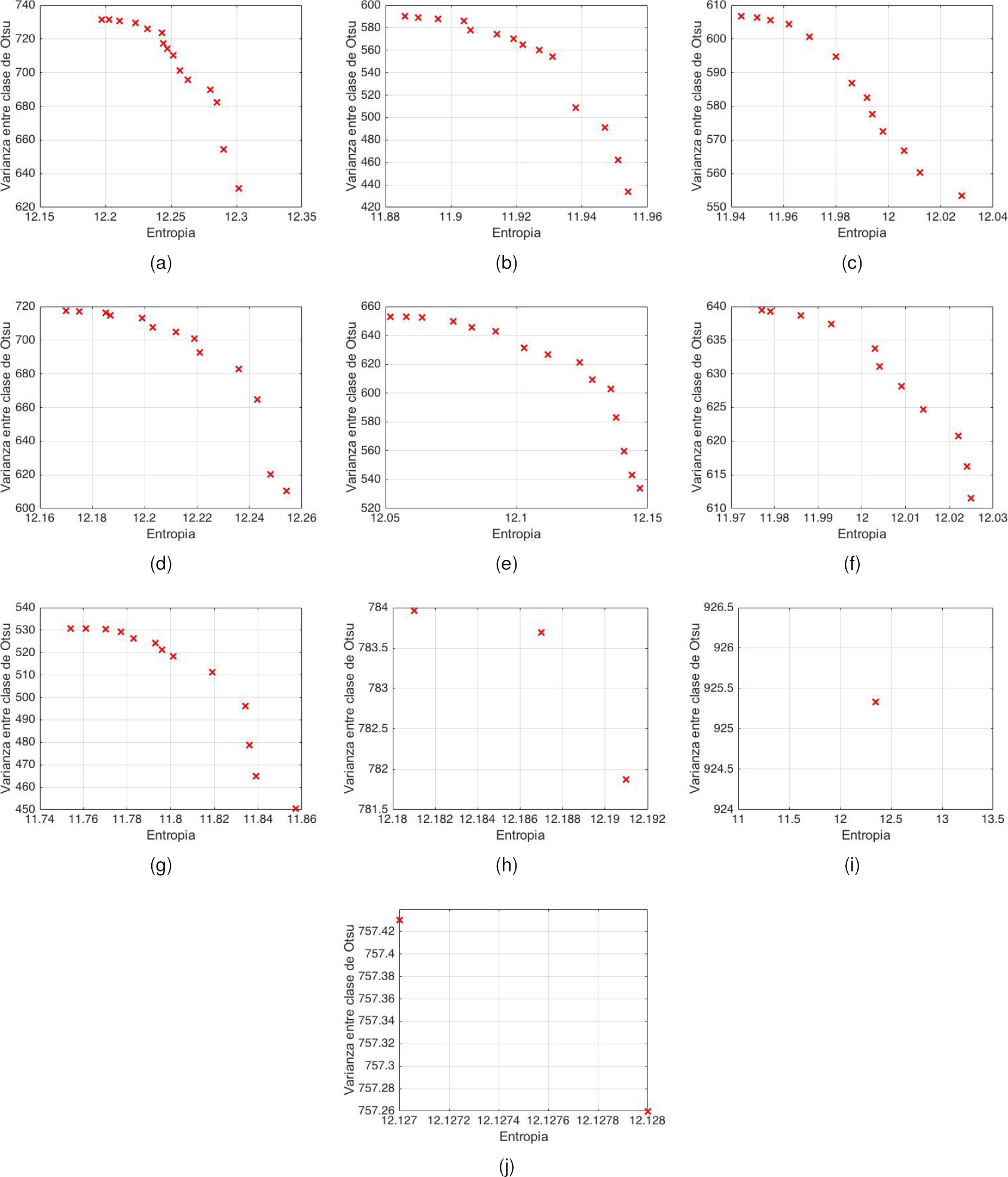
En las Figuras 12, 13 y 14 se muestran los frentes de Pareto generados para 10 imágenes de cada categoría, respectivamente. Por limitaciones de espacio, no se incluyen los frentes de Pareto de las 183 imágenes restantes.
En la Tabla 2 se muestran los valores de umbral para la segmentación de 10 imágenes obtenidos por NSGA-II a partir del conjunto de Pareto para cada categoría de imágenes. Para evaluar la segmentación y considerando que los elementos que constituyen el conjunto de Pareto representan las soluciones óptimas (umbrales), se tomaron tres elementos del conjunto en los casos que tuvieran 3 o más elementos dentro del conjunto de Pareto.
Tabla 2 Umbrales obtenidos por cada conjunto de Pareto
| Imágenes | |||
| Número | Preprocesamiento | Con ruido Gaussiano | |
| No | Sí | ||
| 1 | 81, 82, 83, 84, 85, 86, 87, 88, 89, 90, 91, 92, 93, 95 | 81, 82, 83, 84, 85, 86, 87, 88, 89, 90, 91, 92 | 65, 68, 72, 73, 74, 75, 77, 78, 79, 81, 82, 84, 85, 86, 87 |
| 2 | 76, 77, 78, 79, 80 | 81, 82, 83, 84, 85, 86, 87, 88, 89, 90, 91, 92 | 54, 57, 60, 62, 69, 70, 71, 72, 73, 74, 77, 78, 79, 81 |
| 3 | 82, 84 | 81, 82, 83, 84, 85, 86, 87, 88, 89, 90, 91, 92 | 70, 71, 72, 73, 74, 75, 76, 78, 80, 82, 83, 84, 85 |
| 4 | 84, 85, 86, 87, 89, 91 | 84, 85, 86, 87, 89, 91, 92 | 66, 67, 73, 76, 78, 80, 81, 82, 84, 85, 86, 87, 88 |
| 5 | 80, 81, 82, 84, 85 | 79, 80, 81, 82, 83, 84, 87 | 62, 63, 65, 68, 71, 72, 74, 75, 76, 79, 80, 82, 84, 85, 86 |
| 6 | 74, 75, 76, 77, 78 | 74, 75, 76, 77, 78, 79 | 66, 67, 68, 69, 70, 71, 72, 74, 75, 76, 77 |
| 7 | 75 | 74, 75 | 58, 60, 62, 65, 68, 70, 71, 72, 73, 75, 76, 77, 78 |
| 8 | 75, 76, 77, 78, 79, 80 | 75, 76, 77, 78, 79, 80 | 73, 75, 76 |
| 9 | 78, 79, 80, 81, 82, 83 | 77, 78, 79, 80, 81, 82 | 78 |
| 10 | 73 | 73 | 72 73 |
Estos elementos fueron los 2 de los extremos (izquierdo y derecho) y el situado en la parte media del conjunto. Así, por ejemplo en el caso de la imagen 1 del grupo de imágenes sin preprocesamiento fueron utilizados los valores 81, 87 y 95 (izq, med, der).
En las Tablas 3, 4 y 5 se enlistan los umbrales seleccionados para 10 imágenes. Estos corresponden a los umbrales seleccionados a partir del conjunto de Pareto generado por NSGA-II para cada imagen, y a las funciones objetivo varianza entre-clase de Otsu y entropía.
Tabla 3 Umbrales seleccionados para imágenes Sin preprocesamiento
| Valores de Umbral | |||||
| Num. Img. | NSGA-II | Varianza entre clase de Otsu | Entropía | ||
| Izq | Med | Der | |||
| 1 | 81 | 87 | 95 | 81 | 96 |
| 2 | 76 | 78 | 80 | 80 | 77 |
| 3 | 82 | - | 84 | 82 | 85 |
| 4 | 84 | 86 | 91 | 84 | 92 |
| 5 | 80 | 82 | 85 | 80 | 86 |
| 6 | 74 | 76 | 78 | 74 | 79 |
| 7 | 75 | - | - | 75 | 76 |
| 8 | 75 | 77 | 80 | 75 | 81 |
| 9 | 78 | 80 | 83 | 78 | 84 |
| 10 | 73 | - | - | 73 | 74 |
Tabla 4 Umbrales seleccionados para imágenes Con preprocesamiento
| Valores de Umbral | |||||
| Num. Img. | NSGA-II | Varianza entre clase de Otsu | Entropía | ||
| Izq | Med | Der | |||
| 1 | 81 | 87 | 92 | 81 | 93 |
| 2 | 76 | 78 | 80 | 80 | 77 |
| 3 | 81 | 82 | 84 | 81 | 85 |
| 4 | 84 | 87 | 92 | 84 | 93 |
| 5 | 79 | 82 | 87 | 79 | 88 |
| 6 | 74 | 76 | 78 | 74 | 79 |
| 7 | 74 | - | 75 | 74 | 76 |
| 8 | 75 | 77 | 80 | 75 | 81 |
| 9 | 77 | 79 | 82 | 77 | 83 |
| 10 | 73 | - | - | 73 | 74 |
Tabla 5 Umbrales seleccionados para imágenes con ruido Gaussiano
| Valores de Umbral | |||||
| Num. Img. | NSGA-II | Varianza entre clase de Otsu | Entropía | ||
| Izq | Med | Der | |||
| 1 | 65 | 78 | 78 | 87 | 66 |
| 2 | 54 | 71 | 81 | 81 | 55 |
| 3 | 70 | 76 | 85 | 85 | 49 |
| 4 | 66 | 81 | 88 | 88 | 67 |
| 5 | 62 | 75 | 86 | 86 | 63 |
| 6 | 66 | 71 | 77 | 77 | 67 |
| 7 | 58 | 71 | 78 | 78 | 59 |
| 8 | 73 | 75 | 76 | 76 | 68 |
| 9 | 78 | - | - | 78 | 80 |
| 10 | 72 | - | 73 | 73 | 75 |
En las Figuras 15, 16 y 17 se pueden observar los resultados de la segmentación con los valores de umbral mostrados en las Tablas 3, 4 y 5, respectivamente. En todos los casos, las imágenes que aparecen en negro dentro de la respectiva figura representan la ausencia de un umbral, por lo que no existe salida visible.
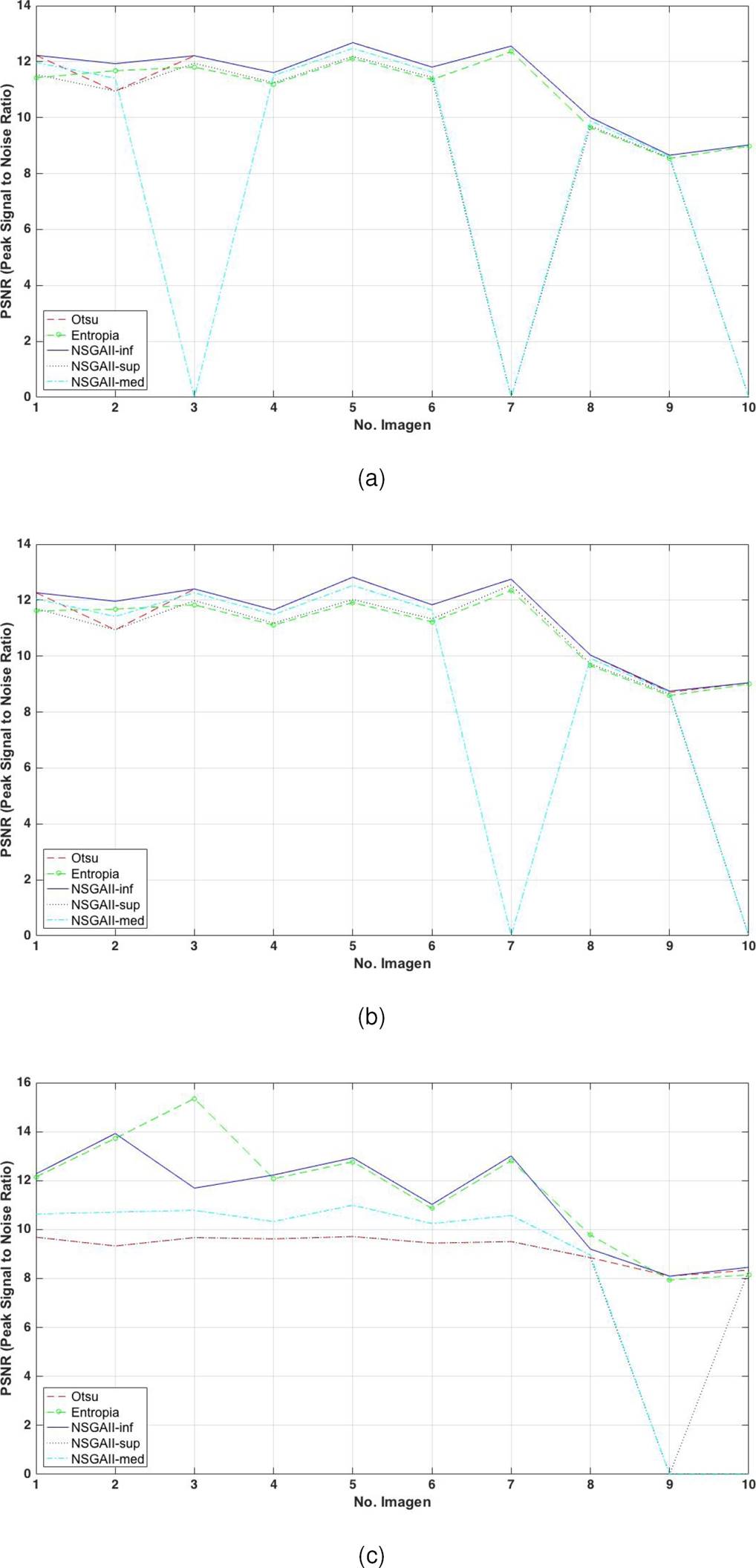
Para evaluar el desempeño de la segmentación en cada caso se utilizó el Peak Signal to Noise Ratio (PSNR), un método de evaluación no supervisado cuyo propósito es obtener una medida cualitativa de la imagen segmentada. Un valor alto de PNSR significa que la imagen es mejor cualitativamente. Las ecuaciones utilizadas para su cálculo están dadas en [45]. En las Tablas 6, 7 y 8 se muestran los valores de PSNR correspondientes a la segmentación utilizando los umbrales incluidos en las Tablas 3, 4 y 5.
Tabla 6 Resultados de PSNR para imágenes Sin preprocesamiento
| Valores de PSNR | |||||
| Num. Img. | NSGA-II | Varianza entre clase de Otsu | Entropía | ||
| Izq | Med | Der | |||
| 1 | 12.226 | 11.9690 | 11.5377 | 12.2263 | 11.4236 |
| 2 | 11.9293 | 11.4077 | 10.9474 | 10.9474 | 11.6767 |
| 3 | 12.2112 | - | 11.9424 | 12.2112 | 11.8037 |
| 4 | 11.6060 | 11.5054 | 11.2471 | 11.6060 | 11.1975 |
| 5 | 12.6796 | 12.4736 | 12.1864 | 12.6796 | 12.1114 |
| 6 | 11.8062 | 11.6286 | 11.4407 | 11.8062 | 11.3572 |
| 7 | 12.5553 | - | - | 12.5553 | 12.3565 |
| 8 | 10.0048 | 9.8821 | 9.6959 | 10.0048 | 9.6385 |
| 9 | 8.6564 | 8.6139 | 8.5604 | 8.6564 | 8.5398 |
| 10 | 9.0215 | - | - | 9.0215 | 8.9787 |
Tabla 7 Resultados de PSNR para imágenes Con preprocesamiento
| Valores de PSNR | |||||
| Num. Img. | NSGA-II | Varianza entre clase de Otsu | Entropía | ||
| Izq | Med | Der | |||
| 1 | 12.2666 | 12.0463 | 11.6974 | 12.2666 | 11.6131 |
| 2 | 11.9598 | 11.4152 | 10.9402 | 10.9402 | 11.6776 |
| 3 | 12.4013 | 12.2630 | 11.9850 | 12.4013 | 11.8310 |
| 4 | 11.6503 | 11.4827 | 11.1803 | 11.6503 | 11.1113 |
| 5 | 12.8224 | 12.5240 | 12.0247 | 12.8224 | 11.9126 |
| 6 | 11.8352 | 11.6412 | 11.3296 | 11.8352 | 11.2243 |
| 7 | 12.7498 | - | 12.5441 | 12.7498 | 12.3434 |
| 8 | 10.0365 | 9.9154 | 9.7229 | 10.0305 | 9.6624 |
| 9 | 8.7482 | 8.7055 | 8.6408 | 8.7055 | 8.5857 |
| 10 | 9.0459 | - | - | 9.0459 | 9.0011 |
Tabla 8 Resultados de PSNR para imágenes con ruido Gaussiano
| Valores de PSNR | |||||
| Num. Img. | NSGA-II | Varianza entre clase de Otsu | Entropía | ||
| Izq | Med | Der | |||
| 1 | 12.2795 | 10.6358 | 9.6880 | 9.6880 | 12.1487 |
| 2 | 13.9237 | 10.7133 | 9.3311 | 9.3311 | 13.7260 |
| 3 | 11.6944 | 10.7860 | 9.6723 | 9.6723 | 15.351 |
| 4 | 12.2299 | 10.3280 | 9.6204 | 9.6204 | 12.0766 |
| 5 | 12.9316 | 10.9956 | 9.7155 | 9.7155 | 12.7697 |
| 6 | 11.0267 | 10.2439 | 9.4484 | 9.4484 | 10.2439 |
| 7 | 13.0106 | 10.5773 | 9.5078 | 9.5078 | 12.8162 |
| 8 | 9.2035 | 8.9597 | 8.8521 | 8.8521 | 9.7779 |
| 9 | 8.097 | - | - | 8.0970 | 7.9423 |
| 10 | 8.4598 | - | 8.3419 | 8.3419 | 8.1505 |
Como se puede observar en la Figura 18, en la mayoría de los casos para todas las categorías de imágenes los mejores resultados en la evaluación cualtitativa se obtienen al utilizar el valor inferior del conjunto de Pareto generado con NSGA-II. Cabe resaltar que el desempeño de Otsu también es bueno en las imágenes sin ruido, ocurriendo lo contrario en el caso de la entropía donde se distinguen resultados satisfactorios en algunas imágenes con ruido. En relación a las imágenes con preprocesamiento, no se percibe alguna mejora con respecto a las imágenes sin preprocesamiento. Esto sugiere que la cantidad de ruido presente en las imágenes originales no es significativo.
6. Conclusiones
En este trabajo se ha descrito el uso de NSGA-II para la segmentación binivel de imágenes microscópicas utilizando un enfoque de umbraliza-ción. Dos funciones objetivo, varianza entre-clase de Otsu y entropía, fueron utilizados en el proceso de optimización. Aunque el uso independiente de estas funciones objetivo para encontrar el umbral de una categoría de imagen es rápido, su mayor problema radica en que para imágenes ruidosas, su rendimiento es bajo y no es posible, en general, evaluar los compromisos entre las funciones objetivo.
Además, los valores de umbral para cada caso es distinto y como una consecuencia, el número de regiones generadas es diferente en la imagen de salida. Por un lado, una ventaja importante sobre el uso de técnicas de segmentación de imágenes basadas en MOEAs, es la posibilidad de evaluar criterios de segmentación distintos en una sola ejecución. Por otro lado, el frente de Pareto representa un conjunto de soluciones no dominadas y muestran el compromiso entre las funciones objetivo, las cuales son alternativas viables al ser integradas en aplicaciones específicas, como la identificación de regiones en imágenes médicas que pertenecen a patologías específicas, facilitando con ello la labor de interpretación de las muestras observadas.











 nueva página del texto (beta)
nueva página del texto (beta)