Introducción
En México, el uso de variedades sintéticas (VSs) de maíz (Zea mays L.) formadas con líneas puras de rendimiento alto no es común debido a la escasez de este tipo de progenitores (Espinosa et al., 2002; Luna et al., 2012) y a la reducida oferta de semilla barata de híbridos de cruza simple sobresalientes. Pérez-López et al. (2014) mencionan que por esto se recurre a la producción de híbridos trilineales (HTs) o dobles, pero su siembra por un mismo agricultor en forma recurrente es poco frecuente. Algunos agricultores explotan sus generaciones avanzadas en lugar de comprar semilla original nuevamente. Las poblaciones resultantes se podrían considerar como las VSs que se formarían con los progenitores de los híbridos cultivados, pero puede no ser así (Márquez-Sánchez, 2010; Sahagún-Castellanos y Villanueva-Verduzco, 2012). De las diferencias entre estos dos tipos de poblaciones destaca la debida a la participación balanceada del material genético de las líneas en la VS que éstas formarían, lo cual no ocurre cuando los progenitores son HTs formados con las mismas líneas, por ejemplo. En este caso, cada línea de una cruza simple contribuye con la mitad del material genético en relación a lo que aporta la tercera línea de la cruza trilineal. Esta diferencia importante se manifiesta en el coeficiente de endogamia que reporta Márquez-Sánchez (2010) para una VS formada con HTs construidos con líneas puras. Las diferencias también pueden surgir cuando hay erosión genética. Escalante-González et al. (2013) consideran que el azar del mecanismo genético, el número finito de individuos que representan cada progenitor de una VS, y la condición heterocigótica de sus genotipos posibilitan la pérdida de genes durante su desarrollo. Ellos estudiaron la ocurrencia de erosión genética durante la formación de VSs cuyos progenitores son cruzas simples y encontraron que la pérdida de genes no idénticos por descendencia (NIPD) se relaciona inversamente con el coeficiente de endogamia de sus progenitores. Además, por diferencias en las frecuencias génicas con que las líneas participan en las VSs debe haber pérdida de genes NIPD en cuantía diferente entre aquellas formadas por sólo líneas y las generadas por HTs cuyos progenitores son las mismas líneas, cualquiera que sea su coeficiente de endogamia.
Respecto a la formación de VSs a partir de HTs, el objetivo de esta investigación fue determinar la magnitud de la erosión genética que ocurriría durante la formación de la muestra de individuos que representan a cada progenitor.
Materiales y métodos
El modelo de un locus en una especie diploide reproducida por apareamiento aleatorio se consideró en esta investigación. Al respecto se supuso que las L líneas iniciales con las que se forman los HTs no tienen parentesco, que su coeficiente de endogamia es F (0≤F≤1), y que L es múltiplo de tres. De acuerdo con las características de las líneas, independientemente de las frecuencias génicas, se supuso que cada una de ellas contiene 2-F genes NIPD.
El número de genes NIPD que en promedio pierde un HT [(NIPD)pt] se expresó como el número inicial de los que tienen sus tres progenitores [3(2-F)], menos el número promedio de genes NIPD que llegan a sus m representantes. La variable que representó este número se definió como Y m , y su promedio como E(Y m ), o sea, (NIPD) pt = 3(2-F) E(Y m ).
Se asumió que los progenitores de un HT son A 1 A 2, B 1 B 2 y C 1 C 2, y que, sin pérdida de generalidad, éste se representa como (A 1 A 2 x B 1 B 2) x C 1 C 2, con arreglo genotípico [(AGE) CT ]:
Además, sus m representantes son los elementos de una muestra aleatoria tomada con reemplazo de la población formada por sus ocho genotipos (Ecuación 1).
Los resultados posibles del muestreo con reemplazo de la progenie de un HT (Ecuación 1) incluyen desde el caso en que sus m elementos son el mismo genotipo m veces, hasta cuando la muestra contenga los ocho genotipos de la progenie. Para este último caso se requiere que m≥38.
Cuando m es grande hay un gran número de posibilidades de frecuencias y órdenes de ocurrencia de los genotipos que integran cada muestra y en ésta se determinó el número de genes NIPD diferentes (sin importar su frecuencia), y el número de formas (permutaciones) en que este número de genes puede ocurrir. Al calcular el número de eventos igualmente posibles y mutuamente excluyentes que puede producir el muestreo (8 m ), se calcularon E(Y m ) y el número de genes NIPD que se pierden en el paso de la población virtual de un HT (arreglo genotípico de la Ecuación 1) a la muestra que lo representa.
Resultados y Discusión
Para derivar una fórmula para E(Y m ) se consideró que la muestra de tamaño m con reemplazo extrae g genotipos de los indicados en la Ecuación 1 (g=1, 2,..., 8) y se definió:
1)
2)
Con ambos términos se construyeron dos cocientes que permiten calcular:
1) El número de formas en que pueden ocurrir en la muestra g genotipos cualesquiera con el k-ésimo juego de g frecuencias del tipo
El número de permutaciones para asignar los g elementos del k-ésimo juego de frecuencias a sendos genotipos de la muestra
Cualquiera que sea la forma de asociación entre frecuencias y genotipos, el número de genes NIPD que éstos aportan es el mismo.
El producto
Si del r-ésimo grupo de
En una muestra de tamaño m el número de genotipos diferentes (g) puede ser 1, 2, 3, ..., 8 (cuando m38). Además, el número de juegos de frecuencias depende de g (k=1, 2, 3, ..., Ug). Con ambas consideraciones y por la Ecuación 4, el número esperado (promedio) de genes NIPD en los m representantes de un HT [E(Y m )] es:
Como el número de genes NIPD que contiene el arreglo genotípico de un HT es 3(2-F), el promedio de genes NIPD que pierde cada híbrido trilineal [(NIPD)pt] se expresó como:
Como ejemplo, cuando m=12 y g=6, digamos G 1, G 2, ..., G 6, hay 11 juegos de frecuencias posibles en que estos últimos pueden ocurrir (k = 1, 2, ..., 11; Cuadro 1). Si k = 1 (juego de frecuencias para seis genotipos cualesquiera en que cada uno de cinco de ellos ocurre una vez y el sexto lo hace siete veces), las frecuencias pueden asociarse a los seis genotipos en seis formas diferentes [6!/(5! 1!) = 6]. Además, el número de formas en que pueden ocurrir estos seis genotipos con m=12 es 12!/(1!1!1!1!1!7!) = 95 040 (Cuadro 1).
Cuadro 1 Tres características del muestreo aleatorio con reemplazo cuando m=12 y g=6 del arreglo genotípico que produce (A
1
A
2
x B
1
B
2
) x C
1
C
2
: 1) los 11 juegos posibles de frecuencias genotípicas (k), 2) las permutaciones para asociar las frecuencias de cada juegoa los seis genotipos
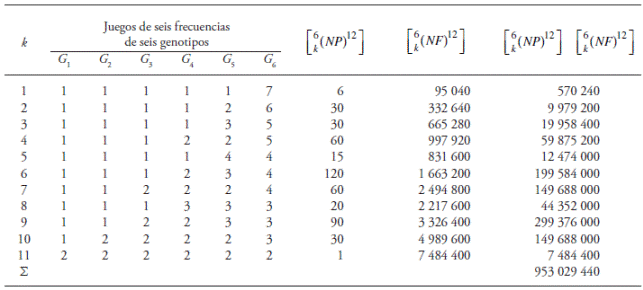
En este ejemplo, para cada uno de los 28 conjuntos diferentes de seis genotipos los juegos posibles de frecuencias son 11. Complementariamente, se muestra los números de formas de asignar las frecuencias de cada juego
Si los seis genotipos que aparecen en la muestra fueran A
1
C
1, A
1
C
2, A
2
C
1, A
2
C
2, B
1
C
1 y B
1
C
2 (Ecuación 1), éstos aportarían 5-2F genes NIPD. Con base en esto, por la información en el Cuadro 1 y aplicando las Ecuaciones 1 a 4, su contribución cuando
Para determinar la contribución total cuando m=12 y g=6 se debe considerar la aportación de los restantes 27 conjuntos de seis genotipos que puede producir el muestreo y la cantidad de genes NIPD que cada uno de éstos contiene (Cuadro 2), así como todos los datos que permiten la determinación de E(Y m ) para cada uno de 11 valores de m (Cuadro 3).
Cuadro 2 Ejemplos de conjuntos de g genotipos (g = 1, 2, 3, ..., 8) que contienen y m genes no idénticos por descendencia y número de casos posibles. Los genotipos se obtienen por muestreo aleatorio de tamaño m con reemplazo del conjunto de los ocho genotipos que produce la cruza (A 1 A 2 x B 1 B 2 ) x C 1 C 2 (Ecuación 1).
| g | Ejemplos | y m | Casos posibles | g | Ejemplos | y m | Casos posibles | |
| 1 | A 1 C 1 | 2 | 8 | 5 | A 1 C 1, A 1 C 2, A2C1, | |||
| A 2 C 2, B 1 C 1 | 5 - 2F | 24 | ||||||
| 2 | A 1 C 1, A 1 C 2 | 3 - F † | 8 | A 1 C 1, A 1 C 2, A 2 C 1, | ||||
| A 1 C 1, B 1 C 1 | 3 | 8 | B 1 C 1, B 2 C 1 | 6 - 3F | 32 | |||
| A 1 C 1, A 2 C 2 | 4 - 2F | 4 | ||||||
| A 1 C 1, B 1 C 2 | 4 - F | 8 | 6 | A 1 C 1, A 1 C 2, A 2 C 1, | ||||
| A 2 C 2, B 1 C 1, B 1 C 2 | 5 - 2F | 4 | ||||||
| 3 | A 1 C 1, A 1 C 2, A 2 C 1 | 4 - 2F | 8 | A 1 C 1, A 1 C 2, A 2 C 1, | ||||
| A 1 C 1, A 1 C 2, B 1 C 1 | 4 - F | 24 | A2C 2, B 1 C 1, B 2 C 1 | 6 - 3F | 24 | |||
| A 1 C 1, A 2 C 1, B 1 C 2 | 5 - 2F | 24 | ||||||
| 7 | A 1 C 1, A 1 C 2, A 2 C1, | |||||||
| 4 | A 1 C 1, A 1 C 2, A 2 C 2, B 1 C 1 | 5 - 2F | 50 | A 2 C 2, 1 | ||||
| A 1 C 1, A 1 C 2, B 1 C 1, B 1 C 2 | 4 - F | 4 | B 2 C 1 | 6 - 3F | 8 | |||
| A 1 C 1, A 2 C 1, B 1 C 1, B 2 C 2 | 6 - 3F | 14 | ||||||
| A 1 C 1, A 1 C 2, A 2 C 1, A 2 C 2 | 4 - 2F | 2 | 8 | A 1 C 1, A 1 C 2, A 2 C 1, | ||||
| A 2 C 2, B 1 C 1, B 1 C 2, | ||||||||
| B 2 C 1, B 2 C 2 | 6 - 3F | 1 |
† F: coeficiente de endogamia de las líneas progenitoras de los híbridos trilineales.
Cuadro 3 Número de formas de ocurrencia de g genotipos en muestras aleatorias de tamaño m tomadas con reemplazo de los ocho genotipos que produce la cruza trilineal (A 1 A 2 x B 1 B 2 ) x C 1 C 2 . Si m = g, además del número de formas, entre paréntesis está la suma de genes no idénticos por descendencia que aportan los 8!/[(8 - g)!g!] conjuntos posibles de g genotipos. No se incluye el caso m =11.
| m | g | |||||||
| 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | |
| 1 | 8 (2) | |||||||
| 2 | 8 | 2!(96-24F)† | ||||||
| 3 | 8 | 6 | 3!(248-88F) | |||||
| 4 | 8 | 14 | 36 | 4!(358-150F) | ||||
| 5 | 8 | 30 | 150 | 240 | 5!(312-144F) | |||
| 6 | 8 | 62 | 540 | 1560 | 1800 | 6!(164-80F) | ||
| 7 | 8 | 126 | 1800 | 8400 | 16 800 | 15 120 | 7!(48-24F) | |
| 8 | 8 | 254 | 5796 | 40 824 | 126 000 | 191 520 | 141 120 | 8!(6-3F) |
| 9 | 8 | 510 | 15 882 | 186 480 | 834 120 | 1 905 120 | 2 328 480 | 1 451 520 |
| 10 | 8 | 1022 | 55 980 | 818 520 | 5 103 000 | 16 435 440 | 20 635 200 | 30 240 000 |
| 12 | 8 | 4094 | 519 156 | 14 676 024 | 165 528 000 | 953 029 440 | 3 162 075 894 | 6 411 968 640 |
† F: coeficiente de endogamia de las líneas progenitoras de los híbridos trilineales.
De los 28 conjuntos diferentes de seis genotipos, cuatro contienen 5-2F genes NIPD cada uno y cada uno de los 24 restantes aporta 6-3F (Cuadro 2). Con las aportaciones de los 11 conjuntos de frecuencias genotípicas posibles (k = 1, 2, 3, ..., 11; Cuadro 1) se puede calcular la contribución total cuando m = 12 y g=6 a E(Y m ). Ésta es (Cuadros 1 a 3):
La obtención de este resultado involucró la consideración ya evidenciada de que las 28 combinaciones de seis de los ocho genotipos de la progenie de un HT (Ecuación 1) tienen la misma probabilidad de ocurrencia en la muestra. Además, con m≥8, los resultados en cuanto a genotipos obtenidos en la muestra aleatoria con reemplazo son todas las combinaciones posibles que pueden ser formadas con g de los ocho genotipos para cada uno de los valores posibles de g (1, 2, ..., 8).
La información de los Cuadros 2 y 3 hace posible el cálculo del numerador del cociente que permite calcular el promedio de genes NIPD que llegan a los m representantes de la cruza trilineal. El denominador (8m) es el número de resultados igualmente posibles y mutuamente excluyentes que puede producir el muestreo; para definir el numerador se requiere calcular el número de genes NIPD que aporta cada uno de los 8 m casos posibles (con 8≤m). Por ejemplo, de acuerdo con la información del Cuadro 3, para m=12, que permite la obtención de muestras que pueden contener 1, 2, 3, 4, ..., 7 u 8 genotipos:
Este resultado, E(Y 12) = 5.8728-2.8733F, es importante para los fitomejoradores y especialistas en recursos genéticos y es un indicador de estar próximo a lo que se debe esperar cuando m es “grande”: ningún gen NIPD debe perderse; es decir, su número en la muestra debe ser 6-3F. Del Cuadro 4 se desprenden varios hechos relevantes: para un valor fijo de F, cuando m aumenta, E(Y m ) crece, más rá pidamente cuando F es menor. Además, con líneas puras (F=1) basta una muestra de 10 individuos de una cruza trilineal para que se alcance el equilibrio en tres genes NIPD y, por lo tanto, cero genes perdidos. Con F=0.5, en cambio, esta condición empieza a ser ostensible con m=12. También se infiere que cuando F=0.75, a partir de m=15 no hay pérdida de genes NIPD.
Cuadro 4 Números de genes no idénticos por descendencia esperados [E(Y m )] y perdidos [(NIPD) pt ] en muestras de tamaño m con reemplazo tomadas del arreglo genotípico de una cruza trilineal hecha con líneas no emparentadas (Ecuación 1) para cinco coeficientes de endogamia (F). El caso m=11 no se incluye.
| m | F = 0.00 | F = 0.50 | F = 0.75 | F = 0.875 | F = 1.00 | ||||||
| E(Y m ) | (NIPD) pt | E(Y m ) | (NIPD) pt | E(Ym) | (NIPD) pt | E(Y m ) | (NIPD) pt | E(Y m ) | (NIPD) pt | ||
| 1 | 2.00 | 4.00 | 2.00 | 2.50 | 2.00 | 1.75 | 2.00 | 1.38 | 2.00 | 1.00 | |
| 2 | 3.25 | 2.75 | 2.88 | 2.38 | 2.69 | 1.06 | 2.59 | 0.78 | 2.50 | 0.50 | |
| 3 | 4.06 | 1.94 | 3.41 | 1.09 | 3.08 | 0.67 | 2.91 | 0.46 | 2.75 | 0.25 | |
| 4 | 4.61 | 1.39 | 3.74 | 0.76 | 3.31 | 0.44 | 3.09 | 0.28 | 2.88 | 0.13 | |
| 5 | 4.99 | 1.01 | 3.96 | 0.54 | 3.45 | 0.30 | 3.19 | 0.18 | 2.94 | 0.06 | |
| 6 | 5.26 | 0.74 | 4.11 | 0.39 | 3.54 | 0.21 | 3.25 | 0.12 | 2.97 | 0.03 | |
| 7 | 5.45 | 0.55 | 4.22 | 0.28 | 3.60 | 0.15 | 3.29 | 0.08 | 2.98 | 0.02 | |
| 8 | 5.59 | 0.41 | 4.29 | 0.21 | 3.64 | 0.11 | 3.32 | 0.06 | 2.99 | 0.01 | |
| 9 | 5.69 | 0.31 | 4.34 | 0.16 | 3.67 | 0.08 | 3.33 | 0.04 | 2.99 | 0.01 | |
| 10 | 5.77 | 0.23 | 4.39 | 0.11 | 3.69 | 0.06 | 3.34 | 0.03 | 3.00 | 0.00 | |
| 12 | 5.87 | 0.13 | 4.44 | 0.06 | 3.72 | 0.03 | 3.36 | 0.02 | 3.00 | 0.00 | |
Con una muestra cada vez más grande su número de genes NIPD tiende a aumentar porque cada individuo adicional que se incluya, es una nueva oportunidad de que tenga un genotipo que aporte genes que aún no eran parte de esa muestra. Esto puede repercutir en el coeficiente de endogamia; MárquezSánchez (2010) encontró que una VS formada con t HTs (Sin t ) construidos con líneas puras no emparentadas tiene coeficiente de endogamia (FSin t ) igual a (3m + 1)/(8tm). Según este resultado m se relaciona inversamente con FSin t y se reduce el número de genes NIPD perdidos, como se prediciría, cuando FSin t es más grande.
Para Busbice (1970), el coeficiente de endogamia de una VS se relaciona lineal e inversamente con su media genotípica. En este contexto, los coeficientes de endogamia de una variedad sintética formada con un número par de L líneas puras (Sin L ) y otra formada con L/2 cruzas simples (Sin CS ) hechas con las mis mas L líneas puras, no dependen de m, son iguales a 1/L, y las medias genotípicas de las dos variedades sintéticas deben ser iguales. Sin embargo, si L es un número par y múltiplo de tres, el coeficiente de endogamia de un Sin t hecho con L/3 cruzas trilineales sería mayor que 1/L según el resultado reportado por Márquez-Sánchez (2010). Además, en consistencia con la igualdad de sus coeficientes de endogamia, si F=1 el Sin CS y el Sin L nunca pierden genes NIPD cuando se forma la muestra de los m representantes de cada progenitor; también, los m representantes de cada progenitor tienen el mismo genotipo. El Sin t , en cambio, sí pierde genes NIPD (Cuadro 4). Sahagún Castellanos y Villanueva-Verduzco (2012) reportan pérdidas de genes NIPD en sintéticos formados únicamente con cruzas dobles o simples (F<1).
Conclusiones
Desde la formación de los m individuos de cada una de las cruzas trilineales progenitoras de una variedad sintética, puede ocurrir pérdida de genes no idénticos por descendencia (NIPD); ésta es mayor cuando m es más pequeña y sus líneas progenitoras tienen un coeficiente de endogamia (F) menor. En cambio, si F=1 basta una muestra de tamaño ocho (m=8) para que esta pérdida se reduzca a cero. Si F=0.5 y m=12 el número esperado de genes NIPD perdidos es 0.06. Además, el desbalance de las frecuencias génicas de los progenitores de cada cruza trilineal produce un coeficiente de endogamia mayor en el Sin t que en el Sin L y esto afecta negativamente la media genotípica del primero.











 texto em
texto em 


