Introducción
Un objetivo principal de la biología molecular es saber cómo está organizada la información genética y cómo se regula su expresión. Así, al extraer el ADN de cualquiera de los órganos de un organismo y en cualquier etapa de su desarrollo no cambia en su secuenciación; es decir, permanece estable e intacto en su secuencia original. Las zonas activas de esta macromolécula con capacidad codificante, llamadas exones, estimulan la síntesis de enzimas específicas en las distintas etapas del desarrollo de un organismo. La propiedad catalítica de estas enzimas revela su presencia mediante métodos histoquímicos y desde 1957 los genetistas han usado la electroforesis para identificar miles de proteínas contenidas en un extracto crudo. Biomoléculas que tengan la misma propiedad catalítica se denominan isoenzimas.
Con isoenzimas o marcadores bioquímicos es posible realizar estudios de variabilidad genética, donde la variabilidad tisular (VT) se refiere a que varios tejidos u órganos de un mismo individuo muestran diferentes formas moleculares o distintas cantidades relativas de las mismas isoenzimas. Además, la variabilidad ontogénica (VO) se refiere a que en el mismo órgano en determinados momentos de su desarrollo (etapas fenológicas) se muestran diferentes formas moleculares o distintas cantidades relativas de la misma isoenzima. Así, se puede inferir que la VT y la VO son consecuencia de una regulación diferencial. De manera que cada tejido de un organismo tiene diferentes genes expresándose y en un mismo tejido en distintos momentos de su ciclo fenológico se expresan varios grupos de genes. En un corrimiento electroforético cada banda que aparece sobre el medio de soporte corresponde a una proteína diferente cuya síntesis puede estar controlada por uno o varios genes. Debido a que el orden y naturaleza de los aminoácidos determina la estructura primaria de una proteína, esta secuencia depende sólo de la información contenida en el ADN, la cual codifica para un polipéptido en particular. Así, los fenotipos observados sobre el medio de soporte pueden interpretarse en términos de genotipos, o sea en términos de genes y sus alelos.
Algunas características de las isoenzimas importantes para los genetistas son: 1) Una proporción de estos genes son polimórficos, o sea existen en la forma de uno o más alelos; y 2) estos alelos son codominantes, es decir ambos alelos se expresan en los organismos heterocigóticos; este fenómeno permite relacionar los fenotipos observados con un genotipo dado. Al interpretar de los fenotipos isoenzimáticos en un medio de soporte debe considerarse su estructura cuaternaria, en función de si tiene una, dos, tres o cuatro subunidades polipeptídicas que permiten clasificarlos como monómeros, dímeros, trímeros o tetrámeros, respectivamente, cuyo patrón de bandeo para el homocigótico queda representado, en todos los casos, por un solo alelo.
Sin embargo, para los heterocigóticos los transcritos codifican para proteínas asociadas con dos alelos (monómero), tres alelos (dímero), cuatro alelos (trímero) y cinco alelos (tetrámero). Entonces, un estudio a través de isoenzimas puede detectar la presencia de diferentes formas moleculares de estas proteínas, en etapas específicas del desarrollo de un organismo, de lo cual se infiere que la presencia de una nueva o diferente isoenzima en un estado o etapa específica del desarrollo podría estar controlando un proceso por aquella(s) isoenzima(s). Por ello, para aumentar la eficiencia de la técnica, podrían identificarse las etapas de desarrollo de la planta durante los cuales la proteína se manifiesta (Kephart, 1990).
Para calcular y detectar productos génicos se usa electroforesis en geles de poliacrilamida o de almidón. El análisis isoenzimático por electroforesis es una técnica sólida, reproducible y poderosa con numerosas aplicaciones debido a que las isoenzimas presentan expresión codominante (Kephart, 1990), y porque no exhiben efectos ambientales (Marquard y Chan, 1995). Además de confiables, son relativamente más económicos que aquellos de tipo de ADN y permiten obtener información enzimática de diferentes tejidos (Vicente y Fulton, 2003). El análisis de las isoenzimas se ha realizado con éxito en varias especies vegetales: Nicotiana tabacum L. (Stafford y Galston, 1970), Zea mays L. (Scandalios, 1964; Efron, 1970; Rao et al., 1997), Hordeum vulgare L. (Pedersen y Simonsen, 1987), Cereus peruvianus (Machado et al., 1993), Cucurbita pepo L. (Carpin et al., 1999), Brassica oleraceae (Bellani et al., 2002), plantas pertenecientes a la familia Asteraceae (Ros-Barceló y AznarAsensio, 2002), Mammillaria gracillis Pfeiff. (Balen et al., 2003), Tigridia pavonia (Arzate-Fernández et al., 2005a) y Lilium spp. (Arzate-Fernández et al., 2005b).
Sprekelia formosissima (L.) Herbert, también conocida como lirio azteca, es una especie originaria de México que aún se considera silvestre, pertenece a la familia Amaryllidaceae y se caracteriza por ser bulbosa, anual y de ciclo corto. Crece en pastizales y pedregales de los estados de Durango, Jalisco, Guanajuato, Hidalgo, Estado de México, Distrito Federal, Puebla, Veracruz, Guerrero y Oaxaca (López-Ferrari y Espejo-Serna, 2002). El valor ornamental de esta especie se debe al color rojo escarlata de sus flores, por lo que en México es común cultivarla en jardines y como planta medicinal (Vázquez-García et al., 1998). Pese al potencial ornamental de esta especie, no se han realizado estudios empleando isoenzimas para S. formosissima. Por lo tanto, el objetivo de este estudio fue evaluar la expresión diferencial (la variación tisular y ontogénica) en esta planta silvestre mediante marcadores bioquímicos para determinar las diferentes formas enzimáticas que se expresan en la etapa vegetativa y reproductiva.
Materiales y métodos
En este estudio se usaron plantas de lirio azteca (S. formosissima (L.) Herbert cv. Terciopelo, obtenidas de clones de bulbos, cultivadas 78 d en un sustrato compuesto por hojarasca, arena y estiércol de bovino en proporción 1:1:1, en un invernadero rústico en la Facultad de Ciencias Agrícolas de la Universidad Autónoma del Estado de México (UAEMex).
El ciclo de cultivo de S. formosissima se dividió en dos etapas fenológicas para evaluar su expresión diferencial: etapa vegetativa y etapa reproductiva (floración). Durante la etapa vegetativa sólo se obtuvieron muestras de tejido de hoja (H). En la etapa reproductiva, que se presentó después de la brotación de las primeras hojas, con una duración de unos 40 d, se recolectaron muestras de tejido proveniente de tépalos (T), estigma-estilo (G), estambre (E) y hoja (H).
Una vez que emergieron las plántulas, todos los muestreos se realizaron cada 8 d y en cada uno de ellos se tomaron muestras de los tejidos disponibles. En la etapa reproductiva se realizaron tres muestreos: el primero sólo fue de H, el segundo de T, G y E, y el tercero de H, T, G y E. En la etapa vegetativa se realizaron siete muestreos, todos ellos de H (Cuadro 1). En cada muestreo se recolectó muestra de tejido, de al menos tres plantas, colocarlo dentro de una bolsa Ziploc, y depositarlo en una hielera para conservarlo a -20 °C en un congelador doméstico. Todas las muestras se procesaron en el Laboratorio de Biología Molecular Vegetal de la Facultad de Ciencias Agrícolas de la UAEMex.
Cuadro 1 Patrones de bandeo isoenzimáticos (PBI) observados en las etapas de desarrollo fenológico (reproductiva y vegetativa) en Sprekelia fromosissima (L) Herbert.
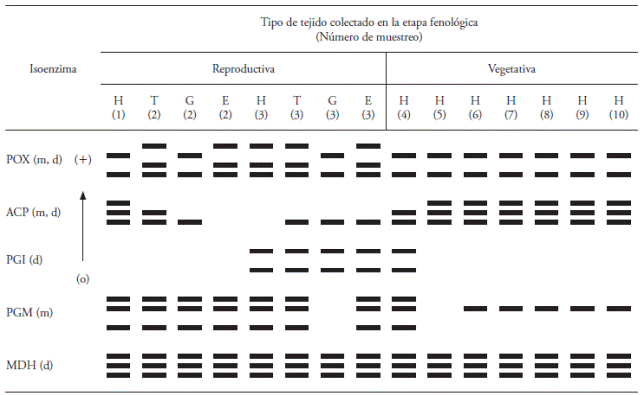
Del (1) al (10) representa el número de muestreo; “o”: Origen de la electroforesis; H: Hoja; T: Tépalo; G: Estigma-Estilo; E: Estambre; m: monómero; d: dímero.
Procedimiento electroforético
La extracción de proteínas totales se realizó usando aproximadamente 50 mg de tejido fresco, cada muestra se homogeneizó con una barreta de plástico en tubos Eppendorf de 2 mL, con 50 µL de solución amortiguadora extractora de TrisHCl (hidroximethil aminometano) 0.1M, EDTA-2Na (etilen diamino tetracético) 2 %, glicerol 50.4 %, tween-80 3.2 % y DTT (ditiotreitol) 0.8 %, pH 7.5 (Arzate-Fernández et al., 2005a). Una vez extraídas las proteínas, cada muestra se conservó a -20 °C hasta su análisis. Dos sistemas amortiguadores se usaron: histidina-ácido cítrico, pH 5.7 y pH 6.5 (H-AC) y Tris-citrato/Tris-histidina, pH 7.8 (T-C/T-H), según los procedimientos de Glaszmann et al. (1988) y Stuber et al. (1988). Los cinco sistemas isoenzimáticos evaluados fueron: peroxidasa (POX; EC 1.11.1.7), fosfatasa ácida (ACP; EC 13.1.3.2), fosfoglucosa isomerasa (PGI; EC 5.3.1.9), fosfoglucomutasa (PGM; EC 2.7.5.1) y malato deshidrogenasa (MDH; EC 1.1.1.37). Las muestras se cargaron usando el sobrenadante en un gel hidrolizado de almidón de papa al 12 %, realizando el corrimiento electroforético a 4 °C, y una duración media de 3 h, 40-50 mA y 100-150 v. Cada muestra se corrió un mínimo de tres veces para verificar su reproducibilidad. Los procedimientos de tinción histoquímicos se realizaron según las técnicas descritas por Torres et al. (1978) en citrus, Vallejos (1983), Stuber et al. (1988) usaron extractos de Zea mays L., Wendel y Weeden (1990), Glaszmann et al. (1988) e Ishikawa (1994) en arroz (Oriza sativa L.).
Para la expresión diferencial se consideró la presencia o ausencia de bandas en cada etapa fenológica (vegetativa y reproductiva) de las plantas muestreadas.
Resultados y discusión
En cinco sistemas electroforéticos se observaron bandas con suficiente resolución y nitidez para su registro y análisis (Cuadro 1). Las isoenzimas PGI, PGM y MDH mostraron mejor resolución con el sistema histidina-ácido cítrico a pH 5.7; mientras que la isoenzima ACP mostró una mejor resolución en el mismo amortiguador, pero a un pH de 6.5. En el caso de POX, hubo mejor resolución en el sistema tris-citrato/tris-histidina a un pH de 7.8.
Las cinco isoenzimas produjeron 15 bandas y nueve patrones de bandeo isoenzimáticos (PBI) (Figura1):dospara POX, trespara ACP, unopara PGI, dos para PGM y uno para MDH. El PBI obtenido con cada isoenzima en ambas etapa fenológicas se describe a continuación.

Figura 1 Representación esquemática de los patrones de bandeo isoenzimáticos (PBI) en Sprekelia formosissima utilizando marcadores bioquímicos: (A) POX en sistema Tris-citrato, pH 7.8; (B) ACP en sistema Histidina-Ácido-cítrico, pH 6.5; (C) PGI en sistema Histidina-Ácido-cítrico, pH 5.7; (D) PGM en sistema Histidina-Ácido-cítrico, pH 5.7; (E) MDH en sistema Histidina-Ácido-cítrico, pH 5.7. Números en pie de figura representan el número de patrón de bandeo isoenzimático (PBI), mientras “o” representa el origen de la electroforesis.
Peroxidasa (POX)
Las plantas tienen numerosas enzimas POX las cuales difieren en su localización dentro de la planta y entre especies. Las POX tienen una función importante en la biosíntesis de lignina (Conroy et al., 1982), generación de hormonas y desintoxicación por peróxido de hidrogeno. Además, se encuentran asociadas con procesos físicos y bioquímicos como crecimiento, formación celular, desarrollo de frutos, síntesis de etileno y también en mecanismos de respuesta a diferentes formas de estrés (Matamoros et al., 2003). La expresión de esta enzima está en un amplio intervalo de tipos tisulares vegetales durante la etapa reproductiva (Passardi et al., 2004). En nuestro estudio, en el sistema POX se observaron cuatro bandas activas con diferentes tasas de migración; las cuales fueron agrupadas en dos PBI, uno con dos bandas (1) y otro con tres bandas (2) (Figura 1A). En el patrón 1, la banda cercana al origen fue más intensa en comparación con la segunda. Ambos PBI se observaron en la etapa reproductiva mientras que en la etapa vegetativa sólo se observó el patrón 1. Durante la etapa reproductiva se recolectaron únicamente dos muestras de H; en la primera se observó el patrón 1 y en el extracto del tercer muestreo de H se observó el patrón 2, el cual también se expresa en tejidos de T y E de la misma etapa, mientras que en los dos extractos de G se observó el patrón 1, como se muestra en el Cuadro 1. Estos resultados coinciden con los reportados por McInnis et al. (2006) en Senecio squalidus, durante la etapa reproductiva de esta especie identificaron la presencia de isoformas específicas de peroxidasa las cuales probablemente participan en el complejo proceso de reconocimiento celular de la etapa de polinización.
Fosfatasa ácida (ACP)
En las plantas, las ACP están descritas en varias especies, tejidos y compartimentos celulares, y su función principal es la obtención y reciclaje de fosfato (Li et al., 2002; Bozzo et al., 2006). Además, las ACP exhiben actividad durante diferentes etapas de desarrollo como floración y germinación (Duff et al., 1994; Bozzo et al., 2002). En nuestro estudio, usando ACP se observaron tres bandas activas y tres PBI, el primero con tres bandas, el segundo con dos y el tercero con una sola banda (Figura 1B). La existencia de expresión diferencial se constató a través del sistema ACP, pues durante la etapa reproductiva se observaron los tres PBI. En esta etapa, el primer muestreo de tejido de H exhibió primer patrón y en el tercer muestreo no se observó ninguna banda (Cuadro 1). En la muestra de T se observó el patrón 2 y 3 (Cuadro 1), mientras que el patrón 3 se expresó en tejido de las muestras de G y en la tercera muestra de E. En el segundo muestreo de E no se observó ninguna banda.
Durante la etapa vegetativa, el sistema ACP, generó los PBI 1 y 2. El tejido de H de la cuarta muestra exhibió el patrón 2 y todas las muestras restantes de H mostraron el patrón 1. Estos resultados son similares a los reportados en una monocotiledónea por Pedersen y Simonsen (1987), quienes evaluaron tejidos de hojas de diferentes posiciones en la planta.
Fosfoglucosa isomerasa (PGI)
La PGI es una enzima que participa activamente en el metabolismo de azúcares en la mayoría de los organismos eucariotas; además, se localiza en plastidios y citosol de angiospermas. En nuestro estudio la PGI mostró dos bandas activas y un solo PBI (Figura 1C). Esto indica la existencia de expresión diferencial a través del sistema PGI, es decir, esta isoenzima no mostró especificidad tisular pero sí especificidad ontogénica en su expresión, pues en la etapa reproductiva se observó sólo en el tercer muestreo en tejidos de H, T, G y E, y sólo en tejido de H al inicio de la etapa vegetativa (cuarto muestreo) (Cuadro 1). Por lo tanto, es posible que esta isoenzima tenga una función preponderante durante el proceso de floración de S. formosissima.
Fosfoglucomutasa (PGM)
La PGM participa activamente en el metabolismo de azúcares, es decir cataliza la conversión de glucosa-1 y glucosa-6 fosfato en la síntesis y el consumo de sacarosa. Mühlbach y Schnarrenberger (1978) indican que PGM está presente en plantas y es posible obtener un patrón con dos bandas activas que con frecuencia se localizan en el citosol y cloroplastos. En nuestro estudio, con el sistema PGM se observó expresión diferencial porque se encontraron tres bandas activas y dos PBI, el primero con tres bandas y el segundo con una banda (Figura 1D). PGM mostró actividad enzimática con poca variabilidad en ambos etapas fenológicas de desarrollo. Durante la etapa reproductiva sólo se observó el patrón 1 en los tejidos muestreados, pero en los tejidos G, del tercer muestreo no se observaron bandas que evidenciaran actividad enzimática. En la etapa vegetativa el patrón 1 sólo se observó en tejidos del cuarto muestreo, mientras que en los tejidos de H se observó el patrón 2 del sexto al décimo muestreo, posiblemente por la mayor presencia de cloroplastos en tejido de H (Cuadro 1).
Malato deshidrogenasa (MDH)
La MDH es una enzima que, según su localización subcelular, participa activamente en el ciclo de Krebs, la fotorrespiración, y otras rutas catabólicas y anabólicas (Machado et al., 1993). En nuestro estudio, MDH presentó tres bandas activas con un solo PBI (Figura 1E), todas ellas presentes en ambas etapas fenológicas del desarrollo y en todos los tejidos muestreados (Cuadro 1). MDH fue el único sistema isoenzimático con el cual no se observó expresión diferencial en ambas etapas fenológicas.
En los organismos vivos el ADN es el compuesto más importante porque contiene la información hereditaria que determina la estructura de las proteínas, las cuales, para su expresión durante el proceso de desarrollo y diferenciación celular, involucran factores como hormonas, tipo de luz, longitud del día, temperatura, etc., que son determinantes en la regulación de la expresión génica o en su expresión diferencial (Grierson y Covey, 1991). Las isoenzimas pueden usarse como herramientas en el mejoramiento genético (Arzate-Fernández et al., 2005b) y el proceso de expresión diferencial se ha estudiado en maíz (Scandalios, 1964; Efron, 1970; Rao et al., 1997), tabaco (Stanford y Galston, 1970), cebada (Pedersen y Simonsen, 1987) y Brassicca oleraceae (Bellani et al., 2002), utilizando electroforesis en geles de almidón con muestras de diferentes partes de la planta como la hoja, el tallo, la raíz, las espigas jóvenes y maduras, la vaina, los granos de polen, los tejidos de escutelo, y los brotes obtenidos in vitro.
Conclusiones
Con la técnica de electroforesis horizontal sobre geles de almidón, se identificaron los patrones de bandeo isoenzimáticos diferenciales durante dos etapas fenológicas de Sprekelia formosissima. Esta técnica presenta ventajas respecto a los marcadores moleculares de ADN porque pueden obtenerse varios geles al mismo tiempo y éstos teñirse hasta con siete enzimas, reduciendo tiempos y costos.
Los PBI obtenidos en nuestro estudio con los marcadores bioquímicos peroxidasa, fosfatasa ácida, fosfoglucosa isomerasa y fosfoglucomutasa, indican que esta técnica es útil en investigaciones de expresión diferencial en la etapa vegetativa y reproductiva para S. formosissima, así como una herramienta de diagnóstico en un programa de mejoramiento genético o en un estudio de variabilidad o diversidad genética.











 texto em
texto em 


