Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Agrociencia
versión On-line ISSN 2521-9766versión impresa ISSN 1405-3195
Agrociencia vol.48 no.5 Texcoco jul./ago. 2014
Protección vegetal
Detección de Cymbidium mosaic virus (CymMV) y Odontoglossum ringspot virus (ORSV) en orquídeas en México
Detection of Cymbidium mosaic virus (CymMV) and Odontoglossum ringspot virus (ORSV) from orchids in Mexico
M. Siboney López-Hernández, P. Eloisa Palacios-Popo, Rodolfo De La Torre-Almaraz*
Laboratorio de Microbiología. UBIPRO. FES-Iztacala, Universidad Nacional Autónoma de México, Avenida de los Barrios Número 1. Los Reyes Iztacala. 54090. Tlalnepantla, Estado de México. * Autor responsable (drodolfo@unam.mx).
Recibido: julio, 2013.
Aprobado: julio, 2014.
Resumen
En recorridos realizados en 2011 para la detección de enfermedades en orquídeas en invernaderos comerciales en el Estado de Morelos, se observaron plantas dañadas con moteados cloróticos, rayados tenues de color amarillo y manchas cloróticas y necróticas anulares. Hojas dañadas fueron recolectadas de plantas de los géneros Brassia, Brassocattleya, Cattleya, Encyclia, Epidendrum, Guariathe, Laelia, Oncidium, Shomburghia, Vainilla y Xilobium. Pruebas de detección serológica de proteína viral de doble anticuerpo (DAS)-ELISA) fueron realizadas con antisueros específicos para diversos virus que afectan a orquídeas. Al menos una planta de cada género presentó los virus Cymbidium mosaic virus (CymMV; Potexvirus) y Odontoglossum ringspot virus (ORSV; Tobamovirus), en infecciones individuales o mezcladas. Las plantas de Encyclia y Laelia estuvieron infectadas con mayor frecuencia con ambos virus. El patrón electroforético se determinó en geles de poliacrilamida (PAGE) del genoma del CymMV, con extractos de ARN viral de doble cadena de origen viral de las muestras positivas en los ensayos serológicos de DAS-ELISA. La identidad del CymMV y ORSV se confirmó mediante secuenciación directa y por clonación de productos diferentes de la transcriptasa inversa ligada a la PCR (RT-PCR) punto final, con oligonucleótidos degenerados, que amplifican dos regiones consenso del gene de la replicasa (RdRp) del género Potexvirus, y oligonucleotidos específicos, que amplifican una región conservada del gen de la proteína de la cápside (CP) de CymMV y ORSV. Clones del gen RdRp de tres plantas de Laelia (números de acceso Genbank HQ393958, HQ393959, HQ393960, HQ393961 y HQ393962), del gen CP de CymMV de dos plantas de Oncydium (números de acceso Genbank HQ393956 y HQ393957) y del gen CP de ORSV de tres plantas de Encyclia (números de acceso Genbank HQ393953, HQ393954 y HQ393955), fueron obtenidos y secuenciados. Las secuencias nucleotídicas del gen de la proteína de la cápside (CP) del CymMV y del CP del ORSV, obtenidos de las muestras de orquídeas que se cultivan en México, mostraron similitud de 96 a 97% con CymMV y de 99 a 100% con ORSV con secuencias disponibles en el Genbank, lo cual confirmó que los aislados de ambos virus de México son idénticos a los de otras partes del mundo.
Palabras clave: Replicasa (RdRp), RT-PCR, ornamentales.
Abstract
In a field inspection of commercial greenhouse orchids in 2011 in the state of Morelos, plants were observed to exhibit damage in the form of chlorotic mottle, faint yellow stripes and ring-shaped chlorotic and necrotic spots. Damaged leaves were collected from plants of the genera Brassia, Brassocattleya, Cattleya, Encyclia, Epidendrum, Guariathe, Laelia, Oncidium, Shomburghia, Vainilla and Xilobium. Viral protein serological detection tests (double antibody sandwich enzyme-linked immunosorbent assays, DAS-ELISA) were done with specific antiserums for diverse viruses that affect orchids. At least one plant of each genus had Cymbidium mosaic virus (CymMV; Potexvirus) and Odontoglossum ringspot virus (ORSV; Tobamovirus) in individual or mixed infections. Encyclia and Laelia plants had the highest incidence of infection by both viruses. The electrophoretic pattern of the CymMV genome was determined in polyacrylamide gels (PAGE) with double chain viral RNA from the samples that were positive in the DAS-ELISA serological assays. Identity of CymMV and ORSV was confirmed by direct sequencing and by cloning products different from the reverse transcriptase linked to PCR (RT-PCR) final point with degenerated oligonucleotides, which amplify two consensus regions of the replicase gene (RdRp) of the genus Potexvirus, and specific oligonucleotides, which amplify a conserved region of the capsid protein gene (CP of CymMV and ORSV). Clones of the gene RdRp from three Laelia plants (GenBank access numbers HQ393958, HQ393959, HQ393960, HQ393961 and HQ393962), of the CP gene of CymMV from two Oncydium plants (GenBank access numbers HQ393956 and HQ393957), and from three Encyclia plants (GenBank access numbers HQ393953, HQ393954 and HQ393955), were obtained and sequenced. The nucleotide sequences of the capsid protein gene (CP) of CymMV and of the ORSV CP, obtained from samples of orchids cultivated in México, were 96 to 97 % similar to CymMV and 99 to 100 % to ORSV, with sequences available in the GenBank, confirming that both viruses isolated in México are identical to those found in other parts of the world.
Key words: Replicase (RdRp), RT-PCR, ornamentals.
INTRODUCCIÓN
En México se cultivan numerosas especies o híbridos de orquídeas (Orchidaceae, monocotiledóneas), nativas o introducidas, para comercializar sus flores (Ramírez, 1996; INEGI, 1998; Dole y Wilkins, 1999).
Durante enero y febrero de 2011 en invernaderos comerciales del estado de Morelos, México, se recolectaron plantas de géneros diversos de orquídeas, mexicanas e introducidas desde otros países, que mostraron en sus hojas moteados cloróticos (Figura 1A), manchas irregulares cloróticas (Figura 1B), manchas cloróticas con márgenes necróticos (Figura 1C) o manchas irregulares necróticas (Figura 1D). Las plantas con estos daños reducen su vida útil y la calidad de las flores.

Análisis fitopatológicos del material recolectado no mostraron la presencia de microorganismos con los que se pudiera relacionar los daños. Por lo tanto, se consideró la hipótesis de que los daños observados son causados por virus, los cuales son patógenos comunes en los centros grandes de producción de orquídeas comerciales en el mundo y causan pérdidas económicas altas (Rees, 1992; Albouy y Devergne, 2000; La Croix, 2008).
La información de la identidad de los virus que infectan a las orquídeas en cultivo comercial en México parece inexistente. Por lo tanto, el objetivo de este estudio fue identificar a los virus relacionados con los daños observados en diferentes géneros de orquídeas que se comercializan en México.
MATERIALES Y MÉTODOS
Recolecta de muestras
Tres hojas se recolectaron de una o varias plantas de orquídeas, que manifestaban daños de probable origen viral, de los siguientes géneros o sus híbridos (número de plantas indicado): Brassia (1), Brassocattleya (1), Cattleya (2), Encyclia (6), Epidendrum (2), Guariathe (1), Laelia (3), Oncidium (5), Shomburgkia (2), Vainilla (1) y Xylobium (1). Todas las plantas se cultivaban en invernaderos comerciales de Morelos. Las muestras se colocaron en bolsas plásticas y se transportaron al Laboratorio de Microbiología de la UBIPRO de la Facultad de Estudios Superiores, Iztacala, de la Universidad Nacional Autónoma de México, para la detección serológica e identificación molecular con ácidos nucleicos de origen viral (Dijkstra y De Jager, 1998).
Detección serológica de virus por ensayo inmunológico ligado a enzimas (DAS-ELISA)
Se emplearon anticuerpos comerciales (Agdia, Inc., Elkhart, IN) específicos, diluidos 1 en 200 μL y 1 g de tejido con daños macerado en 10 mL de solución amortiguadora de extracción, según las instrucciones del fabricante. Las reacciones se consideraron positivas por la presencia de color amarillo en los pocillos e intensidad del color de los testigos positivos, incluidos en todos los antisueros probados. Los anticuerpos utilizados fueron: Cucumber mosaic virus (CMV; Cucumovirus), Tobacco mosaic virus (TMV; Tobamovirus), Alfalfa mosaic virus (AMV; Alfamovirus), Tobacco etch virus (TEV; Potyvirus), Tomato bushy stunt (TBSV; Tombusvirus), Tobacco ringspot virus (TRSV; Nepovirus) y Tomato spotted wilt virus (TSWV; Tospovirus). Además se utilizaron los anticuerpos específicos comerciales de Cymbidium ringspot virus (CyRSV; Tombusvirus), Odontoglossum ringspot virus (ORSV; Tobamovirus) y Cymbidium mosaic virus (CymMV; Potexvirus) que comúnmente infectan a las orquídeas en el mundo (Brunt et al., 1996).
Extracción y electroforesis en geles de poliacrilamida (PAGE) de ARN de doble cadena de origen viral
La extracción y la electroforesis en geles de acrilamida del ARN de doble cadena de origen viral (ARN-dc) se realizó con 3.5 g de tejido, de acuerdo al protocolo propuesto por Valverde et al. (1990), que resultó positivo en las pruebas de serología.
Detección y caracterización molecular de virus por clonación y secuenciación de productos de transcripción inversa ligada a la reacción en cadena de la polimerasa (RT-PCR)
Las pruebas de RT-PCR se realizaron usando como templado de la RT-PCR el ARN de doble cadena de origen viral (ARN-dc) de las muestras de orquídeas que fueron positivas a virus en pruebas de serología. Se emplearon dos pares de oligonucleótidos que amplifican dos segmentos consenso del gen de la replicasa (RdRp) específicos para el grupo Potexvirus, que incluye al CymMV, el más frecuente en las orquídeas infectadas (Conti, et al., 2001; Ajjikuttira, et al., 2002; Grisoni, et al., 2004). Los oligonucleotidos POTEX 1F/R5 y POTEX 2F/R5 amplifican un fragmento de 735 pb y de 584 pb (Ryu y Park, 1995; Van der Vlugt y Berendesen, 2002).
Para conocer la especie de los virus detectados por serología se utilizaron oligonucleótidos específicos que amplifican un fragmento del gen de la proteína de la cápside (CP) del CymMV (CymMV CPF/CPR) que amplifica un fragmento de 669 pb (Lim et al., 1993). También se usaron oligonucleótidos específicos para amplificar el gen de la CP de ORSV (ORSV CPF/CPR) que rinde un amplicón de 474 pb (Ryu y Park, 1995; Ajjikuttira et al., 2002). Las reacciones de RT-PCR se llevaron a cabo en tres pasos, según las indicaciones del fabricante (Promega Corp.). Los productos de la RT-PCR se fraccionaron por electroforesis en geles de agarosa al 1 %, a 100 voltios por 30 min, y para calcular el peso de los productos obtenidos se incluyó un marcador de peso molecular de 1 kb (Invitrogen, Carlsbad, CA) (Dijkstra y De Jager, 1998).
Clonación y secuenciación
Los productos de la RT-PCR de interés fueron separados de los geles de agarosa y purificados utilizando el kit WIZARD (Promega Corp.). Los productos obtenidos de la RT-PCR de la replicasa y de la CP de los virus CymMV y ORSV se ligaron en el vector pDrive y se transformaron bacterias competentes (Escherichia coli, cepa DH5-alfa). Las bacterias se cultivaron en medio LB-agar + ampicilina, adicionado con IPTG (isopropil-β-D-1-tiogalactopiranósido) + X-Gal (50 mg mL-1) (Sambrook et al., 1989). La presencia del inserto se verificó mediante un ensayo con la enzima de restricción (EcoRI) y electroforesis en gel de agarosa. La secuencia de nucleótidos del inserto se determinó con los mismos oligonucleotidos de la RT-PCR, en un secuenciador (Applied Biosystems, Foster City, CA, modelo ABI 3100) de 16 capilares, con el método BIG DYE Terminator fluorescence based sequencing para análisis de secuencia. Las secuencias obtenidas se compararon con las disponibles en el GenBank (NCBI).
RESULTADOS Y DISCUSIÓN
Detección serológica de virus por ensayo inmunológico ligado a enzimas (DAS-ELISA)
Infecciones simples con CymMV se detectaron únicamente en tres muestras, con ORSV en cinco y con ambos virus en 15; esto confirma la incidencia alta de infección por virus en las muestras recolectadas. En todas las muestras, con excepción de una (Encyclia sp 3), se detectó la infección por uno o ambos virus (Cuadro 1), lo cual confirmó que los daños tienen origen viral.

Con los antisueros utilizados no se detectaron otros virus, pero es posible que estén presentes.
Extracción y electroforesis en geles de poliacrilamida (PAGE) de ARN de doble cadena de origen viral
El patrón electroforético del CymMV consistió en una banda de aproximadamente 13.0 x 106 Da que corresponde al componente genómico viral y dos fragmentos subgenomicos, típico de Potexvirus (Figura 2). Sin embargo, no fue posible obtener regularmente el patrón ARN-dc del ORSV, un Tobamovirus, que consta de una banda principal de 4.3 x 106 Da (Valverde et al., 1986). Los geles de poliacrilamida con todas las muestras no presentaron las bandas adicionales que indicaran la presencia de otros virus, excepto del CymMV en algunas muestras.

El análisis de ARN-dc por electroforesis en PAGE facilita la identificación, con cierta seguridad, de numerosos virus que infectan plantas y también permite detectar virus desconocidos en infecciones latentes. Sin embargo, la limitante principal del análisis de ARNdc viral es que requiere concentraciones altas del virus en su fase replicativa, para obtener, visualizar e identificar las bandas correspondientes al genoma del virus, por lo que podrían pasar desapercibidas las infecciones por los virus, por lo que se requieren otras técnicas de diagnóstico virales complementarias.
Es posible que la concentración de dsARN del ORSV fuera demasiado baja para detectar su patrón electroforético típico en PAGE al 6 %.
Detección y caracterización molecular de virus por clonación y secuenciación de productos de transcripción inversa ligada a la reacción en cadena de la polimerasa (RT-PCR)
Ampliaciones del tamaño esperado se obtuvieron en las pruebas de RT-PCR, con los oligonucleótidos POTEXF1/R5 (735 pb) en 15 muestras (Figura 3), con los oligonucleótidos POTEX F2/R5 (584 pb) en 18 muestras (Figura 4) y 19 de las 25 muestras analizadas fueron positivas con ambos oligonucleotidos.

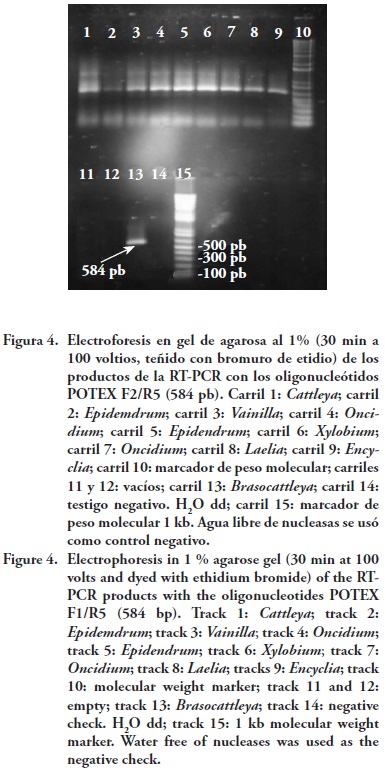
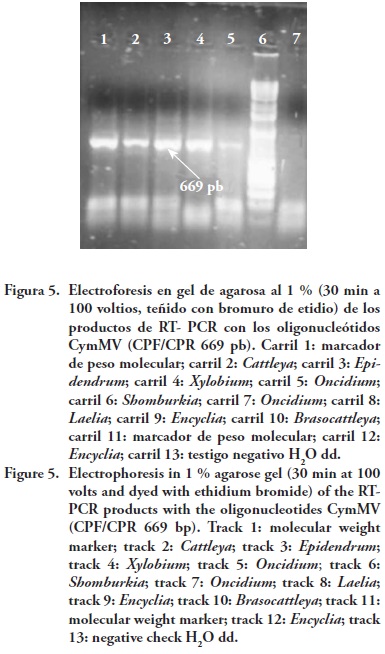
Con los oligonucleótidos específicos para la CP CymMV se detectaron nueve muestras infectadas con el virus (CPF/CPR. 669 pb) (Figura 5) y con los oligonucleótidos específicos para la CP de ORSV (CPF/CPR. 474 pb) nueve infectadas (Figura 6).

Los resultados de la detección por RT-PCR mostraron eficacia mayor para obtener amplicones positivos con los oligonucleótidos que amplifican parte del gen de la replicasa de Potexvirus y que con los oligonucleotidos específicos para la CP de CymMV y de ORSV.
La técnica ELISA fue más eficiente que las otras técnicas para detectar ambos virus, en infecciones simples y en mezcla; la detección fue similar con la RT-PCR específica para la replicasa de Potexvirus, como CymMV. La eficiencia menor se obtuvo con la RT-PCR específica para la CP de ambos virus, aunque no se identificaron las causas de estos resultados. Sin embargo, los amplicones obtenidos permitieron precisar la identidad de ambos virus por clonación y secuenciación de ácidos nucleicos (Cuadro 1).
Además de la eficiencia de la técnica, parece conveniente evaluar el coste de cada técnica para evaluar su utilidad en el diagnóstico eficiente de ambos virus.
Clonación y secuenciación
Con los insertos de las pruebas de RT-PCR para un fragmento del gen de la replicasa del género de Potexvirus y un fragmento de los genes de la CP de CymMV y de ORSV se seleccionaron y secuenciaron 10 clonas. Los productos de la RT-PCR de ambos virus para clonar fueron obtenidos de las plantas infectadas de Encyclia sp., Laelia sp. y Oncidium sp.
Los números de acceso del GenBank se obtuvieron por cada clona seleccionada y se confirmó la identidad de cada aislado por análisis y comparación de secuencias similares disponibles en el GenBank (www.ncbi.nlm.nih.gov, consultada en enero de 2013).
La comparación de las secuencias obtenidas para el gen de la replicasa de las clonas de CymMV, Cymclon 1 (Número de acceso Genbank HQ393958), Cymclon 2 (Número de acceso Genbank HQ393959), Cymclon 3 (Número de acceso Genbank HQ393960), Cymclon 4 (Número de acceso Genbank HQ393961) y Cymclon 5 (Número de acceso Genbank HQ393962), mostraron entre 96 y 100 % de similitud con el gen de la replicasa de las secuencias con número de acceso AY571289 y AF016914.1, disponibles en el Genbank, correspondientes con aislados de CymMV de Taiwan y Corea.
La comparación de las secuencias de las clonas de la CP de CymMV MorCym 1 (Número de acceso Genbank HQ393956) y MorCym 2 (Número de acceso Genbank HQ393957) mostraron similitud del 96 al 98 % con el gen de la CP de las secuencias AF405722.1 y AB541542.1 disponibles en el Genbank, procedentes de Corea.
La comparación de las secuencias de las clonas de la CP de ORSV MorOdon 1 (Número de acceso Genbank HQ393953), MorOdon 2 (Número de acceso Genbank HQ393954) y MorOdon 3 (Número de acceso Genbank HQ393955), mostraron similitud de 98 a 100 % con la secuencia JN584484.1 disponible en el Genbank, del gen de la CP de un aislado de ORSV procedente de Taiwan (Soto et al., 2012).
El análisis y comparación de las secuencias obtenidas con las disponibles en el GenBank confirmaron la identidad de los virus CymMV y ORSV, detectados en las plantas de distintos géneros de orquídeas con síntomas de mosaico, moteados, manchas anulares y necrosis, que se cultivan en invernaderos del estado de Morelos.
La prueba serológica de DAS-ELISA permitió detectar rápidamente y con cierta eficiencia la presencia de CymMV y ORSV en la mayoría del material recolectado, por lo que considerando los síntomas observados en orquídeas, estos están relacionados con infecciones individuales y en mezcla de ambos virus. La detección de Potexvirus por RT-PCR, en este caso CymMV, fue similar a la serológica, al contrario de la RT-PCR con oligonucleotidos para la CP de CymMV y ORSV. Por el momento se desconoce la causa de la diferencia de eficiencia de estas pruebas.
La clonación y secuenciación de los productos de la RT-PCR, con dos juegos de oligonucleotidos degenerados para amplificar dos fragmentos del gen de la replicasa del genero Potexvirus, y los específicos para amplificar los fragmentos de los genes de la CP de CymMV y ORSV, respectivamente, confirmaron la identidad de ambos virus en el material recolectado. La comparación de las secuencias de los fragmentos clonados, confirmó que los aislados de CymMV y ORSV son 96 a 100 % similares a los aislamientos comunes de orquídeas cultivadas en el mundo, particularmente los de Corea y Taiwan. Esto confirma que los aislados de CymMV y ORSV de México, probablemente proceden de todas las regiones donde se cultivan orquídeas. El material usado en el presente estudio, como Encyclia, Laelia y Oncidium, está distribuido ampliamente en México, como variedades o híbridos introducidos de otras regiones del mundo.
CymMV pertenece al género Potexvirus, del orden Tymovirales y familia Alphaflexiviridae. Los virus que pertenecen a los Potexvirus consisten de partículas en forma de varilla flexible, que miden de 470 a 580 nm de largo y 11 a 13 nm de diámetro. El CymMV es el virus que infecta con más frecuencia las especies de orquídeas en el mundo y causa pérdidas elevadas (Francki, 1991).
El genoma de CymMV es de cadena sencilla lineal (6227 nt) de ARN, de sentido positivo, con cinco marcos de lectura abierta (ORF). El ORF1 codifica la ARN replicasa dependiente de ARN (RdRp), los tres siguientes ORF's (triple block) forman parte de la proteína del movimiento (MP), encargada del transporte viral entre las células, y el ORF5 codifica para la proteína de la cápside (CP). CymMV expresa su genoma con la producción de dos ARNm subgenómicos. Este virus se transmite principalmente en forma mecánica o por contacto entre plantas y no se conoce algún biovector (Brunt et al., 1996).
ORSV pertenece al genero Tobamovirus con partículas en forma de varilla rígida con longitud de 300 nm. Este virus es frecuente en numerosas especies de orquídeas y se asocia con lesiones necróticas severas en el material que infecta. El genoma del ORSV consiste de una cadena de ARN, de una sola banda de sentido positivo, con cuatro marcos de lectura abierta (ORF's). Los ORF1 y ORF2 codifican y regulan la traducción de la replicasa (RdRP), el ORF3 codifica para la MP y el ORF4 codifica para CP. Los dos últimos genes se expresan mediante dos ARNm subgenómicos. Este virus se transmite mecánicamente y por contacto entre plantas, y no se conocen sus biovectores (Brunt et al., 1996).
Más de 30 virus que infectan a múltiples géneros de orquídeas se conocen en el mundo, pero CymMV y ORSV son los más frecuentes, principalmente en plantas cultivadas, lo cual está relacionado con la facilidad de dispersión y su capacidad infectiva alta (Jensen, 1970).
En el presente estudio la detección e identificación de CymMV y ORSV fue frecuente con los métodos y técnicas ya descritas. Pero se podrían detectar otros virus con otros métodos de diagnóstico o incrementando las especies de orquídeas (Brunt et al., 1996; You et al., 2005).
CONCLUSIONES
La identidad de CymMV y ORSV se confirmó mediante la clonación y secuenciación de productos de la RT-PCR obtenidos de distintos géneros de orquídeas infectadas por estos virus. Los análisis de comparación de las secuencias nucleotídicas obtenidas con secuencias similares y disponibles en el GenBank mostraron que los aislados de CymMV y ORSV son similares a los encontrados en otras regiones del mundo.
AGRADECIMIENTOS
La presente investigación fue apoyada económicamente por el PROGRAMA DE APOYO A PROYECTOS DE INVESTIGACIÓN E INNOVACIÓN TECNOLÓGICA (PAPIIT- IN 218411) de la UNAM.
LITERATURA CITADA
Ajjikuttira, P. A., C. L. Lim-Ho, M. H. Woon, K. H. Ryu, C. A. Chang, C. S. Loh, and S. M. Wong. 2002. Genetic variability in the coat protein genes of two orchid viruses: Cymbidium mosaic virus and Odontoglossum ringspot virus. Arch. Virol. 147: 1943-1954. [ Links ]
Albouy, J., and J. C. Devergne. 2000. Enfermedades Producidas por Virus de las Plantas Ornamentales. Ediciones Mundi-Prensa. pp: 204-208. [ Links ]
Conti, M. D. Gatelli, V. Lisa, O. Lovisolo, G. Martelli P., A. Ragozzino, G. Rana L., y C. Volvas. 2001. Principales Virus de las Plantas Hortícolas. Ediciones Mundi-Prensa. pp: 11-56. [ Links ]
Brunt, A. A., K. Crabtree, M. J. Dallwitz, A. J. Gibbs, L. Watson, and E. J. Zurcher (eds). 1996. Plant Viruses Online: Description and Lists from the VIDE Database. Versión: 16th January 1997. URL. http://biology.anu.edu.au/Groups/MES/vide. (Consulta: enero-febrero 2013). [ Links ]
Clark, M. F., and A. M. Adams. 1977. Characteristics of microplate method of enzyme-linked immunosorbent assay for the detection of plant viruses. J. Gen. Virol. 34: 475-483. [ Links ]
Dijkstra, J., and P. C. De Jager. 1998. Practical Plant Virology. Protocols and Exercises. Edit. Springer, Berlin. 459 p. [ Links ]
Dole, J. y H. Wilkins.1999. Principios de Floricultura y Especies. Edit. Printice-Hall. Inc. pp: 438-445. [ Links ]
Francki, R. I. B. 1991. Classification and nomenclature of viruses. Arch. Virol. Suppl. 2: 392-401. [ Links ]
Grisoni, M., F. Davidson, C. Hyrondelle, K. Farreyrol, M. L. Caruana, and M. Pearson. 2004. Nature, incidence, and symptomatology of viruses infecting Vanilla tahitensis in French Polynesia. Plant Dis. 88: 119-124. [ Links ]
INEGI. 1998. La Horticultura Ornamental en México. INEGI. México. pp: 1-81. [ Links ]
Jensen, D. D. 1970. Virus diseases of orchids in the Netherlands. Neth. J. Pl. Path. 76: 135-139. [ Links ]
La Croix, I. F. 2008. La Nueva Enciclopedia de las Orquídeas. Editorial Timber Press. pp: 22. [ Links ]
Lim, S. T., S. M. Wong, C. Y. Yeong, S. C. Lee, and C. J. Goh. 1993. Rapid detection of Cymbidium mosaic virus by the polymerase chain reaction (PCR). J. Virol. Meth. 41: 37-46. [ Links ]
NCBI, 2002. http://www.ncbi.nlm.nih.gov/BLAST/
Rees, A. R. 1992. Ornamental Bulbs, Corms and Tuber. CAAB International. England. pp: 75-78. [ Links ]
Ramírez, J. 1996. Orquídeas de México. CONABIO. Biodiversitas 5:1-5. [ Links ]
Ryu, K. H., and W. M. Park. 1995. Rapid detection and identification of Odontoglossum ringspot virus by polymerase chain reaction amplification. FEMS Microbiol Lett. 133: 265-269. [ Links ]
Sambrook, J., E. F. Fritsch, and T. Maniatis. 1989. Molecular Cloning a Laboratory Manual. Cold Spring Harbor Laboratory Press, New York. [ Links ]
Soto, V. A. G., R. De La Torre A., B. Xoconostle C., R. Ruíz M. 2012. First report of Cymbidium mosaic virus and Odontoglossum ringspot virus in orchids in Mexico. Plant Dis. 96: 464. [ Links ]
Valverde, R. A., J. A. Dodds, and J. A. Heick. 1986. Doublestranded ribonucleic acid from plants infected with viruses having elongated particles and undivided genomes. Phytopathology 76: 459-465. [ Links ]
Valverde, R. A., Nameth, S. T. and R. L. Jordan. 1990. Analysis of double-stranded RNA for plant virus diagnosis. Plant Dis. 74: 255-258. [ Links ]
Van der Vlugt, R. A. A. and M. Berendesen. 2002. Development of a general Potexvirus detection method. Eur. J. Plant Pa-thol. 108: 367-371. [ Links ]
Wong, S. M., P. H. Mahtani, K. C. Lee, H. H. Yu, Y. Tan, K. K. Neo, Y. Chan, M. Wu, and C.G. Chang. 1997. Cymbidium mosaic potexvirus RNA: complete nucleotide sequence and phylogenetic analysis. Arch. Virol. 142: 383-391. [ Links ]
You, X. Z., C. C. Ching, K. Ch. Yuh, and J. J. Fuh. 2005. Identification and characterization of a potyvirus causing chlorotic spots on Phalaenopsis orchids. Eur. J. Plant Pathol. 121: 87-95. [ Links ]














