Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Agrociencia
versión On-line ISSN 2521-9766versión impresa ISSN 1405-3195
Agrociencia vol.47 no.8 Texcoco nov./dic. 2013
Ciencia animal
Conectividad en evaluaciones genéticas de animales: 2. Comparación de metodologías
Connectedness in animal genetic evaluations: 2. Comparison of methodologies
Fabián Magaña-Valencia1, Rafael Núñez-Domínguez1*, Rodolfo Ramírez-Valverde1, Felipe A. Rodríguez-Almeida2
1 Departamento de Zootecnia, Posgrado en Producción Animal, Universidad Autónoma Chapingo. Carretera México-Texcoco Km 38.5. 56230. Chapingo, Estado de México. *Autor responsable. (rafael.nunez@correo.chapingo.mx).
2 Facultad de Zootecnia y Ecología, Universidad Autónoma de Chihuahua. Periférico Francisco R. Almada Km 1. 31031. Chihuahua, Chihuahua.
Recibido: agosto, 2013.
Aprobado: noviembre, 2013.
Resumen
El propósito de este estudio fue comparar los principales métodos pata estimar la conectividad entre grupos contemporáneos (GC) en una población simulada, considerando una misma estructura de datos. Se simuló una población de 37 bovinos, de los cuales 15 tuvieron registros de peso al destete en uno de tres GC, en siete escenarios con diferente grado y tipo de conectividad (directa e indirecta). En cada escenario se realizaron contrastes entre los GC y se determinaron sus conectividades con 10 métodos cuantitativos. Los criterios para comparar los métodos fueron: capacidad para identificar GC desconectados, y conectados directa e indirectamente; y consistencia, definida como la relación entre el grado de conectividad obtenida y la simulada al aumentar progresivamente el número de reproductores en común entre GC, a través de los escenarios. Además, se obtuvieron coeficientes de correlación entre los grados de conectividad. Los métodos varianza del error de predicción de las diferencias en valores genéticos entre animales, flujo de genes, número de lazos genéticos directos totales debido a sementales y hembras comunes, e índice de conectividad mostraron tendencias en los valores de conectividad consistentes con el incremento en el número de sementales o vientres en común a través de los siete escenarios simulados. De estos métodos, sólo el de número de lazos genéticos directos detectó grupos contemporáneos desconectados, además de estar altamente correlacionado (—0.97) con la varianza del error de predicción de las diferencias en valores genéticos entre animales. Por tanto, se sugiere usar el método de número de lazos genéticos directos en evaluaciones genéticas rutinarias de bovinos.
Palabras clave: métodos de conectividad genética, evaluación genética, modelo animal, simulación.
Abstract
The purpose of this study was to compare the main methods to estimate connectedness among contemporary groups (CG) in a simulated population, considering a single structure of data. A population of 37 cattle was simulated, of which 15 had records of weaning weight in one of three CG, in seven scenarios with different degrees and types of connectedness (direct and indirect). In each scenario contrasts were performed among the CG and their connectedness was determined with 10 quantitative methods. The criteria for comparing the methods were: capacity to identify CG disconnected, and connected directly or indirectly; and consistency, defined as the relationship between the degree of obtained vs. simulated connectedness by progressively increasing the number of sires and dams in common among CG, through the scenarios. In addition, correlation coefficients were obtained between the degrees of connectedness. The prediction error variance methods of the differences in genetic values among animals, gene flow, total number of direct genetic links due to common sires and dams, and connectedness index showed trends in connectedness values consistent with the increase in the number of sires or dams in common through the seven simulated scenarios. Of these methods, only that of number of direct genetic links detected disconnected contemporary groups, besides being highly correlated (-0.97) with the prediction error variance of the differences in genetic values among animals. Therefore, it is suggested to use the method of total number of direct genetic links in routine genetic evaluations of cattle.
Key words: genetic connectedness methods, genetic evaluation, animal model, simulation.
INTRODUCCIÓN
Cuando los valores genéticos de los animales en la población base están distribuidos al azar e idénticamente en toda la población, los grupos contemporáneos (GC) desconectados no producen predicciones sesgadas de valores genéticos (Fernando et al., 1983). Sin embargo, esto no ocurre cuando ha habido selección o deriva genética antes de iniciar el control genealógico y de comportamiento de los animales, lo cual es común en poblaciones ganaderas y causa diferencias genéticas entre GC (Kuehn et al., 2007). El modelo animal permite que todas las relaciones genéticas entre los animales contribuyan a la conectividad de los datos (Kennedy y Trus, 1993), aunque la calidad de las evaluaciones genéticas depende de numerosos factores, en particular de la estructura de los datos (Tosh and Wilton, 1994; Hanocq et al., 1999; Carneiro et al., 2001) y la confiabilidad de la comparación entre animales de diferentes GC (Laloë et al., 1996; Mathur et al., 2002; Roso et al., 2004). Si estos GC no están conectados de manera apropiada, existe el riesgo de cuantificar diferencias ambientales entre GC como diferencias genéticas (Clément et al., 2001; Kuehn et al., 2007; Tarrés et al., 2010). Lo anterior es muy importante cuando se consideran varias unidades de producción, como en las evaluaciones genéticas regionales, nacionales o internacionales, donde un paso preliminar rutinario es la determinación de conectividad entre los GC.
En México, durante la década más reciente se iniciaron programas de control de producción y de evaluaciones genéticas nacionales, principalmente en bovinos. Estas evaluaciones suponen la existencia de conectividad entre los GC, por lo que en esta etapa de los programas es pertinente verificar el grado de conectividad y el método más apropiado para evaluarla. Magaña-Valencia et al. (2012) presentaron un ensayo sobre los principales métodos disponibles para determinar los GC conectados genéticamente. Aunque se han comparado algunos métodos para determinar conectividad genética entre GC, no hay consenso en el método más eficiente y práctico (Kennedy y Trus, 1993; Kuehn et al., 2007; Soga et al., 2010). Por tanto, se debe compararlos considerando escenarios que varíen en el grado y tipo de conectividad, y con una misma estructura de datos, con el fin de identificar los más sensibles para detectar las diferencias entre escenarios.
El objetivo de este estudio fue comparar los principales métodos para estimar la conectividad genética entre GC en una población simulada, considerando una misma estructura de datos.
MATERIALES Y MÉTODOS
Una población fue simulada para peso al destete en bovinos para carne h2= 0.28, σ2f = 642 kg2, σ2e / σ2a = λ = 2.22; donde, σ2f es varianza fenotípica, σ2e es varianza residual, α2a es varianza genética aditiva; promedio de parámetros estimados en evaluaciones genéticas nacionales de bovinos para carne en la Universidad Autónoma Chapingo. La población tenía 37 bovinos, de los cuales 13 fueron animales base (progenitores desconocidos) y 15 fueron crías con registro de comportamiento en uno de tres GC. Para contrastar diferentes grados y tipos de conectividad (directa e indirecta) entre los GC, siete escenarios se simularon aumentando a priori el número de sementales y vacas en común (Cuadro 1). Los pedigríes de los escenarios se muestran en el Apéndice 1.
En el escenario 1 (E1), los GC estuvieron desconectados; en el E2, GC1 y GC2 se conectaron vía descendientes del semental 2 (Apéndice 1); en E3 y E4, además de las conexiones anteriores, GC2 y GC3 se conectaron vía descendientes de uno y dos sementales (GC1 y GC2, y GC2 y GC3 estuvieron conectados directamente, y GC1 y GC3 indirectamente). En E5 a E7 los GC tuvieron dos, tres y cuatro sementales en común, y para ciertos contrastes, tuvieron una, dos y tres vacas en común.
Para comparar los grados de conectividad entre los GC, en los siete escenarios propuestos se usaron 10 métodos cuantitativos: promedio de las relaciones genéticas aditivas (PRGA; Banos y Cady, 1988), índice de conectividad (IC; Foulley et al., 1992), coeficiente de determinación (CD; Laloë, 1993), varianza del error de predicción (VEP) de las diferencias en valores genéticos entre animales (Kennedy y Trus, 1993), flujo de genes (FG; Kennedy y Trus, 1993), varianza de la deriva genética (VDG; Kennedy y Trus, 1993), varianza de las diferencias entre efectos de GC (VdGC; Kennedy y Trus, 1993), grado de conectividad (GrC; Mathur et al., 2002), correlación de las varianzas de los errores de predicción entre los valores genéticos (rij; Lewis et al., 1999), y número de lazos genéticos directos totales debido a sementales y hembras comunes (TLGD; Fries y Roso, 1997). Una descripción de los fundamentos y características de estos métodos fue publicada por Magaña-Valencia et al. (2012).
El modelo estadístico usado fue: y = Xb + Za + e, donde y es el vector de observaciones, b es el vector de efectos fijos de GC, a es el vector de efectos aleatorios de valores genéticos aditivos de los animales, e es el vector de efectos aleatorios residuales, y X y Z son matrices que asocian y con b y a, respectivamente. La varianza de e es Iσ2, y la varianza de a es Aσ2a, donde I es la matriz idéntica y A es la matriz de relaciones genéticas aditivas entre los animales. Estas matrices se derivan de la información en el Apéndice 1 y las operaciones matriciales se realizaron con Microsoft Excel®.
Los criterios para comparar los métodos aplicados a los escenarios simulados fueron: capacidad para identificar GC desconectados, y conectados directa e indirectamente; y consistencia, definida como la relación entre el grado de la conectividad obtenido y la simulada al aumentar el número de reproductores en común entre GC desde E1 hasta E7. Además, con el propósito de tener una aproximación de la relación entre los métodos, se estimaron coeficientes de correlación entre los grados de conectividad obtenidos por cada par de métodos.
RESULTADOS Y DISCUSIÓN
En la Figura 1 se muestra el patrón de los grados de conectividad para los tres contrastes entre GC, a través de los siete escenarios, obtenidos mediante el método VEP, donde los valores pequeños indican mayor conectividad. Según Kennedy y Trus (1993) y Roso et al. (2004), el promedio de la VEP entre animales de diferentes GC es la medida teórica más apropiada para estimar la conectividad entre GC. En el presente estudio, el método mostró cambios en los grados de conectividad que fueron consistentes con los simulados. Sin embargo, tuvo el inconveniente de no permitir identificar GC desconectados, pues por ejemplo para E1, donde los tres GC están desconectados, el método generó valores no indicativos de ausencia de conectividad. Roso et al. (2004) al evaluar la conectividad genética entre GC con los métodos VEP, GrC, TLGD y VdGC, encontraron que el método VEP fue el más adecuado; pero el cómputo de este indicador fue lento y difícil de usar rutinariamente en evaluaciones genéticas nacionales.
Una tendencia similar a la de VEP fue observada en los métodos FG y VdGC, con relación a la consistencia entre los cambios en los grados de conectividad de acuerdo con los escenarios simulados; sin embargo, también tuvieron problemas para identificar apropiadamente GC desconectados (Cuadro 2). El método FG no permitió detectar GC conectados indirectamente, pues el grado de conectividad fue igual a 10.0, en E3 y E4, para el contraste GC1 vs. GC3. Además, en E3 el método de FG indicó que los GC2 y GC3 fueron los mejor conectados, contrario a lo observado para la mayoría de los métodos aplicados. Aunque FG no requiere invertir matrices, en la práctica puede ser difícil identificar animales fundadores (matriz Q) y obtener la matriz T (matriz triangular inferior que traza el flujo de genes de una generación a la siguiente), sobre todo para bases de datos grandes, por lo que este método no es recomendable para datos de campo. El método VdGC fue inconsistente entre contrastes de GC, ya que con excepción de E2, la magnitud de los grados de conectividad sugirió que los mejor conectados fueron GC1 y GC3, lo que es contrario a lo simulado. Con este método se pueden inferir los GC desconectados desde la matriz de varianzas de los errores de predicción (elementos fuera de la diagonal = cero), e identificar los grupos conectados indirectamente; sin embargo, el valor de conectividad para éstos es algunas veces mayor que el de los GC conectados directamente, como se observó en E3 y E4 del presente estudio.
En la Figura 2 se muestra el patrón del grado de conectividad para los tres contrastes entre GC, a través de los siete escenarios, obtenidos mediante el método rij, el cual presentó grados de conectividad consistentes con los escenarios simulados. Sin embargo, al pasar del E6 a E7 hubo un descenso en los grados de conectividad, a pesar de que E7 se simuló con mayor conectividad que E6 (un semental y una vaca más), lo que refleja inconsistencia del método. El método rij sí detectó conectividades indirectas, por ejemplo entre GC1 y GC3, pues aunque con valores bajos, el grado de conectividad fue diferente de cero en E3 (-0.0018) y E4 (0.002), lo cual fue consistente con estos escenarios simulados. Kuehn et al. (2008) evaluaron cambios de conectividad en varios escenarios con ovinos, comparando VEP y rij, y encontraron que r¡j fue más consistente para medir el riesgo de sesgos en las comparaciones de valores genéticos predichos entre rebaños; valores de 0.05 fueron de buena conectividad y 0.10 excelente, permitiendo tomar decisiones de selección entre rebaños. Con estos valores de conectividad se eliminaron aproximadamente 80 y 90 % de los sesgos iniciales debidos a diferencias en las medias genéticas entre los GC, respectivamente. Similarmente, Soga et al. (2010) compararon cuatro medidas de conectividad (VEP, GrC, rij y porcentaje de sementales comunes) para determinar la más apropiada, al evaluar los riesgos asociados en la comparación de valores genéticos predichos en cerdos Duroc a través de piaras; aunque GrC, rij y porcentaje de sementales comunes estuvieron altamente correlacionadas (>0.96), rij fue considerada la más apropiada por su facilidad de cómputo.
Una tendencia similar a la de rij fue observada en los métodos PRGA, VDG y GrC, con relación a la consistencia entre los cambios en los grados de conectividad de acuerdo con los escenarios simulados, de E1 a E6 (Cuadro 3). Adicionalmente, los métodos PRGA y VDG no permitieron detectar GC conectados indirectamente, pues el grado de conectividad fue igual a cero en E3 y E4 para el contraste GC1 vs GC3; por el contrario, el método GrC sí detectó conectividades indirectas entre GC1 y GC3, ya que el grado de conectividad fue diferente de cero en E3 ( — 0.2) y E4 (0.3), lo cual fue consistente con los escenarios simulados. El método PRGA no requiere la matriz inversa CZZ y Hao et al. (2004) recomiendan su uso porque es una medida directa de conectividad y se puede implementar fácilmente, pero los valores de conectividad en el presente estudio no fueron consistentes del E6 al E7. Respecto al método GrC, Hao et al. (2004) consideran que su desventaja es la capacidad computacional requerida para su determinación; además, según Mathur et al. (2002) y Roso et al. (2004), el GrC decrece linealmente cuando disminuye el tamaño de grupo, lo que puede favorecer a los grupos grandes.
En la Figura 3 se muestran los grados de conectividad generados con el método CD, los cuales tuvieron un patrón inconsistente con los escenarios de conectividad simulados; por ejemplo, para el contraste entre GC1 y GC3 en E3 y E5, la conectividad disminuyó con respecto a E2 y E4, contrario a lo esperado. Otro inconveniente fue que el método no permitió identificar GC desconectados; por ejemplo, en E1 todos los GC están desconectados, pero el método generó valores mayores que cero y diferentes entre sí. A pesar de estas desventajas observadas en el presente estudio, algunos autores reportan ventajas considerando otros atributos. Laloë et al. (1996) compararon los métodos IC, VEP y CD como indicadores de la calidad de las evaluaciones genéticas y encontraron que el CD combinó la estructura de los datos y la cantidad de información, proveyendo un balance entre la reducción de la varianza de errores de predicción y la pérdida de variabilidad debida a las relaciones genéticas entre animales, por lo cual señalan que CD fue el mejor método para evaluar la precisión de una evaluación genética. Kuehn et al. (2007) simularon varios escenarios para identificar estrategias, establecer conectividad y reducir los sesgos potenciales en la comparación de valores genéticos predichos de animales en diferentes GC. Estos autores usaron rij, GrC y CD y encontraron que sólo CD mantuvo una relación con los sesgos entre escenarios, siendo una medida directa de la reducción proporcional de sesgos; por el contrario, rij y GrC variaron dependiendo del tipo de conexión.
Además, la aplicación rutinaria de CD en las evaluaciones genéticas con muchos datos puede ser difícil porque se requiere la matriz inversa CZZ de las ecuaciones de modelos mixtos; sin embargo, Fouilloux y Laloë (2001) sugirieron métodos de muestreo y análisis para superar esta dificultad. Fouilloux et al. (2008) propusieron otro método que incluye: 1) calcular el CD de comparaciones entre grupos de animales usando muestreo para estimar varianzas empíricas y predecir valores genéticos de una muestra simulada; y 2) formar grupos de hatos conectados. Este procedimiento fue utilizado por Tarrés et al. (2010) para determinar conectividad en poblaciones de bovinos para carne que usan monta natural.
En la Figura 4 se muestran los grados de conectividad con el método TLGD, para los contrastes entre los tres GC y a través de los siete escenarios simulados. El método generó valores de conectividad consistentes con los escenarios simulados, por lo que muestra ser un buen indicador de conectividad; pero no permitió identificar GC conectados indirectamente. Según Roso et al. (2004), TLGD demanda menos capacidad de cómputo y se puede usar en evaluaciones genéticas para medir la conectividad. Estos autores compararon los métodos VdGC, GrC y TLGD en términos del coeficiente de determinación para predecir la VEP promedio de cada GC con los demás, y encontraron que TLGD (76 %) fue mejor predictor que GrC (73 %) y VdGC (50 %); cuando los modelos predijeron la VEP promedio para cada par de GC, TLGD (71 %) fue mejor predictor que GrC (49 %) y VdGC (51 %). Además, depende menos del tamaño de grupo que otros métodos, como el GrC, pero según Hao et al. (2004) tiene desventajas teóricas que se deben estudiar para su aplicación en datos de campo.
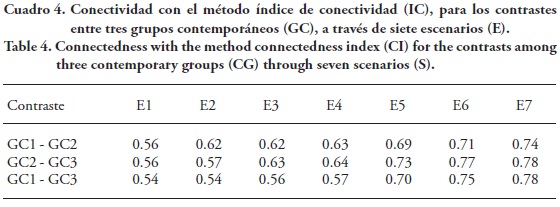
En el Cuadro 4 se muestran los grados de conectividad con el método IC, para los contrastes entre los tres GC y a través de los siete escenarios simulados. Similar a lo observado con TLGD, el método IC generó valores de conectividad consistentes con los escenarios simulados, pero no permitió identificar GC desconectados. Por ejemplo, en E1 todos los GC están desconectados, pero el método generó valores mayores que cero (0.54 a 0.56). A diferencia de los métodos PRGA, VDG, GrC y rij, los métodos IC y TLGD sí mostraron valores de conectividad mayores al pasar de E6 a E7, lo que es consistente con el aumento en el número de animales emparentados. Al comparar dos estructuras de datos completas y balanceadas, pero diferentes en el número de crías, el IC fue de uno en ambos casos, por lo que sólo responde a la estructura de los datos, sin considerar la cantidad de información (Hanocq et al., 1999).
Con el propósito de tener una aproximación de la relación teórica entre los métodos aplicados en este estudio, en el Cuadro 5 se muestran los estimadores de coeficientes de correlación simple entre los métodos de conectividad. El coeficiente de correlación entre la mayoría de los métodos fue alto (>|0.9|), excepto para el método CD y algunas combinaciones con el método VdGC. La correlación entre los valores de conectividad de VEP con TLGD e IC fue alta ( — 0.97) y menor con FG (0.91); este último valor es mayor que el reportado (0.67) por Kennedy y Trus (1993). La correlación entre GrC y rij en el presente estudio fue alta (0.99) y similar a la estimada por Soga et al. (2010). El CD fue el menos consistente con los cambios en conectividad simulados; además, su correlación con la VEP fue baja ( — 0.58). La correlación en el presente estudio entre TLGD y el GrC fue 0.94, superior a la reportada (0.86) por Roso et al. (2004); además, TLGD mostró una alta asociación con VEP y VdGC (—0.97 y —0.85), valores superiores a los publicados (— 0.66 y — 0.64) por Roso et al. (2004).
Un método para determinar conectividad debe identificar GC desconectados, y conectados directa e indirectamente; además, debe generar valores consistentes de conectividad, ser fácil de aplicar e interpretar. Magaña-Valencia et al. (2012) describieron los atributos de los 10 métodos cuantitativos comparados en el presente estudio, en términos de la escala de medición, valores que representan mejor conectividad, requerimientos de inversión de matrices, y su capacidad para identificar GC desconectados y conectados indirectamente. De los 10 métodos estudiados, cinco, PRGA, VDG, GrC, rij, y TLGD identificaron GC desconectados, pero tres PRGA, VDG, y TLGD no estimaron conectividad en forma indirecta. El problema de detección de conectividad indirecta puede superarse mediante algoritmos que consideran el método geométrico simple (Searle, 1987) como se hace para el método TLGD (Fries y Roso, 1997). En el presente estudio, la capacidad de los métodos para identificar GC desconectados y conectados indirectamente fue confirmada mediante su aplicación en los escenarios simulados.
Respecto a la consistencia de los métodos, en el presente estudio sólo VEP, FG, TLGD e IC mostraron patrones en los valores de conectividad acordes con el aumento en el número de sementales o vientres en común a través de los siete escenarios simulados. Sin embargo, debido a que los métodos VEP, FG e IC no permitieron identificar GC desconectados, que el problema de detección de conectividad indirecta para TLGD puede resolverse como ya se indicó (Fries y Roso, 1997), y que TLGD mostró una asociación alta (—0.97) con VEP, se considera que el método de número de lazos genéticos directos es el más apropiado para determinar conectividad en evaluaciones genéticas rutinarias.
CONCLUSIONES
Considerando los criterios de consistencia, definida como la relación entre el grado de conectividad obtenido y la conectividad simulada al aumentar progresivamente el número de sementales o vacas entre grupos contemporáneos a través de los escenarios simulados, y la capacidad para identificar grupos contemporáneos desconectados, y conectados directa e indirectamente, el método de número de lazos genéticos directos totales debido a sementales y hembras comunes es el más apropiado. Además, está altamente correlacionado con la varianza del error de predicción de las diferencias en valores genéticos entre animales. Se sugiere usar el método de número de lazos genéticos directos en evaluaciones genéticas rutinarias de bovinos.
AGRADECIMIENTOS
Los autores agradecen al Consejo Nacional de Ciencia y Tecnología y al Consejo Mexiquense de Ciencia y Tecnología por el financiamiento otorgado para los estudios de Maestría en Ciencias del primer autor.
LITERATURA CITADA
Banos, G., and R. A. Cady. 1988. Genetic relationship between the United States and Canadian Holstein bull populations. J. Dairy Sci. 71: 1346-1354. [ Links ]
Carneiro S., A. P., R. A. Torres, R. F. Euclydes, M. A. Silva, P. S. Lopes, P. L. S. Carneiro, e R. A. T. Filho. 2001. Efeito da conexidade de dados sobre a acuracia dos tested de progenie e performance. Rev. Bras. Zoot. 30: 342-347. [ Links ]
Clément V., B. Bibé, E. Verrier, J. M. Elsen, E. Manfredi, J. Bouix, and E. Hanocq. 2001. Simulation analysis to test the influence of model adequacy and data structure on the estimation of genetic parameters for traits with direct and maternal effects. Genet. Sel. Evol. 33: 369-395. [ Links ]
Fernando, R. L., D. Gianola, and M. Grossman. 1983. Identifying all connected subsets in a two-way classification without interaction. J. Dairy Sci. 66: 1399-1402. [ Links ]
Fouilloux, M. N., and D. Laloé. 2001. A sampling method for estimating the accuracy of predicted breeding values in genetic evaluation. Genet. Sel. Evol. 33: 473-486. [ Links ]
Fouilloux, M. N., B. Clément, and D. Laloë. 2008. Measuring connectedness among herds in mixed linear models: from theory to practice in large-sized genetic evaluations. Genet. Sel. Evol. 40: 145-159. [ Links ]
Foulley, J. L., E. Hanocq, and D. Boichard. 1992. A criterion for measuring the degree of connectedness in linear models of genetic evaluation. Genet. Sel. Evol. 24: 315-330. [ Links ]
Fries, L. A., e V. M. Roso. 1997. Conectabilidade em avaliacóes genéticas de gado de corte: uma proposta heurística. In: Anais XXXIV Reun. Soc. Bras. Zoot., 28 de Julho a 1 de Agosto. Juiz de Fora. pp: 159-161. [ Links ]
Hanocq E., D. L. Tiphine, et B. Bibë. 1999. Le point sur la notion de connexion en génétique animale. INRA Prod. Anim. 12(2): 101-111. [ Links ]
Hao, Z., L. Xiao-Hong, W. Chong, L. Jia-Qi, and C. Yao-Sheng. 2004. Study on the measures of connectedness between herds. Agric. Sci. China 3(2): 143-148. [ Links ]
Kennedy, B. W., and D. Trus. 1993. Considerations on genetic connectedness between management units under an animal model. J. Anim. Sci. 71: 2341-2352. [ Links ]
Kuehn, L. A., R. M. Lewis, and D. R. Notter. 2007. Managing the risk of comparing estimated breeding values across flocks or herds through connectedness: a review and application. Genet. Sel. Evol. 39: 225-247. [ Links ]
Kuehn, L. A., D. R. Notter, G. J. Nieuwhof, and R. M. Lewis. 2008. Changes in connectedness over time in alternative sheep sire referencing schemes. J. Anim. Sci. 86: 536-544. [ Links ]
Laloë, D. 1993. Precision and information in linear models of genetic evaluation. Genet. Sel. Evol. 25: 557-576. [ Links ]
Laloë, D., F. Phocas, and F. Ménissier. 1996. Considerations on measures of precision and connectedness in mixed linear models of genetic evaluation. Genet. Sel. Evol. 28: 359-378. [ Links ]
Lewis, R. M., R. E. Crump, G. Simm, and R. Thompson. 1999. Assessing connectedness in across-flock genetic evaluations. In: Proc. Br. Soc. Anim. Sci., March 22-24. Scarborough, England. p: 121. [ Links ]
Magaña-Valencia, F., R. Núñez-Domínguez, R. Ramirez-Valverde, y F. A. Rodriguez-Almeida. 2012. Conectividad en evaluaciones genéticas de animales: 1. Metodologías. Agrociencia 46: 677-692. [ Links ]
Mathur, P. K., B. Sullivan, and J. Chesnais. 2002. Measuring connectedness: concept and atphplication to a large industry breeding program. In: Proc. 7 World Congr. Genet. Appl. Livest. Prod., August 19-23. Montpellier, France, p. 13 (abstract). [ Links ]
Roso, V. M., R. S. Schenkel, and S. P. Miller. 2004. Degree of connectedness among groups of centrally tested beef bulls. Can. J. Anim. Sci. 84: 37-47. [ Links ]
Searle, S. R. 1987. Linear Models for Unbalanced Data. John Wiley & Sons, New York. USA. 536 p. [ Links ]
Soga, N., M. L. Spangler, C. R. Schwab, P. J. Berger, and T. J. Baas. 2010. Comparison of connectedness measures and changes in connectedness of the U. S. Duroc population. In: Proc. 9th World Congr. Genet. Appl. Livest. Prod., July 31 to August 6, Leipzig, Germany. 4 p. [ Links ]
Tarrés, J., M. Fina, and J. Piedrafita. 2010. Connectedness among herds of beef cattle bred under natural service. Genet. Sel. Evol. 42: 6-15. [ Links ]
Tosh, J. J., and J. W. Wilton. 1994. Effect of data structure on variance of prediction error and accuracy of genetic evaluation. J. Anim. Sci. 72: 2568-2577. [ Links ]














