Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Agrociencia
versión On-line ISSN 2521-9766versión impresa ISSN 1405-3195
Agrociencia vol.46 no.7 Texcoco oct./nov. 2012
Ciencia animal
Conectividad en evaluaciones genéticas de animales. 1. Metodologías
Connectedness in animal genetic evaluations. 1. Methodologies
Fabián Magaña–Valencia1, Rafael Núñez–Domínguez1*, Rodolfo Ramírez–Valverde1, Felipe A. Rodríguez–Almeida2
1 Departamento de Zootecnia, Postgrado en Producción Animal, Universidad Autónoma Chapingo. 56230. Carretera México–Texcoco, Km 38.5. Chapingo, Estado de México. 2 Facultad de Zootecnia y Ecología, Universidad Autónoma de Chihuahua. 31031. Periférico Francisco R. Almada, Km 1. Chihuahua, Chihuahua. *Autor responsable: (rafaelndguez@yahoo.com.mx).
Recibido: febrero, 2012.
Aprobado: septiembre, 2012.
Resumen
La predicción de valores genéticos a través de las ecuaciones de modelos mixtos con el modelo animal es la herramienta más confiable para la selección de sementales y vientres, y el grado de conectividad de los datos es uno de los factores que incide en su calidad. En poblaciones donde la selección y la deriva genética repercuten en las medias genéticas de los grupos contemporáneos (GC), la predicción de valores genéticos puede ser sesgada si los GC están desconectados, disminuyendo la confiabilidad de sus comparaciones. El propósito del presente ensayo fue realizar una recopilación y descripción de los principales métodos disponibles en la literatura para evaluar la conectividad genética entre GC. Se describieron 12 métodos para determinar conectividad genética entre GC, dos cualitativos y 10 cuantitativos. Un método para determinar la conectividad idealmente debe identificar GC desconectados, y conectados directa e indirectamente. Además, debe evaluar el grado de conectividad, ser práctico, y fácil de aplicar e interpretar. Las metodologías cualitativas para evaluar la conectividad entre GC sólo permiten separar los GC que están conectados de los que no lo están, mientras que los métodos cuantitativos evalúan adicionalmente el grado de conectividad. Los métodos descritos son alternativas que se deben comparar considerando una misma estructura de los datos para escenarios comunes en evaluaciones genéticas de animales.
Palabras clave: conectividad genética, métodos de conectividad, evaluación genética.
Abstract
Predicting genetic values with mixed model equations together with the animal model is the most reliable tool for selection of sires and dams, and the degree of data connectedness is one of the factors that influence selection quality. In populations where selection and genetic drift have an influence on the genetic means of contemporary groups (GC), predictions of genetic values may be biased and less reliable if the GCs are disconnected. The objective of this essay was to present a compilation and description of the main methods available in the literature for evaluating genetic connectedness among GC. Twelve methods that determine genetic connectedness among GC were described, two qualitative and 10 quantitative. A method for determining connectedness should ideally identify disconnected and directly and indirectly connected GC. It should also evaluate the degree of connectedness, and it should be practical and easy to apply and interpret. The qualitative methods for evaluating connectedness among GC only allow separating GC that are connected from those that are not, while quantitative methods additionally evaluate the degree of connectedness. The methods described are alternatives that need to be compared considering a single structure of data for common scenarios in genetic evaluations of animals.
Key words: genetic connectedness, connectedness methods, genetic evaluation.
INTRODUCCIÓN
Las ecuaciones de modelos mixtos (EMM) de Henderson con el modelo animal se usan extensivamente para predecir los valores genéticos (VG) de los animales, ya que utilizan todas las relaciones genéticas aditivas de la población a evaluar, maximizando la exactitud de las predicciones (Henderson, 1973). Esta metodología supone que al incorporar los efectos ambientales en el modelo, incluyendo los del grupo contemporáneo (GC), las comparaciones entre los méritos genéticos de animales nacidos en diferentes GC son apropiadas. Sin embargo, si estos GC no están conectados genéticamente de manera apropiada, existe el riesgo de cuantificar las diferencias genéticas entre GC como diferencias ambientales (Clément et al., 2001; Kuehn et al., 2007; Tarrés et al., 2010).
La calidad de las evaluaciones genéticas depende de numerosos factores, en particular de la estructura de los datos (Hanocq et al., 1999) descrita según las matrices de diseño y las matrices de (co)varianzas. Al considerar la estructura de los datos, el grado de conectividad entre los GC puede afectar la exactitud de la evaluación genética (Kennedy y Trus, 1993; Tosh y Wilton, 1994; Carneiro et al., 2001b), y la confiabilidad de la comparación entre animales de diferentes GC (Mathur et al., 2002a; Roso et al., 2004; Fouilloux et al., 2006). La exactitud de los VG se afecta por la sub o sobreestimación de parámetros genéticos, mientras que el efecto en la confiabilidad de las comparaciones puede reflejarse en la jerarquización incorrecta de animales (Fouilloux et al., 2006; Kuehn et al., 2009; Soga et al., 2010) y por consiguiente en la respuesta a la selección (Lewis y Simm, 2000; Simm et al., 2001; Kuehn et al., 2008a).
Para predecir los VG, teóricamente no se requiere de conectividad (Fernando et al., 1983; Kennedy y Trus, 1993; Fries y Roso, 1997) y los GC desconectados no causan predicciones sesgadas, siempre y cuando los VG de los animales base estén aleatoria e idénticamente distribuidos en toda la población (Kennedy y Trus, 1993; Tosh y Wilton, 1994). Sin embargo, esto difícilmente se cumple para poblaciones reales sujetas a fuerzas como la selección o la deriva genética, que repercuten en las medias genéticas de los GC (Kuehn et al., 2007; Tarrés et al., 2010) y en la validez y precisión de las estimaciones de los componentes de (co)varianza (Tosh y Wilton, 1994; Clément et al., 2001).
Los sesgos en VG por GC desconectados pueden reducirse al incluir el efecto de grupo genético en el modelo para representar el valor genético medio de los sementales en cada grupo; sin embargo, esta alternativa no es viable en todos los escenarios. La inclusión de grupos genéticos debería considerarse sólo para grupos con muchos animales, con alta conectividad y alta exactitud, y en caso de detectar grandes diferencias genéticas entre las subpoblaciones de animales base (Phocas y Laloë, 2004). Para características con baja heredabilidad, estos sesgos se pueden disminuir mediante la evaluación conjunta con características de mayor heredabilidad y altamente correlacionadas, por una mejoría en la conectividad genética (Tarrés et al., 2010).
Al considerar un modelo de semental, la conectividad entre GC puede conocerse por la estimabilidad de las funciones lineales entre sementales y GC. Según Hanocq et al. (1999), para determinar la conectividad es mejor usar un modelo de semental que considera la mayor parte de las fuentes de conexión; así, otras fuentes ignoradas en el análisis son adicionales y pueden eventualmente mejorar la conectividad. Sin embargo, con un modelo animal donde todas las relaciones de parentesco contribuyen a conectar los GC, la forma de medir la conectividad es diferente y generalmente se indica su grado.
La conectividad de los GC se puede estimar por métodos cualitativos o cuantitativos. En el primer caso sólo es posible identificar grupos conectados y desconectados, mientras que en el segundo es posible determinar el grado de conectividad ofreciendo una medida de confiabilidad o riesgo de las comparaciones entre animales en diferentes grupos (GC, hatos, regiones, países). Para evaluar el grado de conectividad genética entre GC, Kennedy y Trus (1993), Lewis et al. (1999) y Mathur et al. (2002b) proponen varios métodos, mientras que Laloë et al. (1996), Roso et al. (2004) y Soga et al. (2010) compararon las ventajas y desventajas de sólo algunos métodos. Además se usan algunos métodos para cuantificar el impacto de la implementación de estrategias orientadas a mejorar la conectividad genética de poblaciones reales o simuladas (Huisman et al., 2006; Veselá et al., 2007; Fouilloux et al., 2008) y para evaluar la conectividad genética de poblaciones reales (Nakaoka et al., 2009; Sun et al., 2009; Pegolo et al., 2012).
En las evaluaciones genéticas nacionales rutinarias de algunos países se usan diferentes métodos: el método de coeficiente de determinación en Francia para bovinos para carne y cabras (Pegolo et al., 2012), el método de grado de conectividad en Canadá para cerdos (Mathur et al., 2002a), el método de correlación entre las varianzas de los errores de predicción de VG en el Reino Unido para borregos (Simm et al., 2001), y el método de número total de lazos genéticos directos entre GC en Canadá para bovinos (Roso et al., 2004). Para algunas poblaciones de animales (como razas de bovinos para carne, ovinos y caprinos), los hatos están dispersos en varias regiones geográficas, los tamaños de los hatos y GC muchas veces son pequeños en relación con otras especies, y la inseminación artificial se usa en forma reducida. Por tanto, un problema común para la evaluación genética nacional en este tipo de poblaciones es el bajo grado de conectividad entre los GC, lo cual requiere de métodos precisos y prácticos para su determinación.
En general, la mayoría de los métodos propuestos, comparados o utilizados usan la varianza del error de predicción (VEP) de las diferencias en VG entre animales o medidas relacionadas con ésta. Sin embargo, no hay consenso en recomendar el uso generalizado de alguno de ellos. Para facilitar la comparación de los métodos propuestos, considerando diversos escenarios y una misma estructura de datos, es importante recopilar información que describa los principios y cálculos necesarios para estimar conectividad genética. Por tanto, el propósito de este ensayo fue realizar una recopilación y descripción de los principales métodos disponibles en la literatura para evaluar la conectividad genética entre GC.
MÉTODOS PARA EVALUAR LA CONECTIVIDAD GENÉTICA
Métodos cualitativos
Método geométrico simple (MGS)
Este método, presentado por Searle (1987), se basa en el producto de las matrices XZ, donde X y Z son matrices de incidencia para GC y sementales en los modelos mixtos. De manera esquemática se construye una tabla de doble entrada donde se representan, por un lado a los sementales (i) y por el otro a los GC o hatos (j), y las intersecciones (celdas) muestran la progenie del i–ésimo semental en el j–ésimo hato. La conectividad de los datos se indica por el trazo de líneas continuas horizontales o verticales, las cuales pueden cambiar de dirección sólo si las celdas tienen valores; si la celda no tiene datos, el trazo no es posible. Las celdas unidas mediante líneas representan grupos de datos conectados.
Algoritmo para identificar los grupos conectados (AIGC)
Tomando como base el método geométrico simple, Fernando et al. (1983) propusieron un algoritmo para identificar los GC (hato–año–estación) conectados, en una clasificación de dos vías sin interacción. No requiere la absorción de efectos principales, por lo que es computacionalmente simple. El álgebra se limita a dos vectores, por lo que el algoritmo se puede aplicar cuando el número de clases de cada uno de los efectos principales sea grande. El resultado es un vector y los elementos con el mismo número corresponden a grupos conectados.
El algoritmo involucra cuatro pasos para cada subgrupo (G): 1) forma un vector nx1 (Gi) con sus j–ésimos elementos, siendo Gij=i para j=1,...,n, si un semental j tiene progenie en el subgrupo i, y Gij=0 de otro modo; 2) toma ci = ci — 1+ Gi, donde c0= 0; 3) examina cada elemento de este vector iniciando con el primer elemento de ci, ci1; para el primer elemento donde cij>i, cambia a "i" aquellos elementos de ci que son iguales que cij y también los que son iguales que cij – i; y 4) repite el paso 3 hasta que cada n elemento de ci es menor o igual que i. Finalmente, los elementos de cm con el mismo número corresponden a sementales conectados a través de GC.
Métodos cuantitativos
Promedio de relaciones genéticas aditivas (PRGA)
Banos y Cady (1988) muestran que el PRGA se puede usar como una medida de conectividad entre GC. La mejor conectividad genética entre GC está dada por el mayor PRGA entre animales de diferentes GC. El PRGA entre animales de diferentes GC se diferencia de los demás métodos en que es una medida directa de conectividad genética entre GC y es fácil de obtener.
EL PRGA se obtiene de la inversa de la matriz de relaciones genéticas aditivas (A–1). La A (y A–1) se puede subdividir de la siguiente manera:

donde A11, A22 ... Ann es igual a A para animales del GC1, GC2,... GCn, respectivamente, y los elementos fuera de la diagonal (A12,... A1n y A(n_1)n) son submatrices de relaciones genéticas aditivas (aij) para animales de dos GC. La media de dj para las submatrices de la diagonal (Axx) para i≠j es:

donde n = orden de Axx. La media de aij para las sub–matrices de fuera de la diagonal (Axy) es:

donde r= número de filas, y c= número de columnas en Axy.
Índice de conectividad (IC) e índice general de conectividad (IGC)
Foulley et al. (1992) presentaron el concepto de grado o nivel de no conectividad, relacionando la varianza del error de predicción (VEP) de los efectos genéticos de un modelo completo con la VEP de un modelo reducido que excluye los efectos fijos. Estos autores propusieron el índice de conectividad (IC), que se basa en el uso de un vector de coeficientes de contrastes (x):
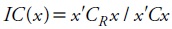
donde CR es una parte de la inversa de la matriz de coeficientes en un modelo reducido, y C es una parte de la inversa de la misma matriz de coeficientes en un modelo completo. Este índice refleja la reducción en la VEP cuando los efectos fijos se conocen exactamente o no existen. El IC (x) varía entre 0 y 1, siendo cercano a 1 cuando los animales están bien conectados.
Foulley etal. (1992) plantearon la hipótesis de que una base de datos bien conectada es necesariamente ortogonal (balanceada). Si dos factores son ortogonales entre sí, no hay sesgo en uno de los factores si el otro se quita del modelo. Si un factor aleatorio es totalmente ortogonal a un factor fijo eliminado en el modelo reducido, la VEP es la misma para efectos aleatorios en el modelo completo y en el reducido. Por tanto, las formas cuadráticas sobre la base de la inversa de la matriz de coeficientes del modelo completo y del reducido deberían ser equivalentes, y el IC(x) sería igual a uno.
Adicionalmente, Foulley et al. (1992) propusieron un índice general de conectividad (IGC) entre GC y VG, que es la media geométrica de los eigenvalores (µi) de [CRzz – µiCzz]ci = 0, determinada por:

donde |C| y |CR| son los determinantes de la inversa de las matrices de VEP con y sin efectos fijos en el modelo, y n es el número de animales o rango de las columnas de la matriz de incidencia de los parámetros a los que relacionan las subunidades CR y C.
Coeficiente de determinación (CD) e índices generales de precisión
Aunque la ortogonalidad de los datos propuesta por Foulley et al. (1992) es deseable, Laloë (1993) menciona que una medida de precisión es más apropiada para determinar si los animales de diferentes ambientes pueden ser comparados, y propone el coeficiente de determinación (CD) de un vector de contrastes (x) de VG, que es la forma general del CD individual o exactitud de la predicción del VG. El CD(x) corresponde al cuadrado de la correlación entre la diferencia predicha y verdadera de valores genéticos; también varía de cero a uno y se interpreta igual (mide la exactitud de la diferencia del valor genético entre dos animales o GC). Así, la exactitud o confiabilidad de la comparación del VG entre el animal i y el j, está dada por:

donde xij el contraste entre el animal i y el j, λ = σe2/σa2, Aii, Aij y Ajj son los elementos de la matriz de relaciones genéticas aditivas, Ciizz, Cijzz, y Cjjzz son los elementos de la inversa de la matriz de coeficientes de las ecuaciones de modelos mixtos. En forma matricial, el CD es:
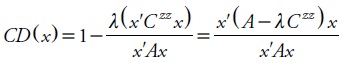
donde x es un vector cuyos elementos suman cero, Czz = (Z'MZ + λA–1)–1 σe2,y M = I – X(X'X)–X'.
Laloë (1993) también propone dos índices generales de precisión usando las formas cuadráticas del CD, y relacionándolas con los eigenvalores (µi) y los eigenvectores (ci) resultantes de la solución al problema generalizado de los eigenvalores:

El número de eigenvalores es igual al número de VG predichos. El eigenvalor menor siempre será cero; el resto corresponde a todos los contrastes independientes posibles. Los µ deben ser ordenados en forma ascendente. Los dos estadísticos propuestos están en función de esos eigenvalores:

El primer índice (p1) es la media aritmética de los n — 1 eigenvalores, y el segundo índice es la media geométrica de los mismos eigenvalores; (p2) es más sensitivo a eigenvalores bajos. Un valor nulo de un eigenvalor da un p2 nulo, indicando que existe por lo menos un contraste sin información suplementaria (desconexión).
Para bases de datos grandes, Boichard et al. (1992) obtuvieron una aproximación razonable (sesgo <4 %) de tr(A–1Ω) para un modelo animal con una clase de efectos fijos y una de efectos aleatorios, donde tr= traza y Ω = C–1 =(Z'MZ + λA–1)–1 Ya que p1 es una función de tr (A–1Ω)(p1 = [1 –λ tr (A–1Ω)]/ (n –1)), este método se puede usar para aproximarse a p1 en bases de datos grandes.
VEP de las diferencias en VG entre animales ( VEP(x))
La conectividad genética entre GC reduce la VEP de las comparaciones entre los animales en diferentes GC, porque la VEP de las comparaciones entre animales emparentados es menor que la obtenida entre animales no emparentados, y porque las conexiones genéticas reducen la variabilidad de las estimaciones de las diferencias entre los efectos de GC. Así, Kennedy y Trus (1993) propusieron el promedio de la VEP de las diferencias en VG (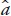 ) entre animales con registro en diferentes GC, o la VEP de la comparación entre animales, como una medida estadística lógica y apropiada del grado de conectividad de los datos. La VEP de la diferencia entre dos animales (VEPD), uno del i–ésimo y otro del j–ésimo grupo, está dada por:
) entre animales con registro en diferentes GC, o la VEP de la comparación entre animales, como una medida estadística lógica y apropiada del grado de conectividad de los datos. La VEP de la diferencia entre dos animales (VEPD), uno del i–ésimo y otro del j–ésimo grupo, está dada por:

Ambas, el promedio de la VEP y la VEP de las diferencias en VG entre animales de diferentes GC, pueden obtenerse para un contraste (x):

donde C zz es una parte de la inversa de la matriz de coeficientes de las ecuaciones de modelos mixtos. Esta medida no tiene un rango definido y está muy relacionada con el CD, que es más fácil de interpretar. Además, la obtención de esta varianza para bases de datos grandes, como ocurre en muchas evaluaciones genéticas, es prácticamente imposible (Carneiro et al., 2001a; Mathur et al., 2002a), por lo que la aplicación rutinaria de este método es poco factible (Roso et al., 2004).
Flujo de genes (FG)
Este método es una extensión, con el modelo animal, de la matriz X'Z usada para medir conectividad con el modelo de semental, y simplemente traza la transferencia de genes de un GC al siguiente. El FG fue propuesto por Kennedy y Trus (1993) su virtud principal es la facilidad de cómputo y mide conectividad histórica (a través de ancestros) mediante la multiplicación de las matrices X'ZTQ. El FG puede aplicarse como una medida geométrica de conectividad o como una medida cuantitativa entre dos GC, al tomar la diferencia absoluta entre la suma de los elementos de la diagonal y la suma de los elementos fuera de la diagonal (un valor pequeño indica alta conectividad). La matriz Q identifica animales fundadores con respecto a su GC de origen, y T es una matriz triangular inferior que traza el flujo de genes de una generación a la siguiente; así A = T'WT; donde W es la diagonal de la matriz de las varianzas de muestreo mendeliano. Para obtener la matriz T es necesario ordenar los animales por generación y seguir las instrucciones: 1) todos los elementos de la diagonal son uno; 2) todos los elementos a la derecha de la diagonal son cero; 3) los elementos a la izquierda de la diagonal que corresponden a las filas de los animales base son cero; y 4) los elementos de las filas a la izquierda de la diagonal de los animales con padres conocidos son el promedio de los elementos correspondientes a la fila del padre y de la madre. No es necesario usar toda la matriz T generada, normalmente sólo se usan las columnas que pertenecen a los animales fundadores. Los elementos dentro de todas las filas para animales con padres conocidos, muestran la fracción de genes de los animales fundadores y suman uno.
Varianza de la deriva genética (VDG)
Este método se basa en el promedio de las relaciones genéticas entre y dentro de los GC y no requiere la identificación de animales base o ancestros (Kennedy y Trus, 1993). Considera el cómputo de la matriz A o partes de ella, y aunque es más difícil que la obtención de A–1, su cómputo es factible para bases de datos grandes.
La matriz X'ZAZ'X mide la suma de las relaciones genéticas entre y dentro de GC. El promedio de relaciones genéticas, entre y dentro de GC, se obtiene al dividir cada elemento de la diagonal entre el número de registros al cuadrado de cada GC, y los elementos fuera de la diagonal entre el producto del número de registros en los GC involucrados. La matriz resultante se puede interpretar como los componentes genéticos de la (co)varianza de la deriva genética entre los GC; así, la VDG entre grupos sería igual a la suma de los elementos de la diagonal menos la suma de los elementos fuera de la diagonal, donde un valor pequeño indica un alto grado conectividad (Kennedy y Trus, 1993).
Varianza de las diferencias entre efectos de GC (VdGC)
Este método, propuesto por Kennedy y Trus (1993), se basa en el cómputo de la matriz de (co) varianzas de los efectos de GC, representada por {XX– X'Z(Z'Z + λA–1)–1 Z'X}–1 σe2. Los elementos positivos fuera de la diagonal son el resultado de las relaciones genéticas entre animales de diferentes GC e indican la conectividad genética; para grupos no conectados estos elementos son cero. De acuerdo con Mathur et al. (2002a) y Roso et al. (2004), la varianza de la diferencia entre el i–ésimo y el j–ésimo grupo (gc) está dada por:

La VdGC mide la precisión con que son estimadas las diferencias en las medias genéticas entre GC (Kuehn et al., 2008b); sin embargo, para poblaciones grandes con muchos GC, el cálculo de la matriz de (co)varianzas de los efectos de GC puede ser difícil (Kennedy y Trus, 1993). Además del desafío computacional, la VEP de la diferencia entre las estimaciones de efectos de GC tiene otra desventaja; es dependiente del tamaño y la estructura de los GC, así como de la naturaleza de las conexiones entre ellos (Mathur et al., 2002a).
Grado de conectividad (GrC)
Para separar los efectos de tamaño de GC y estructura en la conectividad, Mathur et al. (2002a,b) definieron este método como la correlación entre los efectos estimados de dos GC. Así, el GrC entre el i–ésimo y el j–ésimo GC está dado por:

donde los componentes de (co)varianzas con un modelo animal simple se obtienen de la matriz de VEP, que está dada por el producto de la varianza del error y la submatriz Czz de las EMM.
De acuerdo con Mathur et al. (2002a), para superar el problema en la obtención de la inversa directa de la matriz de coeficientes en bases de datos grandes se puede usar este procedimiento:
CC–1 = I así, CCi–1 = Ii
donde, C= matriz de coeficientes de las EMM, I = matriz identidad, Ii = un vector de la matriz identidad correspondiente al i–ésimo GC (un vector con uno para el GC y ceros), y Ci–1 = un vector de elementos de la inversa para el i–ésimo GC.
El vector Ci–1 se puede obtener para un GC a la vez por iteraciones. Esos vectores se combinan y el bloque de elementos de la inversa correspondientes al GC más reciente es extraído. Los elementos de la inversa son las (co)varianzas del error de predicción de los efectos de GC de interés. Este método puede usarse para obtener elementos de la inversa en las filas o columnas de cualquier matriz grande donde la inversa directa no es posible.
Correlación entre las varianzas de los errores de predicción de VG (rij)
Tomando en cuenta que la covarianza del error de predicción (CEP) entre los VG de dos animales debería ser cero si no están conectados, Lewis et al. (1999) propusieron como criterio de conectividad una correlación estadística similar al GrC, definida como la correlación entre las varianzas de los errores de predicción de VG (rij):

donde 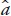 i es el VG predicho para el i–ésimo animal. Los autores sugieren promediar este estadístico para todos los pares de animales en diferentes GC con el fin de evaluar su conectividad.
i es el VG predicho para el i–ésimo animal. Los autores sugieren promediar este estadístico para todos los pares de animales en diferentes GC con el fin de evaluar su conectividad.
Número total de lazos genéticos directos entre GC (TLGD)
Este método fue desarrollado por Fries y Roso (1997), Fries (1998) y Roso et al. (2004) para medir el grado de conectividad entre GC a través de lazos genéticos directos debidos a sementales y hembras comunes. Los pasos básicos para el algoritmo y el criterio usado para el cómputo de TLGD son: 1) calcular el número de lazos genéticos directos entre pares de GC debido a sementales (LGs) y hembras (LGh) comunes; luego, para cada GC se calcula el número total de lazos genéticos (LGT) con todos los otros GC; 2) el GC con mayor LGT es llamado "grupo principal", y los sementales y hembras con progenie en este grupo forman el núcleo inicial conectado; 3) todos los GC se ponen en ceros (LGT) y son analizados recurrentemente contando los LGs y LGh generados por el núcleo inicial conectado; al final de cada corrida los GC con LGT>9 y por lo menos tres progenitores (sementales o hembras) son considerados conectados con el "grupo principal"; y 4) el resto de los GC (desconectados) se ponen en ceros y se repite el proceso hasta que el número de GC conectados sea igual al de la corrida anterior.
Las parejas de GC que no tienen algún lazo genético directo pueden estar conectadas indirectamente a través de otros GC y consecuentemente tener alta exactitud de comparación de VG estimados entre ellos. Por esta razón, TLGD es inadecuado para indicar el grado de conectividad entre pares de GC. La media aritmética también es inadecuada, ya que un par de GC con igual media aritmética puede tener un grado de conectividad diferente. Potencialmente se sugiere el uso de la media armónica de los LGT(2/[(1/ LGTi) + (1/ LGTj)]) (Roso et al., 2004). Para superar estas deficiencias, Roso y Schenkel (2006) desarrollaron el programa AMC – A Computer Program to Assess the Degree of Connectedness Among Contemporary Groups para medir el grado de conectividad entre los GC. El total de lazos genéticos es función del número y tipo de relaciones de parentesco entre animales en los diferentes GC.
Principales atributos de los métodos cuantitativos para determinar conectividad
Un método para determinar conectividad idealmente debe identificar GC desconectados, y conectados directa e indirectamente; además, debe medir el grado de conectividad, ser práctico, y fácil de aplicar e interpretar. En el Cuadro 1 se muestra la escala de los métodos de conectividad, requerimientos de matrices (Czz y A), y su capacidad para identificar GC desconectados y conectados indirectamente. De los 10 métodos cuantitativos descritos, cinco identifican GC desconectados, de los cuales tres estiman conectividad en forma directa e indirecta (GrC, rij y TLGD). Para bases de datos grandes, las matrices más complejas para obtener son la inversa de la matriz de coeficientes (Czz) y en algunas ocasiones la matriz A, y de los 10 métodos considerados seis requieren Czz y dos la matriz A. De los tres métodos que pueden identificar GC desconectados y conectados indirectamente, sólo el método TLGD no requiere las matrices complejas mencionadas. La interpretación de los valores de conectividad depende de la escala de medición; para cinco métodos esta interpretación se facilita dado que su escala es un intervalo definido, mientras que para el resto la escala es mayor o igual que cero, sin un límite superior, lo que dificulta la interpretación de los valores de conectividad.
La elección del mejor método para evaluar la conectividad genética entre GC depende del tamaño y estructura de los datos de poblaciones animales, por lo que se requiere comparar los métodos descritos, considerando una misma estructura de datos para escenarios representativos en evaluaciones genéticas.
CONCLUSIONES
Un método para determinar la conectividad, idealmente debe identificar grupos contemporáneos desconectados, y conectados directa e indirectamente; además, debe evaluar el grado de conectividad, ser práctico, y fácil de aplicar e interpretar. Las metodologías cualitativas para evaluar la conectividad entre grupos contemporáneos, sólo permiten separar los grupos que están conectados de los que no lo están y son útiles cuando se utiliza el modelo de semental para la predicción de los valores genéticos. Debido al uso generalizado de las ecuaciones de modelos mixtos de Henderson con el modelo animal, se utilizan métodos cuantitativos que evalúan adicionalmente el grado de conectividad, acercándose a las características de un método ideal. Los métodos cuantitativos descritos en este ensayo son alternativas que se deben comparar considerando una misma estructura de los datos, para escenarios comunes en evaluaciones genéticas de animales.
AGRADECIMIENTOS
Los autores agradecen al Consejo Nacional de Ciencia y Tecnología y al Consejo Mexiquense de Ciencia y Tecnología por el financiamiento otorgado para los estudios de Maestría en Ciencias del primer autor.
LITERATURA CITADA
Banos, G., and R. A. Cady. 1988. Genetic relationship between the United States and Canadian Holstein bull populations. J. Dairy Sci. 71: 1346–1354. [ Links ]
Boichard, D., L. R. Schaeffer, and A. J. Lee. 1992. Approximate restricted maximum likelihood and approximate prediction error variance of the Mendelian sampling effect. Genet. Sel. Evol. 24: 331–343. [ Links ]
Carneiro S., A. P., R. A. Torres, R. F. Euclydes, M. A. Silva, P. S. Lopes, P. L. S. Carneiro, e R. A. T. Filho. 2001a. Efeito da conexidade de dados sobre o valor fenotípico médio e a variancia genética aditiva. Rev. Bras. Zoot. 30: 336–341. [ Links ]
Carneiro S., A. P., R. A. Torres, R. F. Euclydes, M. A. Silva, P. S. Lopes, P. L. S. Carneiro, e R. A. T. Filho. 2001b. Efeito da conexidade de dados sobre a acurácia dos tested de progenie e performance. Rev. Bras. Zoot. 30: 342–347. [ Links ]
Clément V., B. Bibé, E. Verrier, J. M. Elsen, E. Manfredi, J. Bouix, and E. Hanocq. 2001. Simulation analysis to test the influence of model adequacy and data structure on the estimation of genetic parameters for traits with direct and maternal effects. Genet. Sel. Evol. 33: 369–395. [ Links ]
Fernando, R. L., D. Gianola, and M. Grossman. 1983. Identifying all connected subsets in a two–way classification without interaction. J. Dairy Sci. 66: 1399–1402. [ Links ]
Fouilloux, M. N., S. Minery, S. Mattalia, and D. Laloë. 2006. Assessment of connectedness in the international genetic evaluation of Simmental and Montbéliard breeds. Interbull Bulletin 35: 129–135. [ Links ]
Fouilloux, M. N., B. Clément, and D. Laloë. 2008. Measuring connectedness among herds in mixed linear models: from theory to practice in large–sized genetic evaluations. Genet. Sel. Evol. 40: 145–159. [ Links ]
Foulley, J. L., E. Hanocq, and D. Boichard. 1992. A criterion for measuring the degree of connectedness in linear models of genetic evaluation. Genet. Sel. Evol. 24: 315–330. [ Links ]
Fries, L. A., e V. M. Roso. 1997. Conectabilidade em avaliações genéticas de gado de corte: uma proposta heurística. In: Anais XXXIV Reun. Soc. Bras. Zoot., Juiz de Fora, Brasil. pp: 159–161. [ Links ]
Fries, L. A. 1998. Connectability in beef cattle genetic evaluation: the heuristic approach used in MILC.FOR. In: Proc. 6th World Congr. Genet. Appl. Livest. Prod., Armidale, Australia 27: 449–450. [ Links ]
Hanocq E., D., L. Tiphine, et B. Bibë. 1999. Le point sur la notion de connexion en génétique animale. INRA Prod. Anim. 12(2): 101–111. [ Links ]
Henderson, C. R. 1973. Sire evaluation and genetic trends. In: Proceedings of the Animal Breeding in Genetic Symposium in Honor of Dr. Jay L. Lush. American Society of Animal Science. Champaign, IL. pp: 10–41. [ Links ]
Huisman, A. E., B. Tier, and D. J. Brown. 2006. On assessing contrasts between groups of animals. Livest. Sci. 104: 254–267. [ Links ]
Kennedy, B. W., and D. Trus. 1993. Considerations on genetic connectedness between management units under an animal model. J. Anim. Sci. 71: 2341–2352. [ Links ]
Kuehn, L. A., R. M. Lewis, and D. R. Notter. 2007. Managing the risk of comparing estimated breeding values across flocks or herds through connectedness: a review and application. Genet. Sel. Evol. 39: 225–247. [ Links ]
Kuehn, L. A., D. R. Notter, and R. M. Lewis. 2008a. Assessing genetic gain, inbreeding, and bias attributable to different flock genetic means in alternative sheep sire referencing schemes. J. Anim. Sci. 86: 526–535. [ Links ]
Kuehn, L. A., D. R. Notter, G. J. Nieuwhof, and R. M. Lewis. 2008b. Changes in connectedness over time in alternative sheep sire referencing schemes. J. Anim. Sci. 86: 536–544. [ Links ]
Kuehn, L. A., R. M. Lewis, and D. R. Notter. 2009. Connectedness in Targhee and Suffolk flocks participating in the United States national sheep improvement program. J. Anim. Sci. 87: 507–515. [ Links ]
Laloë, D. 1993. Precision and information in linear models of genetic evaluation. Genet. Sel. Evol. 25: 557–576. [ Links ]
Laloë, D., F. Phocas, and F. Ménissier. 1996. Considerations on measures of precision and connectedness in mixed linear models of genetic evaluation. Genet. Sel. Evol. 28: 359–378. [ Links ]
Lewis, R. M., R. E. Crump, G. Simm, and R. Thompson. 1999. Assessing connectedness in across–flock genetic evaluations. In: Proc. Br. Soc. Anim. Sci., Scarborough, England. p. 121. [ Links ]
Lewis R. M., and G. Simm. 2000. Selection strategies in sire referencing schemes in sheep. Livest. Prod. Sci. 67: 129–141. [ Links ]
Mathur, P. K., B. Sullivan, and J. Chesnais. 2002a. Estimation of the degree of connectedness between herds or management groups in the Canadian swine population. http://www.ccsi.ca/include/docs/connectedness/connectedness_asds_1998.PDF. (consultado: marzo, 2007). [ Links ]
Mathur, P. K., B. Sullivan, and J. Chesnais. 2002b. Measuring connectedness: concept and application to a large industry breeding program. In: Proc. 7th World Congr. Genet. Appl. Livest. Prod., August 19–23. Montpellier, France. p. 13 (abstract). [ Links ]
Nakaoka, H., C. Gaillard, K. Fujinaka, N. Watanabe, M. Ito, K. Kawada, T. Ibi, Y. Sasae, and Y. Sasaki. 2009. The use of link provider data to improve national genetic evaluation across weakly connected subpopulations. J. Anim. Sci. 87: 62–71. [ Links ]
Pegolo, N. T., D. Laloë, H. N. de Oliveira, R. B. Lôbo, and M. N. Fouilloux. 2012. Trends on the genetic connectedness measures among Nelore beef cattle herds. J. Anim. Breed. Genet. 129: 20–29. [ Links ]
Phocas, F., and D. Laloë. 2004. Should genetic groups be fitted in BLUP evaluation? Practical answer for the French AI beef sire evaluation. Genet. Sel. Evol. 36: 325–345. [ Links ]
Roso, V. M., R. S. Schenkel, and S. P. Miller. 2004. Degree of connectedness among groups of centrally tested beef bulls. Can. J. Anim. Sci. 84: 37–47. [ Links ]
Roso, V. M., and R. S. Schenkel. 2006. AMC – A computer program to assess the degree of connectedness among contemporary groups. In: Proc. 8th World Congr. Genet. Appl. Livest. Prod., August, 13 to 18, Belo Horizonte, Brazil. Communication no. 27–26. [ Links ]
Searle, S. R. 1987. Linear Models for Unbalanced Data. John Wiley & Sons, New York. USA. 536 p. [ Links ]
Simm, G., R. M. Lewis, J. E. Collings, and G. J. Neiuwhof. 2001. Use of sire referencing schemes to select for improved carcass composition in sheep. J. Anim. Sci. 79 (E. Suppl): E255–E259. [ Links ]
Soga, N., M. L. Spangler, C. R. Schwab, P. J. Berger, and T. J. Baas. 2010. Comparison of connectedness measures and changes in connectedness of the U. S. Duroc population. In: Proc. 9th World Congr. Genet. Appl. Livest. Prod., July 31 to August 6, Leipzig, Germany. 4 p. [ Links ]
Sun, C. Y., C. K. Wang, Y. C. Wang, Y. Zhang, and Q. Zhang. 2009. Evaluation of connectedness between herds for three pig breeds in China. Animal 3(4): 482–485. [ Links ]
Tarrés, J., M. Fina, and J. Piedrafita. 2010. Connectedness among herds of beef cattle bred under natural service. Genet. Sel. Evol. 42: 6–15. [ Links ]
Tosh, J. J., and J. W. Wilton. 1994. Effect of data structure on variance of prediction error and accuracy of genetic evaluation. J. Anim. Sci. 72: 2568–2577. [ Links ]
Veselá, Z., J. Přibyl, L. Vostry, and L. Stole 2007. Stochastic simulation of the influence of insemination on the estimation of breeding value and its reliability. Czech J. Anim. Sci. 52(8): 236–248. [ Links ]














