Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Agrociencia
versión On-line ISSN 2521-9766versión impresa ISSN 1405-3195
Agrociencia vol.46 no.5 Texcoco jul./ago. 2012
Recursos naturales renovables
Variación genética y ambiental en dos ensayos de progenies de Pinus patula
Genetic and environment variation in two progeny tests of Pinus patula
José M. Salaya-Domínguez1, Javier López-Upton2 , J. Jesús Vargas-Hernández2
1 División Académica de Ciencias Agropecuarias, Universidad Juárez Autónoma de Tabasco. 86040. km. 25 carretera Villahermosa-Teapa, Ranchería La Huasteca, Centro, Tabasco. 2Campus Montecillo, Colegio de Postgraduados. 56230. km. 36.5 carretera México-Texcoco. Montecillo, Texcoco, Estado de México. *Autor responsable. (uptonj@colpos.mx).
Recibido: octubre, 2011.
Aprobado: junio, 2012.
Resumen
Pinus patulaSchiede ex Schltdl. et Cham. es un pino endémico de México mejorado en el extranjero por ser de crecimiento rápido y debe evaluarse al ser reintroducido. En este estudio se comparó el desarrollo de progenies con diferentes niveles de mejora genética en dos ensayos en ambientes contrastantes: Aquixtla, Puebla, a 2930 m (S1) y Acaxochitlán, Hidalgo, a 2260 m de elevación (S2). A los 6 años de edad se evaluó la supervivencia y a los 5 y 6 años el diámetro normal (DN), la altura total (ALT) y el volumen del tronco (VOL) de 36 familias mejoradas en Colombia y Sudáfrica (grupo M) y de 36 nativas seleccionadas fenotípicamente (grupo S). A los 6 años, la supervivencia fue 72.7 % en S1 y 90 % en S2, con mejores crecimientos en este último sitio. Hubo diferencias significativas (p≤0.05) entre los dos grupos de familias en todas las variables evaluadas en ambos años. Las progenies del grupo M fueron superiores en el volumen del tronco en S1 (4.3 vs. 3.6 dm3) pero no en S2 (10.7 vs. 13.5 dm3). En S1, las heredabilidades (hi2) para las características de crecimiento variaron de 0.09 a 0.12 y en S2 de 0.26 a 0.35. Las familias del grupo M presentaron mayores valores de hi2 en S1 pero en S2 los valores fueron similares. Las correlaciones genéticas (rg) y fenotípicas (rp) entre las variables de crecimiento para S1 y S2 variaron de 0.80 a 1.0 (≥0.80) en las dos edades. La interacción genotipo X ambiente (GXA) fue alta porque los valores de rB en ambas edades fueron menores a 0.70 y 0.0 en algunos casos. El grupo de familias M presentó menor interacción G X A en todas las variables evaluadas.
Palabras claves: Pinus patula, G X A, progenies, parámetros genéticos, reintroducción.
Abstract
Pinus patula Schiede ex Schltdl. et., Cham. is an endemic pine of México improved abroad because of its fast growth rate and should be evaluated to be reintroduced into the country. In this study the development of progenies with different breeding levels was compared in two trials in contrasting environments: Aquixtla, Puebla, at 2930 m (S1) and Acaxochitlán, Hidalgo, at 2260 m elevation (S2). Survival was evaluated at 6 years of age and at 5 and 6 years normal diameter (ND), total height (ALT), and stem volume (VOL) of 36 families improved in Colombia and South Africa (group M) and of 36 native families phenotypically selected (group S). At 6 years, survival was 72.7 % in S1 and 90 % in S2, with better growth in the latter site. There were significant differences (p≤ 0.05) between the two groups of families in all variables in both years. Progenies of the M group were higher in the volume of the trunk in S1 (4.3 vs. 3.6 dm3) but not in S2 (10.7 vs. 13.5 dm3). In S1, heritabilities (hi2) for growth traits ranged from 0.09 to 0.12 and in S2 from 0.26 to 0.35. Families of M Group had higher values of hi2 in S1 but in S2 values were similar. Genetic (rg) and phenotypic (rp) correlations between the growth variables for S1 and S2 ranged from 0.80 to 1.0 (≥ 0.80) at both ages. The genotypeX environment interaction (GX A) was high because the values of rB in both ages were lower than 0.70 and 0.0 in some cases. The group of M families had lower GX A interaction in all variables evaluated.
Key words: Pinus patula, G X A, progenies, genetic parameters, reintroduction.
INTRODUCCIÓN
Pinus patula Schiede ex Schltdl. et Cham. es el pino mexicano más plantado dentro y fuera de México. Desde su inclusión en los programas de plantaciones en Sudáfrica, Zimbabwe, Australia, Nueva Zelanda y Colombia, el interés en la especie aumenta por su crecimiento rápido, calidad de madera y facilidad de manejo (Wormald, 1975; Wright et al., 1995; Dvorak et al., 2000). Las plantaciones comerciales inician con un plan de mejoramiento genético, que incluye la selección de las procedencias y las familias con adaptación y crecimiento mayor.
El mejoramiento genético forestal se enfoca en el aumento y calidad de los árboles y su adaptabilidad a cambios ambientales (White et al., 2007). Varias empresas forestales investigan la mejora genética de P. patula y desarrollan individuos con características de crecimiento mejores que los silvestres (Dvorak et al., 2000; CAMCORE, 2007). Losárboles han aumentado su productividad y tasa de crecimiento, pero es posible que se haya modificado su adaptación (Sáenz et al., 1994), especialmente si los árboles mejorados inician más temprano y terminan más tarde su crecimiento en el año, exponiéndose a temperaturas bajas o deficiencias de humedad en el ambiente de reintroducción (Gómez et al., 1998).
Un programa de mejora genética inicia con la selección de árboles superiores con base en su fenotipo, por lo que es esencial determinar la calidad de estos árboles a través de la evaluación del crecimiento de su progenie en los terrenos donde se establecen las plantaciones (White et al., 2007). Sin embargo, cuando los ensayos de evaluación se realizan en varias localidades se genera un comportamiento diferencial entre las progenies de diferentes árboles, según el ambiente particular, conocido como interacción genotipo X ambiente (White et al., 2007). Debido a la longevidad y tasa de crecimiento de los árboles se requieren muchos años para la evaluación y para acelerar el proceso se usa la selección temprana, esto es, seleccionar genotipos con base en una característica manifestada a edad joven con el fin de mejorar otra en edades posteriores (Vargas y Adams, 1992).
Pinus patula se usa intensivamente en plantaciones con árboles mejorados en países de África y América, pero en su área de origen se aprovecha sólo del bosque natural y hay esfuerzos aislados como el establecimiento de áreas y huertos semilleros (Sáenz et al., 1994; Rebolledo et al., 1999; Valencia y Vargas, 2001). En Sudáfrica y Colombia, a través de la cooperativa CAMCORE (Programa Internacional para el Mejoramiento Genético y Conservación de Especies Forestales) se establecieron ensayos de progenies y se seleccionaron individuos con crecimiento rápido y forma superior (Dvorak et al., 2000; CAMCORE, 2007). Es posible utilizar árboles mejorados reintroduciéndolos a su lugar de origen en áreas donde el bosque original está deteriorado o ya desapareció. Para esto se debe conocer el desempeño de los árboles reintroducidos, comparándolos con árboles generados de semillas recolectadas en el bosque natural del mismo sitio u otros similares. Además, con evaluaciones a edades tempranas se puede dejar en pie los mejores individuos de las mejores familias, raleando los inferiores para convertir el estudio en un área de producción de semillas (huerto semillero sexual).
Para evaluar el desempeño de árboles mejorados fuera de su hábitat natural y reintroducidos a sus condiciones naturales, se establecieron dos ensayos de progenie de P. patula en diferente elevación. Otros objetivos fueron comparar el desarrollo de dos grupos de árboles entre localidades con ambientes diferentes y estimar algunos parámetros genéticos útiles en un programa inicial de mejora genética de la especie.
MATERIALES Y MÉTODOS
Se usaron dos grupos de árboles con diferente niveles de selección genética, cada uno con 36 familias de polinización libre; un grupo mejorado (M) por medio de selección en ensayos de progenie establecidos fuera de México y otro, de árboles sin mejora genética, seleccionados en campo por su fenotipo superior (S). Los árboles mejorados provienen de Colombia (18 familias) y de Sudáfrica (18 familias), de los miembros de la cooperativa CAMCORE seleccionados por su crecimiento en volumen, adaptación y rectitud de fuste. Los árboles del grupo S fueron seleccionados por el método comparativo en rodales naturales en los estados de Hidalgo, Veracruz y Puebla (Cuadro 1), considerando su mayor talla, rectitud de fuste y sanidad. En ambos sitios se usó un diseño experimental de bloques completos al azar con 17 (en S1) y 6 repeticiones (en S2), con parcelas de un solo árbol por familia. Se usó un arreglo en parcelas divididas con los grupos (M y S) en las parcelas grandes y las familias en las chicas.
Variables evaluadas
Se determinó la supervivencia acumulada de los árboles desde 2005 a 2010 considerando la mortalidad por heladas (evaluada a fines de febrero en cada año) y por sequía (en mayo al inicio del periodo de lluvias). En febrero del 2009 y 2010 (a los 5 y 6 años de edad desde la siembra de la semilla) se determinó: el diámetro del tallo en cm (DN5 y DN6) medido a 1.3 m de altura con un vernier digital y la altura total del árbol en m (ALT5 y ALT6) medida con un estadal graduado. Con estos datos se estimó el volumen del fuste en dm3 a las dos edades (VOL5 y VOL6) para lo cual se usó la ecuación de volumen para fuste total con corteza de P. patula desarrollada por Carrillo et al. (2004):
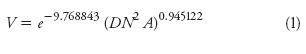
donde V es volumen (dm3), e es la función exponencial, DNes el diámetro del tallo a 1.3 m de altura y A es la altura total del árbol.
Análisis de datos
El análisis de la varianza para los dos sitios en conjunto y para cada sitio se realizó con el procedimiento MIXTO de SAS, obteniéndose los componentes de varianza a través del método REML (SAS Institute, 2002). Se consideró el efecto de familias anidado en grupos de mejora.
El modelo lineal utilizado para el análisis de los ensayos en conjunto fue:

donde Yijkl es el valor observado del individuo de la k-ésima familia, dentro del j-ésimo grupo de mejora, en el i-ésimo bloque anidado en el l-ésimo sitio; μ es la media poblacional; Sl es el efecto fijo del l-ésimo sitio; βi(l) es el efecto aleatorio de bloque anidado en sitio ~ NID (0, σ2bs)) ; Gj es el efecto fijo del j-ésimo grupo (selecto vs. mejorado); SGjl es el efecto fijo de la interacción sitio por grupo de mejora; βGijl es el efecto aleatorio de la interacción bloque por grupo ~ NID(0,σ2bg) ; Fk(j) es el efecto aleatorio de la k-ésima familia anidada en grupo ~ NID (0,σ2f(g)); SFjkl es el efecto aleatorio de la interacción sitio por familia dentro de grupo de mejora ~ NID (0,σ2sfg);y εijkl es el error asociado a dichos efectos ~ NID(0,σ2e), l = Aquixtla y Acaxochitlán; i = 1,2,...,17 o 1,2,...,6 bloques; j = mejorados y selectos; k = 1,2,...,36 familias por grupo de mejora.
Para el análisis de cada sitio se utilizó el mismo modelo general, eliminando todos los términos con efecto de sitios.

Para comparar el comportamiento dentro de cada grupo de mejora, en la estimación de heredabilidad y correlación genética los análisis se hicieron eliminando el efecto de grupos de mejora de las ecuaciones 2 y 3. Debido a la heterogeneidad de varianzas entre sitios y bloques, las variables se estandarizaron dividiendo cada observación entre la raíz cuadrada de su respectiva varianza total para cada combinación sitio-bloque (Hodge etal., 1996).
Correlaciones genéticas y fenotípicas
Las correlaciones fenotípicas y genéticas entre DN, ALT y VOL se calcularon a la misma edad y entre las dos edades. Las correlaciones fenotípicas entre cada par de variables se estimaron con base en el coeficiente de correlación de Pearson, usando las variables estandarizadas. Las correlaciones genéticas (tipo A)
se calcularon con la siguiente ecuación (Falconer y Mackay,1996):

donde rgxy es la correlación genética entre x e y evaluada sobre un mismo individuo, σfxy es la covarianza entre Χ e y, σfx y σfy son las desviaciones estándar de familias de las variables x e y. La covarianza σfxy se estimó con la siguiente ecuación (White y Hodge, 1989):

donde σ2f ( x+y ) es la varianza de familias de la variable X + Y, σ2fx es la varianza de familias de la variable X, σ2fy es la varianza de familias de la variable Y.
El error estándar de las correlaciones genéticas (EE(rg) ) se estimó con la siguiente ecuación (Falconer y Mackay, 1996):

donde r2gxy es la correlación genética entre las variables x e y, EE (h2x) y EE (h2y) son los errores estándar de las heredabilidades individuales de la variable x e y, hx2 y hy2 son las heredabilidades de las variables x e y.
Estimación de heredabilidades
En especies forestales la correlación intraclase de hermanos obtenidos por polinización libre en general es mayor de V (Squillace, 1974) lo cual genera una sobreestimación de la varianza aditiva (σ2A) y por tanto de la heredabilidad (Namkoong, 1966). Por lo anterior, se empleó un coeficiente de determinación genética de 3 para calcular la varianza genética aditiva, suponiendo que la correlación genética entre hermanos es 0.33. Con las varianzas estimadas para cada característica se calculó la heredabilidad en sentido estricto a nivel individual y de medias de familias por sitio individual y en conjunto (Falconer y Mackay, 1996).
Sitios individuales:

Sitios en conjunto:

donde hi2 es la heredabilidad individual, h2f es la heredabilidad de medias de familias, σ2f(g) es la varianza de familias, σ2sf(g)es la varianza de la interacción sitio-familia anidada en grupos de mejora, σ2e es la varianza residual, b es la media armónica del número de repeticiones (parcelas) existentes para cada familia, calculada con la siguiente ecuación:

donde x1, x2 y xn indican las observaciones de cada variable.
El error estándar de la heredabilidad individual (EE (hi2 )) se calculó con la ecuación de Dickerson (1969), que es un método conservador de estimación:

El coeficiente de variación genética (CVg) y el coeficiente de variación ambiental (CVa), se calcularon con las siguientes ecuaciones:

donde σ2g es varianza genética, σ2a es la varianza ambiental y X es la media general.
Correlaciones Tipo B
Para medir la interacción genotipo X ambiente entre los sitios se estimó la correlación genética Tipo B (rB) (Yamada, 1962), a nivel de grupos de mejora y de familia en conjunto sin separar grupos de mejora:

donde σ2f(g)es varianza de familias, σ2 sf(g)es la varianza estimada de la interacción sitio-familia anidada en grupos de mejora.
RESULTADOS Y DISCUSIÓN
Supervivencia de los árboles
En el sitio 1 la supervivencia fue 72.7 %, mientras que en el sitio 2 alcanzó 90.0 % a los 6 años de edad. En el sitio 1, el de mayor elevación (Aquixtla, 2930 m), hubo mortalidad mayor por temperaturas bajas y casi el doble que en el sitio 2 (10.9 vs. 5.8 %), de menor elevación (Acaxochitlán, 2200 m). La mitad de la mortalidad por heladas en el sitio 1 y la mayor parte de los daños en el sitio 2 ocurrieron durante el primer invierno (evaluación hacia fines de febrero 2005). Además, en el sitio 1 hubo mayor porcentaje de plantas muertas debido a la falta de humedad en el suelo, el triple de lo registrado en el sitio 2 (16.4 vs. 4.4 %). El año 2005 fue muy seco en Aquixtla, con un inicio de lluvias muy tardío en la primavera. En el sitio 1 no hubo diferencias de mortalidad entre los grupos M (27.2 %) y S (27.4 %) pero en el sitio 2 fue notoria la diferencia entre el grupo M (15.8 %) y el S (4.2 %) (Figura 1). Las heladas ocasionaron mortalidades de 11.4 y 8.9 % en las familias del grupo M, y de 10.4 y 2.8 % en las familias del grupo S, en los sitios 1 y 2. Pero las sequías ocasionaron pérdidas de 15.8 y 6.9 % en las familias del grupo M, y de 17.1 y 1.4 % en las familias del grupo S, en los sitios 1 y 2.
No hay registros climáticos para los sitios durante el periodo de evaluación, pero los datos históricos muestran que la temperatura media en S1 fue 15.6 °C y 15.1 °C en S2 (Servicio Meteorológico Nacional, 1971-2000). La precipitación total anual fue 718 mm en S1 y 920 mm en S2. Estos factores climáticos son condicionantes importantes que influyen sobre la mortalidad de los árboles en las plantaciones y también afectan los lugares de origen de cada familias, dado que provienen de una área geográfica amplia (Cuadro 1) y se encuentran adaptadas a condiciones ambientales diferentes a las de los sitios donde se establecieron los ensayos.
Análisis de varianza para las variables de crecimiento
Debido a las diferencias entre sitios y a la significancia de sus interacciones con grupos y familias, se realizó el análisis separado de cada sitio (Cuadro 2). En el análisis por grupos de mejoras para sitios en conjunto, sólo VOL5 presentó significancia.

El análisis de varianza para el sitio 1 (Aquixtla) mostró diferencias significativas (p≤0.05) en todas las variables de crecimiento para los factores grupos y familias dentro de grupos. Además hubo diferencias significativas para el sitio 2. En el análisis por sitio se encontraron diferencias significativas entre los dos grupos de mejora en todas las variables en ambos años (Cuadro 2). Sin embargo, en el análisis conjunto hubo diferencias significativas entre los grupos sólo en VOL5 debido a que el grupo M fue superior en el lugar con elevación mayor, mientras que el grupo S presentó crecimiento mayor en el sitio de elevación menor durante toda la evaluación.
Control genético de las características de crecimiento
El control genético de las características evaluadas en el sitio 1 varió de 0.09 para ALT5 hasta 0.12 para VOL5 y VOL6 y en el sitio 2 fue desde 0.26 hasta 0.35 (Cuadro 3), considerados como una heredabilidad moderada (Stanfield, 1971). Los valores de hi2 en el sitio 1 se asemejan a los obtenidos por Valencia etal. (1995), con h2i de 0.09, 0.14 y 0.15 para DN, ALT y VOL, en un ensayo de P. patula de seis años. Pero los valores de h2f , de 0.17 a 0.28 fueron menores a los obtenidos por dichos autores ( h2f ≤0.55) para características de crecimiento similares.
En un ensayo de P. patula de ocho años en Kenya los valores de h2f fueron 0.69, 0.61 y 0.70 para DN, ALT y VOL (Kariuki, 1998), mientras que en Colombia hi2fue 0.31 y 0.71 y h2f 0.59 y 0.84 para altura y volumen con un coeficiente de determinación de 0.4 para P. patula de siete años (Ladrach y Lambeth, 1991). De acuerdo con Stanfield (1971), los caracteres se consideran de heredabilidad alta cuando hi2 es mayor que 0.50, media entre 0.50 y 0.20, y baja si hi2 <0.20. Sin embargo, la heredabilidad de medias de familia depende del número promedio de individuos por familia y de la supervivencia en los ensayos, pues a mayor tamaño promedio de familias, mayor h2f (White etal., 2007).
En cada grupo de familias también se encontró control genético mayor en todas las características en el sitio 2 (Cuadro 4), aunque tuvo número menor de repeticiones. Esto se puede deber a una variabilidad ambiental menor porque en este sitio se realizó mejor control de la maleza, es más uniforme y casi plano y tuvo supervivencia mayor. En Aquixtla no se hizo un control adecuado de la maleza y hay afloramientos rocosos, lo cual aumenta la variabilidad dentro y entre bloques, y reduce la probalidad de encontrar variación genética significativa al aumentar la varianza ambiental. El manejo cultural mejora la estimación de los parámetros genéticos, aumentando sus valores (López et al., 1999). Además, la tasa de mortalidad relativamente alta pudo aumentar la heterogeneidad ambiental, provocando en algunos individuos crecimiento mayor al que hubiera ocurrido si todos los árboles tuvieran un ambiente de competencia similar. Las familias del grupo M presentaron mayores valores de heredabilidad en el sitio Aquixtla, como se esperaba, porque el efecto del mejoramiento es aumentar la uniformidad dentro de las familias (White et al., 2007). Sin embargo, esto no ocurrió en el sitio Acaxochitlán donde el control genético estimado fue mayor en las familias del grupo S.
En el análisis conjunto, el control genético de las características de crecimiento fue muy bajo, tanto para las familias mejoradas como para las selectas (Cuadro 5), lo cual significa que hay un importante efecto de la interacción genotipo X ambiente entre los dos sitios, que aumentó la variación ambiental. Los valores de hi2 estimados en análisis conjunto de sitios son siempre menores a los estimados a partir de análisis de sitios individuales, por lo que se les denomina heredabilidades insesgadas (White et al., 2007).
Coeficientes de variación genética (CVg) y ambiental (CVa)
Los valores de los CVg por sitio y por grupo de familias fueron bajos para todas las características analizadas, por lo cual la relación CVgICVa fue menor o igual a 0.36 en todos los casos (Cuadro 6). Según Vencovsky (1987), hay mayor ganancia por selección cuando la relación CVgICVa tiende a uno porque la variación genética es alta. En forma conjunta, el CVg y la heredabilidad dan una idea del potencial para efectuar una posible selección en los genotipos analizados. Las familias del grupo M tuvieron una mayor relación CVgICVa que las del grupo S.

Correlaciones genéticas y correlaciones fenotípicas entre características a los 5 y 6 años de edad de los árboles
Las correlaciones genéticas (rg) estimadas entre las características de crecimiento para el sitio 1 fueron positivas y entre 0.80 y 1.0. Las correlaciones fenotípicas (rp) variaron de 0.76 a 0.94. Para el sitio 2, las rg fluctuaron de 0.87 a 1.0 y las rp oscilaron entre 0.82 y 0.95 (Cuadro 7).
El comportamiento de rg y rp fue similar en ambos sitios; en todos los casos, al realizar la selección de un carácter se afectaría positivamente el comportamiento de otro carácter. El valor alto de la rg estimada entre todas las características se puede atribuir a genes comunes que influyen en ambas características (Falconer y Mackay, 1996). En P. patula hay valores de rg≥0.89 y rp≥0.78 para diámetro, altura y volumen en plantaciones con seis años de edad (Valencia y Vargas, 2001). Como señalan Farfán et al. (2002), en P. ayacahuite Ehren. ex Schltdl. var. ayacahuite se puede usar el diámetro normal para mejorar la altura y el volumen, incluso un año antes, debido a los valores altos de la rg. Las correlaciones (rgy rp ) entre las características entre las dos edades son cercanas a 1 (Cuadro 7), esto es, al seleccionar un carácter a los cinco años también se selecciona alta y positivamente a los seis años, por lo cual es posible acortar el tiempo para la selección de las mejores familias. La correlación fenotípica señala que la combinación de correlaciones debidas a efectos genéticos y ambientales comunes es alta.
Correlaciones genéticas Tipo B
La interacción genotipo X ambiente fue alta porque los valores de rs en ambas edades son bajos (White et al., 2007). Para todas las características el grupo de familias M presentó valores de correlación genética Tipo B mayores respecto a las familias del grupo S (Cuadro 8), lo cual implica que el grupo M es una colección de familias más estables al plantarse en diferentes ambientes, lo que puede ser producto del mejoramiento. El grupo de progenies de las familias S es propiamente de árboles silvestres muy inestables al cambiar las condiciones ambientales, así, la determinación de los genotipos superiores para plantaciones comerciales dependerá del ambiente de establecimiento. Según, Molina (1992), el común denominador de todos los programas de evaluación de genotipos es su comportamiento inestable en los diferentes ambientes de prueba; los resultados pueden discrepar de lo esperado; y difícil y dudoso tomar una decisión respecto a cuál es el mejor genotipo.
Implicaciones para el mejoramiento genético
La utilización de dos ambientes disímbolos para establecer los ensayos de evaluación provocó una fuerte interacción genotipo X ambiente en el comportamiento de los grupos de familias M y S. La mortalidad de los árboles, la heterogeneidad del ambiente y el manejo relativamente diferente en los dos sitios contribuyeron a ello. También influyó que las familias en ambos grupos (M y S) vienen de una región geográfica muy amplia y diversa (Cuadro 1) y es evidente que tienen un comportamiento muy distinto en estos ambientes contrastantes.
El sitio de menor elevación (Acaxochitlán, Hidalgo) presentó un ambiente más favorable, con mayor supervivencia y con el doble de crecimiento en volumen del fuste a los seis años, comparado con el sitio de mayor elevación (Aquixtla, Puebla). En Aquixtla, la mortalidad entre grupos de familia fue similar y cercana a 11 %, mientras que en Acaxochitlán el grupo M tuvo tres veces más fallas que el grupo de progenies de las familias S.
El grupo de familias M fue una colección de familias más estable al plantarse en diferentes ambientes, lo cual se puede deber a su ciclo de mejoramiento. En cambio, el grupo de familias S consta de árboles silvestres, inestables al modificar las condiciones ambientales del sitio de evaluación, por lo que la determinación de los genotipos superiores para usar como fuentes de germoplasma en plantaciones comerciales, dependerá del ambiente de establecimiento.
CONCLUSIONES
En el sitio de elevación baja, la supervivencia y crecimiento de los árboles fueron superiores a los del sitio a mayor altitud. En el primero hubo mayor mortalidad por heladas y menor crecimiento en el material mejorado que en las familias selectas. En el sitio alto la mortalidad fue similar, pero el grupo mejorado presentó crecimiento superior en diámetro normal, altura y volumen.
Las heredabilidades individual y de medias de familia para las variables de crecimiento en el sitio elevado son menores comparadas con las del sitio bajo, lo cual en parte se atribuye a la diferencia en el manejo cultural dentro de cada plantación. Los árboles mejorados presentaron mayores valores de heredabilidad en el sitio alto pero en el sitio bajo los valores fueron de moderados a altos en ambos grupos de mejora a pesar del menor número de repeticiones. El comportamiento de rg y rp en ambos sitios fue similar, con valores adecuados para tomar el mejor criterio de selección por pares de características. En todos los casos, al realizar la selección de un carácter se afectaría positivamente el comportamiento de otro carácter. Las correlaciones tipo B (rb ) fueron bajas, es decir, hay interacción genotipo X ambiente y son más interactivas las familias selectas. La inconsistencia en los resultados puede tener su origen en el grado de diversidad de los genotipos o de los ambientes.
AGRADECIMIENTOS
Al Fondo CONAFOR-CONACYT, 2003-C03-10714 por el apoyo al proyecto "Establecimiento de huertos semilleros con material genéticamente superior de Pinus patula y P. greggii", al Fideicomiso 2009 y a la Línea Prioritaria de Investigación No. 1 del COLPOS. A los Ingenieros León Jorge Castaños Martínez y Salvador Castro Zavala. A Wiliam S. Dvorak, Director, y a Juan López, Supervisor Técnico, de CAMCORE, y a Smurfit Kappa Cartón de Colombia en Colombia, y Sappi en Sudáfrica, por la donación de la semilla.
LITERATURA CITADA
CAMCORE (Programa Internacional para el Mejoramiento Genético y Conservación de Especies Forestales). 2007. Boletín de noticias CAMCORE para México y Centroamérica. Vol. 1 No. 2. Raleigh. 6 p. [ Links ]
Carrillo A., F., M. Acosta M., G. Tenorio G., y F. Becerra L. 2004. Tabla de volumen para Pinus patula Schl. et Cham. en el estado de Hidalgo. Folleto Técnico No. 2. Centro de Investigación Regional del Centro, Campo Experimental Pachuca. INIFAP. 16 p. [ Links ]
Dickerson, G. E. 1969. Techniques for research in quantitative animal genetics. In: Techniques and Procedures in Animal Science Research. Am. Soc. Anim. Res. Albany. pp: 36-79. [ Links ]
Dvorak, W. S., G. R. Hodge, J. E. Kietzka, F. Malan, L. F. Osorio, and T. K. Stanger. 2000. Pinus patula. In: Conservation & Testing of Subtropical & Subtropical Forest Tree Species by the CAMCORE Cooperative College of Natural Resources, NCSU. Raleigh. pp: 149-173. [ Links ]
Falconer, D. S., and T. F. C. Mackay. 1996. Introduction to Quantitative Genetics. Addison Wesley Longman Limited, Edinburgh. 484 p. [ Links ]
Farfán V., E. G, J. Jasso M., J. López Upton, J. J. Vargas Hernández, y C. Ramírez Herrera. 2002. Parámetro genéticos y eficiencia de la selección temprana en Pinus ayacahuite Ehren. var. ayacahuite. Revista Fitotec. Mex. 25: 239-246. [ Links ]
Gómez, C., M., J. J. Vargas Hernández, J. Jasso M., A. Velázquez M,. y C. Rodríguez F. 1998. Patrón de crecimiento anual del brote terminal en árboles jóvenes de Pinus patula. Agrociencia 32: 357-364. [ Links ]
Hodge, G. R., P. W. Volker, B. M. Potts, and J. U. Owen. 1996. A comparison of genetics information from open-pollinated and control-pollinated progeny tests in two eucalypt species. Theor. Appl. Genet. 92: 53-63. [ Links ]
INEGI. 2009. Prontuario de información geográfica municipal de los Estados Unidos Mexicanos. Acaxochitlán, Hidalgo. Clave geoestadística 13002. s.p. [ Links ]
Kariuki, J. G. 1998. Provenance and family-within-provenance variation in Pinus patula, Pinus patula subspecie tecunumanii and Pinus oocarpa planted at Turbo, Kenya. For. Ecol. Manage. 107: 127-133. [ Links ]
Ladrach, W. E., and C. Lambeth. 1991. Growth and heritability estimates for a seven-year-old open-pollinated Pinus patula progeny test in Colombia. Silvae Genetica 40: 169-173. [ Links ]
López U., J., T. L. White, and D. A. Huber. 1999. Effects of site and intensive culture on family differences in early growth and rust incidence of loblolly and slash pine. Silvae Genetica 48: 284-293. [ Links ]
Molina G., J. D. 1992. Introducción a la Genética de Poblaciones y Cuantitativa (Algunas Implicaciones en Genotecnia). AGT Editor. México. 349 p. [ Links ]
Namkoong, G. 1966. Inbreeding effects on estimation of genetic additive variance. For. Sci. 12: 8-13. [ Links ]
Rebolledo C., V., L. del C. Mendizábal H. y J. Alba L. 1999. Evaluación de familias comunes de Pinus patula Schl. et Cham. en dos ensayos de progenie. Foresta Veracruzana 1: 19-24. [ Links ]
Sáenz R., C., H. Nienstaedt, and J. J. Vargas H. 1994. Performance of Pinus patula genotypes selected in South Africa and growing in their native Mexican environment. Silvae Genetica 43: 73-81. [ Links ]
SAS (Statistical Analysis System) Institute. 2002. SAS/STAT Computer Software. Release 9.00. SAS Institute Inc. Cary. [ Links ]
Squillace, A. E. 1974. Average genetic correlations among offspring from open-pollinated forest trees. Silvae Genetica 23: 149-156. [ Links ]
Stanfield, W. D. 1971. Genética. Teoría y 400 Problemas Resueltos. Serie Schaum. McGraw Hill. México. 405 p. [ Links ]
Valencia M., S., y J. J. Vargas H. 2001. Correlaciones genéticas y selección simultanea del crecimiento y densidad de la madera en Pinus patula. Agrociencia 35: 109-120. [ Links ]
Valencia M., S., C. Ramírez H., y J. J. Vargas H. 1995. Comparación de diferentes métodos de selección en un ensayo de progenie de polinización libre de Pinus patula. In: Salazar, R. (ed). Avances en la producción de semillas forestales en América Latina: memoria del simposio. Managua. pp: 63-69. [ Links ]
Vargas H., J. J., and W. T. Adams. 1992. Age-age correlation and early selection for wood density in young coastal Douglas-Fir. For. Sci. 38: 467-477. [ Links ]
Vencovsky, R. 1987. Herança quantitativa. In: Paterniani, E. (ed). Melhoramento e Produçâo do Milho no Brasil. Ediçâo da Fundaçâo Cargill. Instituto de Genética, Esc. Sup. Agricultura Luis de Queiroz, Universidad de Säo Paulo. Piracicaba. Säo Paulo. pp: 122-199. [ Links ]
White, T. L., and G. R. Hodge. 1989. Predicting Breeding Values with Applications in Forest Tree Improvement. Kluwer Academic Publishers. The Netherlands. 368 p. [ Links ]
White, T. L., W. T. Adams and D. B. Neale. 2007. Forest Genetics. CABI. Oxfordshire. 682 p. [ Links ]
Wormald, P. J. 1975. Pinus patula. Tropical Forestry Paper No. 7. Tropical Forestry Institute. Commonwealth Forestry Institute. Oxford, England. 172 p. [ Links ]
Wright, J. A., L. F. Osorio, and W. S. Dvorak. 1995. Recent developments in a tree improvement program with Pinus patula in Colombia. For. Ecol. Manage. 72: 229-234. [ Links ]
Yamada, Y. 1962. Genotype by environment interaction and genetic correlation of the same trait under different environments. Japan J. Genet. 37: 498-509. [ Links ]














