Serviços Personalizados
Journal
Artigo
Indicadores
Links relacionados
-
 Similares em
SciELO
Similares em
SciELO
Compartilhar
Agrociencia
versão On-line ISSN 2521-9766versão impressa ISSN 1405-3195
Agrociencia vol.44 no.6 Texcoco Ago./Set. 2010
Recursos naturales renovables
Variación isoenzimática de Pinus hartwegii Lindl. en un gradiente altitudinal en Michoacán, México
Isoenzymatic variation of Pinus hartwegii Lindl. along an altitudinal gradient in Michoacan, Mexico
Héctor Viveros–Viveros1,2, Blanca L. Tapia–Olivares3, Cuauhtémoc Sáenz–Romero3*, J. Jesús Vargas–Hernández1, Javier López–Upton1, Amalio Santacruz–Varela4, Gustavo Ramírez–Valverde5
1 Forestal, Campus Montecillo, Colegio de Postgraduados. 56230. Montecillo, Estado de México.
2 Instituto de Investigaciones Forestales, Universidad Veracruzana. Parque Ecológico El Haya, Colonia Benito Juárez, 91070. Xalapa, Veracruz.
3 Instituto de Investigaciones Agropecuarias y Forestales, Universidad Michoacana de San Nicolás de Hidalgo (IIAF–UMSNH). Km 9.5 Carretera Morelia–Zinapécuaro, Tarímbaro, Michoacan. 58880. * Autor responsable: (csaenzromero@gmail.com).
4 Genética, Campus Montecillo, Colegio de Postgraduados. 56230. Montecillo, Estado de México.
5 Estadística, Campus Montecillo, Colegio de Postgraduados. 56230. Montecillo, Estado de México.
Recibido: Noviembre, 2009.
Aprobado: Julio, 2010.
RESUMEN
Para conocer la variación genética en un gradiente altitudinal y generar lineamientos para la conservación de Pinus hartwegii Lindl., se investigó la variación genética isoenzimática entre poblaciones de esta especie a diferentes altitudes en el Parque Nacional Pico de Tancítaro, Michoacán, México. Se recolectaron semillas de cuatro localidades ubicadas a 3000, 3200, 3400 y 3600 m de altitud. Se encontró polimorfismo en 11 de los 12 loci examinados. La heterocigosidad esperada promedio (He) fue 0.12. En tres loci existió desequilibrio de Hardy–Weinberg (p<0.001), con un déficit de heterocigotos. La diferenciación genética entre poblaciones fue significativa (FST=0.111). La distancia genética promedio (0.108) fue baja, y el flujo génico (Nm=2.0) fue relativamente bajo. El agrupamiento de las poblaciones separa a la población de 3600 m del resto (a 3400 m) (p<0.05). Se sugiere establecer una Unidad de Conservación de Recursos Genéticos Forestales (UCRGF) a 3600 m de altitud y un tamaño mínimo de población viable (Ne) de 4157 individuos, y otra entre los 3000 y los 3350 m de altitud y un Ne de 3055 árboles.
Palabras clave: Pinus hartwegii, conservación de recursos genéticos, diferenciación genética, flujo génico, isoenzimas, variación genética.
ABSTRACT
To determine the genetic variation along an altitudinal gradient and generate guidelines for the conservation of Pinus hartwegii Lindl., genetic isoenzymatic variation was studied in populations of the species at different altitudes in Pico de Tancítaro National Park, Michoacán, México. Seeds were collected from four locations at altitudes of 3000, 3200, 3400 and 3600 m. Polymorphism was found in 11 of the 12 loci examined. The expected average heterozygosity (He) was 0.12. At three loci there was Hardy–Weinberg non equilibrium (p<0.05), with a deficit of heterozygotes. Genetic differentiation among populations was significant (FST=0.111). Average genetic distance (0.108) was low, and gene flow was relatively low (Nm=2.0). Grouping populations separated the 3600 m population from the other three (3000 to 3400 m) (p<0.05). It is recommended that a forest genetic resource conservation unit be established at an altitude of 3600 m with a minimum viable population size (Ne) of 4157 individuals, while another should be established between 3000 and 3350 m with Ne of 3055 trees.
Key words: Pinus hartwegii, genetic resource conservation, genetic differentiation, gene flow, isoenzymes, genetic variation.
INTRODUCCIÓN
Pinus hartwegii Lindl. es una especie confinada a las montañas más altas de México y de América Central, y crece entre 3000 a 4000 m de altitud (Perry, 1991). Es una especie de gran importancia por ser uno de los componentes principales de los bosques de las montañas altas en México (Lauer, 1978). Para México se calcula un aumento de la temperatura promedio anual de 1.5 °C para el 2030, 2.3 °C en el 2060 y 3.7 °C en el 2090, y un decremento de la precipitación anual de 6.7%, 9.0% y 18.2% (Sáenz–Romero et al., 2009); además, una disminución del hábitat de P. hartwegii hasta en 42% (Arriaga y Gómez, 2004). El aumento de la temperatura en los 100 años recientes ha causado una reducción de 10.6% en el crecimiento relativo de la especie (Ricker et al., 2007).
El primer paso de una estrategia de conservación de la diversidad genética es evaluar el estatus y la estructura genética de la especie (Yeh, 2000). Las isoenzimas son los marcadores moleculares más usados para ello en especies forestales (El–Kassaby y Ritland, 1996). A partir de la heterocigosidad estimada con isoenzimas, es posible establecer el tamaño mínimo de población genéticamente viable, lo que permite determinar el tamaño necesario de Unidades de Conservación de Recursos Genéticos Forestales (Sáenz–Romero et al. , 2003).
En caracteres cuantitativos con valor adaptativo se ha encontrado un patrón clinal bien definido de variación altitudinal entre poblaciones de P. hartwegii en México (Viveros–Viveros et al., 2009). En dos estudios isoenzimáticos en P. hartwegii en México se examinó la variación entre y dentro de poblaciones (Solís–Ramos e Iglesias–Andreu, 2001; Iglesias–Andreu y Luna–Rodríguez, 2008), pero no se ha investigado la variación a través de un gradiente altitudinal. En otras especies forestales, usando marcadores neutrales a la selección se ha determinado que es más común encontrar algún patrón de variación asociado a la altitud que la ausencia de un patrón (Ohsawa e Ide, 2007).
La hipótesis del presente trabajo fue que probablemente hay un patrón de variación genética altitudinal, donde las poblaciones de la parte central de la distribución altitudinal de P. hartwegii sean las que tengan mayor diversidad genética, mientras las de los extremos altitudinales superior e inferior tengan menor diversidad, ya que ése es el patrón más común (Ohsawa e Ide, 2007).
Los objetivos de esta investigación fueron: 1 ) cuantificar mediante isoenzimas la diversidad genética en poblaciones de P. hartwegii distribuidas en un gradiente altitudinal del estado de Michoacán; 2) determinar la existencia de variación asociada con el gradiente altitudinal en cuanto a parámetros de diversidad genética; 3) estimar el tamaño de población genéticamente viable necesario para establecer Unidades de Conservación de Recursos Genéticos Forestales.
MATERIALES Y MÉTODOS
Material genético
En enero de 2005 se recolectó semilla de polinización libre de P. hartwegii en cuatro poblaciones en un gradiente altitudinal (3000, 3200, 3400 y 3600 m) en el Parque Nacional Pico de Tancítaro, estado de Michoacán (Cuadro 1). La definición de población fue relativamente arbitraria: un grupo de individuos en una condición de rodal relativamente homogénea, distribuidos en un rango altitudinal de ±20 m desde el punto altitudinal designado (3000, 3200, 3400 y 3600 m); esta categoría podría ser considerada por algunos como subpoblación. Las poblaciones mostraron una misma exposición (noreste). En cada población se seleccionaron al azar 19 a 30 árboles, separados al menos 30 m entre ellos. De cada árbol se recolectaron tres a cinco conos y se identificaron por árbol y por población.
Electroforesis de isoenzimas
Se realizó el análisis de electroforesis de isoenzimas en geles de almidón. Se evaluaron 12 loci en total; en ocho loci (ACP, G6P, MDH–1, MDH–2, PGI–1, PGI–2, 6PG–1 y 6PG–2) se usó la metodología de Yamada y Guries (1989), y en cuatro (EST, GOT, LAP y PGM) la de Soltis et al. (1983). Los loci de un mismo sistema isoenzimático se denominaron como locus 1 al de mayor movilidad y 2 al de menor movilidad. Para cada locus se denominó como 1 al alelo más común y para los demás alelos del mismo locus se asignó un número sucesivo según su movilidad, de mayor a menor. La interpretación de los patrones de bandeo se realizó usando como referencia la migración de muestras de Pinus resinosa Aiton, una especie monomórfica. Las muestras de P. hartwegii consistieron en seis megagametofitos por cada individuo; con ese tamaño de muestra, la probabilidad de error de una identificación de heterocigotos como homocigotos es: 2 × 0.56 =0.03 (Conkle, 1981).
Análisis estadístico
Las frecuencias alélicas, la heterocigosidad observada y esperada, los alelos raros, los alelos privados, la proporción de loci polimórficos (criterio <0.99) y las diferencias significativas entre la heterocigosidad observada y esperada se estimaron mediante el software Tools for Population Genetic Analysis (Miller, 1997). El número promedio de alelos por locus, y el número efectivo de alelos se obtuvieron mediante el programa POPGENE versión 1.31 (Yeh et al., 1999). Se estimó la endogamia total (FIT), la proporción de la diversidad genética total entre poblaciones (FST) y la endogamia dentro de poblaciones (FIS); los intervalos de confianza al 90% para los estadísticos de F de Wright se calcularon por la técnica de muestreo con reemplazo (bootstrappig) sobre los loci con 10,000 repeticiones (Miller, 1997). Con el intervalo de confianza para FST se determinó la existencia de diferenciación genética significativa entre poblaciones. Se estimó el flujo génico como el número de migrantes por generación (Nm) (Slatkin 1987):
Nm = ((1/FST)–1)/4
Para conocer el patrón de diferenciación genética entre poblaciones se estimaron las distancias genéticas entre pares de localidades mediante la distancia modificada de Rogers (Miller, 1997). Con base en la matriz de distancias genéticas, las poblaciones se agruparon usando el método de agrupamiento con promedios no ponderados (UPGMA) (Swofford y Olsen, 1990). Se comparó la frecuencia de alelos entre pares de poblaciones mediante la prueba de probabilidad combinada de Fisher (Miller, 1997). El tamaño efectivo de población genéticamente viable se calculó a partir del valor estimado de heterocigosidad esperada, usando una fórmula de regresión ( Sáenz–Romero et al., 2003), obtenida al ajustar un modelo de regresión lineal entre valores de tamaño efectivo de población viable (N ) y valores de heterocigosidad esperada (He) que se desea mantener, considerando una tasa de mutación de 1×10–5 con datos de Millar y Libby (1991) para varias especies de coníferas. La fórmula fue:
Ne = –984.58 + (36723 He)
Se evaluó el grado de asociación de la heterocigosidad esperada, la proporción de loci polimórficos y el número promedio de alelos por locus respecto a la elevación de las localidades usando los procedimientos CORR y RES (SAS Institute, 1988).
Se estimaron las distancias geográficas entre poblaciones con cartas topográficas (1:50 000, INEGI 1998); con éstas y las altitudinales se obtuvieron las distancias espaciales entre pares de poblaciones. Se estimó la correlación entre las distancias genéticas y las distancias espaciales mediante la prueba de Mantel (Miller, 1997).
RESULTADOS Y DISCUSIÓN
Diversidad genética dentro de poblaciones
Se encontraron 30 alelos en 12 loci muestreados. El número promedio de alelos por locus y de alelos efectivos por locus de las poblaciones se presentan en el Cuadro 2. El número de alelos por locus (2.5) fue ligeramente menor al de 2.8 al reportado por Solís–Ramos e Iglesias–Andreu (2001) en P. hartwegii y cercano al de 2.4 en P. oocarpa (Sáenz–Romero y Tapia–Olivares, 2003). El promedio de alelos por locus fue 1.9 a nivel de población, valor cercano al de 1.8 en P. pinceana Gordon (Ledig et al., 2001).
El análisis conjunto de todas las poblaciones mostró que 11 loci fueron polimórficos (91.7%) y un locus fue monomórfico (PGI–1). En P. hartwegii, Solís–Ramos e Iglesias–Andreu (2001) reportan 100% de loci polimórficos en esterasas (EST1, EST2, EST3, y EST4), mientras que Iglesias–Andreu y Luna–Rodríguez (2008) muestran 86% de loci polimórficos en la misma especie. El mayor número promedio de alelos por locus, el mayor nivel de polimorfismo y el mayor número de alelos privados (3 de un total de 5) se presentaron en la población de mayor altitud (Cuadro 2).
La heterocigosidad esperada (He) fue ligeramente mayor en las poblaciones extremas que en las intermedias del transecto altitudinal, con el valor máximo en la población de mayor altitud (Cuadro 2). La He promedio fue mayor que la Ho, con diferencias significativas entre ellas en tres de los loci muestreados (MDH1, 6PG2 y PGM), lo cual indica la ausencia de equilibrio de Hardy–Weinberg (Chi–cuadrada, p<0.0001) en ellos. Esto sugiere un ligero déficit de heterocigotos, debido posiblemente al efecto de la endogamia o a que las poblaciones en algún momento de su vida atravesaron por un cuello de botella (Le–dig et al., 1999; 2001). Algo similar se ha reportado para otras especies de pino con distribución natural restringida como P. maximartinezii Rzedowski (Ledig et al., 1999) y P. pinceana (Ledig et al., 2001).
Estructura de la diversidad genética
El valor medio del coeficiente de endogamia fue no significativamente diferente de cero, ya que el valor estimado está dentro del intervalo de confianza que incluye al cero (Cuadro 3). La no significancia de FIS sugiere que la mayoría de los loci están en equilibrio de Hardy–Weinberg. Sin embargo, el valor promedio de FIS es relativamente alto (FIS = 0.361), lo que coincide con el déficit de heterocigotos mencionado antes; ambos valores son indicadores de endogamia en estas poblaciones. En este sentido es probable que la amplitud de los intervalos de confianza refleje el número reducido de poblaciones muestreadas (cuatro) y no necesariamente de la existencia de equilibrio de Hardy–Weinberg. En forma similar, el valor estimado promedio de FIT a pesar de ser relativamente alto (0.438), no fue significativamente diferente de cero porque su intervalo de confianza incluye al cero (Cuadro 3). El valor de FIT indica que a nivel de todas las poblaciones también existen indicios de endogamia.

La diferenciación genética entre poblaciones (FIS=0.111) fue significativamente diferente de cero (el intervalo de confianza no incluye al cero; Cuadro 3). Valores de FST entre 0.051 y 0.150 indican una moderada diferenciación genética entre poblaciones (Yeh, 2000). En algunas especies de coníferas con distribución restringida o fragmentada como en P. greggii Engelm. (Parraguirre–Lezama et al., 2002), se han encontrado valores mayores de diferenciación genética entre poblaciones.
El número promedio de migrantes por generación (Nm) fue 2.002, valor considerado por Hartl y Clark (1989) suficientemente bajo como para que el flujo génico no pueda contrarrestar totalmente la diferenciación genética causada por la deriva génica. Respecto a otras coniferas, el valor encontrado es menor que la generalidad de valores reportados, que van de 4.6 a 17.2 (Ledig, 1998); sólo es cercano a lo estimado para especies con poblaciones reducidas o fragmentadas, como P. maximartinezii (Nm de 1.39 a 2.09) y P. pinceana (1.39 a 2.09)(Ledig et al, 2001).
Distancias genéticas
La distancia genética promedio fue 0.108, pero varió desde 0.042 entre las poblaciones de 3200 y 3400 m, hasta 0.166 entre las localidades extremas en el transecto altitudinal (Cuadro 4). Estos valores son mayores a los encontrados en otras coníferas en gradientes altitudinales, como en P. oocarpa (Sáenz–Romero y Tapia–Olivares, 2003), pero son menores a la de especies de latifoliadas en gradientes similares, como en Quercus aquifolioides Rehder & Wilson (Zhang et al., 2006).

La prueba de probabilidad combinada de Fisher mostró que la población de mayor altitud es significativamente diferente (p<0.05) de las otras estudiadas, lo que indica una mayor diferenciación genética de esta localidad. En el UPGMA se observa la separación de la población de 3600 m de las otras poblaciones (Figura 1). La mayor diversidad genética de la población de mayor altitud podría deberse a que sus individuos reciben flujo génico de árboles de otras exposiciones, particularmente de la ladera oeste, ya que la parte superior de la montaña es más estrecha y colinda con el parteaguas del volcán, lo que induciría una mayor diferenciación y diversidad genética de esa población en comparación con las de menores altitudes (ubicadas en la ladera este).
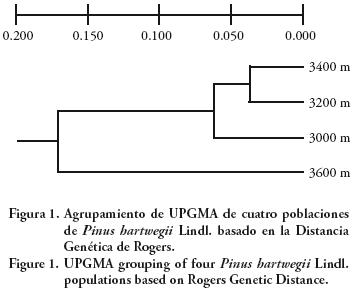
Otro elemento que contribuye a la diferenciación de la población de mayor altitud es el elevado número de alelos raros y privados. Ello se debería a los efectos de flujo génico o a los de la selección natural que favorecen la presencia de un mayor número de alelos en frecuencias bajas como un mecanismo de adaptación a ambientes más desfavorables (Kawecki, 2000; Schaberg et al., 2003). Zhang et al. (2006) muestran que el agrupamiento de cinco poblaciones de Q. aquifolioides separa la población de mayor altitud (3600 m) de las otras poblaciones (2000, 2400, 2800 y 3200 m).
Asociación entre variación genética y altitud de la población
La mayor diversidad genética mostrada por la heterocigosidad esperada, proporción de loci polimórficos, número de alelos por locus y número de alelos privados, se encontró en la población de mayor altitud. Este patrón altitudinal coincide con 24% de los casos analizados por Ohsawa e Ide (2007).
Estructura genética espacial
La prueba de Mantel indica que no hubo asociación significativa entre las distancias espaciales y las genéticas (R=0.0003; p=0.332) de las poblaciones. Por tanto no hubo un efecto de aislamiento genético por la distancia entre ellas.
Implicaciones para la conservación de la especie
En un estudio en el Pico de Tancítaro con las mismas poblaciones de P. hartwegii, basado en caracteres cuantitativos de valor adaptativo, se propuso crear dos Unidades de Conservación de Recursos Genéticos Forestales (UCRGF), una entre 3000 y 3350 m, y la segunda entre 3350 y 3700 m (Viveros–Viveros et al., 2009). Los resultados de la presente investigación apoyan dicha estrategia. En términos de variación isoenzimática, la población ubicada a 3600 m tiene el mayor nivel de diversidad genética, proporción de alelos privados, y diferenciación genética respecto a las poblaciones ubicadas a menor altitud. Para preservar una porción representativa de la diversidad genética actual, es recomendable establecer al menos una UCRGF a 3600 m, y otra a una altitud menor (3000 a 3350 m). Se estima que la UCRGF de mayor altitud deberá tener un tamaño mínimo de población efectiva viable (Ne) de 4157 individuos, y la de menor altitud una Ne de 3055 árboles.
Sin embargo, de continuar la tendencia actual de calentamiento global (Sáenz–Romero et al. , 2009), será necesario realizar una migración asistida hacia altitudes mayores en la misma montaña o en otras montañas. Es decir, recolectar semilla de las poblaciones actuales, producir planta en vivero y hacer plantaciones de conservación ex situ en un sitio ubicado aproximadamente a 400 m de mayor altitud, respecto al sitio en donde se recolectó la semilla. Esto permitiría reacoplar las poblaciones al clima para el cual están adaptadas, clima que ocurrirá en el año 2030 aproximadamente a 400 m de mayor altitud de donde ocurre actualmente, según estimaciones para la especie basadas en el Modelo Canadiense de Circulación Global, y el escenario de emisiones A2 (Sáenz–Romero et al., 2009). Las poblaciones de mayor altitud no tendrían a dónde ir en el Pico de Tancítaro, debido a que la altitud máxima es de 3800 m. Por ello, la migración asistida tendría que realizarse a otras montañas o volcanes del Eje Neovolcánico de mayor altitud, como el Iztaccíhuatl, Popocatépetl y Citlaltépetl (Pico de Orizaba). Se podría argumentar que las poblaciones locales de P. hartwegii en estos volcanes pudieran sufrir de depresión por exogamia, al recibir el flujo génico de las plantaciones con procedencias foráneas. Sin embargo, debe considerarse que las poblaciones locales también se verán gradualmente desadaptadas debido al cambio climático, y tal desacoplamiento seguramente será un riesgo mayor en el largo plazo.
CONCLUSIONES
Pinus hartwegii en el Parque Nacional Pico de Tancítaro, Michoacán, muestra una moderada, pero estadísticamente significativa, diferenciación genética entre poblaciones, fundamentalmente entre la de mayor altitud (3600 m) con las de 3000, 32000 y 3400 m. La población ubicada a 3600 m tiene la mayor diversidad genética y proporción de alelos privados.
Se propone el establecimiento de dos Unidades de Conservación de los Recursos Genéticos Forestales, una a 3600 m y otra entre 3000 y 3350 m de altitud, con un tamaño mínimo de población efectivo de 4157 y de 3055 individuos, a fin de preservar la mayor diversidad genética existente en estas poblaciones. Estas medidas de conservación deben acompañarse de estrategias de conservación ex situ, como la migración asistida altitudinalmente hacia arriba, sobre todo para la población de mayor altitud, para mitigar los posibles efectos negativos del calentamiento global. La población de 3600 m es la más vulnerable al cambio climático, ya que a pesar de contener la mayor diversidad genética, no podrían migrar hacia arriba en el Pico de Tancítaro cuya altitud máxima es 3800 m.
AGRADECIMIENTOS
Se agradece al Consejo Nacional de Ciencia y Tecnología por el financiamiento vía Fondos Sectoriales de Ciencia Básica (proyecto 2004–C01–47777) y de la Comisión Nacional Forestal (proyecto 2005–C02–14783), y de la Coordinación de la Investigación Científica, Universidad Michoacana de San Nicolás de Hidalgo. Se agradece la ayuda de Felipe Aguilar, Rafael Echevarría y Reyes Aguilar, Dirección Técnica Forestal de la Comunidad Indígena de Nuevo San Juan Parangaricutiro, y de Rodrigo Niniz, Víctor H. Cambrón y Miguel A. Silva por su ayuda en la recolecta y obtención de semilla.
LITERATURA CITADA
Arriaga, L., y L. Gómez. 2004. Posibles efectos del cambio climático en algunos componentes de la biodiversidad de México. In: Martínez J. y A. Fernández–Bremauntz (comps.). Cambio Climático: una Visión desde México. Instituto Nacional de Ecología. SEMARNAT. México, D. F. pp: 255–265. [ Links ]
Conkle, M. T. 1981. Isozyme variation and linkage in six conifer specie. In: Proc. Symp. Isozymes of North American Forest Trees and Forest Insects. USDA For. Serv. Gen. Tech. Rep., PSW–48. pp: 11–17. [ Links ]
El–Kassaby, Y. A., and K. Rittland. 1996. Genetic variation in low elevation Douglasfir of British Columbia and its relevance to gene conservation. Biodiversity and Conservation 5: 779–794. [ Links ]
Hartl, D. L., and A. G. Clark. 1989. Principles of Population Genetics. Massachussets, Sinauer, 682 p. [ Links ]
Iglesias–Andreu, L. G., y M. Luna–Rodríguez. 2008. Polimorfismo isoenzimático en la población de Pinus hartwegii Lindl. del Cofre de Perote, Ver., México. Ecosistemas 17: 115–122. [ Links ]
INEGI (Instituto Nacional de Estadística, Geografía e Informática) 1998. Carta Topográfica 1:50000 Uruapan E13B39 Michoacán. México, INEGI. [ Links ]
Kawecki, T.J. 2000. Adaptation to marginal habitats: contrasting influence of the dispersal rate on the fate of alleles with small and large effects. Proc. R. Soc. Lond. B 267: 1315–1320. [ Links ]
Lauer, W. 1978. Timberline studies in central Mexico. Artic and Alpine Res. 10: 383–396. [ Links ]
Ledig, F.T. 1998. Genetic variation in Pinus. In: Richardson, D. M. (ed). Ecology and Biogeography of Pinus. Cambridge University Press. pp: 251–280. [ Links ]
Ledig, F. T., M. T. Conkle, B. Bermejo–Velázquez, T. Eguiluz–Piedra, P.D. Hodgskiss, D.R. Johnson, and W.S. Dvorak. 1999. Evidence for an extreme bottleneck in a rare Mexican pinyon: genetic diversity, disequilibrium, and mating system in Pinus maximartinezii. Evolution 53: 91–99. [ Links ]
Ledig, F. T., M. A. Capó–Arteaga, P.D. Hodgskiss, H. Sbay, C. Flores–López, M.T. Conkle, and B. Bermejo–Velázquez. 2001. Genetic diversity and the mating system of a rare Mexican pinyon, Pinuspinceana, and a comparison with Pinus maximartinezii (Pinaceae). Am. J. Bot. 88: 1977–1987. [ Links ]
Millar, C. I., and W. J. Libby. 1991. Strategies for conserving clinal, ecotypic, and disjunct population diversity in widespread species. In: Falk, D. A., and K.E. Holsinger (eds). Genetic and Conservation of Rare Plants. Biology and Genetics. Oxford University Press. New York. pp: 149–170. [ Links ]
Miller, M.P. 1997. Tools for Population Genetic Analyses (TFP–GA) 1.3: A windows program for the analysis of allozyme and molecular population genetic data. Computer software distributed by author http://herb.bio.nau.edu/~miller (consulta: diciembre de 2008). [ Links ]
Ohsawa, T., and Y. Ide. 2007. Global patterns of genetic variation in plant species along vertical and horizontal gradients in mountains. Global Ecol. and Biogeography 17: 153–162. [ Links ]
Parraguirre–Lezama, C., J. J. Vargas–Hernández, P. Ramírez–Vallejo, H. S. Azpíroz–Rivero, y J. Jasso–Mata. 2002. Estructura de la diversidad genética en poblaciones naturales de Pinus greggii Engelm. Rev. Fitotec. Mex. 25: 279–287. [ Links ]
Perry, J. P. 1991. The Pines of Mexico and Central America. Timber Press. Portland, Oregon, USA. 231 p. [ Links ]
Ricker, M., G. Gutiérrez–García, and D. C. Daly. 2007. Modeling long–term tree growth curves in response to warming climate: test cases from a subtropical mountain forest and a tropical rainforest in México. Can. J. For. Res. 37: 977–989. [ Links ]
Sáenz–Romero, C., and B.L. Tapia–Olivares. 2003. Pinus oocarpa isoenzymatic variation along an altitudinal gradient in Michoacán, México. Silvae Genet. 52: 237–240. [ Links ]
Sáenz–Romero, C., A. Snively, and R. Lindig–Cisneros. 2003. Conservation and restoration of pine forest genetic resources in México. Silvae Genet. 52: 233–237. [ Links ]
Sáenz–Romero, C., G. E. Rehfeldt, N.L. Crookston, P. Duval, R. St–Amant, J. Beaulieu, and B. A. Richardson. 2009. Spline models of contemporary, 2030, 2060 and 2090 climates for Mexico and their use in understanding climate–change impacts on the vegetation. Climatic Change. In press. DOI 10.1007/s10584–009–9753–5. [ Links ]
SAS Institute. 1988. SAS/STAT Users's; release 6.03. SAS Institute. Cary, N.C. 1028 p. [ Links ]
Schaberg, P. G., G. J. Hawley, D.H. DeHayes, and S. E. Nljensohn. 2003. Silvicultural management and the manipulation of rare alleles. In: Beaulieu, J. (ed). Silviculture and the Conservation of Genetic Resources for Sustainable Forest Management. Proc. Symp. North American Forest Commission, Forest Genetic Resources and Silviculture Working Groups, and the International Union of Forest Research Organizations (IUFRO). Quebec, Canada. pp: 67–74. [ Links ]
Slatkin, M. 1987. Gene flow and geographic structure of natural populations. Science 236: 787–792. [ Links ]
Solís–Ramos., L. Y., y L. G. Iglesias–Andreu. 2001. Variación en la composición isoenzimática en la población de Pinus hart–wegii Lindl. del Pico de Orizaba, Veracruz. Cuadernos de Biodiversidad 3: 4–7. [ Links ]
Soltis, D. E, C. H. Haufler, D. C. Darrow, and G. J. Gastrony. 1983. Starch gel electrophoresis of ferns: a compilation of grinding buffers, and staining schedules. Am. Ferns J. 73: 9–27. [ Links ]
Swofford, D. L., and G. J. Olsen. 1990. Phylogeny reconstruction. In: Hillis, D. M., and C. Mortiz (eds). Molecular Systematics. Sunderland, Sinauer Associates. MA, USA. pp: 411–501. [ Links ]
Viveros–Viveros, H., C. Sáenz–Romero, J. J. Vargas–Hernández, J. López–Upton, G. Ramírez–Valverde, and A. Santacruz–Varela. 2009. Altitudinal genetic variation in Pinus hartwegii Lindl: I. Height growth, shoot phenology and cold damage in seedlings. For. Ecol. Manag. 257: 836–842. [ Links ]
Yamada, M., and R. P. Guries. 1989. Manual for starch gel electrophoresis: new chocolate lovers edition. Staff Paper Series No. 39. Depart. of Forestry, College of Natural Resource, Univ. of Wisconsin–Madison. Madison, WI. 29 p. [ Links ]
Yeh, F. C. 2000. Population genetics. In: Young A., D. Boshier, and T. Boyle (eds). Forest Conservation Genetics: Principles and Practice.CSIRO Publishing & CABI Publishing. Collingwood, Australia. pp: 21–37. [ Links ]
Yeh, F. C., R.–C. Yang, and T. Boyle. 1999. POPGENE Version 1.31. Microsoft Window–based Freeware for Population Genetic Analysis. Quick User Guide. Univ. of Alberta & Centre for International Forestry Research. Edmonton, Canada. http://www.ualberta.ca/~fyeh/ (consulta: diciembre 2008). [ Links ]
Zhang, X., H. Korpelainen, and C. Li. 2006. Microsatellite variation of Quercus aquifolioides populations at varying altitudes in the Wolong Natural Reserve of China. Silva Fennica 40(3): 407–415. [ Links ]














