Serviços Personalizados
Journal
Artigo
Indicadores
Links relacionados
-
 Similares em
SciELO
Similares em
SciELO
Compartilhar
Agrociencia
versão On-line ISSN 2521-9766versão impressa ISSN 1405-3195
Agrociencia vol.44 no.2 Texcoco Fev./Mar. 2010
Fauna silvestre
Diversidad genética y contribución reproductiva de una progenie de Brycon orbignyanus en el sistema reproductivo seminatural, usando marcadores microsatélites
Genetic diversity and reproductive contribution of Brycon orbignyanus offspring in the semi–natural reproductive system, using microsatellites markers
N. Mauricio Lopera–Barrero1,*, Lauro Vargas1, Rodolfo Nardez–Sirol2, Ricardo Pereira–Ribeiro1, Jayme Aparecido–Povh1, D. Pedro Streit Jr1, Patrícia Cristina–Gomes1
1 Universidade Estadual de Maringá, Grupo de Pesquisa Peixegen. Av. Colombo 5790, CEP 87020–900. Maringá, Paraná–Brasil, *Autor responsable: (nelson.peixegen@gmail.com), (lvargas@uem.br), (rpribeiro@uem.br), (jayme.peixegen@gmail.com), (danilo.streit@ufrgs.br), (patricia.peixegen@gmail.com).
2 Duke Energy International, Geracáo Paranapanema S.A. Diretoria de Meio Ambiente, Saúde e Seguranca. Rodovia Chavantes–Ribeiráo Claro, Km 10,CEP 18970–000, Chavantes – SP. Brasil, (rnsirol@duke-energy.com).
Recibido: Marzo, 2008.
Aprobado: Enero, 2010.
Resumen
La conservación de la variabilidad genética es prioritaria en lotes de peces usados en programas de repoblación de ríos, especialmente para especies amenazadas o en peligro de extinción como es el caso del Brycon orbignyanus. El objetivo del presente estudio fue estimar la diversidad genética y la contribución reproductiva de una progenie de B. orbignyanus usada en programas de repoblación, en un sistema reproductivo seminatural usando marcadores microsatélites. Se analizaron muestras de aleta caudal de 24 reproductores (12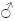 y 12
y 12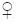 ) y 95 larvas de su progenie. Reproductores y progenie mostraron dos a leí os, ausencia de alelos de baja frecuencia y una heterocigosis media de 0.470 y 0.510. El coeficiente Fis mostró que hubo ausencia de endogamia en los reproductores y progenie (–0.116 y –0.242) y no hubo diferencias (p>0.01) en el equilibrio de Hardy–Weinberg. De acuerdo con el análisis de varianza molecular (AMOVA), la mayor parte de la variación observada estuvo dentro de cada grupo y no entre los grupos, corroborado por el valor de índice de Shannon (reproductores = 0.477; progenie = 0.491). La diversidad genética en la progenie fue preservada y se verificó la presencia de paternidad múltiple y dominancia reproductiva en 50 % de la progenie mediante la utilización del sistema reproductivo seminatural.
) y 95 larvas de su progenie. Reproductores y progenie mostraron dos a leí os, ausencia de alelos de baja frecuencia y una heterocigosis media de 0.470 y 0.510. El coeficiente Fis mostró que hubo ausencia de endogamia en los reproductores y progenie (–0.116 y –0.242) y no hubo diferencias (p>0.01) en el equilibrio de Hardy–Weinberg. De acuerdo con el análisis de varianza molecular (AMOVA), la mayor parte de la variación observada estuvo dentro de cada grupo y no entre los grupos, corroborado por el valor de índice de Shannon (reproductores = 0.477; progenie = 0.491). La diversidad genética en la progenie fue preservada y se verificó la presencia de paternidad múltiple y dominancia reproductiva en 50 % de la progenie mediante la utilización del sistema reproductivo seminatural.
Palabras clave: Brycon orbignyanus, conservación, manejo reproductivo, microsatélite, paternidad, programas de repoblación.
Abstract
The conservation of genetic variability is a priority for fish lots used in restocking programs in rivers, especially for threatened or endangered fish species as is the case oí Brycon orbignyanus. The objective of this study was to estimate the genetic diversity and reproductive contribution of an offspring of B. orbignyanus used in stock programs in a semi–natural reproductive system using microsatellite markers. Samples of caudal fin from 24 breeders (12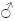 and 12
and 12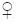 ), and 95 larvae of their offspring. Breeders and offspring showed two alíeles, absence of low frequency alíeles and an average heterozygosity of 0.470 and 0.510. The coefficient Fis showed that there was no inbreeding in breeders and offspring (–0.116 and –0.242) and there were no differences (p>0.01) in the Hardy–Weinberg equilibrium. According to the analysis of molecular variance (AMOVA), most of the variation observed was within each group and not among groups, being confirmed by the Shannon index value (breeders = 0.477; offspring = 0.491). The genetic diversity in the offspring was preserved and the presence of multiple paternity and reproductive dominance was verified in 50 % of the offspring through the use of the semi–natural reproductive system.
), and 95 larvae of their offspring. Breeders and offspring showed two alíeles, absence of low frequency alíeles and an average heterozygosity of 0.470 and 0.510. The coefficient Fis showed that there was no inbreeding in breeders and offspring (–0.116 and –0.242) and there were no differences (p>0.01) in the Hardy–Weinberg equilibrium. According to the analysis of molecular variance (AMOVA), most of the variation observed was within each group and not among groups, being confirmed by the Shannon index value (breeders = 0.477; offspring = 0.491). The genetic diversity in the offspring was preserved and the presence of multiple paternity and reproductive dominance was verified in 50 % of the offspring through the use of the semi–natural reproductive system.
Key words: Brycon orbignyanus, conservation, reproductive management, microsatellite, paternity, stock programs.
INTRODUCCIÓN
La deforestación en los márgenes de los ríos, la construcción de represas hidroeléctricas y canales de riego para producción agrícola (Hatanaka et al., 2006), así como la contaminación del agua y la falta de conocimiento taxonómico (Agostinho et al., 2005) han llevado a la desaparición de varias especies de peces. Brycon orbignyanus, conocido en Brasil como piracanjuba o bracanjuba (Orden Characiformes, familia Characidae, subfamilia Bryconinae), es una especie migratoria nativa de las cuencas formadas por los ríos Uruguay (Zaniboni–Filho y Schulz, 2003) y Paraná (Ganeco et al, 2001). Sus poblaciones naturales se han reducido en respuesta a modificaciones ambientales generadas por cambios climáticos y principalmente por acciones humanas (Zaniboni–Filho et al., 2006), siendo catalogada como especie en peligro de extinción (Machado, 2005). La repoblación es una práctica cada vez más común (Hilsdorf et al., 2006) para reducir dichos impactos desde hace unas tres décadas en el Brasil, pero sin respaldo científico (Agostinho et al., 2005). Por tanto, los estudios genéticos y reproductivos son necesarios para orientar esos programas (Lopera Barrero et al., 2009).
El monitoreo genético y reproductivo en lotes de piscicultura es importante para aumentar la producción y conservación de peces (Feng et al., 2007), ya que la disminución de la variabilidad genética por un inadecuado manejo genético (Frost et al., 2006) y reproductivo (Porta et al., 2006) puede traer problemas de endogamia, adaptabilidad y supervivencia de progenies usadas en programas de repoblación. Esos problemas pueden afectar las poblaciones naturales de peces (Sonstebo et al., 2007) y el ecosistema en general. Para este monitoreo se usan marcadores micro–satélites con éxito (Machado–Schiaffino et al., 2007).
El objetivo del presente estudio fue estimar la diversidad genética y la contribución reproductiva de una progenie de B. orbignyanus usada en programas de repoblación de el río Paranapanema, en un sistema reproductivo seminatural, utilizando marcadores microsatélites.
MATERIALES Y MÉTODOS
Se usaron 24 reproductores (12 y 12
y 12 ) de B. orbignyanus seleccionados de un lote de 136 individuos, mantenidos en cautiverio por seis años en las instalaciones de la Estación de Acuicultura e Hidrología de la Duke Energy International, Geracáo Paranapa–nema, ciudad de Salto Grande, SP (49° 59' 13" O y 22° 53' 32" S), Brasil. Estos individuos provienen de la primera generación de un lote de una piscicultura en la ciudad de Castilho, SP (52° 27' 24" O y 22° 11' 31"S) (Figura 1).
) de B. orbignyanus seleccionados de un lote de 136 individuos, mantenidos en cautiverio por seis años en las instalaciones de la Estación de Acuicultura e Hidrología de la Duke Energy International, Geracáo Paranapa–nema, ciudad de Salto Grande, SP (49° 59' 13" O y 22° 53' 32" S), Brasil. Estos individuos provienen de la primera generación de un lote de una piscicultura en la ciudad de Castilho, SP (52° 27' 24" O y 22° 11' 31"S) (Figura 1).

El experimento se realizó en noviembre de 2006 en la Estación de Acuicultura de la Duke Energy International. Los peces fueron inducidos a reproducción con extracto de hipófisis de carpa. Las hembras recibieron 5.5 mg kg–1 en dos aplicaciones: 10 % en la primera aplicación y 90 % 12 h después. Los machos recibieron 2.5 mg kg–1 en dosis única. Los reproductores se colocaron en un tanque circular (5.1 m radio; 1.85 m profundidad) y abastecido por un flujo continuo de agua (131 L s–1). Un tubo en la parte central del tanque permitió enviar los huevos a una estación recolectora. Los huevos fueron vertidos en una incubadora cilindro–cónica (200 L) con flujo continuo de agua (7 L s–1).
Aproximadamente 6 h después de la última inducción (160 horas–grado, 27 °C) se inició la recolección de huevos con un período máximo de 6 h; los huevos se retiraron cada hora y fueron conducidos hacia incubadoras cilindro–cónicas. Después de las 6 h se verificó por presión abdominal si los machos y hembras desovaron completamente. Una muestra de 0.5 cm2 de la aleta caudal se tomó de cada pez y las muestras se colocaron en micro–tubos de 1.5 mL conteniendo etanol para la extracción del ADN. La mortalidad (%) de los reproductores se determinó 1 d después de la reproducción.
Tres días después de la eclosión de los huevos, aproximadamente 200 larvas se recolectaron aleatoria y manualmente (20 larvas de cada incubadora), y se colocaron en micro–tubos de 1.5 mL conteniendo etanol. Para extraer ADN se recolectaron 95 larvas, de acuerdo con Lopera Barrero et al. (2008). En los micro tubos se adicionaron 550 mL de solución amortiguadora (50 mM Tris–HCl, 50 mM EDTA, 100 mM NaCl, 1 % SDS) y 200 mg mL–1 de proteinasa K. Las muestras se incubaron en baño maría (50 °C) por 12 h. El ADN se precipitó con 600 mL de solución de NaCl (5M) y se centrifugó 10 min a 12000 rpm. El ADN se transfirió a micro tubos vacíos, se precipitó con 700 mL de etanol y se incubó por 1 ha —20 °C. Enseguida se lavó con 700 mL de etanol 70 %, se suspendió en TE amortiguador TE – 10 mM Tris pH 8.0, 1 mM EDTA (80 mL para aleta y 35 mL para larva), y se trató con 30 mg mL–1 de ARNasa. Las muestras se incubaron en baño maría a 37 °C por 60 min y se conservaron a–20 °C.
El ADN se cuantificó en un espectrofotómetro UV 1601 (Shimadzu, EE.UU.) y se diluyó a 10 ng µL–1 (aletas) y 5 ng, µL–1 (larvas). La integridad del ADN se verificó en electroforesis horizontal en un gel de agarosa al 1 %, a 70 V por 60 min usando una solución amortiguadora TBE IX (500 mM Tris–HCl, 60 mM ácido bórico, 83 mM EDTA). El gel se marcó con bromuro de etidio (0.5 µg mL–1) por 30 min y la imagen se capturó con el sistema fotográfico EDAS (Kodak ID Image Analysis 3.5, EE.UU.) (Figura 2).
El ADN se amplificó en un volumen final de reacción de 15 mL usando la solución amortiguadora Tris–KCl, 2.0 mM MgCl2, 0.8 µM de cada cebador (Forwardy Reverse), 0.2 mM de cada dNTP, una unidad de Platinun Taq DNA Polimerasa, 10 ng de ADN para larvas y 20 ng de ADN para las aletas. Se amplificaron cuatro loci descritos por Barroso et al. (2003) para Brycon opalinus (No. Acceso GeneBank AF513621–BoMl, AF513622–BoM2, AF513626–BoM7 y AF513628–BoM13). Las reacciones de amplificación se realizaron en un termociclador Eppendorf Mastercycler* Gradient (EE.UU.), programado para 30 ciclos, con un paso inicial de desnaturación a 94 °C por 4 min y un paso final de extensión a 72 °C por 10 min. Cada ciclo consistió de 1 min a 94 °C, 1 min a 48 °C, 58 °C, 52 °C y 50 °C para cada locus, y 1 min a 72 °C.
Las muestras amplificadas fueron separadas en electroforesis en gel de poliacrilamida al 10 % (acrilamida:bisacrilamida – 29:1) desnaturada (6 M de urea), en solución amortiguadora TBE IX (90 mM Tris–Borato, 2 mM EDTA) a 300 V por 4 h. Para visualizar los alelos microsatélites se usó nitrato de plata según lo descrito por Bassam et al. (1991). El gel se puso en una solución de fijación (10 % etanol, 0.5 % ácido acético) por 20 min, se tiñó con nitrato de plata (6 mM) por 10 min y se reveló (0.75 M NaOH, 0.22 % formol–40 %). Cada gel se fotografió con cámara digital Nikon (E5200). El tamaño de los alelos se calculó por el programa Kodak – EDAS 290 (EE.UU.) usando marcadores de ADN de 10 pb y 50 pb (DNA ladder – Invitrogen*) (Figura 3).
El número de alelos, la heterocigosis observada (Ho) y esperada (He), el equilibrio de Hardy–Weinberg, la deficiencia o exceso de heterocigotos y el coeficiente de endogamia (Fis) se calcularon usando el programa GENEPOP 3.3 (Raymond y Rousset, 1995). Las frecuencias alélicas y el índice de Shannon se calcularon con el programa PopGene 1.31 (Yeh et al., 1999). El análisis de variancia molecular AMOVA se calculó con el programa
Arlequim 3.1 (Excoffier et al, 2005). La prueba de Duncan se usó para determinar las diferencias entre las medias. La determinación de la paternidad de la progenie se realizó con el programa PAPA versión 2.0 (Duchesne et al., 2002).
RESULTADOS Y DISCUSIÓN
Los cuatro loci produjeron ocho alelos con tamaños entre 140 pb y 200 pb, observados en los reproductores y en la progenie (Cuadro 1). Se observó presencia de los mismos alelos en los reproductores y en la progenie y ausencia de alelos de baja frecuencia (menor que 0.100) que indicaría pérdida de alelos. Según Innes y Elliott (2006), la pérdida de alelos es la primera señal de la disminución de la variabilidad genética en una población; así los resultados sugieren que se preservó de la variabilidad genética en la progenie.
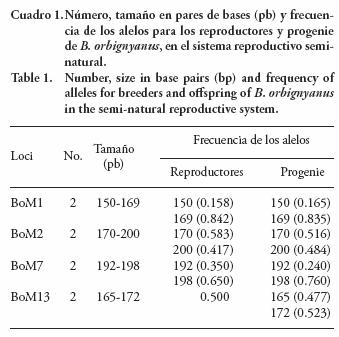
No se encontraron diferencias (p>0.01) en el equilibrio de Hardy–Weinberg en los reproductores y en la progenie. Los valores estimados de heterocigocidad observada (Ho) y de coeficiente de endogamia (Fis), no indicaron aparición de endogamia en los reproductores y exceso de heterocigotos para los loci BoM1 BoM7 y BoM13. El locus BoM2 presentó deficiencia de heterocigotos. Igualmente, los resultados de Fis mostraron pérdida de heterocigosis en el locus BoM2 (0.452) (Cuadro 2).
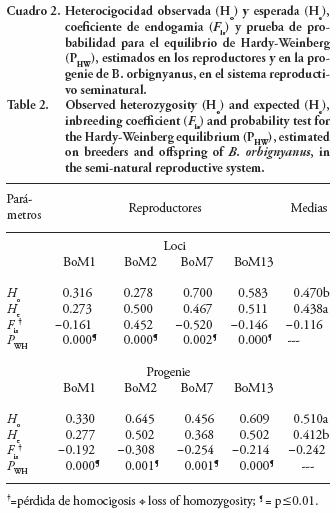
La deficiencia de heterocigotos observado en los reproductores para el locus BoM2 (Cuadro 2) se puede deber a endogamia, presencia de alelos nulos, muestreo no aleatorio o al efecto Wahlund (Hassanien y Gilbey, 2005). Sin embargo, por el Fis negativo encontrado en los otros loci y la falta de diferencias (p>0.01) en el equilibrio de Hardy–Weinberg, es improbable que el efecto Wahlund sea una posible causa.
En la progenie, los valores de heterocigocidad y el Fis mostraron que hubo exceso de heterocigotos, indicando ausencia de endogamia. La progenie obtenida por el sistema reproductivo seminatural presentó una heterocigosis media mayor (0.510) que la encontrada en los reproductores (0.470), mostrando que la variabilidad genética fue preservada (Cuadro 2).
De acuerdo con la AMOVA la mayor parte de la variación observada está dentro de cada grupo y no entre los grupos, mostrando que a pesar de ser Fl (reproductores) y F2 (progenie) no hubo pérdida de variabilidad genética durante el proceso reproductivo (Cuadro 3). Este resultado es corroborado por el valor de índice de Shannon (Reproductores=0.477; Progenie=0.491).
La preservación de la variabilidad genética en la progenie fue debida posiblemente a la utilización del sistema reproductivo seminatural. Este sistema reproductivo, cuando se realiza correctamente, tiende a reducir el direccionamiento y la selección sin intención en el proceso que ocurre normalmente en el sistema reproductivo por extrusión. Además disminuye significativamente la mortalidad causada por el estrés, posibilitando que un mayor número de reproductores genere progenie durante los cruzamientos y que exista una mayor preservación de la variabilidad genética. En el presente trabajo se verificó que todos los reproductores quedaron vacíos (por presión abdominal al final del proceso reproductivo), no hubo mortalidad de los reproductores y la variabilidad genética se preservó en la progenie.
Además, es común en los programas de repoblación se usen pocos individuos (por ejemplo, un macho para varias hembras) ya que las especies migratorias, como el B. orbignyanus, son muy prolíficas y la utilización de numerosos individuos está limitado por falta de infraestructura adecuada. En el presente estudio, cuando fueron cruzados 24 reproductores (12 y 12
y 12 ) con alta variabilidad genética, es probable que la contribución reproductiva en la fertilización de los huevos también fuera alta, favorecida por la utilización del sistema seminatural y de un número similar de hembras y machos, preservando la variabilidad genética. Estos resultados concuerdan con las recomendaciones de Pante et al. (2001), Oota y Matsuishi (2005) y Frost et al. (2006), donde el uso de un adecuado número de reproductores (Ne) junto con la formación correcta de cruzamientos (1:1) y el uso de sistemas reproductivos apropiados, puede controlar el aparecimiento de endogamia en las futuras generaciones y permitir una alta variación y contribución reproductiva entre los individuos y sus progenies (Qin et al., 2007).
) con alta variabilidad genética, es probable que la contribución reproductiva en la fertilización de los huevos también fuera alta, favorecida por la utilización del sistema seminatural y de un número similar de hembras y machos, preservando la variabilidad genética. Estos resultados concuerdan con las recomendaciones de Pante et al. (2001), Oota y Matsuishi (2005) y Frost et al. (2006), donde el uso de un adecuado número de reproductores (Ne) junto con la formación correcta de cruzamientos (1:1) y el uso de sistemas reproductivos apropiados, puede controlar el aparecimiento de endogamia en las futuras generaciones y permitir una alta variación y contribución reproductiva entre los individuos y sus progenies (Qin et al., 2007).
Se determinó sólo 50 % de la paternidad. Esta baja determinación está correlacionada con el bajo número de alelos encontrados (ocho alelos) provocada posiblemente por la utilización de loci heterólogos (desarrollados para Brycon opalinus). Estos resultados divergen del estudio realizado por Barroso et al. (2003) en el que se reportan 12 y 31 alelos entre 78 a 212 pb en B. orbignyanus; pero fueron similares a los encontrados por Sanches y Galetti Jr. (2006), los cuales investigaron siete loci para Brycon hilarii con la presencia de pocos alelos para B. orbignyanus (tres a ocho entre 138 a 220 pb). No obstante, los alelos obtenidos en el presente estudio fueron informativos y permitieron calcular la variabilidad genética y determinar la participación y comportamiento reproductivo de los reproductores en un porcentaje de la progenie usando el sistema reproductivo seminatural.
De los 12 machos y 12 hembras usados sólo cuatro machos (MI, M8, M9 y M10) y nueve hembras (F1,F2, F3, F5, F6, F8, F9, F10 y F11) fueron responsables por 50 % de la progenie. El macho 1 (MI) y la hembra 5 (F5) fueron responsables de la mayor contribución en la progenie (75–51 % y 28.57 %). Como consecuencia, el número efectivo de posibles progenitores (24) se redujo al determinar que el Ne fue realmente 11.08. Sin embargo, por sólo tener datos del 50 % de la paternidad este valor no es representativo para toda la progenie (Figura 4).
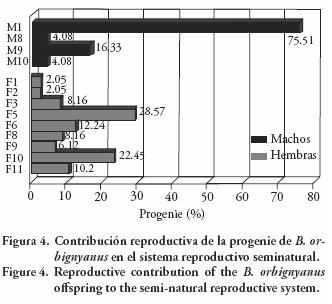
Todos los machos que contribuyeron con 50 % de la progenie fertilizaron más de una hembra, caracterizando la paternidad múltiple. El MI fertilizó la mayor cantidad de hembras (100 %), seguido del macho M9 (33–33 %) y de los machos M8 y M10 (22.22 %). La composición de las familias fue afectada directamente por la mayor participación del M1, y los cruzamientos F5 x M1 y F10 x M1 tuvieron mayor contribución en la progenie (44.69 %) (Figura 5). Este comportamiento determina que el sistema reproductivo seminatural pudo haber influenciado en la variación y en la contribución reproductiva. A pesar de que sólo se determinó 50 % de la paternidad, es evidente que el sistema reproductivo seminatural permitió la participación activa de los reproductores en la fertilización y el posible efecto de dominancia durante el cruzamiento, comportamiento que no es obtenido en el sistema reproductivo por extrusión, donde el cruzamiento es direccionado.
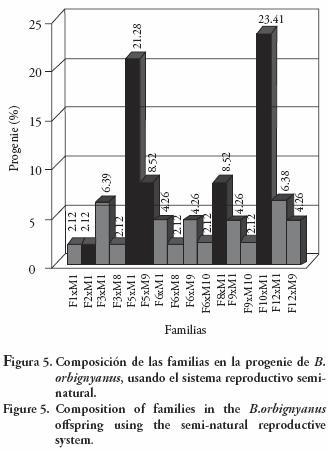
La dominancia reproductiva, también conocida como hipótesis de la calidad intrínseca del macho o hipótesis del buen esperma (Sivinski, 1984), puede afectar la variabilidad genética cuando se usan sistemas reproductivos seminaturales, ya que la dominancia de algunos machos (supuestamente los más fuertes y con mejores características reproductivas) en la fertilización de los óvulos puede influenciar la variabilidad genética de la progenie (Nordeide, 2007)– Este fenómeno ya se observó en peces exóticos y nativos del Brasil. Porta et al. (2006) para Solea senegalensis observaron que dos machos de 11 fueron responsables en mayor porcentaje por la progenie. Igualmente, Sekino et al. (2003) señalan que 99 % de la progenie fue originada por la participación reproductiva de un macho, lo que redujo la variabilidad genética en lotes de Paralichthys olivaceus.
Los resultados del presente estudio son importantes, porque con ellos se puede orientar objetivamente el manejo reproductivo y genético usado en programas de repoblación de B. orbignyanus. Por esta razón, durante la formación de cruzamientos destinados para producción de nuevos lotes o en la realización de programas de conservación en B. orbignyanus usando el sistema reproductivo seminatural, es recomendable usar la mayor cantidad posible de reproductores en proporciones iguales de sexo y así preservar la variabilidad genética, al disminuir la correlación negativa entre la similitud genética y el número de generaciones (Freitas y Galetti Jr., 2005). Sin embargo, se debe analizar la eficiencia del sistema reproductivo seminatural en la preservación de la variabilidad genética, incluyendo estudios de su efecto sobre la paternidad y la contribución reproductiva.
De esta forma, el monitoreo de la diversidad genética de lotes de peces mantenidos en piscícolas es fundamental para la conservación de las especies y de la ictiofauna (Lopera Barrero et al., 2007; Lopera Barrero et al., 2009), siendo necesario para que los individuos liberados durante los programas de repoblación puedan adaptarse y reproducirse sin provocar alteraciones en las poblaciones naturales (Sonstebo et al, 2007). Es necesario efectuar análisis de variabilidad genética en poblaciones naturales de B. orbignyanus en el río Paranapanema para determinar el potencial genético de esta importante especie migratoria y la orientación de programas efectivos de repoblación.
CONCLUSIONES
El sistema reproductivo seminatural fue efectivo en la conservación de la variabilidad genética de progenies de B. orbignyanus usadas en programas de repoblación. Se encontró paternidad múltiple y una contribución reproductiva diferenciada en la composición de las familias en el 50 % de la progenie.
AGRADECIMIENTOS
A la Duke Energy Internacional (Geracáo Paranapanema) por proveer las muestras de B. orbignyanus y al Conselho Nacional de Desenvolvimento Científico e Tecnológico (CNPq) por la beca de estudio.
LITERATURA CITADA
Agostinho, A. A., S. M. Thomaz, and L. C. Gomes. 2005. Conservation of the biodiversity of Brazil's inland waters. Conserv. Biol. 19: 646–652. [ Links ]
Barroso, R. M., A. W. S. Hilsdorf, H. L. M. Moreira, A. M. Mello, S. E. F. Guimaráes, P. H. Cabello, and Y. M. Traub–Cseko. 2003. Identification and characterization of micro–satellites loci in Brycon opalinus (Cuvier, 1819) (Characiforme, Characidae, Bryconiae). Mol. Ecol. Notes 3: 297–298. [ Links ]
Bassam, B. J., G. Caetano–Anollés, and P. M. Gresshoff. 1991. Fast and sensitive silver staining of DNA in polyacrylamide gels. Anal. Biochem. 196: 80–83. [ Links ]
Duchesne, P., M. H. Godbout, and L. Bernatchez. 2002. PAPA (Package for the analysis of parental allocation): a computer program for simulated and real parental allocation. Mol. Ecol. Notes 2: 191–193. [ Links ]
Excoffier, L., G. Laval, and S. Schneider. 2005. Arlequín Ver. 3.1: an integrated software package for population genetics data analysis. Evol. Bioinform. Online 1:47–50. [ Links ]
Feng, Y., Z. Peijun, W. Keling, and X. Jianhai. 2007. Genetic variation of natural and cultured stocks of Paralichthys olivaceus by allozyme and RAPD. Chin. J. Oceanol. Limnol. 25: 78–84. [ Links ]
Freitas, P. D., and P. M. Galetti Jr. 2005. Assessment of the genetic diversity in five generations of commercial broodstock line of Litopenaeus vannamei shrimp. Afr. J. Biotechnol. 4: 1362–1367. [ Links ]
Frost, L. A., B. S. Evans, and D. R. Jerry. 2006. Loss of genetic diversity due to hatchery culture practices in barramundi (Lates cakarifer). Aquaculture 261: 1056–1064. [ Links ]
Ganeco, L., L. Nakaghi, E. Urbinati, R. Dumont–Neto, e L. H. Vasques. 2001. Análise morfológica do desenvolvimento ovocitário de piracanjuba (Brycon orbignyanus) durante o ciclo reprodutivo. B. Inst. Pesca 27: 131–138. [ Links ]
Hassanien, H. A., and J. Gilbey. 2005. Genetic diversity and differentiation of Nile tilapia (Oreochromis niloticus) revealed by DNA microsatellites. Aquae. Res. 36: 1450–1457. [ Links ]
Hatanaka, T., F. Henrique–Silva, and P. M. Galetti Jr. 2006. Population substructuring in a migratory freshwater fish Prochilodus argenteus (Characiformes, Prochilodontidae) from the Sao Francisco River. Genética 126: 513–517. [ Links ]
Hilsdorf, A. W. S., E. K. Resende, e D. K. S. Marques. 2006. Genética e conservacáo de estoques pesqueiros de aguas continentais no Brasil: situacáo atual e perspectivas. Embrapa Pantanal, Corumbá. 44 p. [ Links ]
Innes, B. H., and N. G. Elliott. 2006. Genetic diversity in a Tasmanian hatchery population of Atlantic salmon (Salmo salar L.) compared with its Canadian progenitor population. Aquae. Res. 37: 563–569. [ Links ]
Lopera Barrero, N. M., R. P. Ribeiro, J. A. Povh, L. Vargas, C. B. Jacometo, y P. C. Gomes. 2009. Diversidad genética de lotes de Piaractus mesopotamicus usados en programas de repoblamiento y sus implicaciones en la conservación. Agrociencia 43: 249–256. [ Links ]
Lopera Barrero, N. M., J. A. Povh, R. P. Ribeiro, P. C. Gomes, C. B. Jacometo, y T. S. Lopes. 2008. Comparación de protocolos de extracción de ADN con muestras de aleta y larva de peces: extracción modificada con sal (NaCl). Cien. Inv. Agr. 35:15–24. [ Links ]
Lopera Barrero, N. M., R. P. Ribeiro, e J. A. Povh. 2007. O repovoamento de peixes: urna estrategia multidisciplinar? Aqüicultura & Pesca 30: 71–74. [ Links ]
Machado, A. B. M. 2005. Lista da fauna brasileira ameacada de extincáo: incluindo as especies quase ameacadas e deficientes em dados. In: Machado, A. B. M., C. S. Martins, and G. M. Drummond (eds). Fundacáo Biodiversitas, Belo Horizonte, 160 p. [ Links ]
Machado–Schiaffino, G., E. Dopico, and E. García–Vazquez. 2007. Genetic variation losses in Atlantic salmon stocks created for supportive breeding. Aquaculture 264: 59–65. [ Links ]
Nordeide, J. T. 2007. Is there more in 'gamete quality' than quality of the gametes? A review of effects of female mate choice and genetic compatibility on offspring quality. Aquae. Res. 38: 1–16. [ Links ]
Oota, T., and T. Matsuishi. 2005. Increase of inbreeding by stocking on wild population assessed by using individual–based life history model. Fish. Sci. 71: 73–78. [ Links ]
Pante, M., B. Gjerde, and I. McMillan. 2001. Inbreeding levels in selected populations in rainbow trout, Oncorhynchus mykiss. Aquaculture 192: 213–224. [ Links ]
Porta, J., J. M. Porta, G. Martínez–Rodríguez, and M.C. Alvarez. 2006. Genetic structure and genetic relatedness of a hatchery stock of Senegal sole (Solea senegalemis) inferred by micro–satellites. Aquaculture 251: 46–55. [ Links ]
Qin, Y., X. Liu, H. Zhang, and G. Zhang. 2007. Effect of parental stock size on F1 genetic structure in the bay scallop, Argopecten irradiam (Lamarck, 1819). Aquae. Res. 38: 174–181. [ Links ]
Raymond, M., and F. Rousset. 1995. Genepop (version 1.2): population genetics software for exact tests and ecumenicism. J. Heredity 86: 248–249. [ Links ]
Sanches, A., and P. M. Galetti Jr. 2006. Microsatellites loci isolated in the freshwater fish Brycon hilarii. Mol. Ecol. Notes 1: 1–2. [ Links ]
Sekino, M., K. Saitoh, T. Yamada, A. Kumagai, M. Hara, and Y. Yamashita. 2003. Microsatellite–based pedigree tracing in a Japanese flounder Paralichthys olivaceus hatchery strain: implications for hatchery management related to stock enhancement program. Aquaculture 221: 255–263. [ Links ]
Sivinski, J. 1984. Sperm in competition. In: Smith, R. L. (ed). Sperm Competition and the Evolution of Animal Mating Systems. Academic Press, London, pp: 86–115. [ Links ]
Sønstebø, J. H., R. Borgstrøm, and M. Heun. 2007. Genetic structure of brown trout (Salmo trutta L.) from the Hardan–gervidda mountain plateau (Norway) analyzed by micro–satellite DNA: a basis for conservation guidelines. Conserv. Genet. 8: 33–44. [ Links ]
Yeh, F. C., T. Y. Z. Boyle, and J. M. Xiyan. 1999. PopGene Version 131: Microsoft Window–based freeware for population genetic analysis. University of Alberta and Center for International Forestry Research, USA. 29 p. [ Links ]
Zaniboni–Filho, E., D. Reynalte–Tataje, y M. Weingartner. 2006. Potencialidad del género Brycon en la piscicultura brasileña. Rev. Col. Cienc. Pec. 19: 233–240. [ Links ]
Zaniboni–Filho E., and U. H. Schulz. 2003. Migratory Fishes of the Uruguay River. In: Carolsfeld, J., B. Harvey, C. Ross, and A. Baer. (Eds.), Migratory Fishes of South America. Biology, Fisheries and Conservation Status. World Fisheries Trust, Victoria, pp: 253–271. [ Links ]














