Servicios Personalizados
Revista
Articulo
Indicadores
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Agrociencia
versión On-line ISSN 2521-9766versión impresa ISSN 1405-3195
Agrociencia vol.44 no.2 Texcoco feb./mar. 2010
Biotecnología
Caracterización morfológica y molecular de nueve variedades botánicas de Tigridia pavonia (L.f.) DC
Morphological and molecular characterization of nine botanical varieties of Tigridia pavonia (L.f.) DC
José Luis Piña–Escutia1, César Vences–Contreras1, M. Guadalupe Gutiérrez–Martínez1, L. Miguel Vázquez–García1, Amaury M. Arzate–Fernández2,*
1 Facultad de Ciencias Agrícolas. Universidad Autónoma del Estado de México. Carretera Toluca–Ixtlahuaca. Km 11.5. entronque al Cerrillo. Centro Universitario El Cerrillo. 50200. Toluca, México.
2 Centro de Investigación y Estudios Avanzados del IPN. Laboratorio Nacional de Genómica para la Biodiversidad. Km. 9.6. Libramiento Norte Carretera Irapuato–León. Apartado Postal 629. 36500. Irapuato, Guanajuato. *Autor responsable: (amaury1963@yahoo.com.mx).
Recibido: Febrero, 2009.
Aprobado: Noviembre, 2009.
Resumen
Tigridia pavonia (L.f.) DC es una planta bulbosa nativa de México con potencial ornamental alto. México es considerado el centro de mayor diversidad de esta especie, por lo cual, el estudio de su diversidad genética y fenotípica es muy importante para establecer estrategias de manejo, conservación y utilización en programas de mejoramiento genético. El objetivo de este estudio fue caracterizar nueve variedades botánicas de Tigridia pavonia (L.f.) DC, con 21 marcadores morfológicos y cinco moleculares ISSR (ínter–Secuencias Simples Repetidas). Las variedades fueron recolectadas en tres municipios del Estado de México: Tenancingo (2100 m), Temascaltepec (2250 m) y Temoaya (2600 m). La distancia genética promedio para morfología fue 0.54, mientras que para ISSR fue 0.49. Así, ambos tipos de marcadores mostraron diversidad genética entre las variedades, distinguiendo a cada una de ellas. Los dendrogramas de los marcadores morfológicos e ISSR fueron notablemente similares, existiendo una correlación positiva entre ellos (R=0.30) y, en ambos casos, hubo una relación de las nueve variedades según su origen geográfico. Sin embargo, los marcadores ISSR fueron más efectivos, pues con un solo iniciador se distinguió a todas las variedades de T. pavonia. Se concluye que los marcadores ISSR son una herramienta útil para la discriminación rigurosa de genotipos y que se pueden usar de manera complementaria a la caracterización morfológica.
Palabras clave: Tigridia pavonia, caracterización morfológica, ínter–Secuencias Simples Repetidas.
Abstract
Tigridia pavonia (L.f) DC is a bulbous native plant of México with high ornamental potential. México is considered the center of greatest diversity of this species, thus the study of its genetic and phenotypic diversity is very important for establishing strategies of management, conservation and use in plant breeding programs. The objective of the present study was to characterize nine botanical varieties of Tigridia pavonia (L.f.) DC, with 21 morphological markers and five ISSR (Inter–Simple Sequence Repeat) markers. The varieties were collected in three municipalities of the Estado de México: Tenancingo (2100m), Temascaltepec (2250 m) and Temoaya (2600 m). The average genetic distance for morphology was 0.54, whereas for ISSR it was 0.49. Thus, both types of markers showed genetic diversity among the varieties, distinguishing each one of them. The dendrograms of the morphological markers and ISSR were notably similar, showing a positive correlation between them (R=0.30), and in both cases, there was a relationship of the nine varieties according to their geographic origin. However, the ISSR markers were more effective, because with a single primer all of the varieties of 77 pavonia were distinguished. It was concluded that the ISSR markers are a useful tool for the rigorous discrimination of genotypes and that they can be used as a complement to morphological characterization.
Key words: Tigridia pavonia, morphological characterization, Inter–Simple Sequence Repeat.
INTRODUCCIÓN
Tigridia pavonia (L.f.) DC, también conocida como flor del tigre u oceloxóchitl, es una especie nativa de México que fue utilizada por los aztecas como ornamental, alimenticia y medicinal (Hernández, 1959) y debido a la gran variabilidad en el colorido de su flor y su potencial ornamental, es uno de los principales recursos fitogenéticos de este país, considerado el centro de mayor diversidad genética de esta especie. Tigridia pavonia se encuentra ampliamente distribuida en diferentes países de Europa, Asia y Australia, donde es comercializada como planta para jardín (Vázquez–García et al., 2001a). Esto puede ocasionar que la extracción de material silvestre y la falta de un manejo racional de la especie conduzca a su erosión genética (Vázquez–García et al, 2001b).
El conocimiento de la diversidad genética de especies ampliamente distribuidas es importante para su conservación y distinción genética y fenotípica, pues generalmente muestran variación morfológica, fisiológica y en la estructura genética de sus poblaciones (Wen y Hsiao, 2001). Esta variación puede deberse a diversos factores como el altitudinal, por lo que es común encontrar diferentes fenotipos de una misma especie, entre poblaciones desarrolladas a diferentes altitudes (Ohsawa e Ide, 2007). Por lo anterior, se ha sugerido el desarrollo de los análisis morfológicos en combinación con los moleculares para mejorar la distinción entre especie o variedad dentro de los géneros o dentro de las especies de un género (Rodríguez y Sytsma, 2005).
Los marcadores morfológicos o fenotípicos han sido utilizados tradicionalmente para distinguir variedades (Tapia et al, 2005a; Adugna et al, 2006). Así, Vázquez–García et al. (2001a) describieron morfológicamente nueve variedades botánicas de T. pavonia, provenientes de tres lugares con diferente altitud. Sin embargo, este tipo de caracterización no siempre refleja la variación genética real, debido a que el fenotipo está determinado parcialmente por la información genética del individuo, e influenciado por el ambiente donde se desarrolla (Valadez–Moctezuma et al, 2001).
Los marcadores bioquímicos o isoenzimas también han sido usados para distinguir genotipos estrechamente relacionados. Así, Arzate–Fernández et al. (2008) caracterizaron nueve variedades de T. pavonia, con nueve isoenzimas. No obstante, aunque esta técnica es reproducible y relativamente barata, generalmente muestra niveles bajos de polimorfismo y puede estar limitada por la influencia del ambiente y los estados de desarrollo de la planta (Vicente y Fulton, 2003).
El uso de marcadores de ADN apoya la selección de plantas, pues permite detectar lugares específicos de las secuencias de genes que difieren entre cultivares o líneas mejoradas (Suslow et al., 2005). La técnica de ISSR (ínter–Secuencias Simples Repetidas) consiste en la amplificación de segmentos de ADN entre dos microsatélites. El microsatélite usado como iniciador, generalmente está formado de secuencias cortas repetidas (1–6 pb) y distribuidas ampliamente en los genomas eucariotas, por lo que pueden mostrar polimorfismos en las plantas. Además, estos iniciadores pueden contener un ancla en los extremos 3' o 5', que contiene una, dos o tres bases adicionales al microsatélite, con lo que aumenta su especificidad (Valadez–Moctezuma et al, 2001).
Los marcadores ISSR han sido utilizados exitosamente para calcular la diversidad genética intra o inter–específica en diferentes especies domesticadas y silvestres (Arzate–Fernández et al., 2005; Escandón et al., 2005b; Tapia et al., 2005b), y son superiores otro tipo de técnicas por su rapidez, reproducibilidad y eficiencia altas para detectar polimorfismos (Pradeep et al, 2002).
Los objetivos del presente trabajo fueron: 1) caracterizar nueve variedades botánicas de T. pavonia mediante marcadores morfológicos y moleculares ISSR; 2) correlacionar la eficiencia de ambos marcadores en la diferenciación de las variedades de T. pavonia evaluadas; 3) conocer la correlación entre los patrones generales de variación genética y la altitud de muestreo de cada variedad.
MATERIALES Y MÉTODOS
Material vegetal
Se utilizaron nueve variedades botánicas de Tigridia pavonia (L.f.) DC, recolectadas en tres localidades del Estado de México con registro en el Servicio Nacional de Inspección y Certificación de Semillas (SNICS). La variedad Sandra (TGD–008–030408) fue recolectada en el municipio de Tenancingo (2100 m), Carolina (TGD–002–030408), Trinidad (TGD–009–030408), Penélope (TGD–006–030408), Ángeles (TGD–001–030408), Dulce (TGD–003–030408) y Mariana (TGD–005–030408), en el municipio de Temascaltepec (2250 m) y Samaria (TGD–007–030408) y Gloria (TGD–004–030408) en el municipio de Temoaya (2600 m). Todas las variedades fueron cultivadas en una mezcla de tierra de monte, arena y estiércol bovino (proporción 1:1:1), en un invernadero tipo rústico, de la Facultad de Ciencias Agrícolas de la Universidad Autónoma del Estado de México, a 2600 m.
Análisis morfológico
Para elaborar la matriz de datos morfológicos se evaluaron 20 caracteres cuantitativos y uno cualitativo (Cuadro 1); de ellos, nueve fueron publicados por Vázquez–García et al. (2001a). Cada carácter se evaluó en 14 individuos de cada variedad.
Análisis molecular ISSR
Extracción de ADN
El ADN genómico fue extraído de aproximadamente 100 mg de tejido fresco de hoja con el método denominado CTAB (Zhou et al, 1999). El ADN fue resuspendido en 50 µL de amortiguador TE (Tris–HCl EDTA) y almacenado a –20 °C hasta su uso.
Amplificación por PCR (reacción en cadena de la polimerasa) y electroforesis del ADN
La PCR se efectuó en un volumen final de 10 µL con: 1 µL de ADN (10 ng), 1 µL de 10X amortiguador PCR con amonio (15 mM), 0.5 µL de MgCl2, (15 mM), 1 µL de dNTPs (10 mM) (APPLIED BIOSYSTEMS*), 1 µL del iniciador (20 µM) (INVITROGENTM) y 0.1 unidades de la enzima Taq ADN polimerasa (MERCURY REAGENTSTM). Se usaron cinco iniciadores de tipo microsatélites anclados (3'–ASSR) (Yamagishi et al., 2002). En cada iniciador el ancla consistió en un triplete de secuencia distinta (Cuadro 2).
Las condiciones de amplificación para los iniciadores 3'–ASSR02 y 3'–ASSR15 fueron las descritas por Arzate–Fernández et al. (2005) y consistieron en un ciclo inicial de 9 min a 94 °C, 1 min a 46 °C y 1 min a 72 °C, seguido por 45 ciclos de 1 min a 94 °C, 1 min a 46 °C y 1 min a 72 °C y un ciclo final de 9 min a 94 °C, 1 min a 46 °C y 10 min a 72 °C. Para los iniciadores 3'–ASSR20, 3'–ASSR29 y 3'–ASSR35, los ciclos para la amplificación fueron los utilizados por Yamagishi et al. (2002) y consistieron en un ciclo inicial de 9 min a 94 °C, seguido por 45 ciclos de 1 min a 94 °C, 1 min a 46 °C y 1 min a 72 °C y un ciclo final de 10 min a 72 °C.
La amplificación de los fragmentos de ADN se realizó en un termociclador de gradiente Mastercycler (EPPENDORF*) modelo HAMBURG 22331. La separación de los fragmentos se realizó en cámaras de electroforesis horizontal, con geles de agarosa tipo II (SIGMA*) al 1 %, a los cuales se agregaron 3 µL de bromuro de etidio (SIGMA*). Se usó un marcador de peso molecular de 100 a 3000 pb (FERMENTASTM). Las condiciones de corrimiento para cada muestra fueron 100 V y 120 mA por 80 min, y la observación de los fragmentos se realizó en un transiluminador (UVPTM) modelo MP20.
Análisis estadístico
Los 21 caracteres morfológicos fueron evaluados mediante un análisis de varianza y un diseño completamente al azar con 14 repeticiones, con el programa estadístico SAS versión 8.0. Con los datos generados de los caracteres morfológicos, se elaboró una matriz binaria siguiendo el criterio descrito por Vicente y Fulton (2003); esto es, se asignó el valor 1 si el carácter estuvo presente y 0 si estuvo ausente.
Para el análisis molecular ISSR, cada banda generada por cada iniciador 3'–ASSR fue considerada como un locus independiente calculado manualmente; es decir, se asignó el valor 1 para la presencia de una banda y 0 para su ausencia. Únicamente las bandas con mayor claridad fueron usadas para el análisis.
El número total de bandas (BT), bandas polimórficas (BP) y porcentaje de bandas polimórficas (%P) fueron calculados usando el programa POPGENE versión 1.32 (Yeh y Boyle, 1999). La capacidad de cada iniciador ASSR para diferenciar a las nueve variedades en estudio fue evaluada mediante el poder de resolución (Rp), según Prevost y Wilkinson (1999).
En ambos casos, para conocer las relaciones genéticas y cómo se agrupaban las variedades analizadas, se usó el método UPG–MA (unweighted pair group method arithmetic average, por sus siglas en inglés) basado en la matriz de distancia genética (DG) de Nei (Nei, 1972), a partir de la cual se construyeron los dendogramas usando el programa POPGENE.
Para calcular la correlación entre los marcadores morfológicos y moleculares ISSR, se hizo un análisis de correlación simple basado en las matrices de distancias genéticas, usando el programa STATGRAPHICS Plus versión 4.0.
Para conocer si la variación genética entre las nueve variedades evaluadas estaba relacionada con un gradiente altitudinal, se correlacionaron los valores de las distancias genéticas derivados del análisis morfológico y molecular, con los de la altitud geográfica correspondientes a cada una de las variedades de T. pavonia, para lo cual, se realizó un análisis de correlación usando el programa STATGRAPHICS.
RESULTADOS Y DISCUSIÓN
Análisis morfológico
Los valores de la media, máximo, mínimo, desviación estándar (DE) y coeficiente de variación (CV), fueron calculados para cada uno de los 21 caracteres morfológicos evaluados en las nueve variedades (Cuadro 1). De acuerdo con los valores del CV, los caracteres número de flores por brote, tipo de tépalo interno, distancia de la base de la antera al estigma, número de frutos por brote, longitud del entrenudo y número de ramas por brote mostraron un nivel de variación ligeramente alto. Por el contrario, los caracteres que presentaron niveles bajos de variación entre las variedades fueron la longitud de la columna estaminal, longitud de la parte reproductiva y anchura de la bráctea.
El análisis estadístico mostró que el número de flores por brote y de nudos por brote no fueron significativos. Sin embargo, para los 19 caracteres restantes hubo diferencias altamente significativas (p<0.0001) (Cuadro 1). Estos resultados muestran la alta diversidad fenotípica entre las variedades analizadas, y pueden ser usadas como características feno–típicas distintivas para la selección de líneas parentales dentro de un programa de mejoramiento.
Las variedades formaron dos grupos con base en sus caracteres morfológicos (Figura 1A). El grupo I con promedio de DG=0.42 consistió de las variedades Carolina, Trinidad, Mariana, Angeles, Sandra, Penélope y Dulce, mientras que en el grupo II estuvieron las variedades Gloria y Samaria, con un promedio de DG =0.32.
Los caracteres morfológicos (18 de 21 iguales) mostraron la estrecha relación genética entre las variedades Trinidad y Mariana (DG = 0.28). Por el contrario, las variedades menos emparentadas fueron Carolina y Samaria, así como Penélope y Samaria (DG=0.80), con 4 y 5 de 21 caracteres morfológicos similares, respectivamente.
Según Vázquez–García et al. (2001a), las variedades Gloria y Samaria pueden ser fácilmente distinguidas por su longitud de brote. En el presente estudio, los resultados fueron similares y ambas variedades mostraron altura de planta, longitud del entrenudo, longitud del escapo floral y longitud de bráctea mayor, así como la anchura de las brácteas.
Análisis molecular ISSR
El número total de bandas reproducibles con los cinco iniciadores ASSR fue 40, con un intervalo de 350 a 1900 pb en el tamaño de los fragmentos amplificados. De las 40 bandas, 35 fueron polimórficas, con un promedio de siete por iniciador. El porcentaje de bandas polimórficas varió del 66.6 al 100 %, con un promedio de 87–5 % (Cuadro 2). Estos resultados coinciden con lo reportado por Yamagishi et al. (2002) y Arzate–Fernández et al. (2005), quienes utilizaron estos iniciadores ASSR y comprobaron su eficiencia para detectar porcentaje de polimorfismo alto en diferentes especies del género Lilium.
Un aspecto que favorece la efectividad de los iniciadores ASSR además del tipo de la repetición motivo en su secuencia, es la secuencia de su ancla. Las secuencias motivo CT producen mayor polimorfismo en comparación con las repeticiones AT (Pradeep et al, 2002; Hu et al, 2003), que, a pesar de ser las más abundantes en los genomas vegetales, tienen la desventaja de que la amplificación de fragmentos de ADN sea baja; esto puede deberse a la semi–complementariedad del iniciador en la etapa de alineación de la PCR (Fang y Roose, 1997). En esta investigación, el nivel de distinción entre las variedades dependió de la secuencia del ancla del iniciador. Así, fue posible obtener 100 % de polimorfismo entre las variedades de T. pavonia con los iniciadores ASSR02 y ASSR35 (Cuadro 2).
Prevost y Wilkinson (1999) describieron el poder de resolución (Rp) como una herramienta útil para evaluar la capacidad de un iniciador en la distinción de varios genotipos. En esta investigación, los valores más altos de Rp fueron para los iniciadores ASSR02 y ASSR35 (Cuadro 2), con los cuales se distinguieron una de otra, las nueve variedades. Escandón et al. (2005a) obtuvieron resultados similares sólo con dos iniciadores ASSR y generaron perfiles específicos para 18 de 21 recolectas de Jacaranda mimosifolia. La eficiencia de los iniciadores ASSR para discriminar genotipos en el nivel inter–varietal también ha sido reportada en Solanum tuberosum (Prevost y Wilkinson, 1999), Jacaranda mimosifolia (Pérez de la Torre et al., 2003), Nierembergia linaeriefolia (Escandón et al., 2005b) y Ficus carica (Guasmi et al., 2006), entre otros.
El dendograma generado con los datos ISSR (Figura 1B) formó dos grupos de variedades: en el grupo I se ubicaron Carolina, Angeles, Trinidad, Sandra, Dulce, Mariana y Penélope (DG=0.19); en el grupo II, Gloria y Samaria (DG=0.39). La asociación genética mayor (DG=0.07) se encontró entre las variedades Carolina y Angeles, mientras que las menos emparentadas fueron Gloria y Dulce (DG=0.91).
Correlación entre marcadores morfológicos y moleculares ISSR
El coeficiente de correlación entre las matrices de los marcadores morfológicos y moleculares ISSR fue 0.30 (p<0.1). Esto indica una relación positiva de ambas variables en la diferenciación genética de las nueve variedades de T. pavonia analizadas. Algo similar mostraron los dendogramas con el agrupamiento de las mismas variedades en cada uno (Figura 1). Los análisis de los caracteres morfológicos e ISSR parecen más eficientes que el de isoenzimas utilizado por Arzate–Fernández et al. (2008) para caracterizar nueve variedades de T. pavonia y con el que sólo diferenciaron cinco de las nueve variedades evaluadas.
La correlación positiva también indica que los marcadores morfológicos pueden proveer una medida de la diversidad genética entre las variedades. Sin embargo, a pesar de que los promedios de DG calculados con los caracteres morfológicos (0.54) (Figura 1 A) e ISSR (0.49) (Figura 1B) indican que los primeros tienen un valor ligeramente más alto para detectar la variabilidad genética, la amplitud de DG para los marcadores ISSR fue mayor. Esto permite asegurar la diferencia entre los individuos con una metodología relativamente rápida y sencilla como es el uso de ISSR. Tapia et al. (2005a; 2005b) y Geleta et al. (2006) también observaron una mayor eficiencia de los marcadores ISSR, respecto a los morfológicos, para discriminar genotipos de Ananas comosus L., Citrus reticulata L., y Sorghum vulgare Pers.
Correlación entre distancia genética y altitud geográfica
De acuerdo con Wen y Hsiao (2001), la diferenciación genética en poblaciones de Lilium longiflorum está correlacionada con la altitud; a menor altitud mayor es la similitud entre individuos y viceversa. Arzate–Fernández et al. (2005) evaluaron la diversidad genética en dos poblaciones de L. maculatum y encontraron que las poblaciones desarrolladas en altitudes de 1000 m presentaron mayor variación genética que los que crecían a 650 m. En la presente investigación también fue posible establecer una correlación positiva y estadísticamente significativa (p<0.001) entre la variación genética y la altitud geográfica de las variedades de T. pavonia evaluadas (Figura 2). Con ambos análisis, las variedades se ubicaron según su origen geográfico, lo cual coincidió con los dos grupos principales en los respectivos dendrogramas.
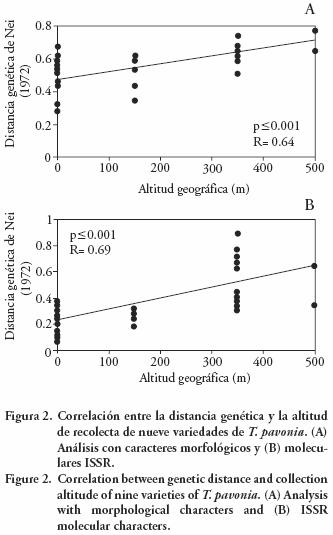
En ambos dendrogramas el grupo I siempre incluyó a la variedad Sandra, recolectada en Tenancingo (2100 m) y las recolectadas en Temascaltepec (2250 m), mientras que en el grupo II permanecieron juntas las variedades Gloria y Samaria, recolectadas en Temoaya (2600 m).
Lesica y Allendorf (1995, citados por Ohsawa e Ide, 2007) mencionan que las poblaciones entre dos límites geográficos experimentan mayor intercambio de genes, mientras que entre las poblaciones periféricas, hay flujo limitado de genes. La diferencia en la altitud de origen de las poblaciones se relaciona con una marcada diferencia en la fenología de cada especie, específicamente en la floración y la fructificación; ambas fases se retardan en altitudes mayores (Jordano y Godoy, 2000). En concordancia con esto, en el presente estudio se observó que la floración de las variedades Samaria y Gloria fue la más tardía y los sitios de recolecta pueden haber causado ese efecto. Similarmente la variedad Sandra, recolectada a 2100 m, estuvo relacionada cercanamente al grupo recolectado a 2250 m y no al recolectado a 2600 m.
CONCLUSIONES
Los marcadores morfológicos y los de tipo ISSR muestran polimorfismo entre las nueve variedades de T. pavonia obtenidas en un gradiente altitudinal, y permiten la distinción entre ellas con el uso de los iniciadores 3'–ASSR02 y 3'–ASSR35.
La relación genética entre las variedades fue visualizada con los dendrogramas generados con caracteres morfológicos, moleculares, y el análisis de correlación.
AGRADECIMIENTOS
Agradecemos al Consejo Nacional de Ciencia y Tecnología (CONACYT) por la beca otorgada al Ing. José Luis Pina Escutia, Proyecto No. 52936 así como a la Universidad Autónoma del Estado de México, Proyecto No. 2106/2005. Por último, a la Q.F.B. Ana Isabel Quintana Carapia, por su valioso apoyo logístico.
LITERATURA CITADA
Adugna, W., M. T. Labuschagne, and C. D. Viljoen. 2006. The use of morphological and AFLP markers in diversity analysis of linseed. Biodiversity and Conservation 15: 3193–3205. [ Links ]
Arzate–Fernández, A. M., M. Miwa, T. Shimada, T. Tonekura, and K. Ogawa. 2005. Genetic diversity of miyamasukashiyuri (Lilium maculatum Thunb. var. Bukosanense), an endemic endangered species at Mount Buko, Saitama, Japan. Plant Species Biol. 20: 57–65. [ Links ]
Arzate–Fernández, A. M., A. Hoyos–Basurto, L. M. Vázquez–García y M. G. Gutiérrez–Martínez. 2008. Caracterización isoenzimática de nueve variedades botánicas de Tigridia pavonia (L.f.) DC. Agrociencia 42: 519–528. [ Links ]
Escandón, A., M. Pérez de la Torre, A. Acevedo, S. Marucci–Poltri, and I. Mijayima. 2005a. Anchored ISSRas molecular marker to characterize accesions of Jacaranda mimosifolia L. Don. Acta Horticulturae 683: 121–127. [ Links ]
Escandón A., M. Pérez de la Torre, M. S. Soto, y N. Zelener. 2005b. Identificación de clones selectos de Nierembergia linariaefolia mediante microsatélites anclados. Rev. Inv. Agrop. INTA. Argentina 34 (1): 5–17. [ Links ]
Fang, D. Q., and M. L. Roose. 1997. Identification of closely related citrus cultivars with inter–simple sequence repeat markers. Theor. Appl. Genet. 95: 408–417. [ Links ]
Geleta, N., M. T Labuschagne, and C. D. Viljoen. 2006. Genetic diversity analysis in sorghum germplasm as estimated by AFLP, SSR and morpho–agronomical markers. Biodiversity and Conservation 15: 3251–3265. [ Links ]
Guasmi, F., A. Ferchichi, K. Fares, and L. Touil. 2006. Identification and differentiation of Ficus carica L. cultivars using Inter simple sequence repeat markers. Afr. J. Biotechnol. 5 (15): 1370–1374. [ Links ]
Hu, J., M. Nakatani, L. A. García, T. Kuranouchi, and T. Fujimura. 2003. Genetic analysis of sweetpotato and wild relatives using inter–simple sequence repeats (ISSRs). Breeding Sci. 53: 297–304. [ Links ]
Hernández F. 1959. Historia natural de la Nueva España. Universidad Nacional Autónoma de México. Tomo II. Vol. I. 476 p. [ Links ]
Jordano, P., and J. A. Godoy. 2000. RAPD variation and population genetic structure in Prunus mahaleb (Rosaceae), an animal–dispersed tree. Molecular Ecol. 9: 1293–1305. [ Links ]
Nei M. 1972. Original measures of genetic identity and genetic distance genetic. Am. Naturalist 106: 283–292. [ Links ]
Ohsawa, T., and Y. Ide. 2007. Global patterns of genetic variation in plant species along vertical and horizontal gradients on mountains. Global Ecol. Biogeography 1–12. [ Links ]
Pérez de la Torre M., A. Acevedo, J. C. Serpa, I. Mijayima, y A. S. Escanden. 2003. Puesta a punto de la técnica de microsatélites anclados para la caracterización de individuos selectos de Jacaranda. In: Mascarini, L., F. Vilella., and E. Wright (eds). Floricultura en la Argentina. Inv. Tecnol. Prod. 3–12. [ Links ]
Pradeep, R. M., N. Sarla, and E. A. Siddiq. 2002. ínter simple sequence repeat (ISSR) polymorphism and its application in plant breeding. Euphytica 128: 9–17. [ Links ]
Prevost, A., and M. J. Wilkinson. 1999. A new system of comparing PCR primers applied to ISSR fingerprint of potato cultivars. Theor. Appl. Genet. 98: 107–112. [ Links ]
Rodríguez, A., and K. J. Sytsma. 2005. Phylogenetics of the "tiger flower" group (Tigridieae: Iridaceae): molecular and morphological evidence. Aliso 22 (1): 412–424. [ Links ]
Suslow, T. V., B. R. Thomas, and K. J. Bradford. 2005. Biotechnology provides new tools for plant breeding. Agrie. Biotechnol. California Series. Publication 8043. pp: 1–19. [ Links ]
Tapia C. E., A. H. Guillen, y E. M. A. Gutiérrez. 2005a. Caracterización genética de materiales de pina (Ananas spp.) mediante RAPD e ISSR. Fitotec. Mex. 28 (3): 187–194. [ Links ]
Tapia, C. E., E. M. A. Gutiérrez, M. L. Warburton, V. A. Santacruz, and M. A. Villegas. 2005b. Characterization of mandarin (Citrus spp.) using morphological and AFLP markers. Interciencia 30 (11): 687–693. [ Links ]
Valadez–Moctezuma E., G. Kahl, J. Ramser, B. Hüttel, y A Rublúo–Islas. 2001. Técnicas moleculares para la caracterización de genomas vegetales (Garbanzo) y algunas aplicaciones potenciales. Fitotec. Mex. 24(1): 103–120. [ Links ]
Vázquez–García, L. M., A. A. Przybyla, T. E. De la Cruz, N. H. Torres, and G. Rodríguez. 2001a. Morphological description of nine botanical varieties of Tigridia pavonia (L.F.) Ker. Gawl. J. Appl. Bot. 75: 14–19. [ Links ]
Vázquez–García L. M., T. H. Norman M., y M. del C. Corona R. 2001b. Oceloxochitl Tigridia pavonia (L.f.) DC. Colección: Ciencias Naturales y Exactas, Serie: Ciencias Agrícolas. Universidad Autónoma del Estado de México. 69 p. [ Links ]
Vicente M. C., y T. Fulton. 2003. Tecnologías de Marcadores Moleculares para Estudios de Diversidad Genética de Plantas: Módulo de Aprendizaje. Illus. Nelly Giraldo. Instituto Internacional de Recursos Fitogenéticos (IPGRI), Roma, Italia. 1:1–52. [ Links ]
Wen, C. S., and J. Y. Hsiao. 2001. Altitudinal genetic differentiation and diversity of Taiwan lily (Lilium longiflorum var. Formosanum; Liliaceae) using RAPD markers and morphological characters. Int. J. Plant Sci. 162(2): 287–295. [ Links ]
Yamagishi, M., H. Abe, M. Nakano, and A. Nakatsuka. 2002. PCR–based molecular markers in Asiatic hybrid lily. Scientia Horticulturae 96: 225–234. [ Links ]
Yeh, F. C., and T. J. B. Boyle. 1999. Population genetic analysis of codominant and dominant markers and quantitative traits. Belgium J. Bot. 129: 157. [ Links ]
Zhou, Z., M. Miwa, and T. Hogetsu. 1999. Analysis of genetic structure of a Suillus grevillei population in a Laris kaemferi stand by polymorphism of inter–simple sequence repeat (ISSR).NewPhytol. 144: 55–63. [ Links ]














