Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Acessos
Acessos
Links relacionados
-
 Similares em
SciELO
Similares em
SciELO
Compartilhar
Agrociencia
versão On-line ISSN 2521-9766versão impressa ISSN 1405-3195
Agrociencia vol.42 no.1 Texcoco Jan./Fev. 2008
Protección vegetal
Estudio del transcriptoma en Capsicum chinense Jacq. resistente al virus huasteco vena amarilla del chile
Study on the transcriptome in Capsicum chinense Jacq. resistant to pepper huasteco yellow vein virus (PHYVV)
Ma. Rosario Gasca–González1 , Yadira Rivera–Herrera3 , Irineo Torres–Pacheco4 , Mario M. González–Chavira2 , Lorenzo Guevara–Olvera1 , Claudia I. Muñoz–Sánchez1 y Ramón G. Guevara–González4
1 Instituto Tecnológico de Celaya. Departamento de Ingeniería Bioquímica. Avenida Tecnológico y Antonio García–Cubas, S/N. Colonia FOVISSSTE. 38010. Celaya, Guanajuato, México.
2 Universidad Autónoma de Querétaro. Facultad de Ingeniería. 76010. Querétaro, Querétaro. México.
3 Programa de Posgrado en Alimentos del Centro de la Republica (PROPAC). Facultad de Química.
4 Facultad de Ingeniería. Universidad Autónoma de Querétaro. 76010. Querétaro, Querétaro, México.
(gerardo@itc.mx).
Recibido: Noviembre, 2006.
Aprobado: Octubre, 2007.
Resumen
Para enriquecer el conocimiento del transcriptoma en la interacción incompatible entre el PHYVV y plantas de chile habanero (Capsicum chinense Jacq.) de la colecta de INIFAP denominada BG–3821, se estudió el perfil de expresión de genes. Estos estudios son fundamentales para el conocimiento a nivel molecular de los mecanismos de señalización para el reconocimiento mutuo y en su caso la respuesta de defensa del hospedante al patógeno, lo que puede contribuir al diseño de nuevas estrategias de protección de los cultivos. Se usó un modelo de expresión diferencial en las plantas infectadas por el PHYVV y la metodología de hibridación sustractiva por supresión (SSH), para obtener una biblioteca de 99 fragmentos de genes (EST) cuya expresión fue inducida específicamente. La secuencia de los EST obtenidos mostró que los genes expresados en esta interacción se pueden agrupar en las siguientes categorías: a) posibles genes de resistencia; b) genes involucrados en rutas de regulación genética; c) genes con función desconocida. Mediante el análisis tipo Northern se confirmó la expresión diferencial de un EST (clona R–100) seleccionado con base en su similitud de secuencia con genes con una función potencial en mecanismos de resistencia. Con nuestros resultados, se presentan las primeras evidencias de genes que posiblemente estén implicados en las rutas de reconocimiento y señalización en C. chinense en respuesta a PHYVV.
Palabras clave: Capsicum chinense, geminivirus, transcriptoma, PHYVV.
Abstract
In order to enrich the knowledge of transcriptome in incompatible interaction between PHYVV and Havana chili pepper plants (Capsicum chinense Jacq.) from the INIFAP collection denominated BG–3821, the profile of gene expression was studied. These studies are essential for the knowledge at molecular level of signaling mechanisms for mutual recognition and, in its case, the defense response of the host to the pathogen, which may contribute to the design of new crop protection strategies. A differential expression model was used in plants infected by PHYVV and subtractive hybridization methodology by suppression (SSH), in order to obtain a library of 99 gene fragments (EST), whose expression was specifically induced. The sequence of the obtained EST's showed that the genes expressed in this interaction may be grouped in the following categories: a) possible resistance genes; b) genes involved in genetic regulation routes; c) genes with unknown function. By Northern–type analysis the differential expression of one EST (R100 clone) was confirmed, selected on the basis of its similarity of sequence with genes playing a potential role in resistance mechanisms. With the results of this research, first evidence of genes is presented, that possibly may be involved in recognition and signaling routes in C. chinense in response to PHYVV.
Key words: Capsicum chinense, geminivirus, transcriptome, PHYVV.
INTRODUCCIÓN
La familia Geminiviridae está integrada por virus de plantas con estructura geminada, su genoma esta formado por uno (monopartita) o dos (bipartita) componentes de ADN de cadena sencilla circular covalentemente cerrado y se dividen en cuatro géneros con base en su rango de hospedantes, insecto vector y organización genómica (Hanley–Bowdoin et al., 1999; Van Regenmortel et al., 2000). El virus huasteco de la vena amarilla del chile (PHYVV, antes PHV) es miembro del género Begomovirus, tiene genoma bipartita, es transmitido por mosquita blanca, e infecta dicotiledóneas incluyendo chile (Garzón–Tiznado et al., 1993; Torres–Pacheco et al., 1996; Anaya–López et al., 2005). El PHYVV es uno de los patógenos virales más importantes en plantas de chile y se ha identificado varias fuentes de resistencia natural en chiles silvestres y criollos recolectados en México (Godínez–Hernández et al., 2001; Anaya–López et al., 2003).
Una fuente identificada de resistencia a geminivi–rus corresponde a la colecta BG–3821 de Capsicum chinense perteneciente al banco de germoplasma del INIFAP (Anaya–López et al., 2003), la cual es un genotipo excelente para estudiar su interacción a nivel molecular con el PHYVV. El mecanismo de resistencia que presenta la colecta BG–3821 de C. chinense a las infecciones del PHYVV se debe probablemente a la restricción del movimiento viral dentro de la planta (Godínez–Hernández et al., 2001). Para evaluar la interacción a nivel molecular entre el PHYVV y C. chinense, se ha estudiado la expresión genética diferencial mediante la técnica de despliegue diferencial de genes, encontrando 45 fragmentos de genes expresados (EST) específicamente en plantas infectadas con PHYVV, entre los que destacaron un EST similar a una metil transferasa (CbiL) y uno similar a una enzima málica de Sorghum bicolor (Anaya–López et al., 2005).
La hibridación sustractiva por supresión (SSH) es una metodología molecular usada para identificar los genes que se expresan diferencialmente (Diatchenko et al., 1996). Con esta técnica se puede identificar genes expresados diferencialmente sin necesidad de clonar previamente el ADN complementario de éstos, así como la normalización y amplificación de los genes expresados a niveles muy bajos (Diatchenko et al., 1996). Asimismo, los fragmentos de ADN complementario (EST) se pueden ordenar en membranas de nylon, lo que mediante un procedimiento de hibridación con sondas moleculares provenientes del ARN de la entidad biológica en estudio, permite estudiar de manera simultánea su transcriptoma.
Mediante el estudio del transcriptoma de C. chinense infectada con el PHYVV será posible identificar los genes que se inducen y probablemente participan en las rutas de reconocimiento y señalización del patógeno en el hospedante. Por tanto, el objetivo del presente trabajo fue ampliar el estudio a nivel molecular de los mecanismos de resistencia en estas plantas de chile a infecciones de PHYVV usando una combinación de SSH y arreglos de DNA en membranas de nylon.
MATERIALES Y MÉTODOS
Material vegetal
Se usaron plantas autopolinizadas de la colecta BG–3821 de C. chinense Jacq., proporcionadas por la Unidad de Biotecnología del Campo Experimental Bajío del CIRCE–INIFAP en Celaya, Guanajuato, México. Antes de usarse en los estudios de expresión diferencial, se verificó la ausencia del PHYVV en ellas mediante PCR usando los oligonucleótidos específicos para este virus: 240 (5'–GGCTTATTTGTAATAAGAG–3') y 241 (5'–GAATTA–AAGGTACATGGAC–3'), los cuales amplifican un fragmento de ADN del componente A del virus de 350 pb (Torres–Pacheco et al., 1996; Anaya–López et al., 2003). Para la PCR se usó ADN genómico de las plantas de C. chinense BG–3821, obtenido de hojas superiores e inferiores siguiendo el protocolo descrito por Dellaporta et al. (1983). En plantas infectadas con PHYVV se realizó la detección del virus 5 d después de su inoculación.
Inoculación del virus
La inoculación del ADN del PHYVV en 20 plantas de C. chinense se hizo mediante biobalística, usando un sistema de bombardeo de micropartículas PDS 1000 (Dupont, Wilmington, Delaware). Se usaron clonas diméricas del ADN de PHYVV o ADN del plásmido bluescript SK+ (Stratagene, La Jolla, California) para la inoculación. La cantidad de ADN del virus, la forma de inocular las plantas y su incubación post–inoculación se hicieron según lo descrito por Anaya–López et al. (2005). Se usaron 10 plantas como testigo y se les administró exclusivamente ADN del plásmido bluescript SK+.
Aislamiento de ARN
El ARN total tanto de las plantas infectadas como de las testigo se obtuvo mediante el protocolo de RNEASY (QIAGEN, Hilden, Alemania). La integridad y tamaño del ARN se analizó mediante electroforesis en geles de agarosa con formaldehido. La cuantificación y pureza se midió espectrofotométricamente mediante la relación de absorbancia (260/280 nm).
Síntesis, amplificación y purificación del ADN complementario (ADNc)
Se usó 1 µg de ARN total de cada condición en las plantas (problema y testigo), como templado para sintetizar la primer cadena de ADNc con la transcriptasa reversa Superscript II (Life Technologies, Rockville, MD, USA) y el protocolo de Síntesis de ADNc SMARTTM, siguiendo las instrucciones del proveedor (Clontech, Palo Alto, CA, USA). El ADNc se amplificó mediante la PCR de larga distancia (LD–PCR) con 15, 18, 21, 24 y 27 ciclos separadamente (Diatchenko et al., 1996) y se analizó mediante electroforesis en geles de agarosa 1.2% para identificar el número de ciclos óptimo para tener la cantidad adecuada de productos de PCR para elaborar la biblioteca de ADNc. Como testigo en este método se usó ARN total de placenta. Para la purificación del ADNc se usaron columnas CROMA–SPIN 1000 (Clontech, Palo Alto, CA, USA).
Aislamiento de fragmentos de ADNc expresados diferencialmente
La hibridación substractiva por supresión (SSH) se hizo con el protocolo de Clontech (Clontech PCR–SelectTM cDNA subtraction kit, Clontech, Palo Alto, CA, USA). Los ADNc problema y testigo fueron digeridos con la enzima Rsa I, la cual es una enzima de restricción que reconoce cuatro pares de bases (5'–GTAC–3') y deja extremos romos en el ADN digerido. El ADN problema se dividió en dos alícuotas y cada una se ligó en forma separada con los adaptadores 1 y 2 resultando en dos poblaciones de ADNc problema. Una pequeña cantidad de cada población problema y un exceso de ADNc testigo (5 µg) fueron mezclados, desnaturalizados por calor, e hibridados por 8 h a 68 °C. Las dos muestras de esta primera hibridación se combinaron e hibridaron con un excedente de ADNc testigo desnaturalizado toda la noche a 68 °C. Luego se hizo una PCR para amplificar los ADNc que representaban genes expresados diferencialmente. Un segundo PCR se hizo con oligonucleótidos anidados (1 R y 2 R) para reducir el nivel de amplificación inespecífica (Clontech PCR–SelectTM cDNA subtraction kit). Los productos de amplificación de la segunda PCR se sometieron a electroforesis y los fragmentos mayores a 500 pb se cortaron del gel de agarosa usando un bisturí estéril y se purificaron con un protocolo QIAEXII (Qiagen, Hilden, Alemania).
Clonación y selección de fragmentos sustraídos de ADNc
Se ligaron 2 µL de los fragmentos de ADNc amplificados por PCR con 1 µL del vector de clonación pCR2.1–TOPO según las instrucciones del proveedor (Invitrogen, Carlsbad, CA, USA). Se transformaron 2 µL de estas reacciones de ligación en 50 µL de células químicamente competentes de Escherichia coli cepa TOP 10. El cultivo de transformación se sembró en cajas de Petri conteniendo medio Luria Bertani (LB)/kanamicina/IPTG/X–gal, y se seleccionaron las colonias blancas. Cada colonia blanca se cultivó en medio LB/kanamicina/ampicilina, incubada 12 h a 37 °C y 250 rpm, y en los plásmidos se analizó la presencia de insertos usando la enzima de restricción Eco RI (Invitrogen, Carlsbad, CA, USA). Cada colonia modificada se cultivó separadamente en el mismo medio y las mismas condiciones, y se almacenó en viales criogénicos conteniendo 500 µL de cada cultivo y 500 µL de glicerol 100% esterilizado. La colección de colonias modificadas (biblioteca de genes expresados diferencialmente) se mantuvo a –80 °C.
Construcción de arreglos de ADNc
Se colocaron 5 µg de cada plásmido recombinante de la biblioteca de genes en membranas de nylon (7X10 cm) cargadas positivamente (BrightStarTM–Plus, Ambion Inc, Austin, TX, USA). Para construir arreglos de clones se usó un dispositivo multicopiador conectado a bomba de vacío (Hoefer PR 648, Amersham Biosciences, Buckinhamshire, UK).
Preparación y marcaje de sondas de ADNc e hibridación de membranas
Las replicas de la biblioteca de genes se hibridaron mediante análisis tipo Southern (Sambrook et al., 1989) con sondas obtenidas de ADNc problema o testigo. Estas sondas se generaron incorporando dUTP–11–fluoresceína con el protocolo descrito en Gene Images CDP–Star Random prime labeling module (Amersham Pharmacia Biotech Inc, Piscataway, NJ, USA). La detección de la sonda se hizo mediante un conjugado antifluoresceína–fosfatasa alcalina y el reactivo de detección CDP–Star (Amersham Pharmacia Biotech Inc, Piscataway, NJ, USA).
Análisis tipo Northern
El análisis tipo Northern se realizó como se describe en Anaya–López et al. (2005).
Secuenciación del ADNc y comparación con bases de datos
Las secuencias de nucleótidos de fragmentos de genes inducidos específicamente por infecciones por PHYVV en las plantas de C. chinense se determinaron usando un secuenciador ABI PRISM 310 (Gene Analyzer; Perkin Elmer, Norwalk, CT, USA). Se hizo análisis de comparación de secuencias con bases de datos realizados con el algoritmo blastx (Altschul et al., 1990) del Centro Nacional para Información en Biotecnología (NCBI). La secuencia de la clona R100, correspondiente a una proteína tipo germina, se depositó en la base de datos del NCBI con el número de accesión DQ677335.
RESULTADOS Y DISCUSIÓN
Detección de PHYVV en plantas inoculadas
La detección de PHYVV en las plantas infectadas y testigo se muestra en la Figura 1. El virus se detectó en la parte basal y en la parte apical de las plantas, lo que indica que 5 d fueron suficientes para que el virus se moviera por la planta. El virus estuvo presente en la planta pero ésta no presentó síntomas, confirmando los resultados de estudios anteriores (Godínez–Hernández et al., 2001; Anaya–López et al., 2003, 2005). La eficiencia de la inoculación por biobalística fue 100%, dado que se detectó el virus en todas las plantas.

Aislamiento de ARN y síntesis de ADNc para SSH
El resultado de la extracción de ARN se observa en la Figura 2. El ARN es de buena calidad y cantidad, y su extracción fue homogénea y reproducible a partir de diferentes individuos. Las extracciones de este tipo fueron adecuadas para la síntesis posterior de ADNc para realizar la SSH.

Para la síntesis de ADNc de cadena sencilla se usó ARN total de la condición testigo y problema (Figura 2); una vez sintetizada la primera cadena de ADNc se procedió con la estandarización del número de ciclos de PCR necesarios para la amplificación de ADNc de doble cadena (Figura 3). Los ciclos de PCR evaluados para tal estandarización referente a las condiciones testigo y problema se muestran en la Figura 3. A los 27 ciclos se obtuvo la condición más adecuada para la sustracción, pues se encontraron mejor representados los ADNc de ambas condiciones ya que el barrido es homogéneo y en concentración adecuada (Figura 3). La condición anterior asegura que los ADNc de doble cadena permanecerán en la fase exponencial de la amplificación, permitiendo que la mayor parte de los ARN mensajeros estén representados. Cuando el número de ciclos de PCR es mayor al óptimo (sobreciclado), el ADNc es un templado muy pobre. Un número de ciclos menor al óptimo (subciclado) resulta en un bajo rendimiento del producto de PCR (Diatchenko et al., 1996).

Hibridación substractiva por supresión por PCR
La población de ADNc de planta testigo y problema a los 27 ciclos mostrados en la Figura 3, se purificaron y se digirieron con la enzima Rsa I de tal manera que cada ADNc queda dividido en múltiples fragmentos. Después de esta digestión el ADNc se volvió a purificar y se sometió a hibridación sustractiva en condiciones de supresión. La condición problema (planta con virus) se dividió en dos alícuotas y en cada una se ligó un diferente adaptador como se describe en Materiales y Métodos. En el carril 2 de la Figura 4 se observa en una corrida electroforética el ADNc resultado de la sustracción y sólo están los fragmentos de genes expresados diferencialmente. Se observan los barridos que corresponden a ADNc de diversos tamaños, enriquecidos principalmente en la zona de 500 pb y 700 pb, lo cual es similar a otros resultados usando la misma metodología para construir bibliotecas de ADNc específicas a estrés osmótico en plantas de Haloxylon ammodendron (Jiang et al., 2004). Para construir los bancos substractivos de C. chinense a partir de los fragmentos de ADNc expresados diferencialmente, se continuó con los protocolos descritos en Materiales y Métodos. Se seleccionaron las zonas de alrededor de 500 y 700 pb; las bandas se cortaron del gel de agarosa con un bisturí para eluir el ADN, se procedió con la ligación en el vector pCR 2.1 TOPO® y se transformaron células químicamente competentes de E. coli.

El banco sustractivo quedó integrado por 99 clonas con tamaños entre 250 y 750 pb.
Expresión diferencial de genes
El ADN de cada clona que constituyó el banco de genes expresados diferencialmente se transfirió a membranas de nylon y se hibridó con ADNc marcado y proveniente tanto de ARN de plantas infectadas como del testigo. Se observó que las 99 clonas obtenidas en el banco de genes mostraron un nivel de expresión mayor en presencia de la sonda proveniente de la planta problema, lo que indica la expresión diferencial de los genes correspondientes en presencia del PHYVV en la planta de C. chinense. En la Figura 5 se muestra el patrón de expresión de arreglos de ADNc de 47 clonas del banco sustractivo obtenido. Las otras 52 clonas mostraron también un patrón de expresión diferencial de inducción en presencia del virus en la planta (no mostrado).
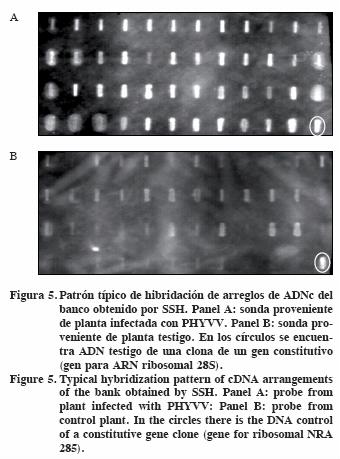
Análisis de homología de secuencia de las clonas del banco sustractivo
Después del análisis de hibridación de arreglos, se seleccionaron 16 clonas con base en su mayor definición en el patrón de expresión inducible por el PHYVV y se secuenciaron. Las secuencias de estas clonas se compararon con las de la base de datos del NCBI mediante el algoritmo blastx, y los resultados de las posibles funciones de los genes encontrados se muestran en el Cuadro 1. Con base en la homología mostrada por las 16 clonas secuenciadas del banco sustractivo, su posible participación en la interacción incompatible de las plantas de C. chinense con el PHYVV se puede agrupar en: 1) posibles genes involucrados en la resistencia y el estrés biótico de la planta (clonas R 21, 22, 31, 35, 52, 55, 80, 81, 100 y YRH 6); 2) posibles genes regulatorios de expresión genética (clonas YRH3, YRH1, YRH5); 3) genes con función desconocida hasta el momento (clonas R 50, 57, 60).
Expresión diferencial de un transcrito seleccionado del banco sustractivo
Con la finalidad de verificar con mayor detalle la expresión inducible por PHYVV en las plantas de C. chinense evaluadas en este trabajo, se seleccionó la clona R100 (número de accesión DQ677335) debido a que presentó similitud con un grupo de proteínas tipo germina que presentan actividad superóxido dismutasa (Khuri et al., 2001; Druka et al., 2002; Kim et al., 2004) y que se han relacionado con una función directa en la respuesta de plantas a diferentes tipos de estrés biótico y abiótico (Carter y Thornburg, 2000; Druka et al., 2002). Se ha sugerido una función directa de proteínas tipo germina en la supresión del mildiú del trigo ocasionado por Blumeria graminis f. sp. tritici por expresión transitoria con un promotor constitutivo (Schweizer et al., 1999).
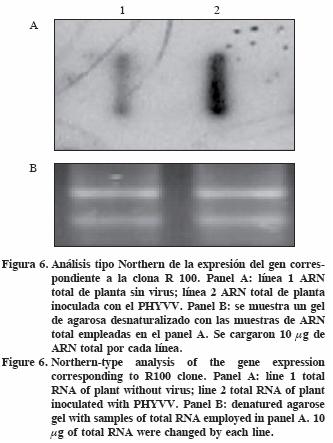
Asimismo, se ha demostrado una función importante de las proteínas tipo germina en la defensa de plantas contra insectos herbívoros (Lou y Baldwin, 2006). En la Figura 6 se muestra el resultado del análisis tipo Northern realizado para corroborar la expresión diferencial del gen correspondiente a la clona R 100. Es evidente la inducción del gen correspondiente a la clona R 100 en plantas inoculadas con el PHYVV, mientras en plantas testigo el nivel de expresión fue comparativamente inferior.
CONCLUSIONES
Para estudiar el transcriptoma de plantas de C. chinense BG–3821 infectadas con el PHYVV bajo un modelo de expresión diferencial, el uso de SSH y arreglos de ADN, resultó ser una herramienta sencilla, rápida y efectiva en el aislamiento de EST inducidos específicamente por el PHYVV. De las 99 clonas que constituyeron el banco de EST obtenido en este trabajo, 16 fueron secuenciados y 56% de estos ESTs mostraron homología con genes que sugieren una participación en mecanismos de resistencia de plantas a patógenos. Dentro de los EST con homología a genes de resistencia destacaron los correspondientes a las clonas R 52 y R 100, que mostraron una similitud con proteínas tipo germina (GLP), las cuales se han relacionado directamente con el control de enfermedades como el mildiú del trigo ocasionado por Blumeria graminis f. sp. tritici. Será interesante estudiar la especificidad de la expresión de los EST encontrados en este trabajo en respuesta a diversos tipos de estrés biótico o abiótico, así como aislar los ADNc completos, para dilucidar con mayor detalle la función de estos genes en las respuestas de C. chinense BG–3821 a diferentes patógenos y agentes abióticos.
AGRADECIMIENTOS
Los autores agradecen el financiamiento por parte de SEP–CONACYT (CONACYT–2002–C01–41818/A–1), Fondo mixto CONACYT–Gobierno del Estado de Guanajuato (FOMIX–GTO–2005–C02–52), PRECI 99999 del INIFAP y al proyecto DGEST (clave 423.05–P). Ma. Del Rosario Gasca–González y Yadira Rivera–Herrera agradecen a CONACYT por el apoyo de beca de posgrado.
LITERATURA CITADA
Altscchul S. F, W. Gish, W. Miller, E. W. Myers, and D. J. Lipman. 1990. Basic local alignment search tool. J. Molecular Biol. 215, 403–410. [ Links ]
Anaya–López, J. L, E. Pérez–Mora, I. Torres–Pacheco, C. I. Muñoz–Sánchez, L. Guevara–Olvera, M. M. González–Chavira, N. Ochoa–Alejo, R. F. Rivera–Bustamante, and R. G. Guevara–González. 2005. Inducible gene expresión by Pepper huasteco virus in Capsicum chinense plants with resistance to geminivirus infections. Can. J. Plant Pathol. 27: 276–282. [ Links ]
Anaya–López, J. L, M. González–Chavira, J. L. Pons–Hernández, J. A. Garzón–Tiznado, I. Torres–Pacheco, R. F. Rivera–Bustamante, S. Hernández–Verdugo, R. G. Guevara–González, C. I. Muñoz–Sánchez, and L. Guevara–Olvera. 2003. Resistance to geminivirus mixed infections in Mexican wild peppers. Hortscience 38: 251–255. [ Links ]
Dellaporta, S. L., J. Word, and J. B. Hicks. 1983. A plant DNA minipreparation: version II. Plant Mol. Biol. Rep. 1: 19–21. [ Links ]
Hanley–bowdoin, L, S. B. Settlage, B. M. Orozco, S. Nagar, and D. Robertson. 1999. Geminiviruses: model for plant replication, transcription and cell regulation. Critical Rev. Plant Sci. 18: 71–106. [ Links ]
Diatchenko, L, Y. F. Lau, A. P. Campbell, A. Chenchik, F. Moqadam, B. Huang, S. Lukyanov, K. Lukyanov, N. Gurskaya, E. D. Sverdlov, and P. D. Siebert. 1996. Suppression subtractive hybridization: a method for generating differentially regulated or tissue–specific cDNA probes and libraries. Proc. Natl. Acad. Sci. USA. 93: 6025–6030. [ Links ]
Druka, A, D. Kudrna, C. G. Kannangara, D. von Wettsein, and A. Kleinhofs. 2002. Physical and genetic mapping of barley (Hordeum vulgare) germin–like cDNAs. Proc. Natl. Acad. Sci. USA. 99: 850–855. [ Links ]
Garzon–Tiznado, J. A., I. Torres–Pacheco, J. T. Ascencio–Ibañez, L. Herrera–Estrella, and R. F. Rivera–Bustamante. 1993. Inoculation of peppers with infectious clones of a new geminivirus by a biolistic procedure. Phytopathology 53: 514–521. [ Links ]
Godínez–Hernández, Y., J. L. Anaya–López, R. Díaz–Plaza, M. González–Chavira, I. Torres–Pacheco, R. F. Rivera–Bustamante, and R. G. Guevara–González. 2001. Characterization of resistance to pepper huasteco geminivirus in chili peppers from Yucatán, México. Hortscience 36: 139–142. [ Links ]
Jiang, X–C., X–H. Guo, X–L. Pan, and S–Q. Song. 2004. Construction and differential screening of a cDNA library specific to osmotic stress of Haloxylon ammodendron seedlings. J. Biochem. Mol. Biol. 37: 527–532. [ Links ]
Khuri, S, F. T. Bakker, and J. M. Dunwell. 2001. Phylogeny, function, and evolution of the cupins, a structurally conserved, functionally diverse superfamily of proteins. Mol. Biol. Evol. 18: 593–605. [ Links ]
Kim, H. J., T. C. Pesacreta, and B. A. Triplett. 2004. Cotton–fiber germin–like protein. II: Immunolocalization, purification, and functional analysis. Planta 218: 525–535. [ Links ]
Lou, Y., and I. T. Baldwin. 2006. Silencing of a germin–like gene in Nicotiana attenuate improves performance of native herbivores. Plant Physiol. 140: 1126–1136. [ Links ]
Sambrook J., E. F. Fritsch, and T. Maniatis. 1989. Molecular Cloning: A Laboratory Manual. Cold Spring Harbor Laboratory Press. 1531 p. [ Links ]
Schweizer, P., A. Christoffel, and R. Dudler. 1999. Transient expression of members of the germin–like gene family in epidermal cells of wheat confers disease resistance. The Plant J. l 20: 541–552. [ Links ]
Torres–Pacheco, I., J. A. Garzón–Tiznado, J. K. Brown, A. Becerra–Flora, and R. F. Rivera–Bustamante. 1996. Detection and distribution of geminiviruses in Mexico and Southern United Status. Phytopathology 86: 1186–1192. [ Links ]
Van Regenmortel, M. H. V., C. M. Fauquet, D. H. L. Bishop, E. Carstens, M. Estes, S. Lemon, J. Maniloff, M. A. Mayo, D. McGeoch, C. R. Pringle, and R. B. Wickner (eds). 2000. Virus Taxonomy. Seventh Report of the International Committee on Taxonomy of Viruses. Academic Press, San Diego, California. pp: 367–369. [ Links ]














