Introducción
El género Cnidoscolus Pohl (Euphorbiaceae) se distribuye desde los Estados Unidos de América hasta el norte de Argentina siendo México, con 25 especies endémicas, su segundo centro de diversidad (Maya-Lastra & Steinmann, 2018, 2019). En la Península de Yucatán existen tres especies: C. aconitifolius (Mill.) I.M. Johnst., C. souzae McVaugh y C. urens (L.) Arthur (Carnevali et al., 2010; Schikorr, 2017). Mientras que C. aconitifolius y C. souzae tienen una distribución restringida al área de la Península de Yucatán, Belice y Guatemala; C. urens se distribuye desde México hasta Argentina (Schikorr, 2017). La Chaya fue clasificada por Breckon (1975) como C. aconitifolius (Mill.) I. M. Johnst. ssp. aconitifolius; como representante de su género, esta posee una reproducción monoica, con flores masculinas y femeninas separadas temporalmente (Schikorr y Rodríguez, 2018). Sin embargo, en la Chaya cada una de estas flores posee órganos reproductivos no funcionales del sexo opuesto (esto es, la flor femenina posee estambres vestigiales y la masculina posee pistilos vestigiales; en ambos casos, estas estructuras no poseen una función reproductiva) lo cual limita la formación de semillas y obliga a la Chaya a una propagación clonal (por esquejes) (Ross-Ibarra y Molina-Cruz, 2002). Solo en pocos casos se ha reportado la formación de frutos y semillas en la Chaya (Cifuentes et al., 2010; Munguía-Rosas et al., 2019; Munguía‐Rosas y Jácome‐Flores, 2020). Con relación a C. aconitifolius silvestre, la Chaya exhibe un aislamiento reproductivo alto, con cierto grado de asimetría: mientras barreras pre-polinización (producción diferencial de polen y especificidad del polinizador) existen solamente en C. aconitifolius silvestre, barreras post-polinización (incompatibilidad polen-pistilo y/o fallas en la producción de frutos) existen tanto en la Chaya como en C. aconitifolius silvestre (Munguía‐Rosas y Jácome‐Flores, 2020). Todas estas características reproductivas de la Chaya y C. aconitifolius silvestre limitan fuertemente el flujo genético entre ambos taxa.
Algunas especies de Cnidoscolus son de interés humano por su potencial nutrimental y medicinal (Markus et al., 2016; Sanchez-Hernandez et al., 2017; Moura et al., 2019), pero es la Chaya la más importante al ser la única planta cuyas hojas son consumidas como verdura (Ebel et al., 2019); estas hojas contienen glucósidos cianogénicos que pueden formar ácido cianhídrico (HCN) al hidrolizarse, generando así toxicidad, pero este compuesto es eliminado con el vapor y no se retiene en el agua de cocción (Fan y Zhou, 2010). La Chaya se distribuye en el sureste de México y la Península de Yucatán, así como en Guatemala, Belice y Honduras (Steinmann, 2002). Es una planta representativa de la cultura Maya, la cual ha sido consumida desde tiempos prehispánicos (Ross-Ibarra y Molina-Cruz, 2002). En la Península de Yucatán, la Chaya es cultivada en los solares mayas, lugar en donde también suelen encontrarse plantas silvestres de C. aconitifolius (Tzin-tzin chay en maya). Las comunidades Mayas reconocen a la Chaya de su contraparte silvestre, ya que esta posee una cantidad mucho menor de pelos urticantes en hojas y tallos (Ebel et al., 2019; Munguía-Rosas et al., 2019). A la Chaya se le considera un cultivo subutilizado debido al conocimiento general limitado que existe sobre ella, su escasa representación en colecciones ex situ y la falta de programas de conservación (Galluzzi y López-Noriega, 2014); sin embargo, en los últimos años su importancia ha incrementado en el mundo gracias a las facilidades de su cultivo, su valor nutrimental alto y sus usos medicinales potenciales (Markus et al., 2016; Moura et al., 2019). Hoy en día sigue existiendo un vacío de información sobre la diversidad genética de la Chaya, cultivo que se considera formado por un solo clon sin que se existan estudios moleculares suficientes que confirmen esto. Este vacío de información abarca también a sus parientes silvestres, los cuales podrían representar importantes reservorios genéticos de utilidad potencial para el mejoramiento de este cultivo (Hajjar & Hodgkin, 2007; Ford-Lloyd et al., 2011; Dempewolf et al., 2014).
Ross-Ibarra y Molina-Cruz (2002) reportaron cuatro variedades locales de Chaya (i.e. agrupaciones de plantas dentro de un solo taxon botánico del rango más bajo conocido, definido por la reproducibilidad de sus características distintivas; FAO, 2009) en la Península de Yucatán: Chayamansa, Redonda, Estrella y Picuda; indicando que: a) Chayamansa es la de mayor distribución y abundancia, b) Redonda es poco conocida y poco abundante y, c) Estrella y Picuda, aunque colectadas en huertos familiares, son morfológicamente similares a las plantas silvestres de C. aconitifolius. Molina-Cruz et al. (1997) y Cifuentes et al. (2010), también reportaron cuatro variedades locales de Chaya en Guatemala: Mansa, Estrella, Picuda y Plegada; aunque varios de estos nombres coinciden con los reportados por Ross-Ibarra y Molina-Cruz (2002), una revisión de las fotos de los ejemplares y las características morfológicas reportadas en estos estudios permitieron observar que estos no coinciden con el mismo taxa descrito. Así, mientras Ross-Ibarra y Molina-Cruz (2002) reconocen a un taxon como Redonda, Molina-Cruz et al. (1997) y Cifuentes et al. (2010) nombran a este taxon como Mansa. Munguía-Rosas et al. (2019) basados en información morfológica, reportaron para C. aconitifolius solo una variedad cultivada (Chay) y otra variedad silvestre (Tzin-tzin chay) en la Península de Yucatán, señalando que ambas variedades presentan diferencias significativas pero que algunas plantas cultivadas cayeron en el rango de variación fenotípica de las plantas silvestres. Ross-Ibarra, (2003) propuso que la Chaya se domesticó en Yucatán, México, a partir de C. aconitifolius, considerando que: a) allí está el mayor conocimiento etnobotánico y usos de la Chaya, b) las plantas domesticadas presentan una reducción del 34.3% en diversidad genética como resultado de un efecto fundador por domesticación, y c) se encontraron solo tres clones en la variedad local Redonda y uno en la variedad local Chayamansa, siendo estas las únicas variedades que pueden considerarse como domesticadas por ser diferentes morfológicamente a las plantas silvestres de C. aconitifolius.
Solo dos estudios han aportado información sobre la diversidad genética de la Chaya y de algunos de sus parientes silvestres utilizando marcadores moleculares. Ross-Ibarra (2003), usando 12 loci de isoenzimas, reportó valores bajos de diversidad genética (H E) para las variedades locales de Chaya de la Península de Yucatán: Chayamansa (H E= 0.20), Redonda (H E= 0.23), Estrella (H E= 0.34), Picuda (H E= 0.30), así como para C. aconitifolius silvestre (H E= 0.30). Cifuentes et al. (2010), usando marcadores AFLP (Amplified Fragment Leght Polimorphism), reportaron que la Chaya en Guatemala muestra una similitud genética del 25% al 35% entre variedades locales y del 50% al 60% dentro de estas variedades, valores que indican variación genética media; los autores no reportaron valores de diversidad genética para estas variedades. Considerando este vacío de información y la importancia cada vez mayor del cultivo de la Chaya y de los parientes silvestres de las especies domesticadas, los objetivos de este trabajo fueron: 1) evaluar la diversidad genética de la Chaya cultivada en 10 pueblos Mayas, 2) realizar un análisis preliminar de la diversidad genética de C. aconitifolius y C. souzae, 3) determinar las relaciones genéticas entre la Chaya y estos dos parientes silvestres. Todos los individuos evaluados fueron colectados en Yucatán, México, el posible centro de domesticación de la Chaya, los cuales fueron caracterizados usando marcadores ISSR (Inter Simple Secuence Repeat).
Material y métodos
Material vegetal
El estudio incluyó a 100 individuos de Chaya colectados en 10 pueblos mayas (10 individuos en promedio por pueblo) de Yucatán, México (Fig. 1, Anexo 1); cada uno de estos pueblos fue considerado como una población de Chaya. El trabajo de campo permitió determinar que la Chaya colectada es propagada por esquejes, los cuales son regalados y/o intercambiados entre parientes o vecinos considerando la frondosidad y la ausencia de plagas/enfermedades en las plantas madre. Considerando esto, y para lograr una mayor representatividad genética, los individuos seleccionados procedieron de 10 solares diferentes ubicados a lo largo y ancho de cada pueblo. También, se incluyeron ocho individuos silvestres de C. aconitifolius y nueve de C. souzae donados por el Dr. Germán Carnevali, curador del Herbario CICY del Centro de Investigación Científica de Yucatán (Anexo 1, Fig. 1). En este artículo usamos el término Chaya para hablar de C. aconitifolius ssp. aconitifolius, sin importar si los individuos analizados son de diferentes variedades locales, a menos que se especifique el nombre de estas; y nos referiremos a C. aconitifolius, cuando hablemos solamente de plantas silvestres de esta especie. Fotos de los taxa analizados se presentan en la Figura 2.

Fig. 1 Sitios de colecta de las plantas de Chaya, Cnidoscolus aconitifolius y C. souzae, en el estado de Yucatán, México.
Extracción de ADN y Técnica de ISSR
El ADN se extrajo a partir de tejido de hojas usando el protocolo de Echevarría-Machado et al. (2005). La calidad del ADN se verificó con electroforesis en geles de agarosa al 1 %, teñidos con bromuro de etidio (10 mg/ml) usando una cámara horizontal LIFE TECHNOLOGIES-Horizon 11-14 y una fuente poder EC-105 a una carga de 100 volts durante 30 minutos, las bandas obtenidas se observaron en un transiluminador de luz UV (Transilluminator UV, Dinco & Rhenium Industries). Se probaron 16 iniciadores de ISSR evaluados por Martínez-Castillo (2005), de los cuales se seleccionaron los seis más polimórficos (Cuadro 2). Cada banda se consideró como un locus independiente y las bandas polimórficas se marcaron como ausente (0) o presente (1). Con el fin de asegurar una lectura correcta de de los geles, solo bandas claramente reproducibles fueron consideradas (sin importar su intensidad) y, cuando fue necesario, se realizaron réplicas independientes para el caso particular de los loci polimórficos.
Cuadro 2 Características de los 6 iniciadores de ISSR usados en la caracterización molecular de la Chaya y sus parientes silvestres (C. aconitifolius y C. souzae) de Yucatán, México.
| Iniciador | Secuencia de iniciador | TA (°C) |
|---|---|---|
| T06 | AGA GAG AGA GAG AGA CT | 51°C |
| C03 | TGT CAC ACA CAC ACA CAC | 51°C |
| A04 | GCA ACA CAC ACA CAC ACA | 52°C |
| P15 | GACA GACA GACA RG | 42°C |
| P16 | YR GACA GACA GACA | 42°C |
| P17 | GACAC GACAC GACAG RG | 40°C |
TA: Temperatura de alineamiento en grados centígrados
La amplificación se realizó con la técnica de Reacción en Cadena de la Polimerasa (PCR) en un volumen final de 20μl de reacción. Cada reacción de amplificación consistió de 1X Tris-HCl, 2 mM MgCl2, 0.2 µM dNTPs, 1 µM de iniciador, 1 unidad de Taq polimerasa (Promega, Madison, WI) y 100 ng de ADN templete. La amplificación se realizó en un termociclador GeneAmp PCR System 9700 (Applied Biosystems, Foster City, USA) bajo las siguientes condiciones: 7 min a 94 °C para una desnaturalización inicial, seguido por 34 ciclos consistentes de 30 segundos a 94 °C de desnaturalización, 45 segundos a 51-54 °C (dependiendo del iniciador) de alineación y 2 minutos de extensión, y 5 minutos a 72 °C para una extensión final. Posteriormente, cuatro microlitros de formamida conteniendo 0.45% de azul de bromofenol y 0.25% de xileno-cianol se agregaron a cada producto PCR. De esta mezcla, cuatro microlitros se cargaron en geles no desnaturalizantes de bis-acrilamida al 6%, conteniendo 3 M urea y buffer TBE 1x (100 mM Tris-borato, pH 8.0, 2 mM EDTA) (Zietkiewicz, et al., 1994). La electroforesis se realizó a 300 V (SQ3 Sequence Hoeffer) y los productos amplificados se visualizaron con la técnica de tinción con plata (Bassam et al., 1991) usando un marcador de 123 pares de bases como referencia de tamaño de las bandas amplificadas.
Análisis de datos
Considerando la naturaleza dominante de los marcadores ISSR, los análisis no tomaron en cuenta equilibrio Hardy-Weinberg (H-W) evitando así los problemas comunes asociados con este tipo de marcadores (Culley y Wolfe, 2001; Lynch y Milligan, 1994); además, considerando que la Chaya se propaga de forma clonal, no hay poblaciones que estén en equilibrio H-W. En este estudio asumimos que: 1) no hubo co-migración de alelos de diferentes loci, 2) alelos compartidos por dos individuos descienden de un ancestro común y, 3) cada locus consiste de solamente dos alelos que segregan por herencia Mendeliana (Martínez-Castillo et al., 2008).
Patrón de agrupamiento y estructura genética
Para evaluar cómo está organizada la diversidad genética de la Chaya y sus parientes silvestres en Yucatán, se aplicaron cinco métodos: 1) Prueba de asignación de individuos con el software Structure (Pritchard et al., 2000), usando el modelo Admixture con frecuencias alélicas correlacionadas, con 100000 iteraciones como periodo de quema y 200000 iteraciones posteriores al periodo de quema. Diez simulaciones independientes se corrieron para cada valor de K examinado (K = 1 a K = 5). El valor óptimo de K se obtuvo con el método de Evanno et al., (2005) implementado en el programa STRUCTURE HARVESTER (Earl y vonHoldt, 2012). Las gráficas finales se obtuvieron con base en la K óptima y en el número de taxa estudiados. 2) Análisis con Neighbor-Joining (Saitou y Nei, 1987), usando 1000 boostraps y el software DARwin v6 (Perrier y Jacquemoud-Collet, 2006). 3) Análisis de Coordenadas Principales (PCoA), técnica que permite explorar los patrones de agrupamiento de las muestras sin un criterio a priori de los datos, usando cada alelo como una variable independiente. 4) Análisis Molecular de Varianza (AMOVA) jerárquico, considerando la existencia de tres grupos: Chaya, Cnidoscolus aconitifolius y C. souzae; este análisis no asume equilibrio Hardy-Weinberg y es recomendado para marcadores dominantes por sobre el uso de F ST. 5) Comparación de distancias genéticas no sesgadas de Nei, (1978) entre la Chaya y sus parientes silvestres. Para los tres últimos análisis se usó el programa GenALEx 6 (Peakall y Smouse, 2012).
Diversidad genética
Considerando que el uso de marcadores ISSR no permite la estimación directa de las frecuencias alélicas, en este trabajo solo se calcularon el porcentaje de loci polimórficos (%P) al nivel del 5% y la Heterocigosidad esperada (H E), usando para ello un enfoque Bayesiano (Zhivotovsky, 1999). Para el cálculo de ambos estimadores se usó el programa AFLP-SURV V.1 (Vekemans, 2002).
Resultados
Patrón de agrupamiento y estructura genética
El método de Evanno mostró un valor óptimo de K=2. La Figura 3-A muestra el patrón de agrupamiento generado con Structure y una K=2, en esta se observa que un grupo integró a todos los individuos de Chaya (en color rojo) y el otro a todos los individuos de C. aconitifolius y C. souzae (en color verde). Al interior del grupo rojo se observan dos individuos (en color rojo/verde) que comparten ancestría con uno de sus parientes silvestres, estos dos individuos fueron colectados en los pueblos de Acanceh y Euan. Una comparación con la nomenclatura empleada por Ross-Ibarra y Molina-Cruz (2002) mostró que 98 individuos de Chaya pertenecen a la variedad local Chayamansa y que los dos individuos con ancestría compartida pertenecen a la variedad local Redonda. La Figura 3-B muestra el patrón de agrupamiento generado con Structure y una K= 3, claramente se observan los tres grupos de plantas analizadas: Chaya en rojo, C. souzae en azul y C. aconitifolius en verde; así como que los dos individuos de la variedad local Redonda comparten ancestría con C. aconitifolius.

Fig. 3 Análisis de asignación de 100 individuos de Chaya, 8 de Cnidoscolus aconitifolius y 9 de C. souzae de Yucatán, México, usando el software Structure (K = 2 y K = 3) y 158 loci de ISSR.
El PCoA mostró una separación clara entre la Chaya y sus parientes silvestres; así como entre las dos variedades locales de Chaya (Fig. 4). El PCoA también mostró una sobreposición casi perfecta de los 98 individuos de la variedad local Chayamansa (solo se observa un ligero desfase de estos individuos); así como una separación ligera, aunque clara, entre los dos individuos de la variedad local Redonda. Para Cnidoscolus aconitifolius y C. souzae, todos los individuos se separaron entre sí de manera visible. La primera y segunda coordenadas explicaron el 80.67% y el 8.1% de la variación total, respectivamente.

Fig. 4 Análisis de Coordenadas Principales de 100 individuos de Chaya, 8 de C. aconitifolius y 9 de C. souzae, colectados en Yucatán, México, usando 158 loci de marcadores ISSRs. Los colores de los símbolos corresponden a los del análisis de Structure (K = 3). Los rombos en bicolor (rojo-verde) corresponden a la Chaya variedad Redonda.
El patrón de agrupamiento generado por el análisis de Neighbor-Joining (N-J) (Fig. 5) apoyó lo observado en PCoA. Además de los dos individuos claramente diferenciados de la variedad local Redonda, el N-J permitió observar un grupo de individuos de la variedad local Chayamansa que también se diferenciaron, aunque en menor grado. Para C. aconitifolius (en color verde), uno de sus individuos se separó del resto; todos los individuos de C. souzae (en color azul) se agruparon juntos.
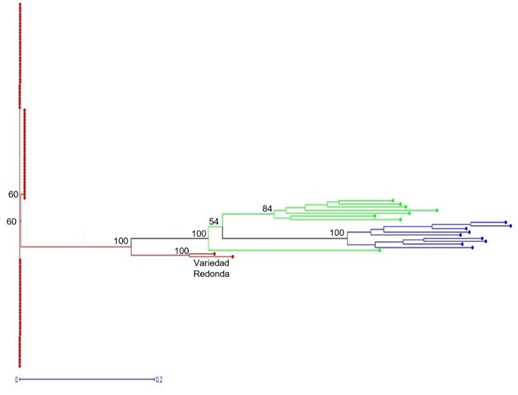
Fig. 5 Análisis Neighbor-Joining de 100 individuos de Chaya, 8 de C. aconitifolius y 9 de C. souzae, colectados en Yucatán, México, usando 158 loci de marcadores ISSRs. Los colores de los símbolos corresponden a los del análisis de Structure (K = 3) y del PCoA. Números por encima de los nodos principales corresponden a porcentajes de valores bootstrap.
El AMOVA indicó que 90% de la variación se encuentra entre los tres grupos considerados (Chaya, C. aconitifolius y C. souzae) y la comparación entre distancias genéticas mostró mayor diferenciación entre la Chaya y sus parientes silvestres (Chaya con C. aconitifolius de 0.459, Chaya con C. souzae de 0.622,), que entre C. souzae y C. aconitifolius (0.287).
Diversidad genética
Todas las poblaciones de Chaya mostraron valores muy bajos de diversidad genética: %P = 0-0.31; H E = 0.01-0.07. Las poblaciones de Euan y Acanceh fueron las que mostraron los valores más altos de %P (31 y 28.5, respectivamente) y de H E (0.1 y 0.07, respectivamente) (Cuadro 3). Agrupando los 100 individuos, la Chaya mostró un %P = 5.95 y una H E= 0.018; considerando solo los 98 individuos de la variedad local Chayamansa, se obtuvieron valores de diversidad genética mucho menores (%P = 0.6, H E= 0.008). Valores mayores de diversidad genética se observaron en Cnidoscolus aconitifolius (%P: 71.5, H E = 0.26) y C. souzae (%P: 56.3, H E = 0.21) (Cuadro 4).
Cuadro 3 Estimadores de diversidad genética de 10 poblaciones de Chaya (Cnidoscolus aconitifolius ssp. aconitifolius) colectados en Yucatán, México, usando 158 loci de ISSR.
| Nombre de la población | N | %P | H E ± E.E |
|---|---|---|---|
| Ekmul | 10 | 0 | 0.02 ± 0.001 |
| Tixcocob | 10 | 0 | 0.02 ± 0.001 |
| Ruinas de Aké | 11 | 0 | 0.02 ± 0.001 |
| Euan | 8 | 31 | 0.10 ± 0.008 |
| Hubilá | 11 | 0 | 0.02 ± 0.001 |
| Chemax | 10 | 0 | 0.02 ± 0.001 |
| Maxcanu | 10 | 0 | 0.01 ± 0.001 |
| Pocoboch | 10 | 0 | 0.01 ± 0.001 |
| Dzilam González | 10 | 0 | 0.02 ± 0.001 |
| Acanceh | 10 | 28.5 | 0.08 ± 0.007 |
N: tamaño de muestra, %P: Porcentaje de loci polimórfico al nivel de 5%, H E: Heterocigosidad Esperada, E.E.: Error Estándar.
Cuadro 4 Estimadores de diversidad genética de la Chaya (Cnidoscolus aconitifolius ssp. aconitifolius), C. souzae y C. aconitifolius de Yucatán, México, usando 158 loci de ISSR.
| Nombre | N | %P | H E ± E.E |
|---|---|---|---|
| Chaya | 100 | 5.95 | 0.02 ± 0.002 |
| Variedad Chayamansa | 98 | 0.6 | 0.01 ± 0.003 |
| C. souzae | 9 | 56.3 | 0.21 ± 0.016 |
| C. aconitifolius | 8 | 71.5 | 0.26 ± 0.015 |
N: tamaño de muestra, %P: Porcentaje de loci polimórfico al nivel de 5%, H E: Heterocigosidad esperada, E.E.: Error Estandar.
Discusión
Es aceptado que la propagación vegetativa tiene un impacto fuerte en la diversidad y estructura genética de las especies (Hamrick y Godt, 1997), en particular en aquellas que son domesticadas (McKey et al., 2010). La propagación clonal es una práctica común en cultivos perennes, sistemas en donde una escasa variabilidad genética podría esperarse entre clones de una variedad determinada; sin embargo, mecanismos genéticos intrínsecos, como las mutaciones somáticas, siguen ocurriendo y acumulándose a lo largo de la existencia de estas variedades (Gaut et al., 2018). La Chaya es una planta de gran importancia para la cultura Maya, la cual fue domesticada en la península de Yucatán (Ross-Ibarra, 2003). Por su reproducción por esquejes, se ha dado por hecho que la Chaya es un cultivo formado por un solo clon y, por ende, que presenta una diversidad genética baja; sin embargo, no existen estudios moleculares suficientes que confirmen esto. Los resultados del presente trabajo mostraron la presencia de dos variedades locales de Chaya diferenciadas genéticamente en Yucatán, México: Chayamansa y Redonda, así como dos clones en cada una de estas variedades. La mayor diversidad genética observada en Redonda, así como la colecta de solo dos individuos de esta variedad (de los 100 individuos colectados de Chaya), recalcan el gran valor de este germoplasma y el riesgo de conservación al cual se enfrenta, al menos en Yucatán. La diversidad genética mayor observada, y reportada por primera vez, en los parientes silvestres de la Chaya, en particular en C. aconitifolius, también indica el valor potencial de estas especies para fines de mejoramiento genético.
Patrón de agrupamiento y estructura genética
El método de Evanno indicó la presencia de solo dos grupos genéticos en los 117 individuos analizados. El patrón de agrupamiento encontrado con el software Structure y una K = 2, mostró la gran diferenciación genética que existe entre la Chaya y Cnidoscolus aconitifolius, su pariente silvestre filogenéticamente más cercano (Breckon, 1975; Maya-Lastra y Steinmann, 2018, 2019) y del cual se domesticó (Ross-Ibarra, 2003). Es tan grande esta diferenciación que impidió que el método de Evanno discriminara entre C. aconitifolius y C. souzae, dos especies taxonómicamente reconocibles. Cuando el software Structure se corrió con una K = 3, se logró diferenciar claramente a los tres taxa estudiados; además, se logró determinar que los dos individuos de la variedad local Redonda comparten ancestría con C. aconitifolius. De acuerdo a Meirmans (2015), analizar diferentes valores de K puede reflejar diferentes procesos genéticos y demográficos, garantizando así una mejor interpretación biológica de los datos; los resultados obtenidos cuando usamos K = 2 y K = 3 apoyan esta recomendación. Sin embargo, la falta de un mayor número de colectas de la variedad local Redonda no permitió realizar un análisis más profundo sobre su diversidad genética y su relación con C. aconitifolius. Como lo indica este trabajo, es difícil encontrar individuos de la variedad local Redonda en el estado de Yucatán; situación que parece extenderse a toda la Península de Yucatán, como lo sugiere el estudio de Ross-Ibarra (2003) quien solo colectó 10 individuos o el trabajo reciente de Munguía-Rosas et al. (2019) quienes no colectaron ningún ejemplar de esta variedad local.
El PCoA mostró una marcada separación no solo entre la Chaya y sus parientes silvestres, apoyando con ello el patrón de agrupamiento obtenido con el software Structure y una K=3; además, mostró una clara separación entre las dos variedades locales de Chaya, apoyando con ello una clasificación varietal que hasta ahora estaba basada principalmente en caracteres morfológicos y nombres comunes (Ross-Ibarra y Molina-Cruz, 2002; Cifuentes et al., 2010). El PCoA también mostró que, aunque los 98 individuos de la variedad local Chayamansa se sobreponen casi de forma perfecta, existe un ligero desfase entre estos (aunque difícil de detectar visualmente); este análisis también mostró que los dos individuos de la variedad local Redonda muestran una ligera, aunque clara, separación entre sí. Estos resultados sugieren la existencia de un clon para Chayamansa y dos clones para Redonda. Ross-Ibarra (2003), usando 12 loci de isoenzimas, reportó un solo clon para Chayamansa (de 30 individuos colectados) y tres clones para Redonda (de 10 individuos colectados). Para el caso de C. aconitifolius y C. souzae, el PCoA pudo discriminar entre todos los individuos de ambas especies.
El análisis Neighbor-Joining (N-J) apoyó, en general, lo encontrado con el software Structure (K = 3) y el PCoA. Sin embargo, el N-J también mostró evidencias de que la variedad local Chayamansa se compone de dos clones y no solo de uno, como lo reportó Ross-Ibarra (2003). El PCoA ya sugería evidencias de la existencia de dos clones en Chayamansa al mostrar un desfase muy ligero en donde, en lugar se observarse un rombo rojo, se observa un rectángulo rojo ligeramente inclinado (Fig. 4). En comparación con el PCoA, el N-J es un método de agrupamiento que permite visualizar discontinuidades (grupos), lo cual permitió detectar más fácilmente la presencia de dos clones en la variedad local Chayamansa. Para el caso de la uva (Vitis vinifera), especie que ha sido propagada durante miles de años de manera clonal, ha sido relativamente fácil detectar la variación genética entre diferentes cultivares, pero no así al nivel intra-cultivar; esta limitación está basada en la variabilidad baja esperada y en que los marcadores moleculares aplicados al estudio de este cultivo han mostrado baja resolución al abordar la diversidad genética intra-cultivar (Gaut et al., 2018). Una explicación similar puede ayudar a entender por qué Ross-Ibarra (2003), usando pocos loci de isoenzimas, reportó solo un clon para la variedad local Chayamansa y dos clones para la variedad local Redonda, aún cuando su estudio abarcó un muestro geográfico más amplio y un número mayor de individuos de la variedad Redonda (Península de Yucatán y 10 individuos de Redonda) con respecto al presente trabajo (estado de Yucatán y solo dos individuos de Redonda). Es probable que el uso de marcadores ISSR en un muestreo que abarque toda el área de distribución de la Chaya, logre detectar un número mayor de clones en estas variedades locales de Chaya, a los reportados hasta ahora.
Los resultados del AMOVA jerárquico y la comparación de distancias genéticas no sesgadas de Nei también apoyaron la existencia de tres grupos principales: el primero mostrando que el 90% de la variación total está entre los tres taxa estudiados (Chaya, Cnidoscolus aconitifolius y C. souzae), el segundo mostrando valores altos de distancias genéticas entre estos taxa. La distancia genética menor entre la Chaya y C. aconitifolius apoya su status taxonómico (Breckon, 1975; Maya-Lastra y Steinmann, 2018, 2019). La colecta de solo dos individuos de la variedad local Redonda, no permitió incorporar a esta variedad como un grupo taxonómico diferente, aspecto que habría enriquecido los resultados del presente trabajo.
Entre los factores principales que afectan los patrones de diferenciación genética en las especies está el modo de reproducción; en particular, la propagación vegetativa tiene profundas consecuencias en la estructura genética de las poblaciones (Hamrick y Godt, 1997). En el caso particular de las especies domesticadas con propagación vegetativa, los individuos con rasgos deseables pueden dar como resultado tasas de cambio rápidas porque estos pueden seleccionarse y reproducirse de forma idéntica y en grandes cantidades (McKey et al., 2010), favoreciendo así la diferenciación genética entre estas y sus parientes silvestres. En el caso de la Chaya, entre las principales características seleccionadas están una cantidad mayor de ramas y hojas, y una menor pilosidad (Munguía-Rosas et al., 2019). Esta diferenciación genética puede incrementarse aún más en plantas domesticadas que muestran alteraciones en sus mecanismos de floración y fructificación, los cuales pueden disminuir las oportunidades de entrecruzamiento; en particular, los cultivos crecidos por sus partes vegetativas exhiben la disrupción más drástica de sus sistemas de floración y fructificación (Zohary, 2004) y la Chaya es un ejemplo de esto (Ross-Ibarra, 2003). También, se ha señalado que cuando las personas han utilizado preferentemente el modo de reproducción vegetativo, algunas plantas cultivadas pierden parcial o completamente su capacidad de reproducirse sexualmente a través de la acumulación de características genéticas (por ejemplo: floración asincrónica, mutaciones somáticas, supresión de semillas y poliploidía) (Denham et al., 2020); ejemplo de esto son especies como el ñame (Dioscoria alata; Alexander y Coursey, 1969) y el enset (Ensete ventricosum; Borrell et al., 2019).
La diferenciación genética observada en este trabajo no concuerda con la evidencia morfológica, la cual ha mostrado individuos de Chaya parecidos a Cnidoscolus aconitifolius (Munguía-Rosas et al., 2019) y a C. multilobus (Cifuentes et al., 2010). Aunque se señala comúnmente que la Chaya no produce polen ni semillas (Ross-Ibarra, 2003), se ha reportado la generación de estas estructuras en algunas plantas de Chaya (Cifuentes et al., 2010; Munguía-Rosas et al., 2019), abriendo con ello la posibilidad de un flujo genético potencial entre la Chaya y sus parientes silvestres. Sin embargo, recientemente Munguía‐Rosas y Jácome‐Flores (2020) reportaron un aislamiento reproductivo casi completo entre la Chaya y C. aconitifolius silvestre que crecen en simpatría. Estos autores encontraron que las barreras de aislamiento reproductivo exhibieron cierto grado de asimetría; por un lado, la falta de polen fue un rasgo clave para alcanzar el nivel de aislamiento observado, particularmente en la dirección de plantas domesticadas a silvestres; en la dirección opuesta, mientras que el fracaso para dar fruto fue la única barrera, este fue lo suficientemente fuerte como para lograr un aislamiento reproductivo completo. Así, el flujo genético silvestre-domesticado parece no explicar la existencia de los fenotipos intermedios reportados, al menos aquellos entre la Chaya y C. aconitifolius. Munguía-Rosas et al. (2019) no reportaron la existencia de variedades de Chaya, lo que hace suponer que Munguía‐Rosas y Jácome‐Flores (2020) no incluyeron en su estudio a la variedad local Redonda, lo cual podría haber ayudado a explicar la ancestría compartida de esta variedad local con C. aconitifolius encontrada en el presente trabajo. Otra posible explicación de la existencia de fenotipos intermedios de Chaya es una asignación incorrecta de las plantas de C. aconitifolius; al respecto, Ross-Ibarra (2003) señaló que las variedades locales de Chaya conocidas como Estrella y Picuda, aunque colectadas en huertos familiares, son fenotipos que se asemejan más a C. aconitifolus.
Diversidad genética
La Chaya mostró una diversidad genética muy baja, la cual se acercó a cero cuando se eliminaron del análisis los dos individuos de la variedad local Redonda. Estos resultados van de acuerdo con la existencia de solo dos clones en la variedad local Chayamansa y de dos clones en la variedad local Redonda. Euan y Acanceh fueron las poblaciones con valores más altos diversidad genética, la colecta de individuos de la variedad local Redonda que presenta ancestría compartida con Cnidoscolus aconitifolius en estas dos poblaciones, podría explicar este resultado. Una diversidad genética mayor se observó en C. aconitifolius y C. souzae, a pesar del tamaño de muestra pequeño analizado en ambos taxa. El modo de reproducción es un factor que puede reducir fuertemente los niveles de diversidad genética de las especies (Abbo et al., 2014; Hamrick y Godt, 1997). La reproducción clonal por esquejes de la Chaya (Ross-Ibarra y Molina-Cruz, 2002), contrario a la reproducción sexual de C. aconitifolius y de C. souzae (Schikorr y Rodríguez, 2018), puede explicar las diferencias tan grandes en diversidad genética observadas en este trabajo. Otro factor que podría explicar estas diferencias en diversidad genética, al menos entre la Chaya y C. aconitifolius, es la existencia de un efecto fundador por domesticación (Ladizinsky, 1985). Al respecto, Ross-Ibarra (2003) reportó una reducción fuerte en diversidad genética en la Chaya, explicando sus resultados con la existencia de un efecto fundador por domesticación en esta especie. Para la Chaya, la combinación de ambos factores (efecto fundador por domesticación y reproducción clonal) son la explicación más probable de sus niveles bajos de diversidad genética. Tomando en cuenta que el tamaño de muestra considerado para C. aconitifolius y C. souzae fue muy pequeño en comparación con el empleado en la Chaya, los valores en diversidad genética encontrados en estas dos especies silvestres deben ser tomados con mucha precaución, hasta que se realicen nuevos estudios que abarquen un tamaño de muestra mayor y geográficamente más representativo. Sin embargo, al ser los primeros valores de diversidad genética reportados para ambas especies, estos pueden ser tomados como base para reconocer el valor potencial de los parientes silvestres de la Chaya (Hajjar & Hodgkin, 2007; Ford-Lloyd et al., 2011).
Ross-Ibarra (2003), usando 12 loci de isoenzimas, reportó valores mayores de diversidad genética (H E) para Chayamansa, Redonda y C. aconitifolius (0.2, 0.225, 0.3, respectivamente) a los encontrados en el presente trabajo. Este autor también reportó la existencia de un solo clon para la variedad local Chayamansa; sin embargo, su valor de H E reportado para esta variedad fue muy alto en comparación al encontrado por nosotros, el cual fue muy cercano a cero. Estas diferencias tan grandes en H E reportadas en ambos trabajos podrían ser el resultado del tipo de marcador molecular empleado en cada estudio. En comparación a los marcadores co-dominantes (p. ej. isoenzimas), se ha señalado que los marcadores dominantes (p. ej. ISSR) no calculan directamente las frecuencias alélicas y con ello no estiman correctamente los valores de heterocigosidad (Lynch y Milligan, 1994). Sin embargo, también ya se ha demostrado que el uso de un número alto de loci (Kimberling et al., 1996) y el empleo de métodos bayesianos robustos (Kremer et al., 2005) compensan estas limitaciones (Martínez-Castillo et al., 2008). Cifuentes et al. (2010) caracterizaron cuatro variedades de Chaya de Guatemala usando marcadores AFLPs, reportando una similitud genética del 25-35% entre variedades y del 50-60% dentro de las variedades. Estos valores indican una variación genética media; sin embargo, debido a que Cifuentes et al. (2010) no reportaron valores de diversidad genética para cada una de las variedades locales evaluadas, no fue posible comparar sus resultados con los nuestros.
La reducción de la diversidad genética en especies cultivadas perennes con reproducción clonal, en comparación a sus parientes silvestres, es un hallazgo que ha sido reportado comunmente en diferentes revisiones sobre el tema (McKey et al., 2010; Miller & Gross, 2011; Denham et al., 2020). Dos cultivos propagados por esquejes que pueden servir de como punto de comparación con la Chaya son el taro [Colocasia esculenta (L.) Schott] y la yuca (Manihot esculenta subsp. esculenta Crantz). Kreike, et al. (2004), usando marcadores dominantes AFLP, reportaron diversidad genética mayor en las poblaciones silvestres de taro colectadas en Tailandia (H E= 0.19) en comparación a la encontrada en las poblaciones cultivadas (H E= 0.007). Por su parte, Olsen y Schaal (2001), usando cinco loci de microsatélites, reportaron que la variación genética de la yuca es solo un subconjunto de la encontrada en sus parientes silvestres ya que, de los 73 alelos encontrados con los cinco loci, solo 15 alelos se hallaron en este cultivo.
Conclusiones
La Chaya presentó una diversidad genética muy baja; Chayamansa fue la variedad local de mayor distribución y la que posee una diversidad genética muy baja, mientras que Redonda fue una variedad local poco abundante pero que posee una diversidad genética mayor. Los resultados de este trabajo indican que la Chaya está compuesta por al menos cuatro clones en el estado de Yucatán, dos de cuales pertenecen a la variedad local Chayamansa (y no solo uno como había sido reportado para la Península de Yucatán). Este trabajo también resaltó la importancia de realizar un programa de rescate y conservación de la variedad local Redonda, así como un estudio más profundo sobre su diversidad genética y su relación con Cnidoscolus aconitifolius. Aunque solo se caracterizaron pocos individuos de C. aconitifolius y C. souzae, estas especies presentaron valores altos de diversidad genética en comparación a la Chaya, lo que resalta su importancia como reservorios genéticos para futuros programas de mejoramiento de la Chaya.











 nueva página del texto (beta)
nueva página del texto (beta)



