INTRODUCCIÓN
El frijol ayocote (Phaseolus coccineus L.) es una leguminosa que tiene su centro de origen y domesticación en México (Llaca et al., 1994; Sicard et al., 2005) el cual, de acuerdo con Salinas (1988) se ubica en las zonas altas de los estados de Puebla, Oaxaca, y Chiapas. Sin embargo, el frijol ayocote en México es poco cultivado y consumido no así en regiones de Europa principalmente España, Holanda y Reino Unido, donde su consumo ha sustituido al frijol común (P. vulgaris L.) y se consume principalmente en su presentación de ejote y de grano seco en el sur de Italia (Santalla et al., 2004; Rodiño et al., 2007). Por otra parte, se ha reportado su consumo en Japón donde se cultiva principalmente en zonas montañosas y su semilla es muy apreciada por su excelente calidad culinaria (Watanabe & Tojo, 2006). De las especies domesticadas del género Phaseolus spp., el frijol ayocote es la segunda especie en importancia económica (Saburido-Álvarez & Herrera-Estrella, 2015). El cultivo tradicional ocurre en pequeña escala (Schwember et al., 2017). En Centroamérica, el frijol ayocote se cultiva casi exclusivamente en asociación con maíz (Zea mays L.) (Vargas-Vázquez et al., 2007). Dado que los frijoles son un elemento importante en el autoabasto de poblaciones rurales, además de ser la leguminosa de grano más importante constituye la fuente principal de proteína de calidad a bajo costo, con diversas variaciones en cuanto a formas de cultivo, uso y maneras de preparación para consumo humano (Svetleva et al., 2003).
El frijol ayocote en México se siembra con variedades nativas o criollas y su cultivo se ha reducido de manera alarmante en los últimos 50 años (Vargas-Vázquez et al., 2007). Además, el manejo agronómico de esta especie es diferente al del frijol común porque presenta aproximadamente 14.7% de cruzamiento natural (Muruaga et al., 1992), lo que dificulta conservar la pureza genética de las variedades criollas. Lo anterior, aunado al manejo en la selección e intercambio de semilla por parte de los productores, impacta directamente en la estructura poblacional de esta especie (Mercati et al., 2015). Sin embargo, se han conducido pocos trabajos que comparen el germoplasma de P. coccineus y de P. vulgaris con respecto a los rasgos morfoagronómicos y genéticos útiles para el mejoramiento (Acampora et al., 2007).
La diversidad genética del frijol ayocote se ha estudiado previamente aplicando diversas estrategias (Nowosielski et al., 2002; Sicard et al., 2005; Boczkowska et al., 2012), principalmente marcadores moleculares de ADN y marcadores morfoagronómicos. Por su parte, Spataro et al. (2011) desarrollaron una colección núcleo de frijol ayocote con 228 accesiones (52 de Mesoamérica y 148 de Europa) y se ha confirmado la diferenciación genética entre ambos acervos genéticos (Rodriguez et al., 2013; Mercati et al., 2015) y diferenciación entre poblaciones debido a su estructuración genética (Catarcione et al., 2013; Mercati et al., 2015). En Mexico también se estructuró una colección nacional de P. coccineus que incluye 798 accesiones, y donde alrededor del 80% proviene de las regiones del eje Neovolcánico y la Sierra Madre Oriental (Vargas-Vázquez et al., 2012). El frijol ayocote del Carso Huasteco se subdivide en dos grupos: uno que incluye germoplasma con ciclo de vida tardío y regiones con temperaturas mínimas de 2-5°C con semillas y vainas grandes y otro grupo con genotipos precoces que se colectaron en sitios con temperaturas mínimas de 0 a -2°C, así como semillas y vainas pequeñas (Ruíz-Salazar et al., 2010; Vargas-Vázquez et al., 2011; 2013; 2014).
Por lo anterior, el objetivo de la presente investigación fue determinar, por primera vez, la variabilidad y la estructura genética de germoplasma de frijol ayocote de la Subprovincia Carso Huasteco de Puebla, México, mediante marcadores moleculares AFLP.
MATERIALES Y MÉTODOS
Extracción de ADN
El presente trabajo se realizó en el Laboratorio de Biotecnología Vegetal del Centro de Biotecnología Genómica del Instituto Politécnico Nacional (CBG-IPN) en Reynosa, Tamaulipas donde se analizaron 117 accesiones de P. coccineus L. colectadas en la Subprovincia Carso Huasteco del estado de Puebla, México, más cinco accesiones empleadas como grupo testigo (cuadro 1). A partir del primer trifolio de cada plántula se tomaron 30 mg de hoja fresca de los cuales se extrajo el ADN de acuerdo con el método propuesto por Doyle & Doyle (1987).
Cuadro 1 Procedencia y número de muestras de las poblaciones de Phaseolus spp. estudiadas.
| Especie | n | Localidad | Estado |
|---|---|---|---|
| P. coccineus | 38 | Zacapoaxtla | Puebla |
| " | 3 | Zacatlán | " |
| " | 18 | Tlatlauquitepec | " |
| " | 7 | Nauzontla | " |
| " | 1 | Tételes de Ávila Castillo | " |
| " | 4 | Zoquiapan | " |
| " | 1 | Huauchinango | " |
| " | 6 | Chignahuapan | " |
| " | 3 | Ahuacatlán | " |
| " | 2 | Xochiapulco | " |
| " | 14 | M‡. de Zacapoaxtla | " |
| " | 4 | M. de Cuetzalan | " |
| " | 6 | M. de Tlatlauquitepec | " |
| " | 6 | M. de Ciudad Serdán | " |
| " | 4 | Atempan | " |
| P. glabellus, P. vulgaris (Pinto Villa y Pinto Zapata) y P. coccineus (P. coccineus tipo y Blanco Tlaxcala) | 5 | Texcoco | Estado de México |
Donde ‡ = Mercado; se visitaron los sitios donde la semilla se comercializa de manera local, las accesiones fueron elegidas de acuerdo con sus características morfológicas de tamaño, color y forma.
Técnica AFLP
El análisis genético mediante AFLP ('amplified fragment length polymorphism' por sus siglas en inglés) se realizó de acuerdo con Vos et al. (1995). Inicialmente se probaron ocho combinaciones selectivas de las cuales se seleccionaron las cuatro que produjeron mayor cantidad de polimorfismos.
Eco RI + AAG con Mse I + 3 (AGG, ACC, ATG y AGT). La reacción en cadena de la polimerasa (PCR) se realizó en termociclador Perkin Elmer modelo Gene Amp 9800 (Applied Biosystem®; Foster City, CA, EU) de acuerdo con las siguientes condiciones de pre-amplificación: temperatura inicial de 94°C por 30 s; 56°C por 60 s y 72°C por 60 s por 20 ciclos, después se realizó la amplificación selectiva con las siguientes condiciones: temperatura inicial de 94°C por 4 min; 6 ciclos de 94°C por 30 s, 65°C por 30 s, 72°C por 1 min; 6 ciclos de 94°C por 30 s, 60°C por 30 s, 72°C por 1 min; 23 ciclos de 94°C por 30 s, 56°C por 30 s, 72°C por 1 min; 72°C por 5 min. Los fragmentos amplificados se separaron en un sistema de secuenciación automática IR2 (modelo 4200-029; LI-COR®; Lincoln, NE, EU). Con los resultados de las lecturas del secuenciador se construyó una matriz binaria donde 1= presencia y 0 = ausencia de banda (producto amplificado).
Análisis estadístico
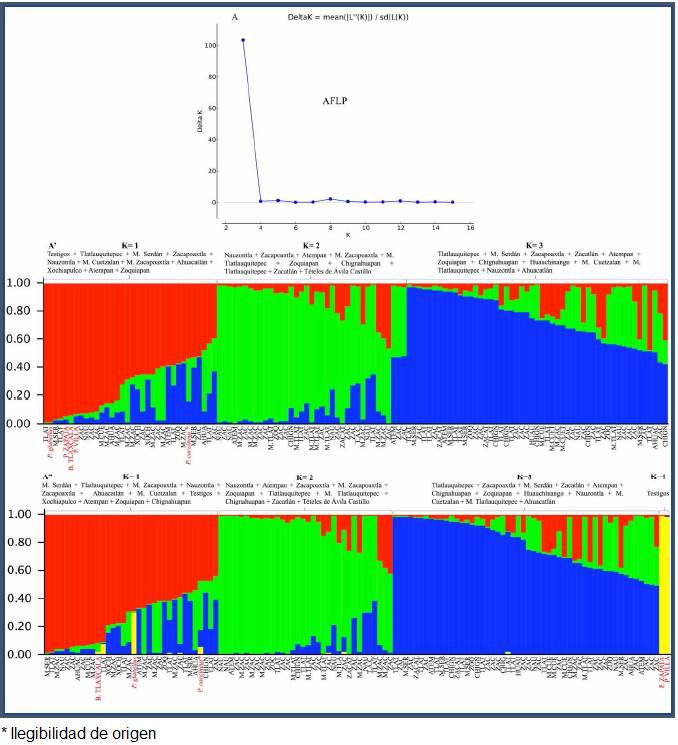
El análisis de conglomerados se desarrolló con el paquete computacional Statistica versión 7 (StatSoft, 2004) mientras que el análisis de varianza molecular (AMOVA) se calculó para luego estimar el estadístico FST que mide el grado de diferenciación genética entre accesiones de frijol ayocote; dicho análisis se llevó a cabo con el software GenoDive 2.0b27 (Meirmans & VanTienderen, 2004). Posteriormente, se construyó una matriz de distancias genéticas mediante el índice de similitud de Jaccard. Dichas distancias sirvieron para generar un dendrograma con el algoritmo UPGMA ('Método de promedio simple con pares no ponderados' por sus siglas en inglés). Para determinar la estructura poblacional del frijol ayocote se llevó a cabo un análisis de inferencia bayesiana con el software Structure 2.3.4 (Pritchard et al., 2000; Hubisz et al., 2009) donde se asumieron 16 poblaciones con parámetros de modelo mixto y frecuencias alélicas correlacionadas; se hizo el cómputo para 30 iteraciones por cada población con pre-tratamiento de datos que consistió en 5000 'alineamientos' seguido de 50 000 cadenas de Markov-Monte Carlo (MCMC) (Evanno et al., 2005). El valor óptimo de delta_K se determinó con el software Structure Harvester versión 0.6.94 (Earl & VonHoldt, 2012).
RESULTADOS
La amplificación de cuatro combinaciones de oligonucleótidos AFLP arrojó 256 bandas, de las cuales 224 fueron polimórficas (87.4%) y 32 monomórficas (12.6%) con estos datos se construyó la matriz de presencia/ausencia de bandas (cuadro 2). El AMOVA presentó un valor FST = 0.067; además, se observó que la mayor proporción de la varianza molecular explicada se encuentra dentro de las accesiones (93.3%) (cuadro 3). El análisis de conglomerados con datos AFLP formó dos grupos principales (A y B) sin embargo no se observaron agrupamientos definidos con base en el origen del germoplasma pues los testigos Pinto Villa, Pinto Zapata (P. vulgaris) L. y P. glabellus L. (grupo B) fueron distintos a P. coccineus (grupo A), donde se observó una mezcla de poblaciones (fig. 1).
Cuadro 2 Productos amplificados por combinación AFLP en genotipos de frijol ayocote.
| Combinación AFLP | Productos amplificados | Polimorfismo (%) | ||
|---|---|---|---|---|
| Monomórficos | Polimórficos | Total | ||
| AAG/AGG | 16 | 47 | 63 | 74.6 |
| AAG/ACC | 5 | 53 | 58 | 91.3 |
| AAG/ATG | 7 | 61 | 68 | 89.7 |
| AAG/AGT | 4 | 63 | 67 | 94.0 |
| Total/Media | 32 | 224 | 256 | 87.4 |
Cuadro 3 Análisis de la varianza molecular de frijol ayocote con base en datos AFLP.
| Fuente de variación | Grados de libertad | Suma de cuadrados | Cuadrados medios | Varianza exp. (%) | Estadístico F | Valor F | Valor P |
|---|---|---|---|---|---|---|---|
| Dentro de accesiones | 106 | 13800.015 | 130.189 | 93.3 | - | - | - |
| Entre accesiones | 15 | 2916.116 | 194.408 | 6.7 | FST | 0.067 | 0.001 |
| Total | 121 | 16716.131 | 138.150 | 100 | - | - | - |
El análisis de la estructura poblacional de accesiones de frijol ayocote determinó un valor de delta K = 3. El primer grupo (K1) incluye accesiones de once localidades: Tlatlauquitepec, M. Serdán, Zacapoaxtla, Nauzontla, M. Cuetzalan, M. Zacapoaxtla, Ahuacatlán, Xochiapulco, Atempan y Zoquiapan y los testigos; el segundo conglomerado (K2) incluye germoplasma de diez localidades (Nauzontla, Zacapoaxtla, Atempan, M. Zacapoaxtla, M. Tlatlauquitepec, Zoquiapan, Chignahuapan, Tlatlauquitepec, Zacatlán y Tételes de Ávila Castillo). Finalmente, el tercer grupo (K3) muestra genotipos de 12 localidades: Tlatlauquitepec, M. Serdán, Zacapoaxtla, Zacatlán, Atempan, Zoquiapan, Chignahuapan, Huauchinango, M. Cuetzalan, M. Tlatlauquitepec, Nauzontla y Ahuacatlán. Lo anterior confirmó los resultados del análisis de conglomerados dado que no se observaron agrupamientos definidos con base en el origen de colecta de cada accesión. Se realizó un segundo análisis donde se consideró un valor de delta K = 4 y se observó que el grupo testigo que incluyó dos accesiones de de P. vulgaris (Pinto Villa y P. Zapata) y una de P. glabellus se agruparon de manera conjunta en el clóster cuatro (K4) (fig. 2).
DISCUSIÓN
En este trabajo se llevó a cabo el análisis de la variabilidad y la estructura genética de accesiones de P. coccineus provenientes del Carso Huasteco de México, una de las regiones más importantes de origen y diversidad de frijol ayocote en Mesoamérica, aplicando la estrategia de marcadores moleculares tipo AFLP. La estrategia molecular fue eficiente para el análisis genético en virtud de que detectó más del 87% de productos polimórficos en el germoplasma analizado y a su condición de estrategia de 'alta eficiencia multiplex al generar alta cantidad de productos amplificados por ensayo o reacción de amplificación (Bitocchi et al., 2015).
El análisis molecular separó claramente al germoplasma de P. coccineus de los testigos pertenecientes a las especies P. vulgaris y P. glabellus (Nowosielski et al., 2002); aunque la separación entre el germoplasma del Carso Huasteco y P. coccineus tipo y la variedad de frijol ayocote Blanco Tlaxcala ya no fue tan clara. Sin embargo, la formación de al menos tres sub-grupos de genotipos de frijol ayocote con diferentes orígenes demuestra la existencia de poblaciones genéticamente similares y adaptadas a rangos climáticos específicos y en constante intercambio debido a la 'captura' de nuevos alelos dada la posible polinización cruzada que ocurre naturalmente en sus zonas de distribución (Spataro et al., 2011; Bitocchi et al., 2015).
Éste es el primer trabajo en México que analiza genéticamente P. coccineus con base en una estrategia molecular altamente confiable e informativa y donde, además, se incluyen poblaciones representativas de la especie. Los estudios anteriores han enfatizado el estudio de P. coccineus con base en rasgos fisiológicos, fenológicos, morfológicos o productivos (Vargas-Vázquez et al., 2011; 2013; 2014). Destaca el hecho de encontrar la mayor proporción de la varianza genética molecular dentro de las accesiones más que entre accesiones o grupos de accesiones con base en el origen (Spataro et al., 2011) mientras que en frijol común se reportan proporciones diferentes (Gill-Langarica et al., 2011). Lo anterior sugiere genotipos de frijol ayocote diversos. Además, esto indica que, aunque el análisis genético de plantas basado en la formación de 'bulks' de DNA reduce el trabajo y costos, puede subestimar la real naturaleza genética de cada accesión o población de genotipos de frijol. Las futuras estrategias de conservación del germoplasma de frijol ayocote deben considerar mecanismos de multi plicación que garanticen preservar la variabilidad genética de cada genotipo.
El análisis genético de P. coccineus ha puesto énfasis en germoplasma de Europa y los resultados coinciden en reportar diversidad genética reducida (Sicard et al., 2005; Boczkowska et al., 2012). Incluso, se han podido diferenciar genotipos de frijol ayocote con base en acervos genéticos (Sicard et al., 2005; Acampora et al., 2007). El análisis de una colección 'mundial' de P. coccineus (228 genotipos de Mesoamérica -52-, Europa -148- y silvestres -28-) volvió a recalcar la diversidad genética reducida del frijol ayocote (Spataro et al., 2011). En este sentido se enfatiza que el germoplasma analizado fue introducido a Europa proveniente de América (Rodriguez et al., 2013; Mercati et al., 2015) sufrió el efecto del 'cuello de botella' que precedió a la expansión de la especie en el continente Europeo (Rodríguez et al., 2013).
Sin embargo, Rodríguez et al. (2013) sugieren la diferenciación genética adaptativa que luego condujo a la diversificación de P. coccineus en Europa, con la consiguiente estructuración genética de poblaciones (Catarcione et al., 2013; (Mercati et al., 2015)en virtud a factores tales como la polinización abierta (≈14% según Muruaga et al., 1992) y el intercambio de germoplasma (semilla) y la selección empírica de genotipos por los agricultores locales (Castillo-Mendoza et al., 2006; Vargas-Vázquez et al., 2011; 2012; 2013; 2014); Así, los estudios previos concluyen que Europa debe considerarse un centro de diversificación secundario de P. coccineus (Mercati et al., 2015).
La variabilidad genética de P. coccineus podrá ser aprovechada para el mejoramiento genético de la propia especie o de otras cercanas como P. vulgaris, en la medida que se intensifiquen los esfuerzos en caracterizar el germoplasma disponible de la especie y se identifiquen así genotipos que puedan servir como fuentes de resistencia a factores como las enfermedades y el frío. También, se propone apoyarse en tecnologías de secuenciación de nueva generación para el análisis de la diversidad genética, el mapeo genético y estudios de sintenia para identificar SNPs o genes funcionales e 'indels' asociados con características agronómicas importantes (Schwember et al., 2017).
El análisis con 42 548 SNPs generados por secuenciación de 242 individuos de P. coccineus y las formas domesticadas de especies cercanas a P. vulgaris (20) y P. dumosus (35) indicó la formación de ocho conglomerados o grupos de genotipos, donde la mitad correspondió a poblaciones silvestres y el resto, a domesticadas. Dado que las poblaciones domesticadas formaron un solo clado monofilético se asume que P. coccineus de Méxio sólo sufrió un evento de domesticación mismo que posiblemente ocurrió en germoplasma del Cinturón Volcánico Trans-Mexicano. Contrario a lo observado previamente, no se observaron evidencias de flujo de genes recientes entre poblaciones silvestres y cultivadas. Finalmente, se identificaron 24 SNPs relacionados con domesticación, 12 con diversificación genética y ocho con selección natural. De los pocos SNPs identificados y que están anotados, se expresan en flores y vainas (Guerra-García et al., 2017).
El germoplasma silvestre de P. coccineus se dividió en cuatro grupos genéticos que exhiben diferenciación genética significativa (Guerra-García et al., 2017). Así como la diversidad contenida en los parientes silvestres es un determinante importante en la evolución de cultivos, el de las poblaciónes domesticadas también lo es. La domesticación reduce la diversidad genética (Schmutz et al., 2014) debido a la deriva genética resultante de los 'cuellos de botella' poblacionales y la selección artificial (Schmutz et al., 2014); además de los tamaños efectivos de las poblaciones o la alta tasa de polinización cruzada (Muruaga et al., 1992).











 nueva página del texto (beta)
nueva página del texto (beta)