Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Polibotánica
versión impresa ISSN 1405-2768
Polibotánica no.40 México ago. 2015
Variabilidad del gen nuclear G3pdh en Jatropha curcas L.
Variability of the nuclear gene G3pdh in Jatropha curcas L.
Castro Guzmán Sonia, Ogata Aguilar Nisao, Cano Asseleih Leticia M., y Sánchez Sánchez Odilón
Centro de Investigaciones Tropicales Ex-Hacienda Lucas Martín Privada de Araucarias s/n, Col. Periodistas, CP 91019 Xalapa, Veracruz, México. Teléfonos: (01-228) 842-17-00 y 842-27-00 Extensiones: 12641, 12642 y 12643. Correo electrónico: odsanchez@uv.mx
Recibido: 21 enero 2014.
Aceptado: 29 mayo 2015.
Resumen
Jatropha curcas L. es una especie nativa de América tropical; en nuestro país se ha venido utilizando principalmente como medicinal y alimenticia desde la época prehispánica. Actualmente el aceite extraído de sus semillas ha adquirido importancia internacional ya que puede ser transformado a biodiesel. El conocimiento que existe sobre la variabilidad genética de la especie es escaso. Un gen utilizado con éxito para el estudio de patrones de variación y origen de la yuca (Manihot esculenta L.) y del caco (Theobroma cacao L.) es el gen nuclear G3pdh (Gliceraldehído 3 fosfato deshidrogenasa) involucrado en la fotosíntesis. Con base en ello, en este trabajo se exploró la variabilidad del gen G3pdh en individuos de J. curcas provenientes de 15 poblaciones de los estados de Veracruz, Campeche, Yucatán y Quintana Roo. Para obtener el gen G3pdh completo de alrededor de 1000 pares de bases, se amplificó utilizando los primeros diseñados por Strand et al. (1997) y los dos primeros internos reverse diseñados por Olsen y Schaal (1999) para obtener segmentos más cortos, de 600 y 800 pares de bases aproximadamente. Por primera vez se amplificaron aproximadamente 500 pb y los resultados demuestran que el gen G3pdh es útil para analizar la variabilidad de J. curcas, y con un importante potencial para evaluar la distribución y evolución de sus poblaciones en México, conocer las relaciones ancestro-descendiente a nivel poblacional y explicar las causas de la distribución de los distintos haplotipos.
Palabras clave: gen G3pdh, diversidad genética, haplotipos.
Abstract
Jatropha curcas L. is native of tropical America; in our country it has mainly been used as a medicinal and food since pre-Hispanic times. Currently the oil extracted from its seeds has gained international importance as it can be transformed into biodiesel. The knowledge on the genetic variability of the species is scarce. A gene used successfully for the study of patterns of variation and origin of cassava (Manihot esculenta L.) and cocoa (Theobroma cacao L.) is the nuclear gene G3PDH (glyceraldehyde 3-phosphate dehydrogenase) involved in photosynthesis. In this paper, we used the same gene to explore its variability in J. curcas. Fifteen populations from different areas of Mexico were examined (Veracruz, Campeche, Yucatán y Quintana Roo). The gene was amplified using primers originally designed by Strand et al. (1977) to isolate around 1000 bp. Two additional internal primers designed by Olsen y Schaal (1999) were used to amplify shorter sections of 600 and 800 bp approximately. As a result a 500 bp section of the G3pdh of J. curcas L. was amplified and tested for variation at population level showing its usefulness for studies of genetic diversity and phylogeographic patterns.
Key words: gene G3pdh, genetic diversity, haplotypes.
Introducción
Jatropha curcas L. es una especie de la familia Euphorbiaceae que recientemente ha cobrado importancia debido al potencial que representa como fuente de biocombustible (Openshaw, 2000). Con usos diversos en la mayoría de los países donde prospera, principalmente se le utiliza como cerca viva y medicinal. Es útil también para reforestación y regeneración de suelos erosionados. En el norte del estado de Veracruz, en la región totonaca, las semillas de J. curcas son muy apreciadas para la alimentación humana, además de que poseen un alto valor nutritivo (Cano y Hernández, 1984). Sin embargo, en las otras regiones donde se localiza, las semillas son tóxicas tanto para animales como humanos, debido a la presencia de inhibidores de tripsina, ésteres de forbol y otros componentes antinutricionales, los cuales, al ser consumidos, causan efectos purgantes, vómito e irritación de la piel (Cano, 1986; Makkar et al., 1998; Schmook y Sánchez, 2000; Openshaw, 2000).
No obstante la importancia de J. curcas, no existe información de esta especie en torno a su variabilidad genética. Entre los genes que se han utilizado con éxito para el estudio de patrones de variación y origen de la yuca (Manihot esculenta L.) (Euphorbiaceae) (Olsen y Schaal, 1999) y del cacao (Theobroma cacao L.) (Malvaceae) (Ogata, 2002) se encuentra el G3pdh, que codifica la enzima Gliceraldehído-3 fosfato deshidrogenasa. Con base en ello, el objetivo de esta investigación fue secuenciar el gen nuclear G3pdh en J. curcas L. y analizar su variabilidad en distintas poblaciones localizadas en diferentes estados de la república mexicana, con el fin de evaluar su utilidad para proponer hipótesis relacionadas con su origen, distribución y posible domesticación a través de su rango de distribución natural.
Metodología
Se colectaron hojas juveniles de 104 individuos en 54 localidades distribuidas en los estados de Veracruz (89 individuos/42 localidades), Puebla (1 individuo/1 localidad), Campeche (2 individuos/1 localidad), Quintana Roo (11 individuos/9 localidades) y Yucatán (1 individuo/1 localidad). Al mismo tiempo se colectaron especímenes de herbario y fueron depositados, un ejemplar por localidad, en el herbario XALU de la Facultad de Biología de la Universidad Veracruzana.
La extracción de ADN se realizó utilizando el paquete de extracción de ADN de plantas de la compañía QIAGEN (USA). Posteriormente se cuantificó el contenido de ADN mediante espectrofotómetro marca JENWAY (UK), modelo Genova. Adicionalmente, se estimó la pureza de los extractos, determinando la absorción a 280 nm y calculando el cociente OD260/OD280 (Sambrook y Russell, 2001). Para aislar el gen G3pdh en J. curcas se utilizaron los primers: GPDX7 (GAT AGA TTT GGA ATT GTT GAG G) y GPDX9R (AAG CAA TTC CAG CCT TGG) (Strand et al., 1997) para amplificar el gen completo, de alrededor de 1000 pb y los dos primers internos reverse diseñados por Olsen y Schaal (1999), para obtener segmentos más cortos de este mismo gen: GPD9R4 (TCC CTT AAG CTT ACC ATA AG) y GPD9R2 (CTT GAT TTC CTC ATA TGT TGC C). Para la amplificación del gen G3pdh y sus segmentos se realizaron varias pruebas utilizando diferentes concentraciones de MgCl2, de primers y dos tipos de Taq polimerasa en la mezcla de reacción y las temperaturas de alineamiento y el número de ciclos para obtener las condiciones óptimas que se presentan en los cuadros 1 y 2.


La amplificación se realizó indistintamente en dos termocicladores AB PCR system 2700 (Aplied Byosystems, USA), y PTC-200 (Peltier Thermal Cycler, MA).
Los productos amplificados se analizaron mediante electroforesis en geles horizontales de agarosa al 2% de 7x 8.3 cm (Barker, 1998). Se utilizó un estándar de oligonucleótidos de Fermentas (Kb ladder) y azul de bromofenol como indicador del corrimiento. Previo a la secuenciación, los productos amplificados se purificaron utilizando el paquete de purificación QIAquick de QIAGEN, de acuerdo a su protocolo.
Las muestras amplificadas fueron enviadas para su secuenciación al laboratorio del Institute for Integrative Genome Biology de la Universidad de California, Riverside, USA. Se utilizó el sistema de secuenciación basado en ciclos fluorescentes. Para corroborar que las secuencias pertenecieran al gen G3pdh, se utilizó el Basic Local Alignment Search Tool (BLAST), uno de los recursos que ofrece el National Center of Biotechnology Information (NCBI) en su sitio web. Para analizar las secuencias generadas en este estudio, se utilizaron los siguientes programas: a) 4peaks versión 1.7.2, (programa para editar secuencias, diseñado para Mac: http://nucleobytes.com/index.php/4peaks) para editar y visualizar las secuencias en formatos ABI, SCF, CTR, ALF y ZTR. b) Phred, Phrap y c) Consed de Red Hat Linux (Universidad de Washington) para ensamblar los archivos de secuencias de ADN (Gordon et al., 1998). Finalmente, para conocer la diversidad y distribución de patrones filogeográficos de las poblaciones muestreadas se utilizó el programa TCS (Clement et al., 2000).
Resultados
De las 104 extracciones realizadas, 82 muestras fueron enviadas a secuenciar (26 del segmento de 962 pb, 20 del segmento de 752-771 pb y 36 del segmento de 610-631 pb). De éstas, 40 tenían la calidad suficiente para ser alineadas y analizadas. Las de mejor calidad correspondieron al segmento de 610-631 pb (26 muestras) en las cuales se secuenciaron 487 pb con la suficiente nitidez para ser analizadas. Le siguió el segmento de 962 pb (12 muestras), en el cual se recuperaron alrededor de 750 pb y, finalmente el segmento de 752-771 pb (dos muestras), donde se obtuvo información hasta aproximadamente 600 pb. Las secuencias de cada individuo obtenidas con cada par de primers fueron alineadas como una forma de corroborar los resultados, confirmando que se trataba del gen G3pdh mediante la búsqueda en el banco de información de NCBI. Pudo comprobarse la veracidad de los resultados hasta las 487 bases, que fueron utilizadas para determinar la estructura del gen, diversidad y distribución de los distintos haplotipos en el área de muestreo.
Al analizar la estructura de este fragmento de gen de J. curcas (fig. 1) y compararla con la del mismo fragmento de M. esculenta y T. cacao, se observó que comparten ciertas características generales.

Se analizaron 487 pb del fragmento de 610-630 pb en el programa TCS (Clement et al., 2000) de 26 individuos, para evaluar las relaciones entre las poblaciones muestreadas y la distribución geográfica de los haplotipos (fig. 2).
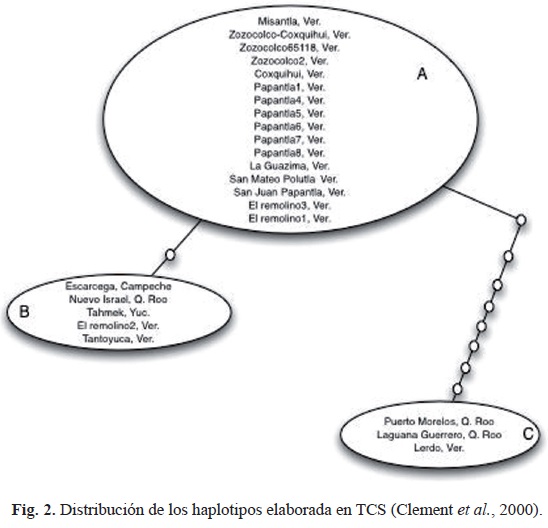

De acuerdo con el análisis de patrones de variación, se diferenciaron tres haplotipos, el más frecuente (A) y dos haplotipos (B y C) con uno y ocho cambios respectivamente. Como es posible observar, el haplotipo "A", el más frecuente de la muestra, conecta al haplotipo "B" por un cambio y al haplotipo "C" por ocho cambios. Estos resultados coinciden con Posada y Crandall (2001) en el sentido de que los haplotipos más frecuentes tienden a conectar a los haplotipos menos frecuentes, y no a que las conexiones se den entre los haplotipos menos frecuentes. Cuando los haplotipos fueron distribuidos geográficamente, el haplotipo más frecuentes se halló concentrado hacia el norte del estado de Veracruz y límites del estado de Puebla (fig. 3). En el cuadro 4 se muestran los sitios de colecta de los 26 individuos incluidos en los haplotipos.

Discusión y conclusiones
En este trabajo se reporta por primera vez la amplificación y secuenciación de 487 pb del gen G3pdh en hojas de J. curcas. En relación con la diversidad y distribución de patrones filogeográficos de las poblaciones de la J. curcas muestreadas se detectaron tres haplotipos, en los cuales la diferencia notable fue en los haplotipos "A" y "B", y 8 adeninas en el haplotipo "C". El haplotipo "A" fue el más frecuente pero no ampliamente distribuido, restringido al norte de Veracruz y límites con Puebla; los haplotipos "B" y "C" derivados, se ubicaron en la periferia de la distribución del haplotipo "A" (Tantoyuca, El Remolino y Lerdo, Ver.) y en la península de Yucatán. Para explicar la restricción en la distribución del haplotipo más frecuente, será necesario incrementar el número de muestras y localidades para saber si el comportamiento de este haplotipo está sesgado debido al tamaño de la muestra. En relación con los haplotipos restantes, es muy probable que su distribución esté ligada a las migraciones humanas recientes ocurridas en la península de Yucatán.
Actualmente, es común que en huertos familiares de Quintana Roo se hallen especies de plantas que no son nativas de la zona. En este sentido, es muy probable que los haplotipos "B" y "C" hallados en la península sean el resultado de la migración humana, hipótesis que se basa en el análisis químico de presencia de ésteres de forbol en las semillas colectadas en esos sitios. En estas muestras se detectaron ésteres de forbol en concentraciones muy bajas (Sánchez y Sánchez, 2014), lo que permite suponer que este material probablemente proviene de plantas no tóxicas de la región norte de Veracruz.
Además de que el gen G3pdh es útil para analizar la variabilidad de esta especie, tiene un importante potencial para evaluar la distribución de poblaciones J. curcas, para conocer las relaciones ancestro-descendientes a nivel poblacional y para explicar las causas de la distribución de los distintos haplotipos. Representa también un modelo adecuado para explorar hipótesis relacionadas con la domesticación de plantas y la migración de éstas asociada con las poblaciones humanas. Esta aproximación al conocimiento de filogeografía de J. curcas representa un área de estudio que hasta la fecha no había sido explorada para conocer más sobre la biología de la especie.
Agradecimientos
La realización de la presente investigación se llevó a cabo gracias al apoyo recibido del proyecto Establecimiento de huerto semillero de Jatropha curcas L.: suplemento proteínico y recurso bioenergético para incrementar la productividad ganadera sustentable en Veracruz, financiado por el Fondo Mixto Conacyt-Gobierno del estado de Veracruz con clave 37035.
Literatura citada
Barker K., 1998. At the bench, a laboratory navigator. Cold Spring Harbor Laboratory Press. Nueva York. 460 pp. [ Links ]
Cano, A.L.M., y A.C. Hernández; 1984. "El piñoncillo, Jatropha curcas L. recurso biótico silvestre del trópico". Cuadernos de divulgación núm. 14. Instituto Nacional de Investigaciones sobre Recursos bióticos. Xalapa, Veracruz, México. 16 pp. [ Links ]
Cano, A.L.M., 1986. Chemical investigation of Jatropha curcas L. seeds. Tesis doctoral. Universidad de Londres. 291 pp. [ Links ]
Clement M.; D. Posada, y K.A. Crandall, 2000. "TCS: a computer program to estimate gene genealogies". Molecular Ecology, 9(10): 1657-1660. [ Links ]
Gordon D.; C. Abajian, y P. Green, 1998. "Consed: A graphical tool for sequence finishing". Genome Research, 8(3): 195-202. [ Links ]
Makkar, H.P.S.; K. Becker, y B. Schmook, 1998. "Edible provenances of Jatropha curcas from Quintana Roo state of México and effect of roasting on antinutrient and toxic factors in seeds". Plant Foods for Human Nutrition, 52: 31-36. [ Links ]
Ogata, N., 2002. "Studies of Mesoamerican tropical trees: trees of the Maya region and a case of study on the ethnobotany and phylogeography of cacao (Theobroma cacao L.)". Tesis Doctoral. Universidad de California, Riverside. 338 pp. [ Links ]
Olsen, K., y B. Schaal, 1999. "Evidence on the origin of cassava: Phylogeography of Manihot esculenta". Proc. Natl. Acad. Sci., 26: 5586-5591. [ Links ]
Openshaw, K., 2000. "A review of Jatropha curcas: an oil plant of unfulfilled promise". Biomass y Bionergy, 19: 1-15. [ Links ]
Posada, D., y K.A. Crandall, 2001. "Intraspecific gen genealogies: trees grafting into networks". Trends in Ecology y Evolution, 16(1): 37-45. [ Links ]
Sambrook, J., y D.W. Russell, 2001. "Molecular cloning. A laboratory manual". vol. 1. Ed. Cold Spring Harbor Laboratory Press. New York. 2344 pp. [ Links ]
Sánchez, S.O., y D.E. Sánchez-Herrera. 2015. El uso de los recursos vegetales autóctonos para impulsar el empoderamiento de las clases marginadas que habitan en las zonas rurales del estado de Veracruz: caso Jatropha curcas L. En: Gobernanza Ambiental: teoría y práctica para la conservación y uso sutentable de los recursos. Ruelas, M.L.C.; B. A. C. Travieso, y S.O. Sánchez (Edit.). Universidad Veracruzana, El Colegio de Veracruz y Editorial Plaza y Valdés, 739-748. [ Links ]
Schmook, B., y S.O. Sánchez, 2000. "Usos y potencial de Jatropha curcas L. en la península de Yucatán, México". Foresta veracruzana, 2(2): 7-11. [ Links ]
Strand, A.E.; J. Leebens-Mack, y B.G. Milligan, 1997. "Nuclear DNA-based markers for plant evolutionary biology". Molecular Ecology, 6: 113-118. [ Links ]














