Introducción
Las especies relictas se definen como aquellas que en el pasado tenían una amplia distribución geográfica y actualmente tienen un espacio geográfico muy restringido (Lomolino, Riddle y Brown, 2006; Habel, Assmann, Schmitt y Avise, 2010). Estas se caracterizan por presentar poblaciones con bajo número de individuos (García-Gonzáles et al., 2008), distribución discontinúa y poblaciones fragmentadas por pérdida de su hábitat (Habel et al., 2010). Son especies susceptibles a procesos de cambio climático (Rodríguez-Sánchez, 2011) y pérdida de diversidad genética (Habel et al., 2010; Dantas et al., 2015) con estructuración genética de sus poblaciones (Tang et al., 2008), por lo que se ha recomendado su estudio, para realizar acciones adecuadas de conservación a mediano y largo plazo (Habel et al., 2010).
La conservación de las especies relictas requiere un enfoque de conservación local in situ (Alfonso-Corrado et al., 2017). El enfoque de conservación in situ, es uno de los nuevos paradigmas de la biología de la conservación, donde el conocimiento biológico de la especie permite la participación y vinculación entre científicos y las comunidades locales. El conocimiento científico y tradicional permite que las estrategias de conservación a escala local sean exitosas sin la necesidad de decretar áreas protegidas (Primack, 2012). En México, las especies relictas están presentes principalmente en los bosques tropicales y de niebla en donde ocurre una alta diversidad de estas especies (Vargas-Rodríguez, 2011). Los bosques de niebla en el país son ecosistemas que albergan especies de origen tropical y templado, entre las que sobresalen las especies endémicas y relictas (Anta-Fonseca et al., 2010; Vargas-Rodríguez, 2011); además, son considerados estratégicos por los servicios ecosistémicos de tipo hidrológico y captación de carbono (Álvarez-Arteaga, García-Calderón, Krasilnikov y García-Oliva, 2013; Alfonso-Corrado et al., 2017).
Entre las especies relictas del bosque de niebla de México, Oreomunnea mexicana (Standl.) J. F. Leroy (Juglandaceae) es de relevancia; esta especie surgió en el Mioceno tardío y Plioceno temprano, hace aproximadamente 23 millones de años (Rzedowski y Palacios, 1977), y es considerada en peligro de extinción en la Lista Roja de árboles del bosque de niebla mexicano (González-Espinosa, Meave, Lorea-Hernández, Ibarra-Manríquez y Newton, 2011). O. mexicana es el elemento arbóreo dominante en diversas áreas del bosque de niebla de la Sierra Juárez localizado en Oaxaca (Rzedowski y Palacios, 1977; Alfonso-Corrado et al., 2017). En este bosque de niebla, uno de los más conservados y extensos de México (Anta-Fonseca et al., 2010), la especie aporta importancia biológica al considerarse un elemento clave para la conservación y mantenimiento de la biodiversidad asociada (e.g. Panthera onca Linnaeus), además que se encuentra inmersa en la cosmovisión de la cultura indígena chinanteca, es utilizada en la construcción de casas (Alfonso-Corrado et al., 2017). Sin embargo, a nivel ecológico las poblaciones en la Sierra Juárez muestran reducción por el efecto de cambio ambiental (Alfonso-Corrado et al., 2017), además de su baja germinación, ya que sus semillas tienen requerimientos de hábitats específicos para lograr un establecimiento exitoso (Pacheco-Cruz et al., 2019).
Dada la importancia biológica y cultural de la especie, para su conservación in situ a mediano y largo plazo, son necesarios los estudios que permitan tomar decisiones adecuadas. El estudio genético de poblaciones permite tomar acciones preventivas, para que la diversidad genética de una especie no se erosione y junto con ella, la capacidad para responder a factores bióticos y abióticos (Dvorak, 2012), pero también son útiles para determinar cuáles son las poblaciones críticas que permite tomar acciones adecuadas de conservación in situ y ex situ (Primack, 2012).
Objetivos
El objetivo de este estudio es evaluar la diversidad y estructura genética de Oreomunnea mexicana en el bosque de niebla de Sierra Juárez, Oaxaca. Además, analizar en una población la estructura genética espacial a escala fina. Por ser una especie relicto amenazada, con poblaciones fragmentadas se espera encontrar niveles bajos de diversidad genética y aislamiento genético entre poblaciones. Dada la dispersión por viento de sus semillas se espera obtener una estructura genética espacial a escala fina aleatoria.
Materiales y métodos
Sitio de estudio
La Sierra Juárez se localiza en una zona de transición biogeográfica al norte de la ciudad de Oaxaca, México, ubicada (17º 19’ de latitud norte y 96º 29’ de longitud oeste) dentro de la Sierra Madre de Oaxaca. El clima del área de estudio es semicálido húmedo con abundantes lluvias en verano, temperatura media anual de 18 ºC, y una precipitación media anual de 1500 mm (Clark-Tapia, Suárez-Mota, Venegas-Barrera y Velasco-Hipólito, 2016). El relieve tiene pendientes muy pronunciadas (> 50 º) que generan gran diversidad de microclimas y difícil acceso (Naranjo-Luna, 2014). El muestreo se realizó en el bosque de niebla ubicado en los municipios de Santiago Comaltepec e Ixtlán de Juárez (Fig. 1).
Especie de estudio
Oreomunnea mexicana (nombre común: caudillo; nombre en Chinanteco: maá hue loó; que significa árbol que se descascara), pertenece al orden Fagales (mismo que los encinos) y familia Juglandaceae, es un árbol relicto de la era (Rzedowski y Palacios, 1977) y se distribuye de forma discontinua en México (Oaxaca, Chiapas y Veracruz), Guatemala, Nicaragua, Panamá y Costa Rica, principalmente entre 1100 m y 2000 m snm (González-Espinosa et al., 2011). Esta especie puede tener hasta 40 m de altura y presenta hojas perennes pinnadas y el fruto es una nuez globosa encerrada por la base de la bráctea abaxial y el lóbulo bracteolar adaxial. La corteza exfoliada que se desprende en pedazos grandes y el fruto con la larga bráctea trilobada son características importantes para distinguir la especie (Russell y Cohn, 2012).
Elección de sitios y recolecta del material biológico
De enero a mayo de 2013, muestras de tejido foliar de 225 individuos (25 por sitio) de O. mexicana se recolectaron en nueve sitios (tres en el municipio de Ixtlán de Juárez y seis en el municipio de Santiago Comaltepec (Fig. 1), a lo largo de un transecto de 1.25 km. Un individuo se recolectó cada 50 m para evitar el parentesco entre individuos cercanos. Adicionalmente, muestras de tejido foliar de 120 individuos se recolectaron en una parcela de 20 m2 × 20 m2 (400 m2) en el sitio de San Bernardo (Fig. 1) y su ubicación espacial (latitud y longitud) se registró con un GPS submétrico Mobilemapper Field y Office v. 10 (Marca Ashtech).
Análisis genéticos
Se extrajo ADN a hojas jóvenes de 345 individuos siguiendo el protocolo de DNeasy Plant Minikit (QIAGEN, 2004). La cantidad y calidad del DNA se visualizaron en geles de agarosa de 1.0%. Se utilizaron para el análisis 12 oligonucleótidos (814, 844A, 844B, 17898A, 17899B, HB8, HB9, HB11, HB12, HB13, HB14 y HB15) de tipo inter simple sequence repeats (ISSRs) diseñados por Wolfe, Xiang and Kephartas (1998).
La reacción de PCR de amplificación utilizada tuvo 25 µl de volumen total; que contuvo 17.63 µl de agua purificada (InvitrogenTM), 2 µl (50 mM) de oligonucleótido, 0.3 µl de taq polimerasa (InvitrogenTM), 2 µl de ADN molde, 5.07 µl de master stock [1.38 µl de agua purificada, 2.6 µl de buffer (InvitrogenTM, pH = 8.4), 0.05 µl de MgCl2 (1M) y 0.26 µl (0.1 mM) de cada dNTPs (0.26 µl × 4 = 1.04 µl) (InvitrogenTM)]. La amplificación se realizó con el termociclador Mycycler Thermal Cycler-BIORAD (2012) y el programa de PCR consistió en pre-naturalización a 94 ºC durante cinco minutos, seguida de 44 ciclos para la etapa de naturalización a 94 ºC por un minuto. Se utilizó una etapa de alineamiento de 38 ºC para cada oligonucleótido con duración de un minuto, una etapa de extensión de 72 ºC de dos minutos y una extensión final de 72 ºC de 13 minutos. Los productos de PCR obtenidos se visualizaron en geles de agarosa a 1.4% a 110 V durante 180 minutos y se tiñeron con bromuro de etidio, para luego visualizarlos con luz ultravioleta.
La base de datos se obtuvo a través de la observación visual directa de cada gel, a partir de la cual se construyó una matriz de presencias y ausencias (ceros y unos) de las bandas para cada locus de cada individuo.
Análisis estadísticos de datos genéticos
Por medio del programa GenAlEx v. 6.501 (Peakall y Smouse, 2012) se determinó el número de bandas polimorficas observadas (I o ), exclusivas (I e ) por sitio, y bandas totales de los sitios (I T ). Los siguientes parámetros de diversidad genética tales como porcentaje de loci polimórficos con el criterio de 95% (% P), heterocigosis esperada (H e ) e índice de Shannon (I) (Hedrick, 2011) se obtuvieron en los nueve sitios muestreados.
La estructura genética se evaluó con el estadístico F st de Wright (1951), el cual es una medida de diferenciación genética entre las poblaciones. Posterior al calculó de F st se estimó número de migrantes por generación (Nm) de Crow y Aoki (1984). También, para entender las relaciones geográficas e interrelaciones genética entre los sitios se realizó un análisis de agrupamiento (clúster análisis) usando el estimador de similitud de distancias genéticas de Nei (1978). Además, para analizar la existencia de una relación entre las distancias geográficas y las distancias genéticas se realizó una prueba de Mantel (1967) calculadas a partir de los valores F st entre los sitios y convertidos a valores de flujo génico, Nm = Fst/(1-Fst) con estimaciones de densidad de Kernel a los gráficos de correlación (Jombart, 2008). Esta prueba identifica la correlación que existe entre dos matrices simétricas y devuelve el valor del coeficiente de correlación de Pearson, en dónde valores cercanos a 1 reflejan completa dependencia positiva, valores cercanos a 0 son independientes y valores cercanos a -1 indican completa dependencia negativa.
Asimismo, para estimar la estructura genética espacial a escala fina en el sitio de San Bernardo se construyó un mapa utilizando las coordenadas geográficas y los genotipos multilocus de cada individuo (n = 120 individuos) utilizando el programa ArcMap v.9.3. (Environmental System Research Institute [ESRI], 2008) y una prueba de autocorrelación espacial se realizó con base en la similitud/disimilitud del patrón de bandeo de los ISSRs usando el índice de distancia de Tanimoto´s (D k ) (Deichsel and Trampisch, 1985). Para probar que la población tuviera desviaciones significativas de la distribución espacial aleatoria de la media del valor D k se realizaron permutaciones utilizando el método de Monte Carlo a intervalos de confianza de 95%. Cada permutación consistió en una redistribución al azar de cada perfil de los ISSRs sobre las coordenadas espaciales de los individuos muestreados, el análisis se realizó utilizando el software de SGS v.1.0c (Degen, 2001). Finalmente, para determinar la relación de parentesco entre los individuos se realizó un análisis de red de expansión mínima (MSN), el cual agrupa genotipos multilocus (MLG) por distancias genéticas entre ellos. Cada MLG es un nodo y la distancia genética es representada por las aristas (líneas que conectan los nodos). Los nodos se encuentran conectados por la distancia mínima entre los individuos, que permite obtener un conjunto de nodos conectados entre ellos por distancias genéticas idénticas con base en un índice de disimilitud (e.g.Kamvar et al., 2015), en este estudio, la de Hamming. El análisis se realizó con el programa R v. 3.5.3 con la librería poppr y las funciones bitwise.dist y poppr.msn.
Resultados
En promedio 122.7 ± 3.5 bandas polimórficas claramente detectables y reproducibles se encontraron para la especie (Tabla 1) y no se detectó ninguna banda única. El intervalo en el número de bandas fue de 117 (El Panteón y El Orquidiario) a 125 (El Relámpago, La Luz, Cueva del Tigre y El Algodón). La heterocigosis esperada promedio fue de H e = 0.405 ± 0.003, mientras que, en promedio se encontró 97.16% ± 1.17% de loci polimórficos y un Índice Shannon promedio de 0.578 ± 0.004.
Tabla 1 Diversidad genética por sitio y promedio de la especie Oreomunnea mexicana en Sierra Juárez, Oaxaca.
| Sitio | n | IS | I o | I e | uH e | %P |
| San Bernardo | 25 | 0.576 ± 0.013 | 123 | 0 | 0.404 ± 0.011 | 97.60 |
| El Ídolo | 25 | 0.535 ± 0.018 | 123 | 0 | 0.366 ± 0.014 | 92.00 |
| El Panteón | 25 | 0.549 ± 0.019 | 117 | 0 | 0.389 ± 0.014 | 91.20 |
| El Orquidiario | 25 | 0.572 ± 0.016 | 117 | 0 | 0.405 ± 0.012 | 93.60 |
| El Relámpago | 25 | 0.632 ± 0.005 | 125 | 0 | 0.451 ± 0.005 | 100.00 |
| La Luz | 25 | 0.617 ± 0.005 | 125 | 0 | 0.436 ± 0,005 | 100.00% |
| El Algodón | 25 | 0.598 ± 0.006 | 125 | 0 | 0.418 ± 0.005 | 100.00 |
| Cueva del Tigre | 25 | 0.532 ± 0.008 | 125 | 0 | 0.358 ± 0.007 | 100.00 |
| La Cañada | 25 | 0.597 ± 0.005 | 125 | 0 | 0.416 ± 0.005 | 100.00 |
| Promedio | 25 | 0.578 ± 0.004 | 122.78 ± 3.18 | 0 | 0.405 ± 0.003 | 97.16 ± 1.27 |
n = número de individuos por muestra; IS = índice de Shannon, I e =número de bandas observadas; I e = número de bandas exclusivas; H e = heterocigosidad esperada; y %P = porcentaje de loci polimórficos. En paréntesis están las desviaciones estándares.
El sitio El Relámpago mostró la mayor diversidad genética en comparación con el sitio La Cueva del Tigre que obtuvo la menor diversidad de acuerdo con He y H´(Shannon). Los sitios El Algodón, La Cañada, La Cueva del Tigre, El Relámpago y La Cañada mostraron un porcentaje de loci polimórficos mayor al promedio, mientras que El Ídolo, El Panteón y El Orquidiario presentaron valores menores al promedio (Tabla 1). El índice de fijación poblacional (F st ) fue de 0.077 ± 0.018 y el flujo génico (Nm) de 2.25 migrantes por generación.
Las relaciones genéticas entre los sitios de O. mexicana mostró dos grupos, uno integrado por las poblaciones de Ixtlán de Juárez asociadas con dos poblaciones de Santiago Comaltepec (El Orquidiario y La Cañada) y el otro integrado por dos subconjuntos de poblaciones de Santiago Comaltepec (Fig. 2). La prueba de Mantel mostró una correlación negativa débil no significativa entre la relación de distancia geográfica y genética (r = -0.123; P = 0.575), no obstante, se observó diversas relaciones entre sitios de Santiago Comaltepec e Ixtlán de Juárez apartados en el diagrama con valores de correlación bajos en la estimación de densidad de Kernel (Fig. 3).
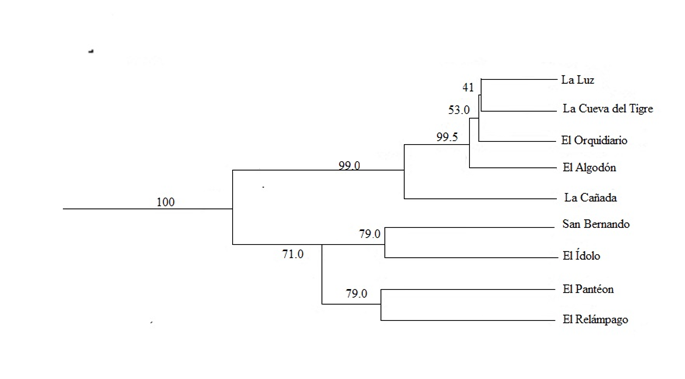
En cada nodo se indica el número de replicas que lo sustentan.
Figura 2 Fenograma de las poblaciones de O. mexicana en Sierra Juárez, Oaxaca.
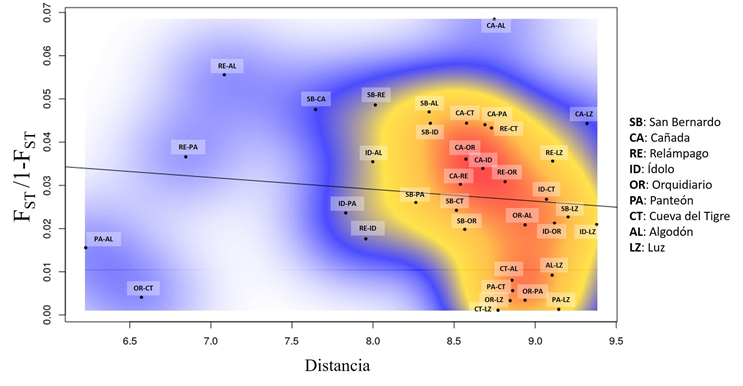
Los colores representan el valor acumulado de la relación entre pares de poblaciones de mayor (rojo) a menor (blanco).
Figura 3 Prueba de Mantel entre una matriz de distancias geográficas (Km) y una de distancias genéticas representadas por flujo génico (Nm) con estimaciones de densidad de Kernel.
El índice de Tanimoto mostró ausencia de autocorrelación significativa, lo cual indicó que la distribución de los genotipos multilocus fue al azar (Fig. 4). El análisis de parentesco con el análisis de red de expansión mínima encontró una similitud mayor a 92%, y una conexión entre nodos inmediatos de 0.01en distancia genética. El mayor número de conexiones a otros genotipos dentro de la población fue registrado por los genotipos o nodos principales 6, 11, 18, 12, 39 y 98 que mostraron una distancia promedio entre ellos de 0.022 (Fig. 5). Los genotipos multilocus que integraron cada nodo principal no se encontraron distribuidos de manera agregada, sino distribuidos espacialmente en el área de estudio.
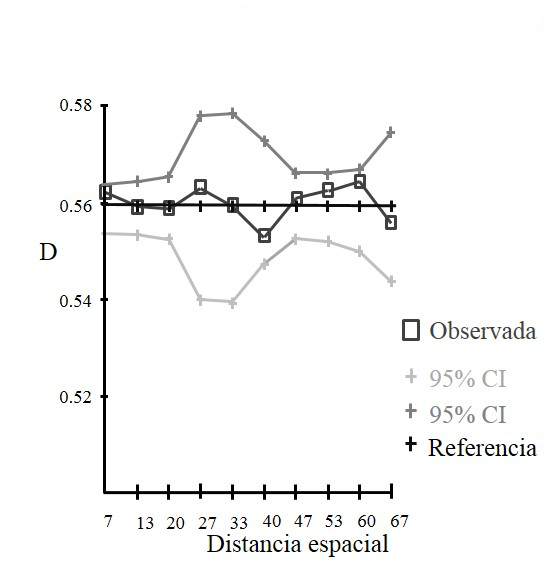
Los puntos representan los valores observados de distancia (D), la línea solida representa los valores esperados para una distribución al azar y las líneas con puntos son los límites de confianza superior e inferior a 95%.
Figura 4 Distograma con coeficientes de autocorrelación espacial y clases de distancia entre los individuos de O. mexicana en la Sierra Juárez, Oaxaca.
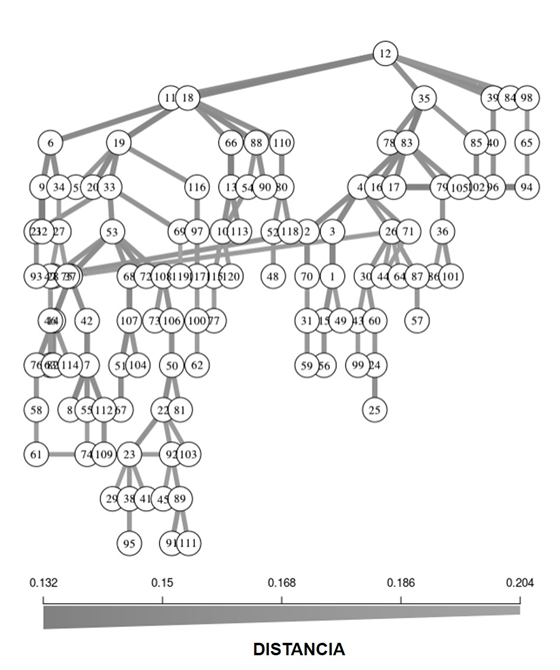
Los nodos (círculos) representan genotipos individuales de multilocus únicos. Los bordes (líneas) representan la distancia genética mínima entre individuos determinada por el algoritmo de Prim.
Figura 5 Red de expansión mínima basada en la distancia genética de Hamming para 120 individuos de O. mexicana del sitio San Bernardo.
Discusión
La diversidad genética obtenida con marcadores dominantes (ISSRs), como los utilizados en las poblaciones de O. mexicana fue intermedia comparable al de especies endémicas con distribución restringida del orden Fagales en Oaxaca (Pacheco-Cruz, 2019), sin embargo es alta si se compara con estudios de especies de árboles relictos, como Podocarpus sellowi (H e = 0.072) (Dantas et al., 2015), Gomortega keule (H e = 0.2465 - 0.4627) (García-Gonzáles et al., 2008) y Abies zyuanensis (H e = 0.136) (Tang et al., 2008).
Se ha sugerido que el mantenimiento de la diversidad genética a través del tiempo en una planta relicto puede ser explicada en parte por la historia de vida común del género (Rodríguez-Barahona y Eguiarte, 2014). Entre las características de historia de vida que podrían amortiguar la pérdida de diversidad genética y que presenta la especie en Sierra Juárez, Oaxaca se encuentran: 1) una alta longevidad y alta abundancia de individuos, aspecto que cumplía la especie al momento de este estudio (cfr. Alfonso-Corrado et al., 2017) y la distingue de otras relicto con bajo número de individuos en sus poblaciones (García-Gonzáles et al., 2008), 2) contar con un sistema de polinización por viento e incompatibilidad en el sistema reproductor (Stone, 1972; Russell y Cohn, 2012), 3) establecimiento constantes de nuevos individuos vía semilla, en cuyo proceso ocurre recombinación genética y nueva diversidad genética (Hedrick, 2011) y 4) estar presente en hábitats de estadios sucesionales tardíos (Naranjo-Luna, 2014).
Un aspecto destacable, que se ha descrito recientemente es el efecto de las oscilaciones climáticas en el tamaño poblacional y variación genética de Alsophyla firma en el bosque de niebla durante el Pleistoceno (Rodríguez-Barahona y Eguiarte, 2014). Este proceso de oscilación climática asociado con las características de historia de vida, podrían explicar los niveles actuales de variación genética en O. mexicana, a futuro un estudio filogeográfico con modelos paleoclimáticos permitiría analizar este suceso.
Por otro lado, las poblaciones de O. mexicana no se encuentran estructuradas, lo cual, en árboles de vida larga, como lo es O. mexicana se ha atribuido al sistema de polinización por viento, que permite que el polen viaje largas distancias, por lo que un flujo de génico mayor a uno es suficiente para homogenizar las frecuencias alélicas y evitar la diferenciación genética entre poblaciones (Wright, 1951; Hedrick, 2011). Esto no es común en especies relictos con poblaciones aisladas y fragmentadas que presentan aislamiento por distancia y poblaciones estructuradas (Tang et al., 2008). No obstante, que las poblaciones de O. mexicana no mostraron un aislamiento por distancia significativo, el resultado en la prueba de Mantel y el análisis de agrupamiento sugieren un proceso de diferenciación entre poblaciones, particularmente entre las poblaciones de Ixtlán de Juárez y Santiago Comaltepec. Este proceso de diferenciación puede ser generado por la fragmentación y discontinuidad entre poblaciones (García-Gonzáles et al., 2008), así como el sistema orográfico conformado por montañas con altitud mayor a 3500 m snm, que en conjunto pueden actuar como barrera geográfica que interfieren con la dispersión de polen.
Un aislamiento por distancia entre poblaciones atribuido a fragmentación e interferencia en la dispersión de polen por montañas es reportado en la Sierra Juárez por Pacheco-Cruz (2019) para el encino microendémico Quercus macdougallii, a pesar de su corta distancia entre el par más distante de poblaciones (< 20 km), mayor que la O. mexicana de 9.2 km. Este patrón que no ocurre en Q. crassifolia, un encino cuya distribución y conectividad entre poblaciones es mayor en la Sierra Juárez (Gorgonio-Ramírez, Clark-Tapia, Campos, Monsalvo-Reyes y Alfonso-Corrado, 2017). Por ello, es necesario explorar el efecto de aislamiento genético a futuro, particularmente ante el proceso de cambio ambiental en el área de estudio que han afectado la distribución y ecología de la especie (Alfonso-Corrado et al., 2017; Pacheco-Cruz et al., 2018), y donde un incremento en la diferenciación genética puede tener efectos en la diversidad genética y en los procesos evolutivos de la especie (Epperson y Álvarez-Buylla, 1997).
A escala de población, la dispersión de semillas es a corta distancia (< 30 m) de la madre, ello a pesar de estar adaptada con brácteas trilobadas para dispersión de las semillas por viento (Russell y Cohn, 2012; Pacheco-Cruz et al., 2019). La ausencia de una estructuración local significativa en el sitio de San Bernardo como consecuencia de una distribución espacial aleatoria de genotipos multilocus, es comparable a especies de pinos cuyo polen y semillas es dispersada por viento (Friedrich, Hernández-Díaz, Leinemann, Prieto-Ruíz y Wehenkel, 2018). No obstante, es común que plantas con dispersión limitada de semillas exhiban una estructura espacial marcada incluso si el polen se dispersa a largas distancias (Epperson y Álvarez-Buylla, 1997). La ausencia de estructura genética espacial a escala fina en O. mexicana, podría ser explicada por un fenómeno conocido como sombra de semillas, donde individuos emparentados, producto de una dispersión de semilla a corta distancia son eliminados con el tiempo por sobrelapamiento temporal y espacial de diferentes cohortes de plántulas (Hamrick, Murawski y Nason, 1993; Epperson y Álvarez-Buylla, 1997; Terra, Shirk y Givnish, 2014). Adicionalmente, el efecto de autoaclareo debido a competencia interespecíficas entre grupos emparentados puede generar espaciamiento entre los individuos dejando pocos parientes cercanos (Hamrick et al., 1993), aspecto observado en este estudio. Un análisis genético espacial en otras áreas podrá a futuro brindar mayor claridad acerca de la estructura espacial de la especie, así como determinar efectos en la diversidad y estructura genética.
Conclusiones
Los niveles moderados de diversidad genética indican que la especie no se encuentra degradada genéticamente. Aun cuando sus poblaciones muestran ausencia de estructuración a escala regional y local, se observó una tendencia a la estructuración debido a procesos de cambio ambiental que afectan el tamaño de las poblaciones, dispersión de semillas a escala local e interferencia en la dispersión de polen por barreras geográficas. Se debe evaluar a futuro con marcadores moleculares más finos y más poblaciones el efecto de cambio en el ambiente e historia de vida de la especie sobre la diversidad genética y procesos evolutivos. Para su conservación in situ, se sugiere dé prioridad para su protección inmediata a las poblaciones de El Relámpago (por su gran riqueza alélica), El Panteón, El Orquidiario y San Bernardo por la alta vulnerabilidad por perturbación antrópica (además de selección de árboles progenitores, distantes entre sí, para la colecta de semillas en programas de reforestación o para tener representado el germoplasma de O. mexicana.











 text new page (beta)
text new page (beta)



