Introducción
Los productos forestales no maderables son todos aquellos recursos de origen biológico que provienen de los bosques, exceptuando la madera. Tienen un gran significado cultural y de valor para habitantes de zonas rurales y urbanas. Entre ellos se encuentran los hongos (Rivera-García, Dávalos-Sotelo y Lascurain-Rangel, 2013). Los hongos silvestres son un recurso forestal no maderable que ha generado un gran impacto sobre los grupos culturales del mundo, incluyendo principalmente su importancia como alimento, medicina, uso recreativo y lúdico (López-García, Jiménez-Ruiz y Pérez-Moreno, 2017). En México, como en otros países, muchas personas están familiarizadas con el cultivo de hongos comestibles y para otros usos (Okhuoya et al., 2010). El número de hongos comestibles registrado en el mundo es de alrededor de 14 000 especies (Chang y Wasser, 2012), mientras que en nuestro país se han registrado cerca de 300 especies comestibles y medicinales provenientes principalmente de los bosques templados (Garibay-Orijel, Ruán-Soto y Estrada-Martínez, 2010). Entre los hongos cultivados comercialmente, 85% es producido por especies de tan solo cinco géneros y en 2013 la producción mundial se estimó en 34 millones de toneladas (Royse, Baars y Tan, 2017). Agaricus L. (Agaricaceae, Agaricomycetes, Basidiomycota) es un género cosmopolita que incluye entre 200 y 450 especies de hongos saprobios (Cappelli, 1984; Bas, 1991; Kirk, Cannon, Minter y Stalpers, 2008; Zhao et al., 2011b; Karunarathna et al., 2016; International Mycological Association [IMA], 2018), algunas de ellas con importancia nutricional e incluso medicinal [e.g. Agaricus bisporus (J.E. Lange) Imbach]. Zhao et al. (2011b) reconocieron 386 especies alrededor del mundo (203 de zonas templadas, 183 de zonas tropicales), y enfatizaron que la diversidad de especies de Agaricus en zonas tropicales está subestimada y que el número de especies presentes en dichas regiones probablemente sea mayor a aquel en regiones templadas. Debe considerarse que la taxonomía de este género es muy compleja (Cappelli, 1984; Karunarathna et al., 2016). A pesar de su gran diversidad taxonómica y de su importancia económica, este género ha sido poco estudiado en México; solo destacan los trabajos de Gutiérrez-Ruíz y Cifuentes (1990) para todo el país y de Medel, Palestina-Villa y Mata (2015) para el estado de Veracruz. Al menos 43 especies de Agaricus han sido citadas para México (Herrera y Guzmán, 1972; Bandala-Muñoz, Guzmán y Montoya-Bello, 1988; inter alia), que equivalen a alrededor de 10% de la diversidad global conocida de este género.
Agaricus subrufescensPeck (1894) es un hongo comestible al que se le atribuyen propiedades medicinales (e.g. antigenotóxicas y antitumorales), por lo que ha sido estudiado y cultivado en varias partes del mundo (Wisitrassameewong et al., 2012b). Fue descrito de Nueva York, EE.UU. Sus poblaciones naturales están ampliamente distribuidas en el mundo, pues se han citado de casi todos los continentes (excepto de Australia y Antártica) y de algunas islas del Pacífico y del Índico (e.g. Peck, 1897; Kauffman, 1918; Murrill, 1922; Zeller, 1922; Kerrigan, 1986; Nauta, 1999, 2001 [como A. rufotegulis Nauta]; Wasser et al., 2002 [como A. brasiliensis Wasser, Didukh, De Amazonas & Stamets]; Peterson, Desjardin y Hemmes, 2000; Arrillaga y Parra, 2006; Ludwig, 2007; Wisitrassameewong et al., 2012a; Parra, 2013; Gui et al., 2015; Thongklang et al., 2016 [como sinónimo de A. bambusae Beeli var. bambusae]). La especie pertenece a la sección Arvenses (Konrad & Maubl.) Konrad & Maubl. del subgénero Flavoagaricus Wasser, y varios estudios filogenéticos han confirmado la posición de A. subrufescens dentro de esta sección (e.g.Zhao et al., 2011b). Los representantes de dicha sección se caracterizan por presentar anillo súpero y doble (dos capas sobre el estípite); contexto que se mancha de amarillo al maltratarse; queilocistidios catenulados frecuentemente presentes; reacción positiva al KOH (coloración amarillenta) y a la mezcla de Schäffer (coloración amarillo-anaranjada a rojo); y olor y sabor a anís o almendra. Particularmente, A. subrufescens se caracteriza por la pigmentación café-rojiza de las fibrillas sobre el píleo (e.g.Nauta, 2001).
Existen discrepancias en cuanto a la circunscripción de esta especie. Por un lado, algunos autores propusieron un concepto restringido para A. subrufescens (i.e.Wasser et al., 2002, 2005; Didukh, Wasser y Nevo, 2003; Didukh, Vilgalys, Wasser, Isikhuemhen y Nevo, 2005), pues reconocieron a otras como especies independientes; a saber: A. brasiliensis (descrita de América del Sur) y A. rufotegulis (descrita de Europa). Por otro lado, la mayoría de los autores aceptan un concepto más amplio (e.g.Kerrigan, 2005, 2007), pues consideran a dichas especies como sinónimos de A. subrufescens, el nombre actualmente aceptado sensu auctorum. Thongklang et al. (2016) agregaron un sinónimo más para A. subrufescens, a saber: A. bambusae var. bambusae). Además, la referencia errónea para este hongo y sus cepas cultivadas bajo el nombre de A. blazei Murrill fue acertada y detalladamente aclarada por Wasser et al. (2002), de manera que actualmente se considera que A. blazei sensu Heinnemman es también un sinónimo de A. subrufescens. En este trabajo se acepta el sensu lato para A. subrufescens, que considera a todas estas especies como sinónimos. De hecho, esta especie es cultivada y comercializada en varias partes del mundo bajo los nombres de A. blazei Murrill y A. sylvaticus Schäeff. (Wasser et al., 2002; Kerrigan, 2005; Wisitrassameewong et al., 2012b; Kerrigan, 2016), pero solamente se trata de denominaciones taxonómicas mal aplicadas; asimismo, es conocida con varios nombres vernáculos según el área geográfica de donde que provenga (e.g. ‘Himematsutake’, ‘Agarikusutake’ y ‘Kawariharatake’ en Japón; Cogumelo-do-sol, ‘Cogumelo de Deus’ o ‘Cogumelo de vida’ en Brasil; Sun Mushroom y Royal Sun Agaricus en la mayor parte de Europa; ‘Mandelpilz’ en Alemania; ‘Almond mushroom’ en EUA; y ‘Champiñón almendra’ en España) (Wasser et al., 2002, 2005; Kerrigan, 2005; Wisitrassameewong et al., 2012b; Parra, 2013).
La importancia nutracéutica, terapéutica y farmacéutica de esta especie está fuertemente sustentada, pues desde 1968 se han publicado al menos 1000 artículos clínicos dedicados a sus propiedades benéficas (e.g.Wisitrassameewong et al., 2012b). Se han aislado e identificado varios compuestos bioactivos a partir de los basidiomas y del micelio de este hongo, los cuales han mostrado actividad contra distintas enfermedades tan relevantes como cáncer de pulmón y de estómago (Itoh, Ito y Hibasami, 2008). Debido a estas propiedades culinario-medicinales, A. subrufescens representa uno de los recursos forestales no maderables más prometedores en cuanto a su alto potencial de aprovechamiento a escala mundial.
A. subrufescens fue registrado de México por Martínez-Carrera et al. (2001) haciendo referencia a una cepa aislada de un espécimen silvestre recolectado en el estado de Puebla. Sin embargo, este reporte no está sustentado ya que dichos autores no proporcionan datos sobre el ejemplar de herbario y/o una secuencia de DNA disponible, ni aportaron una descripción morfológica, por lo que su registro no puede ser confirmado formalmente y tampoco se tiene información sobre su morfología, hábitat, distribución o variabilidad genética. Así, el objetivo de este trabajo es ampliar el conocimiento sobre A. subrufescens mediante la caracterización morfológica y molecular (región ITS nuc-rDNA) de nuevas recolectas silvestres, a fin de respaldar y corroborar la presencia de dicha especie en México, lo cual representaría un potencial económico para el país.
Materiales y métodos
Recolectas y descripción morfológica
Se realizaron exploraciones dirigidas a la búsqueda de especímenes de Agaricus subrufescens en distintas localidades de la porción central del estado de Veracruz durante 2011, 2012 y 2013, incluyendo sitios con bosque de coníferas, bosque de encino, bosque mesófilo de montaña y bosque tropical perennifolio (sensuRzedowski, 1978). Los basidiomas frescos recolectados fueron caracterizados morfológicamente siguiendo la terminología de Largent (1977) y de Parra (2008). La codificación de los colores se basó en el sistema de Munsell (1979). Además, sobre la superficie de las distintas porciones del basidioma se hicieron las pruebas macroquímicas de KOH (al 10%) y reacción de Schäeffer (solución acuosa de anilina + solución de ácido nítrico al 5%) (Arranz et al., 2000; Parra, 2008). Se seleccionaron y separaron los basidiomas para el aislamiento de cepas abajo descrito. El resto de los especímenes fueron secados a temperaturas entre 40 °C y 60 °C. Para el estudio micromorfológico, se montaron preparaciones temporales usando KOH al 5%, solución de floxina ácida, Rojo Congo, y reactivo de Melzer (Largent, Johnson y Watling, 1977) de las distintas partes del basidioma (píleo, estípite, contexto e himenio) en fresco y en seco, y se observaron bajo el microscopio óptico (Carl Zeiss Primo Star). Todas las medidas fueron presentadas en forma de intervalo (valores mínimo-máximo) cuando se hizo más de una medición. Para el caso de las medidas de los elementos microscópicos, el número de mediciones siempre fue de al menos 30; el valor x m corresponde al intervalo de medias aritméticas parciales calculadas a partir de las mediciones realizadas en cada preparación fija (n > 30). El índice Q representa la proporción individual longitud/anchura de cada espora medida y es presentado como un intervalo de valores sin media aritmética. La identificación taxonómica del material mediante morfología se basó en los trabajos de Peck (1897), Kerrigan (1986, 2016) y Parra (2013). El código MB es el número de registro en la base de datos Mycobank (IMA, 2018). El material estudiado se depositó en la colección de hongos del herbario XAL (Instituto de Ecología, A.C., Xalapa, Veracruz, México).
Propagación de cepas
Para la propagación de cepas, se aisló tejido miceliar a partir del contexto del píleo de los basidiomas frescos, usando como medios de cultivo papa-dextrosa-agar (PDA) y medio completo básico (CMB, descrito en Maia et al., 2012). Las cepas aisladas se incubaron a 25 °C, se observaron y caracterizaron en fresco haciendo preparaciones temporales para microscopía óptica usando KOH al 5%, Rojo Congo y reactivo de Melzer (Largent et al., 1977). Posteriormente, las cepas fueron resembradas en los mismos medios de cultivo, además de medio en agar de extracto de composta comercial para Agaricus bisporus, y se mantuvieron en refrigeración a 4 °C. Después, las cepas fueron inoculadas en semilla esterilizada de sorgo y de mijo, e incubadas a 25 °C. Finalmente, el inóculo obtenido en semilla fue sembrado en pacas de 4 kg de composta comercial usada para cultivo de A. bisporus, e incubado a 25 °C.
Caracterización y análisis moleculares
1. Extracción de DNA, PCR y secuenciación. Para el aislamiento de DNA a partir de los basidiomas frescos y de las cepas puras, se colocaron 1 mm2 - 2 mm2 de micelio en tubos Eppendorf de 0.6 μL con el kit Extract-NAmpTM (Sigma-Aldrich, Missouri, EUA), los cuales se agitaron en vortex y se incubaron durante dos minutos a 90 °C. La región ITS se amplificó utilizando los iniciadores ITS1F y ITS4 descritos por Gardes y Bruns (1993) usando 25 μL del kit de PCR USB® RubyTaqTM PCR Master Mix (Sigma-Aldrich, Missouri, EUA) mezclado con 0.5 μL de cada iniciador [concentración 50 μMol/ μL], 11 μL de H2O miliQ y 1 μL de cada muestra de DNA; el termociclador estuvo bajo las condiciones descritas por Izzo, Agbowo y Burns (2005). Los productos positivos de PCR fueron limpiados usando el KIT de purificación GeneJET PCR Purification Kit (Thermo Scientific, Massachusetts, EUA). Los productos de purificación y los iniciadores usados en la PCR fueron enviados a Macrogen Inc. (Seúl, Rép. de Corea) para su secuenciación. La secuencia recibida fue editada en el programa Sequencher (Gene Codes Co., Michigan, EUA) y está disponible en la base de datos GenBank de Ncbi con el número de acceso KY114572.
2. Análisis filogenéticos. Para corroborar la identificación taxonómica y la ubicación filogenética del material estudiado, se hicieron reconstrucciones filogenéticas. Para ello, se hizo una primera matriz de la secuencia obtenida junto con secuencias seleccionadas identificadas como A. subrufescens o cercanas a esta especie, utilizando la herramienta de búsqueda Blast en la base de datos GenBank de Ncbi (Maryland, EUA). Solamente se consideraron secuencias ya publicadas (e.g.Didukh et al., 2005; Geml, Geiser y Royse, 2004; Geml, Laursen y Taylor, 2008; Kerrigan 2005; Zhao et al., 2011b; Gui et al., 2015), y se descartaron secuencias provenientes de cepas de cultivo, híbridos y especímenes de origen incierto. Con base en el estudio de Zhao et al. (2011b), en la matriz también se incluyeron secuencias de algunas especies de la sección Arvenses, y se usaron secuencias de cuatro especies de la sección Minores como grupo externo, ambas secciones representativas del subgénero Flavoagaricus dentro del género Agaricus. Esta matriz se sometió a un alineamiento múltiple con la herramienta Clustal W implementada en el programa BioEdit 7.2.5 (Hall, 1999). El alineamiento resultante se editó manualmente en el mismo programa y se analizó con los métodos de inferencia bayesiana de probabilidades posteriores (BPP), máxima verosimilitud (ML) y parsimonia (MP). El análisis de BPP (Fig. 1) fue realizado usando el programa MrBayes (Huelsenbeck y Ronquist, 2001; Ronquist y Huelsenbeck, 2003), considerando el modelo GTR de sustitución de nucleótidos y una tasa de variación con forma gamma; se montaron cuatro cadenas de 4 Monte Carlo sobre 1 111 000 generaciones con un submuestreo cada 400 generaciones y un calentamiento de cadena de 0.2; la longitud de ramas fue mantenida al estimar su probabilidad posterior, la cual se indica en la figura 1 (a la izquierda por encima de cada rama). El análisis de ML (resultado no mostrado) se realizó en el programa PhyML v3.0 (Guindon et al., 2010) implementado en el sitio ATGC usando el modelo HKY85 de sustitución de nucleótidos y una proporción estimada de sitios invariables de 0.403, suponiendo seis categorías de tasa de distribución gamma para explicar la tasa de heterogeneidad a través de sitios; como árbol inicial se construyó un dendrograma de consenso de neighbor-joining con la misma matriz, usando el modelo de distancia genética de Tamura-Nei con un remuestreo de bootstrap con 1 000 pseudoréplicas; el parámetro de la forma gamma fue estimado directamente desde la matriz (gamma = 0.513); la verosimilitud se incrementó usando el mejoramiento de árbol SPR; se usaron 100 pseudoréplicas de bootstrap para el cálculo del soporte de ramas (Fig. 1, a la derecha por encima cada clado). El análisis de MP (resultado no mostrado) fue realizado en el programa Paup 4.0b10 (Swofford, 2003), sin peso de caracteres; se obtuvieron 68 árboles igualmente parsimoniosos (314 pasos); este análisis reveló 98 sitios variables informativos en la matriz; finalmente se construyó un árbol de consenso estricto con todos los árboles obtenidos; se hicieron 10 000 pseudoréplicas de bootstrap para el cálculo del soporte de ramas (Fig. 1, por debajo de cada clado).
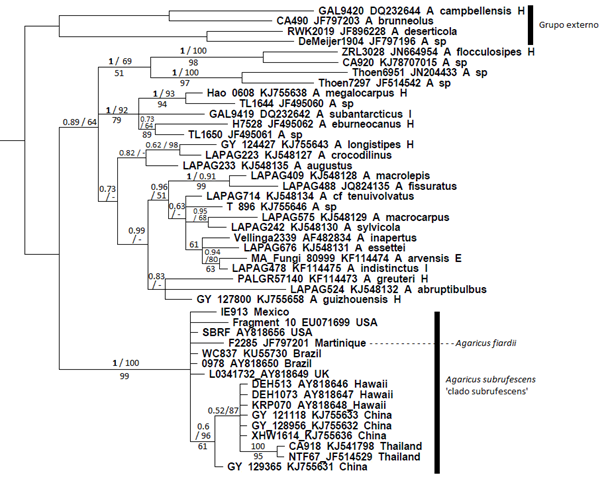
Los valores de probabilidad posterior de cada rama se muestran del lado izquierdo por encima las líneas basales de cada clado; los valores de soporte de ramas por bootstrap de máxima verosimilitud se muestran del lado derecho por encima las líneas basales de cada clado; los valores de soporte por bootstrap del análisis de parsimonia se muestran por debajo de las líneas basales de cada clado.
Figura 1 Cladograma de consenso del análisis filogenético por inferencia bayesiana que muestra la posición filogenética de la cepa IE913 dentro del ‘clado subrufescens’ en la sección Arvenses del género Agaricus.
3. Análisis de distancia genética. En los tres análisis filogenéticos (i.e. BPP, ML y MP) estuvo representado un clado monofilético, aquí nombrado ‘clado subrufescens’, el cual agrupó a 16 secuencias referidas a A. subrufescens o sus sinónimos. Con las 16 secuencias pertenecientes al ‘clado subrufescens’, se construyó una segunda matriz cuyo alineamiento se realizó con herramienta Clustal W implementada en el programa BioEdit 7.2.5 (Hall, 1999), el cual se editó manualmente en el mismo programa. El segundo alineamiento resultante fue analizado mediante métodos de distancia genética: Upgma (Mega 6.06), neighbor-joining (BioEdit 7.2.5), splitts tree (V.4.13.1), minimum spanning tree (WinArlequin 3.5.1.2) y red de haplotipos (TCS 1.21). Los análisis de splitts tree y minimum spanning tree se realizaron considerando la matriz entera como una sola población; en tanto, para el análisis de red de haplotipos se consideró 95% de similitud genética como umbral de conectividad. Debido a que los polimorfismos de la región ITS se han considerado útiles para caracterizar especies y variedades dentro del género Agaricus (Callac et al., 2003; Challen, Kerrigan y Callac, 2003; Gui et al., 2015), estos fueron usados en este estudio para tratar la secuencia de material mexicano.
Resultados
Recolecciones y cepas
De todos los sitios explorados, solo en una localidad con bosque mesófilo de montaña se encontró un espécimen cuyas características tanto macro- como micromorfológicas coincidieron con lo descrito para A. subrufescens en la bibliografía (Peck, 1894; 1897; Kauffman, 1918; Murrill, 1922; Zeller, 1922; Kerrigan, 1986; Nauta, 1999, 2001; Wasser et al., 2002; Peterson et al., 2000; Arrillaga y Parra, 2006; Ludwig, 2007; Wisitrassameewong et al., 2012a; Parra, 2013; Gui et al., 2015; Thongklang et al., 2016). Su respectiva cepa aislada y pura fue depositada en el Cepario de Hongos del Instituto de Ecología, A.C., Xalapa, Veracruz, México (IE) bajo la clave IE913. Se obtuvo crecimiento miceliar de la cepa en todos los medios de cultivo y semilla utilizados; en cambio, el crecimiento del inóculo en composta comercial para A. bisporus fue limitado y no se obtuvo fructificación. Asimismo, el aislamiento de DNA de la cepa y su secuenciación de DNA fueron efectivos.
Análisis filogenéticos
Las topologías obtenidas en los tres métodos filogenéticos (i.e. BPP, ML y MP) fueron congruentes en lo general; por tanto, solamente el cladograma de BPP es presentado en la figura 1, en donde se muestran como soporte de ramas los valores de probabilidad posterior para el análisis de BPP y los de bootstrap para los análisis de MP y ML. Estos análisis fueron coincidentes en ubicar la secuencia obtenida del material proveniente de México (IE913) junto con otras 14 secuencias referidas como A subrufescens, como alguno de sus sinónimos (A. brasiliensis y A. rufotegulis) o como el nombre mal aplicado de A. blazei, además de la secuencia F2285-JF797201 identificada como A. fiardii, todas ellas formando un clado monofilético dentro de la sección Arvenses con un alto soporte de ramas (probabilidades posteriores = 1; ML bootstrap = 100%; MP bootstrap = 99%), el ‘clado subrufescens’.
Particularmente para el ‘clado subrufescens’, no se recobró una estructura histórica interna consistente en los tres análisis filogenéticos (i.e. BPP, ML y MP), sino que prácticamente se presentó como una politomía; el único grupo constantemente definido dentro de este clado corresponde al que agrupa a las secuencias de China, Hawái y Tailandia en dichos análisis con soporte (probabilidades posteriores = 0.6; ML bootstrap = 96%; MP bootstrap = 61%) (Fig. 1).
Análisis de distancia genética
Los análisis de distancia genética (aquellos realizados al segundo alineamiento, el cual incluye solo secuencias agrupadas en el ‘clado subrufescens’) muestran que la secuencia de México tiene dos sitios polimórficos únicos, es decir, que no están representados en el resto de las secuencias (únicamente se muestran los resultados de la red de haplotipos, Fig. 2). Estos análisis también muestran que las secuencias de China, Hawái y Tailandia son las más disyuntas con cuatro sitios polimórficos únicos. La similitud genética mínima para la región ITS del ‘clado subrufescens’ fue 98.52%, por lo que en el análisis de la red de haplotipos no hubo corte de conectividad entre las secuencias; en tanto, la tasa de sustitución promedio por sitio fue 0.008.
Caracterización morfológica (Figs. 3-4)
Basidioma agaricoide y placomicetoide (con tendencia a ser más alto que ancho). Píleo 83 mm de diámetro; convexo a convexo-plano, sin umbo; cubierto de escamas flocoso-fibrilosas, adpresas, de color café rojizo (2.5YR 6/6) a café rosado (2.5RP 6/6), y densamente agrupadas hacia el centro del píleo, sobre un fondo blanquecino a beige (5Y 9/2 - 5Y 8/4); superficie seca. Láminas libres; estrechas a segmentiformes; muy juntas a densamente dispuestas; de color blanquecino a rosáceo (5Y 9/2 - 7.5RP 9/4) cuando inmaduras, y de color café-púrpura negruzco (2.5RP 1/2) al madurar; con lamélulas de tres diferentes tamaños; con arista lisa y concolora. Estípite 108 mm × 18 mm; subcilíndrico; liso a ligeramente fibriloso bajo el anillo; blanco; con base bulbosa (26 mm de ancho), sin rizomorfos. Anillo súpero; blanco; y doble (la capa superior como un anillo membranoso, colgante, más o menos amplio, flácido y con la cara superior lisa; y la capa inferior como parches esquamuloso-fibrilosos sobre la cara inferior del anillo). Contexto carnoso; blanquecino (N10 - 5Y 9/2), con ligero cambio a amarillento al maltrato o al corte (5Y 8/2 - 8/4); olor y sabor dulce (a almendras). Esporada de color café-rojo negruzco (5RP 1/2).
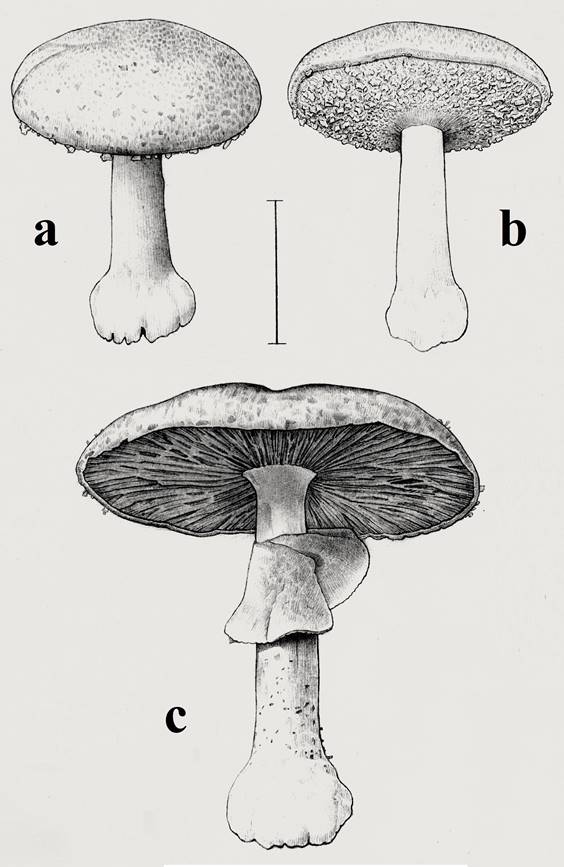
a: patrón de escamas en el píleo y borde del mismo, base del estípite bulbosa. b: aspecto del patrón de escamas en el anillo de aspecto fibriloso. c: patrón de distribución de las láminas en el himenio y anillo colgante.
Figura 3 Esquemas del basidioma del espécimen mexicano de Agaricus subrufescens mostrando distintos aspectos de su morfología (barra = 2 cm).
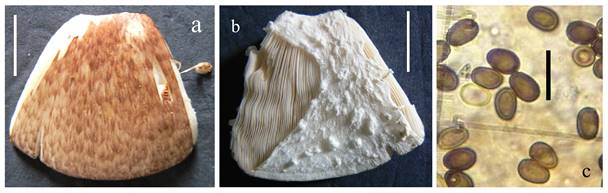
a: detalle de la superficie del píleo mostrando el patrón de las escamas (barra = 20 mm). b: detalle de la superficie inferior del anillo que muestra el patrón de escamas (barra = 20 mm). c: esporas en KOH al 5% bajo microscopía óptica (barra = 10 µm).
Figura 4 Morfología del espécimen mexicano de Agaricus subrufescens.
Basidiosporas 5.6 - 6.9 (7.5) × 3.5 - 4.6 (4.9) µm (x m = 6.1 - 6.4 × 4.1 - 4.4 µm), Q = 1.28 - 1.73, suboblongas u ovoides a subelípticas; de color café oscuro a café rojizo; de pared gruesa (0.4 µm - 0.6 µm); poro germinal no distinguible. Basidios 13.5 - 16 × 5.6 - 7 µm; clavados; tetraspóricos; de pared delgada; hialinos. Queilocistidios 7 - 12 × 6 - 10 µm; subglobosos; simples o catenulados; hialinos; de pared delgada; frecuentes a abundantes. Reacciones macroquímicas: reacción de Schäffer positiva (anaranjado-amarillo a anaranjado-rojo) sobre la superficie del píleo, el contexto (menos evidente) y la superficie del estípite; y reacción al KOH positiva (amarillento con tinte oliváceo) sobre la superficie del píleo y del estípite.
Hábito solitario. Hábitat terrícola al pie de un árbol latifoliado en el interior de bosque mesófilo de montaña a 1340 m snm.
Material estudiado. Veracruz, mpio. Xalapa, Santuario del Bosque de Niebla; 19° 30' 48.71" N, 96° 56' 35.59" O; 31/enero/2012; Velázquez-Narváez 2289 (XAL) (cepa IE913).
Discusión
La presencia de Agaricus subrufescens en México es confirmada, y este es el primer registro respaldado, pues tanto el estudio morfológico como los análisis filogenéticos realizados en este trabajo confirman la identificación taxonómica del material estudiado y su ubicación en el ‘clado subrufescens’ dentro de la sección Arvenses. Dicho clado incluyó todas las secuencias referidas como A. subrufescens, como sus supuestos sinónimos A. brasiliensis y A. rufotegulis o como A. blazei (nombre mal aplicado). El hecho de que dicho clado haya agrupado también a una secuencia referida en GenBank como A. fiardii, sugiere que esta última se trata de una especie filogenéticamente muy cercana a A. subrufescens o incluso de su probable sinónimo. A. fiardii fue descrita por Pegler (1983) de las Antillas Menores en el Caribe, y en general los caracteres morfológicos señalados en el protólogo son bastante semejantes a los de A. subrufescens, salvo que el tamaño de esporas es ligeramente menor en la primera especie: 4.7 - 6 × 3.2 - 4.5 µm (x m = 5.3 ± 0.29 × 3.6 ± 0.27 µm). El análisis filogenético realizado por Zhao et al. (2011b) apoya la idea de la cercanía filogenética de A. fiardii con A. subrufescens, aunque tales autores no lo suscriben.
Existen algunas especies de la sección Arvenses que presentan fibrillas pigmentadas sobre la superficie del píleo, al igual que A. subrufescens; pero el color de la pigmentación es distinto: en A. augustus Fr. y A. salicophilus M. Lange es café-dorado (Cappelli, 1984; Kerrigan, 1986, 2016; Parra, 2013), en A. flocculosipes R.L. Zhao, Desjardin, J. Guinberteau & K.D. Hyde y en A. subtilipes Thongklang, L.J. Chen, Callac & K.D. Hyde es café-gris a café-anaranjado (Zhao et al., 2011a; Thongklang et al., 2016) y en A. nanaugustus Kerrigan es café-anaranjado (Kerrigan, 2016). De ellas, solamente A. augustus está registrada de México (Gutiérrez-Ruíz y Cifuentes, 1990), y también se distingue de A. subrufescens por un tamaño mayor de basidiosporas (7.5 - 10.7 × 4.8 - 6.9 µm; x m = 8.6 × 5.5 µm) (Parra, 2013). A. subantarcticus Geml, Laursen & D. Lee Taylor (descrita de Nueva Zelanda) presenta escamas sobre el píleo con una pigmentación semejante (gris-beige a café-rojizo sobre un fondo amarillo miel), pero estas son mayores a las de A. subrufescens; además, sus basidiosporas son claramente más largas (7 - 9 × 4 - 5.5 µm; x m = 7.9 × 4.8 µm; Q = 1.54 - 1.85) (Geml, Laursen, Nusbaum y Taylor, 2007); filogenéticamente, se acerca más a otras especies (Fig. 1). El resto de las especies de la sección Arvenses se distingue por presentar escamas sin pigmentación o por carecer de ellas. Las especies de la sección Minores macromorfológicamente similares a A. subrufescens [e.g. A. diminutivus Peck, A. porphyrizon P.D. Orton, A. purpurellus (F.H. Møller) F.H. Møller], se distinguen por tener un píleo menor [generalmente menor a 50 (60) mm] y basidiosporas más cortas [x m < 5 (5.5) µm de longitud] (Cappelli, 1984; Parra, 2013); ninguna de ellas se conoce de México.
Los análisis de distancia genética de la región ITS para esta especie aquí realizados indican que la secuencia mexicana presenta dos polimorfismos exclusivos que no están representados en las secuencias de los ejemplares provenientes del resto del mundo, lo que la separa del núcleo genético más común; esto es especialmente visible en el análisis de red de haplotipos (Fig. 2); es importante destacar esto, ya que el hecho de presentar polimorfismos exclusivos, sugiere que el espécimen encontrado en México representa una población nativa, es decir, que dichos polimorfismos son geográficamente locales. Kerrigan (2005) señaló que la mayoría de las cepas de cultivo comerciales provienen de Brasil y mostró que sus respectivas secuencias de ITS son altamente similares o idénticas entre sí; en relación con dichas cepas, el espécimen mexicano tampoco tiene ninguna correspondencia genética, por lo que se puede afirmar que no proviene de alguna cepa cultivada. Kerrigan (op. cit.) también afirmó que entre las secuencias de A. subrufescens (y todos sus sinónimos) hay poca variación en la región del ITS. La similitud genética menor y la tasa de sustitución mayor aquí calculadas, indican que la diferenciación genética para el ITS de esta especie es relativamente baja, lo cual coincide con el resultado de Kerrigan (op. cit.), aun incluyendo a la cepa mexicana; por esta razón, la conectividad de la red de haplotipos debería considerarse como una sola especie. Kerrigan (op. cit.) encontró que las secuencias hawaianas eran las más diferenciadas entre las secuencias de A. subrufescens y sus sinónimos; tal disyunción genética (apoyada por los análisis de distancia genética aquí realizados) explica que el clado que agrupa a las secuencias de China, Hawái y Tailandia sea constantemente soportado por los análisis filogenéticos, aunque esto no implica su separación como especie dado que, al menos en el caso de las cepas tailandesas, su interfertilidad con cepas de Brasil y Francia ha sido positiva (Thongklang et al., 2014).
Freeman (1979a,b) indicó que la reacción de Schäffer es negativa para A. subrufescens; sin embargo, Kerrigan (1986), Arrillaga y Parra (2006), Wisitrassameewong et al. (2012a) y Parra (2013) señalaron que esta especie presenta una reacción de Schäffer positiva, lo cual es confirmado con las observaciones de este estudio, siendo la reacción de color anaranjado-amarillo a anaranjado-rojo. Es posible que Freeman (op. cit.) haya realizado esta reacción sobre especímenes herborizados. Tal reacción también es positiva en la mayoría de las especies de la sección Arvenses, a la cual pertenece A. subrufescens.
A. subrufescens tiene una distribución geográfica muy amplia, que abarca regiones templadas y tropicales. Para América del Norte ha sido reportada de varias partes de Estados Unidos (Peck, 1894; Kauffman, 1918; Murrill, 1922; Zeller, 1922; Freeman, 1979b; Kerrigan, 1986, 2016), por lo que con este trabajo se amplía su distribución conocida al bosque de niebla de la vertiente del Golfo de México. En América del Sur se distribuye en Brasil (Wasser et al., 2002); en Europa de Alemania (Ludwig, 2007), España (Arrillaga y Parra, 2006), Francia (Thongklang et al., 2014) y Reino Unido (Nauta, 1999, 2001); en Asia de China (Gui et al., 2015) y Tailandia (Wisitrassameewong et al., 2012a); en África de Congo y Rep. Dem. del Congo (Thongklang et al., 2016); y en Islas del Pacífico de Hawái (Peterson et al., 2000). Kerrigan (2005) mostró que las cepas japonesas y estadounidenses son interfértiles; en tanto, Thongklang et al. (2014) mostraron que las cepas brasileñas, francesas y tailandesas son interfértiles.
Conclusiones
Hasta ahora, la presencia de A. subrufescens en México está circunscrita al bosque mesófilo de montaña, que es un ecosistema caracterizado por tener vegetación con elementos de origen Neártico y Neotropical con un clima húmedo con alta cantidad de lluvia y subtropical. Los registros de esta especie en China (Gui et al., 2015) y Tailandia (Wisitrassameewong et al., 2012a) también incluyen localidades subtropicales. No obstante, si se consideran los registros de esta especie en el estado mexicano de Puebla (Martínez-Carrera et al., 2001), en América del Norte (Kerrigan, 2016) y en Europa (Parra, 2013), es factible su presencia en las zonas templadas de México; asimismo, si se consideran los registros en zonas tropicales de Brasil, Congo, Hawái y Rep. Dem. del Congo (Peterson et al., 2000; Wasser et al., 2002; Thongklang et al., 2016) también lo es en las zonas tropicales de México. La presencia de A. subrufescens en México (particularmente en Veracruz) y el aislamiento de una cepa logrado en este trabajo, representan la posibilidad de realizar estudios sobre su cultivo y desarrollar el potencial aprovechamiento de un recurso forestal no maderable presente en el bosque mesófilo de montaña, el cual podría ser cultivado en el país, pues la población mexicana de esta especie muy probablemente es nativa.











 nueva página del texto (beta)
nueva página del texto (beta)



