Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista Chapingo. Serie horticultura
versión On-line ISSN 2007-4034versión impresa ISSN 1027-152X
Rev. Chapingo Ser.Hortic vol.29 no.3 Chapingo sep./dic. 2023 Epub 26-Ene-2024
https://doi.org/10.5154/r.rchsh.2023.04.002
Artículo científico
Identificación automática de enfermedades en frutos de aguacate con base en máquinas de aprendizaje y descriptores cromáticos
1Colegio de Postgraduados, Campus Montecillo. Carretera México-Texcoco km. 36.5, Montecillo, Texcoco, Estado de México, C. P. 56264, MÉXICO.
La identificación oportuna de problemas fitosanitarios en cultivos agrícolas es esencial para reducir pérdidas de producción. Los algoritmos de inteligencia artificial facilitan su identificación rápida y confiable. En esta investigación, se evaluaron tres clasificadores de aprendizaje: bosque aleatorio (RF), máquina de soporte vectorial (SVM) y perceptrón multicapa (MLP), para identificar tres clases objetivo (frutos sanos, antracnosis [Colletotrichum spp.] y roña [Sphaceloma perseae]) a partir de imágenes digitales de frutos. Se compararon dos técnicas de extracción de descriptores de color (selección por región y submuestreo de imágenes) con el clasificador RF, y se obtuvo una precisión global de clasificación (ACC) de 98±0.03 % con selección por región, y de 84±0.08 % con submuestreo. Posteriormente, los clasificadores se evaluaron con descriptores de color extraídos con selección por región. RF y MLP fueron superiores a SVM, con una ACC de 98±0.03 %. La roña y la antracnosis se identificaron con un puntaje F1 de 98 %. El alto desempeño de los clasificadores muestra el potencial de aplicación de los paradigmas de inteligencia artificial para identificar problemas fitosanitarios en cultivos agrícolas.
Palabras clave: Persea americana; Sphaceloma perseae; Colletotrichum spp.; aprendizaje automático; inteligencia artificial.
Timely identification of phytosanitary problems in agricultural crops is essential to reduce production losses. Artificial intelligence algorithms facilitate their rapid and reliable identification. In this research, three learning classifiers, namely random forest (RF), support vector machine (SVM) and multilayer perceptron (MLP), were evaluated to identify three target classes (healthy fruit, anthracnose [Colletotrichum spp.] and scab [Sphaceloma perseae]) from digital fruit images. Two color descriptor extraction techniques (region selection and image subsampling) were compared with the RF classifier, and an overall classification accuracy (ACC) of 98±0.03 % with region selection and 84±0.08 % with subsampling was obtained. Subsequently, the classifiers were evaluated with color descriptors extracted with region selection. RF and MLP were superior to SVM, with an ACC of 98±0.03 %. Scab and anthracnose were identified with an F1 score of 98 %. The high performance of the classifiers shows the potential for applying artificial intelligence paradigms to identify phytosanitary problems in agricultural crops.
Keywords: Persea americana; Sphaceloma perseae; Colletotrichum spp.; machine learning; artificial intelligence.
Ideas sobresalientes:
Three machine learning classifiers are evaluated to identify scab and anthracnose diseases in avocado fruits.
The extraction technique of chromatic descriptors, selection by region was more appropriate than the extraction with image subsampling.
The high performance of machine learning classifiers to identify anctranosis and scab in avocado fruits is confirmed.
The classifiers achieved higher performance than a deep learning network applied in a previous study.
Introducción
México es el principal productor y exportador de aguacate del mundo, con una producción superior a dos millones de toneladas en 2021 (Servicio de Información Agroalimentaria y Pesquera [SIAP], 2022); sin embargo, la pérdida en poscosecha es un factor que afecta la comercialización y la seguridad alimentaria. En los frutos de aguacate se reporta la presencia de dos enfermedades fungosas de interés comercial: la roña (Sphaceloma perseae) y la antracnosis (Colletotrichum spp.). El manejo inadecuado de los huertos de aguacate puede ocasionar pérdidas superiores a 70 % antes de la cosecha, y pérdidas totales en poscosecha si existen condiciones ambientales y biológicas para el desarrollo de estas enfermedades (Téliz & Mora, 2019). La detección temprana de problemas fitosanitarios es una etapa esencial para garantizar la seguridad alimentaria. En la literatura, se han propuesto sistemas automáticos con capacidad para detectar enfermedades en especies vegetales con base en imágenes digitales (Saleem, Potgieter, & Arif, 2019).
El paradigma de aprendizaje automático (ML, machine learning) es ampliamente utilizado para detectar enfermedades. Una tarea básica en ML es la clasificación o predicción supervisada de una variable respuesta discreta. Los datos de entrada se asocian con cada clase objetivo para formar un conjunto de entrenamiento, y el modelo aprende a predecir las clases objetivo sobre un conjunto de datos no vistos (Ketkar & Moolayil, 2021).
En la agricultura, se han reportado diferentes trabajos que utilizan algoritmos de ML para la detección automática de enfermedades en plantas. Entre los paradigmas aplicados se tienen los algoritmos máquina de soporte vectorial (SVM, support vector machine) (Sandhya, Balasundaram, & Arunkumar, 2022), bosque aleatorio (RF, random forest) (Srinivasa, Venkata, Anusha, Sai, & Bhanu, 2022) y perceptrón multicapa (MLP, multilayer perceptron) (Chen, Dewi, Huang, & Caraka, 2020). Estos algoritmos se han aplicado a diferentes órganos de la planta, como hojas, raíces o frutos (Doh et al., 2019).
Un clasificador de aprendizaje profundo (deep learning) permite la extracción automática de características a partir de un conjunto de datos de entrada, y reduce el error del usuario para la selección de éstas. En general, con este enfoque se alcanzan altos niveles de precisión global de clasificación cuando se dispone de grandes conjuntos de datos. En la agricultura, esto se ha utilizado en la identificación de enfermedades, clasificación de cultivos y predicción de rendimientos (Alzubaidi et al., 2021).
El objetivo de esta investigación fue identificar frutos infectados con antracnosis, con roña y frutos sanos (tres clases objetivo) por medio de tres modelos de aprendizaje automático (RF, SVM y MLP) y descriptores cromáticos extraídos (selección por región [BD1] y submuestreo de imágenes [BD2]) de imágenes digitales de frutos de aguacate.
Materiales y métodos
Base de datos
El conjunto de imágenes de frutos de aguacate que se utilizaron en este estudio (30 por clase objetivo) se seleccionaron con diferentes niveles de las enfermedades de una base de datos con 569 imágenes digitales de tamaño 256 x 256 píxeles, pertenecientes a frutos de aguacate variedad Fuerte reportadas en un estudio previo (Campos-Ferreira & González-Camacho, 2021) (https://www.kaggle.com/datasets/camposfe1/clasifiacin-de-enfermedades-del-aguacatero).
Las imágenes de los frutos se capturaron en laboratorio y bajo condiciones homogéneas de iluminación. Las clases objetivo fueron: frutos sanos (S), roña (R, Sphaceloma perseae) y antracnosis (A, Colletotrichum spp.). Los frutos se obtuvieron en los municipios de Ocuituco (18° 51’ 47.6” LN y 98° 46’ 49.1” LO) y Tetela del Volcán (18° 53’ 36.3” LN y 98° 43’ 47.7” LO) en el estado de Morelos. Los frutos de la variedad Fuerte, en su periodo de madurez, son de color verde, lo cual permite contrastar las lesiones provocadas por las dos enfermedades mencionadas (Figura 1).
Software y hardware
El lenguaje de programación Python ver. 3.9 se utilizó como plataforma de programación, y la librería scikit-learn ver. 1.0.2 se utilizó para implementar los clasificadores de aprendizaje automático. El entrenamiento y validación de los clasificadores se realizaron en el ambiente de desarrollo Spyder ver. 4.2.1 y el sistema operativo MacOs Monterey (12.6) en una computadora Mac mini (Apple), la cual utiliza un chip ARM (M1) con velocidad de procesamiento de 3.2 GHz y 8 GB de RAM.
Procesamiento de imágenes
El conjunto de imágenes representadas con el modelo de color RGB (red, blue y green) se transformaron al modelo de color HSV (hue, saturation y value). Lo anterior debido a que la representación HSV es más apropiada que RGB para extraer características de color, y permite mejorar el desempeño de los clasificadores de aprendizaje (Abdel-Hamid, 2019).
Extracción de características de color
La extracción de características (descriptores) de color se realizó por medio de dos técnicas: BD1 y BD2. Esto se realizó con la ayuda del programa IDENTO (Ambrosio-Ambrosio, González-Camacho, Rojano-Aguilar, & del Valle-Paniagua, 2023) para crear los conjuntos de datos de entrada a partir de las imágenes por cada clase objetivo. El conjunto de datos BD1 se creó con la técnica de extracción por región. Esta consistió en seleccionar, en cada imagen, una región de interés, y a partir de un píxel semilla, o de inicio, el algoritmo extrae un conjunto de píxeles similares a la semilla, donde cada píxel se representa por los canales de color H, S y V. IDENTO permitió generar, para cada clase objetivo, un conjunto de 45,536 tripletas de valores (H, S y V) (Cuadro 1).
Cuadro 1 Descripción de los conjuntos de datos de entrada (pixeles o descriptores de color H, S y V) obtenidos con las técnicas de extracción por región (BD1) y submuestreo de imágenes (BD2).
| Clase | BD1 | BD2 |
|---|---|---|
| S | 15,213 | 17,746 |
| R | 14,836 | 13,416 |
| A | 15,487 | 8,513 |
| Total | 45,536 | 39,675 |
S = frutos sanos; R = frutos con roña; A = frutos con antracnosis.
El conjunto de datos BD2 se creó con la técnica de extracción por submuestreo de un recuadro de la imagen con el área de interés. Para ello, con la ayuda del programa IDENTO, se seleccionaron tres áreas rectangulares (30
En ambos conjuntos de datos, se eliminaron las tripletas repetidas dentro de clases y entre clases objetivo. Los conjuntos de tripletas de valores (H, S y V), y su clase objetivo asociada, se exportaron y guardaron en un archivo con formato .csv para su uso en el entrenamiento y prueba con el clasificador RF.
Bosque aleatorio (RF)
El clasificador RF es un modelo de aprendizaje supervisado de ensamble. En el caso de un problema de clasificación, este algoritmo utiliza árboles de decisión a partir de una condición, y cada árbol obtendrá un valor para clasificar los datos. Cada árbol emite un voto unitario, y la elección del mejor árbol de decisión se hace de acuerdo con el que tenga el mayor número de votos de todo el bosque (Parmar, Katariya, & Patel, 2019).
En RF, cada árbol se construye a partir de una muestra extraída de manera aleatoria, con reemplazo, del conjunto de datos de entrenamiento (bootstrap); posteriormente, se producen múltiples conjuntos de entrenamiento con valores distintos al conjunto inicial. A partir de cada muestra, se construye un modelo (bagging) y se introducen los datos de la muestra original, se determina su clase predicha y se analiza la diferencia con el valor real, con lo cual se obtiene el error de clasificación. Los modelos de ensamble permiten reducir la varianza del estimador de RF y evitar el sobre-ajuste del modelo (Knauer et al., 2019).
En un modelo RF se busca maximizar la ganancia de información, la cual está definida por:
donde f es la condición que divide el nodo padre, D p y D j pertenecen a los datos del nodo padre y del j-ésimo hijo, I es la métrica de impureza, N p es el número de muestras en el nodo padre y N j es el número de muestras del j-ésimo hijo. La ganancia de información es la diferencia entre la impureza del nodo padre y la suma de las impurezas de los nodos hijo, mientras más bajas sean las impurezas de los nodos hijo, más grande es la ganancia de información (Raschka, Liu, & Mirjalili, 2022).
Una función objetivo que se utiliza en RF es el criterio de Gini (I G ) y se calcula como:
donde K corresponde al número de clases objetivo y
En este trabajo, se utilizó la función randomforestclassifier, que toma como argumentos principales el número de árboles (n_estimators, NE), el criterio (criterion, Cr), la profundidad del árbol (max_depth, MD) y las características máximas (max_features, MF) (Raschka et al., 2022). Estos hiperparámetros se utilizaron para optimizar el modelo RF con base en los siguientes intervalos y valores (seleccionados por prueba y error): NE = 100, 200, 300 y 1000; Cr = ‘gini’ y ‘entropy’; MD = 4, 6 y 8; MF = ‘sqrt’ y ‘auto’.
Máquina de soporte vectorial (SVM)
SVM es un modelo que asocia los datos del conjunto original, a partir de un espacio de entrada con un espacio de características de alta dimensión, volviéndolo más simple en el espacio de características. Esto se realiza para separar, de manera óptima, las clases en un hiperplano y minimizar el error de generalización, además de maximizar el margen.
En caso de que las clases sean separables entre sí a partir de un clasificador lineal, el modelo SVM determina el hiperplano que minimiza el error de generalización (mediante un conjunto de datos de prueba). En caso contrario, cuando al menos una clase no es separable de las otras, SVM intenta buscar el hiperplano que maximice el margen y, al mismo tiempo, minimice a una cantidad proporcional al número de clasificaciones incorrectas. Con base en el hiperplano seleccionado, se tendrá el máximo margen entre clases; es decir, la suma de las distancias entre el hiperplano de separación y los puntos más cercanos para cada clase (Das, Singh, Mohanty, & Chakravarty, 2020).
La ecuación de un hiperplano se define como w × x + b = 0, donde w es un vector normal al hiperplano y b es un sesgo. En el caso de una clasificación multiclase, se utiliza el siguiente problema de optimización:
donde ξ i (i = 1, …, n) son variables de holgura, las cuales pueden permitir algunos datos para eliminar las limitaciones que definen el margen mínimo requerido para los datos de entrenamiento para un caso separable. C es un parámetro de penalización definido por el usuario para controlar el margen de error del conjunto de entrenamiento; mientras más grande sea el valor de C, más grande es la penalización. Por otra parte, el j-ésimo SVM es entrenado con los datos en la j-ésima clase con etiquetas pertenecientes a la clase, y las demás clases con etiquetas diferentes a la clase (Pisner & Schnyer, 2020).
Existe una función kernel, la cual es utilizada para transformar el conjunto de datos de entrenamiento, para que una superficie de decisión no lineal se transforme en una ecuación lineal en una mayor cantidad de espacios dimensionales. De manera general, esta función devuelve el producto interno entre dos puntos en una dimensión de características. Los kernel más utilizados son el lineal:
Y el kernel Radial basis function (RBF), también conocido como kernel Gaussiano:
donde σ > 0 es el parámetro que controla el ancho del kernel. Esta expresión se puede simplificar de la siguiente forma:
donde
En la biblioteca de programas scikit-learn, dentro del módulo svm, se incluyen los métodos para clasificación. En el presente trabajo, se utilizó la función support vector classification (SVC), la cual toma como principales hiperparámetros la penalización o el valor C (C), el valor gamma (γ) y el tamaño de la máscara kernel (K). Mediante prueba y error se definieron los siguientes intervalos y valores: C = 0.01, 0.1, 1, 10 y 100; γ = 0.001, 0.1, 1, 10 y 100; kernels = ‘rbf ’ y ‘linear’ (Raschka et al., 2022).
Perceptrón multicapa (MLP)
En MLP, al elemento básico se le conoce como neurona artificial. Esta neurona artificial, de tipo hacia adelante (feed forward), consta de elementos de entrada y salida que se procesan en la unidad central. Las capas de entrada dependen de los datos utilizados para el entrenamiento, mientras que las capas ocultas y las capas de salida pertenecen al número de clases de interés.
La arquitectura MLP se compone de una capa de entrada, una o más capas ocultas y una capa de salida. La capa de entrada depende del número de datos de entrada, las capas ocultas representan el nivel de complejidad que existe entre la capa de entrada y de salida, y la capa de salida representa el número de clases objetivo y da la clase predicha (Edmond & Girsang, 2020).
La función que transforma los datos de entrada se conoce como función de activación. La más común es la función ReLU (Rectified Linear Unit), la cual se expresa como:
donde la respuesta es z si la entrada es positiva y 0 si es negativa. ReLU se utiliza para filtrar los datos en las capas intermedias. En la capa de salida se utiliza la función de activación softmax, que se expresa como:
donde p(z) es la probabilidad de pertenencia de una entrada z a la clase i-ésima (Chollet, 2018).
Para el entrenamiento de MLP, se utilizó el optimizador adam (Adaptative moment estimation), que es una variante del método gradiente descendente. Este método utiliza el momentum y la varianza del gradiente de la función de pérdida para actualizar los pesos, lo cual permite suavizar la curva de aprendizaje y mejorar el aprendizaje del clasificador (Géron, 2022).
El modulo neural_network de scikit-learn contiene la función MLPClassifier, y toma como argumentos los siguientes hiperparámetros: tamaño de la capa oculta (CO), número de iteraciones (It), función de activación (FA), optimizador (Op), rango de aprendizaje (RA) y tamaño del lote de muestras que entran al modelo en cada paso de iteración (TL) (Raschka et al., 2022). Los intervalos y valores de búsqueda de los hiperparámetros se definieron por prueba y error, y se consideraron los siguientes: CO = 50, 100 y 500; It = 50, 100 y 500; FA = ‘softmax’ y ’ReLU’; Op = aproximación del algoritmo Broyden-Fletcher-Goldfarb-Shanno (lbfgs) y adam; RA = ‘constant’ y ‘adaptative’; TL = 8, 16 y 32.
Métricas de desempeño
Las métricas para determinar el desempeño global y para cada clase objetivo de los clasificadores se obtienen a partir de la matriz de confusión, con base en los datos de prueba. En el caso de una clasificación binaria, esta matriz tiene cuatro posibles resultados: verdadero positivo (VP), verdadero negativo (VN), falso positivo (FP) y falso negativo (FN) (Kulkarni, Chong, & Batarseh, 2020). Con base en estos valores se definen las siguientes métricas:
La precisión (P), la cual mide que tan acertado es el modelo para predecir los valores positivos.
La exhaustividad o sensibilidad (E), que mide la fuerza para predecir muestras positivas.
El puntaje F1, que es la media armónica entre P y E.
Estas tres métricas se utilizan para evaluar el desempeño que tiene el clasificador al predecir cada clase. La evaluación del desempeño global de los clasificadores se realizó con base en la precisión global de clasificación correcta (ACC) y el área bajo la curva (AUC) ROC (curva característica operativa del receptor).
La ACC es la proporción de clasificaciones correctas con respecto al total de muestras, y se expresa como:
La ROC es una gráfica que se construye con valores para diferentes umbrales de probabilidad de la tasa de verdaderos positivos (TVP) versus la tasa de falsos positivos (TFP) (Jiang, Li, & Safara, 2021). AUC varía entre 0 y 1, y mide el desempeño del modelo para cada clase objetivo. TVP se obtiene a partir de la siguiente ecuación:
y TFP se calcula como:
Selección de modelos de aprendizaje automático
En la literatura se reporta que los clasificadores RF, SVM y MLP, en general, alcanzan buena precisión global de clasificación (Yuvali, Yaman, & Tosun, 2022). En pruebas preliminares, estos clasificadores obtuvieron buen desempeño para clasificar imágenes de frutos de aguacate.
Entrenamiento y prueba de los clasificadores
El entrenamiento y prueba de los clasificadores constó de dos etapas. En la primera se entrenó el clasificador RF con base en los conjuntos de datos BD1 y BD2 para comparar las técnicas de extracción de descriptores cromáticos y por submuestreo de imágenes, y para seleccionar la que genere el mejor desempeño de RF en términos de ACC. En la segunda etapa, los tres clasificadores (RF, SVM y MLP) se entrenaron con el conjunto de datos más apropiado de la etapa 1. Los hiperparámetros óptimos de cada clasificador se obtuvieron por medio de una búsqueda por retícula y validación cruzada (Raschka et al., 2022).
Selección de hiperparámetros óptimos
Para optimizar cada clasificador, primero se realizó una partición aleatoria estratificada por cada clase objetivo del conjunto de datos en proporción 80:20 (80 % para entrenamiento y 20 % para la prueba en predicción). Esta proporción establece un balance entre los datos de entrenamiento y de prueba para medir el desempeño de los clasificadores. Los datos de entrenamiento se estandarizaron para homogeneizar los descriptores de entrada mediante la siguiente expresión:
donde x corresponde a cada descriptor del conjunto de entrada, µ x es la media del conjunto de valores de x y σ x es la varianza muestral del conjunto de valores de x.
La selección óptima de hiperparámetros se realizó por medio de una búsqueda por retícula y validación cruzada con k = 10 grupos disjuntos. Para cada clasificador, se definieron intervalos de valores de cada hiperparámetro para seleccionar la combinación de valores que maximicen la ACC promedio del clasificador (Liashchynskyi & Liashchynskyi, 2019).
La validación cruzada consiste en dividir el conjunto de entrenamiento de forma aleatoria en k grupos disjuntos, donde k-1 grupos se utilizan como conjuntos de entrenamiento, y el grupo restante como conjunto de validación. Este proceso se repite k veces para cada combinación de valores de los hiperparámetros, y se obtiene una ACC promedio de k corridas del modelo (Raschka et al., 2022). La combinación de valores óptimos de los hiperparámetros es la que genera el máximo valor de ACC promedio.
Prueba en predicción de los clasificadores
Con el conjunto de hiperparámetros óptimos obtenidos en la etapa de entrenamiento, se realizó un procedimiento de validación cruzada con k = 10 grupos con el conjunto total de datos de entrada (100 %) para recalcular los pesos o parámetros de cada clasificador. En cada corrida k, se determinó el desempeño de predicción ACC para cada conjunto de prueba k-ésimo. Después de k corridas, se obtuvo el desempeño ACC promedio de cada clasificador.
Los códigos implementados para el presente trabajo se encuentran en el siguiente enlace: https://github.com/Camposfe1/Avocado-disease-classification.git.
Resultados y discusión
Selección de hiperparámetros óptimos
Los valores óptimos de los hiperparámetros de cada clasificador fueron NE = 10, MF = ‘auto’, MD = 8 y Cr = ‘entropy’ para RF, C = 10, γ = 10 y K = ‘rbf’ para SVM, y TL = 8, CO = 150, FA = ‘ReLU’, It = 100 y Op = ‘adam’ para MLP.
Comparación de técnicas de extracción de descriptores
La comparación de las técnicas de extracción de características o descriptores de color, por región y submuestreo de imágenes, así como su efecto en el desempeño del clasificador RF, mostró que la selección por región (BD1) permite generar conjuntos de píxeles de color con más información para diferenciar las tres clases objetivo (S, R y A). RF obtuvo mayor precisión para clasificar las tres clases objetivo. Los valores de FP y FN, para cada clase, fueron menores que los correspondientes a BD2. Asimismo, la extracción por región generó tamaños de clase mejor balanceados que por submuestreo (Figura 2).

Figura 2 Matrices de confusión del clasificador bosque aleatorio (RF) con base en los conjuntos de datos generados con extracción por región (BD1) y submuestreo de imágenes (BD2). Clases objetivo: S = frutos sanos; R = frutos con roña; A = frutos con antracnosis. Píxeles de color predichos versus reales por clase objetivo.
De manera similar, el desempeño de RF, a nivel global y a nivel de clase, fue superior con BD1 que con BD2. Con BD1, RF alcanzó una ACC promedio de 98 %, mientras que con BD2 ésta fue de 84 %. A nivel de clase, los puntajes F1 fueron superiores a 97 % con BD1 y superiores a 76 % con BD2 (Cuadro 2).
Cuadro 2 Métricas de desempeño en predicción del clasificador bosque aleatorio (RF) con base en los métodos de extracción de descriptores de color por región (BD1) y submuestreo de imágenes (BD2).
| Métrica | BD1 | BD2 | ||||||
|---|---|---|---|---|---|---|---|---|
| S | R | A | S | R | A | |||
| P | 0.94 | 1.00 | 1.00 | 0.77 | 0.97 | 0.76 | ||
| E | 1.00 | 0.97 | 0.96 | 0.98 | 0.75 | 0.84 | ||
| F1 | 0.97 | 0.98 | 0.98 | 0.86 | 0.84 | 0.76 | ||
| AUC | 1.00 | 1.00 | 1.00 | 0.97 | 0.94 | 0.95 | ||
| ACC | 0.98 ± 0.03 | 0.84 ± 0.08 | ||||||
S = frutos sanos; R = frutos con roña; A = frutos con antracnosis; P = precisión; E = exhaustividad; F1 = puntaje F1; ACC = precisión global de clasificación; AUC = área bajo la curva ROC.
Evaluación de la predicción de los clasificadores RF, SVM y MLP
El desempeño, en cuanto a la predicción de los tres clasificadores, fue alto. En las matrices de confusión de cada clasificador se observa que RF y MLP alcanzaron la mayor ACC para las tres clases. Los tres clasificadores tienen el mayor valor de FP para predecir la clase S; es decir, los clasificadores predicen muestras de las clases R o A como S. Para este análisis, hay muestras de píxeles con roña que se predicen como sanas, y muestras sanas que se predicen con antracnosis (Figura 3).
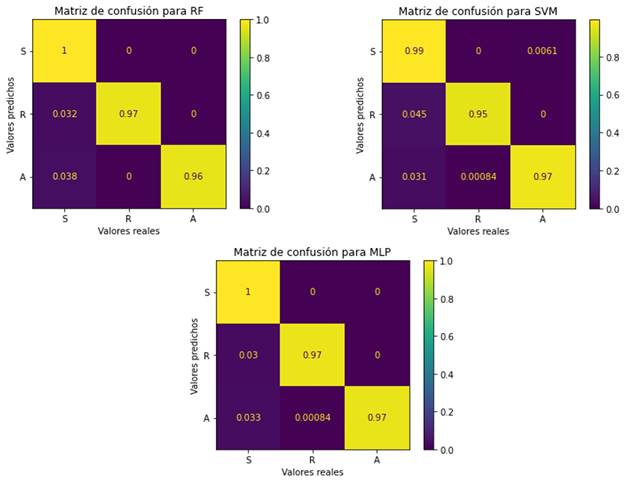
Figura 3 Matrices de confusión de los clasificadores bosque aleatorio (RF), máquina de soporte vectorial (SVM) y perceptrón multicapa (MLP) con base en la extracción de descriptores por región (BD1). Clases objetivo: S = frutos sanos; R = frutos con roña; A = frutos con antracnosis.
En términos del desempeño global de los clasificadores, RF y MLP fueron superiores a SVM con una ACC de 98 %. Asimismo, ambos clasificadores obtuvieron un puntaje F1 superior a 97 % para cada clase objetivo (Cuadro 3).
Cuadro 3 Métricas de desempeño en la predicción de los clasificadores de aprendizaje automático bosque aleatorio (RF), máquina de soporte vectorial (SVM) y perceptrón multicapa (MLP) con base en la extracción de descriptores por región (BD1).
| Métrica | RF | SVM | MLP | ||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|
| S | R | A | S | R | A | S | R | A | |||
| P | 0.94 | 1.00 | 1.00 | 0.94 | 1.00 | 0.99 | 0.95 | 1.00 | 1.00 | ||
| E | 1.00 | 0.97 | 0.96 | 0.99 | 0.95 | 0.97 | 1.00 | 0.97 | 0.97 | ||
| F1 | 0.97 | 0.98 | 0.98 | 0.96 | 0.98 | 0.98 | 1.00 | 0.98 | 0.98 | ||
| AUC | 1.00 | 1.00 | 1.00 | 0.99 | 0.99 | 0.99 | 0.99 | 0.99 | 1.00 | ||
| ACC | 0.98 ± 0.03 | 0.97 ± 0.06 | 0.98 ± 0.03 | ||||||||
S = frutos sanos; R = frutos con roña; A = frutos con antracnosis; P = precisión; E = exhaustividad; F1 = valor F1; AUC = área bajo la curva ROC; ACC = precisión global de clasificación.
El método de extracción de características o descriptores de color BD1 tuvo un efecto importante en el desempeño de los clasificadores. Este método permitió obtener píxeles o muestras de color representativas de cada clase objetivo y tamaños de clase balanceados, lo cual condujo a un mejor desempeño de los clasificadores. El método de extracción de características BD2 generó un conjunto de datos con clases desbalanceadas y se privilegió a la clase S de frutos sanos. El desbalance entre tamaños de clase en BD2 tuvo un efecto negativo en el desempeño del clasificador RF, el cual alcanzó una ACC de 84 %. En algunas publicaciones se menciona que no hay diferencias entre los métodos de extracción de características (Jones, Faiz, Qiu, & Zheng, 2022; Suresh & Mohan, 2020); sin embargo, en este estudio la técnica de selección BD1 permitió obtener una mejor ACC del modelo RF que BD2, el cual depende de la habilidad para seleccionar las áreas de interés (Chen et al., 2020). En el caso de clases desbalanceadas, la métrica F1 (media armónica de P y E) es una medida más adecuada para evaluar el desempeño del clasificador (Fourure, Javaid, Posocco, & Tihon, 2021).
Cuando se tienen clases desbalanceadas, los clasificadores sesgan la predicción a la clase mayoritaria; por lo cual, hay una mala clasificación para la clase minoritaria (Kaur, Singh, & Kaur, 2019). Lo anterior se debe a que es más fácil conseguir frutos sanos que frutos con los síntomas de las enfermedades, ya que se necesitan ciertas condiciones para que se puedan expresar los síntomas en la fruta (Wardhani, Rochayani, Iriany, Sulistyono, & Lestantyo, 2019).
Los tres clasificadores RF, SVM y MLP catalogaron a los frutos con roña y anctracnosis con puntaje F1 de 98 %, y fueron las clases que se clasificaron mejor. Los frutos con roña se diferencian visualmente de los frutos sanos o con antracnosis; por ello, los valores de los píxeles entre clases son más contrastantes.
Los clasificadores de aprendizaje automático utilizados en este estudio muestran que es posible alcanzar altos niveles de desempeño con conjuntos de datos pequeños y tiempos de cómputo menores, en comparación con los clasificadores de aprendizaje profundo; en particular, con las redes neuronales convolucionales, las cuales requieren mayores tiempos de cómputo. No obstante, en problemas de identificación más complejos, con un gran número de clases (mayor de 10) y grandes conjuntos de datos (miles de imágenes), los algoritmos de aprendizaje profundo resultan más apropiados (Alzubaidi et al., 2021).
Conclusiones
La extracción de descriptores de color con el método de selección por región indujo una mejor precisión global de clasificación del clasificador bosque aleatorio (ACC = 98 %), en contraste con el método de extracción con submuestreo de imágenes (ACC = 84 %). Los tres clasificadores (bosque aleatorio, máquina de soprte vectorial y perceptrón multicapa) obtuvieron ACC mayores a 97 % para clasificar la superficie de frutos de aguacate sano, con roya o antracnosis. Asimismo, las clases superficie del fruto con roña y con antracnosis obtuvieron un puntaje F1 de 98 % con los tres clasificadores.
Agradecimientos
Al Consejo Nacional de Ciencia y Tecnología (CONACyT) por la asignación de la beca al primer autor para los estudios de posgrado.
REFERENCIAS
Abdel-Hamid, L. (2019). Glaucoma detection using statistical features: comparative study in RGB, HSV and CIEL*a*b* color models. Tenth International Conference on Graphics and Image Processing , 11069. doi: 10.1117/12.2524215 [ Links ]
Alzubaidi, L., Zhang, J., Humaidi, A. J., Al-Dujaili, A., Duan, Y., Al-Shamma, O., Santamarìa, J., Fadhel, M. A., Al-Amidie, M., & Farhan, L. (2021). Review of deep learning: concepts, CNN architectures, challenges, applications, future directions. Journal of Big Data, 8(53), 1-74. doi: 10-1186/s40537-021-00444-8 [ Links ]
Ambrosio-Ambrosio, J. P., González-Camacho, J. M., Rojano-Aguilar, A., & del Valle-Paniagua, D. (2023). Identification of disease in tomato leaves using machine learning classifiers and digital images. Agrociencia, 57(3), 476-507. doi: 10.47163/agrociencia.v57i3.2462 [ Links ]
Campos-Ferreira, U. E., & González-Camacho, J. M. (2021). Clasificador de red neuronal convolucional para identificar enfermedades del fruto de aguacate (Persea americana Mill.) a partir de imágenes digitales. Agrociencia, 5(8), 695-709. doi: 10.47163/agrociencia.v55i8.2662 [ Links ]
Chen, R. C., Dewi, C., Huang, S. W., & Caraka, R. E. (2020). Selectig critical features for data classification based on machine learning methods. Journal of Big Data, 7(52), 1-26. doi: 10.1186/s40537-020-00327-4 [ Links ]
Chollet, F. (2018). Getting started with neural networks. In: Chollet, F. (Ed.), Deep Learning with Python (pp. 56-92). New York: Manning Publications Co. [ Links ]
Das, D., Singh, M., Mohanty, S. S., & Chakravarty, S. (2020). Leaf disease detection using support vector machine. International Conference on Communication and Signal Processing, 2020. 1036-1040. doi: 10.1109/ICCSP48568.2020.9182128 [ Links ]
Doh, B., Zhang, D., Shen, Y., Hussain, F., Doh, R. F., & Ayepah, K. (2019). Automatic citrus fruit disease detection by phenotyping using machine learning. 25th International Conference on Automatic and Computing, 2019, 1-5. doi: 10.23919/IConAC.2019.8895102 [ Links ]
Edmond, C., & Girsang, A. S. (2020). Classification performance for credit scoring using neural network. International Journal of Emerging Trends in Engineering Research, 8(5), 1592-1599. doi: 10.30534/ijeter/2020/19852020 [ Links ]
Fourure, D., Javaid, M. U., Posocco, N., & Tihon, S. (2021). Anomaly detection: how to artificially increase your f1-score with a biased evaluation protocol. In: Dong, Y., Kourtellis, N., Hammer, B., & Lozano, J. A. (Eds.), Machine learning and knowledge discovery in databases. Applied data science track (pp. 3-18). New York: Springer. doi: 10.48550/arXiv.2106.16020 [ Links ]
Géron, A. (2022). Ensemble learning and random forests. In: Géron, A. (Ed.), Hands-on machine learning with scikit-learn, keras & tensorflow. concepts, tools, and techniques to build intelligent systems (pp. 337-373). Sebastopol: O'Reilly Media. [ Links ]
Jiang, H., Li, X., & Safara, F. (2021). Iot-based agriculture: deep learning in detecting apple fruit diseases. Microprocessors and Microsystems, 14, 1-23. doi: 10.1016/j.micpro.2021.104321 [ Links ]
Jones, M. A., Faiz, R., Qiu, Y., & Zheng, B. (2022). Improving mammography lesion classification by optimal fusion of handcrafted and deep transfer learning features. Physics in Medicine & Biology, 67(5). doi: 10.1088/1361-6560/ac5297 [ Links ]
Kaur, H., Singh, P. H., & Kaur, M. A. (2019). A systematic review on imbalanced data challenges in machine learning: applications and solutions. ACM Comput, 52(4), 1-36. doi: 10.1145/3343440 [ Links ]
Ketkar, N., & Moolayil, J. (2021). Feed-forward neural networks. In: Ketkar, N., & Moolayil, J. (Eds.), Deep learning with python (pp. 93-132). Pune: APress. [ Links ]
Knauer, U., Rekowski, C. S. v., Stecklina, M., Krokotsch, T., Pham, M. T., Hauffe, V., Kilias, D., Ehrhardt, I., Sagischewski, H., Chmara, S., & Seiffert, U. (2019). Tree species classification based on hybrid ensembles of a convolutional neural network (cnn) and random forest classifiers. Remote Sensing, 11(23), 1-15. doi: 10.3390/rs11232788 [ Links ]
Kulkarni, A., Chong, D., & Batarseh, F. A. (2020). Foundations of data imbalance and solutions for a data democracy. In: Batarseh, F. A., & Yang, R. (Eds.), Data Democracy (pp. 83-106). Cambridge: Academic Press. [ Links ]
Liashchynskyi, P., & Liashchynskyi, P. (2019). Grid search, random search, genetic algorithm: A Big Comparison for NAS. arXiv, 12, 1-11. doi: 10.48550/arXiv.1912.06059 [ Links ]
Parmar, A., Katariya, R., & Patel, V. (2019). A review on random forest: an ensemble classifier. In: Hemanth, J., Fernando, X., Lafata, P., & Baig, Z. (Eds), International Conference on Intelligent Data Communication Technologies and Internet of Things, 2018, 758-763. Cham: Springer Nature Switzerland. [ Links ]
Pisner, D. A., & Schnyer, D. M. (2020). Support vector machine In: Mechelli, A., & Vieira, S. (Eds.), Machine learning - methods and applications to brain disorders. Cambridge: Academic Press . [ Links ]
Raschka, S., Liu, Y., & Mirjalili, V. (2022). A tour of machine learning classifiers using scikit-learn. In: Raschka, S., Liu, Y., & Mirjalili, V. (Eds.), Machine Learning with PyTorch and Scikit-Learn. Birmingham: Packt Publishing. [ Links ]
Saleem, M. H., Potgieter, J., & Arif, K. M. (2019). Plant disease detection and classification by deep learning. Plants, 8(11), 1-22. doi: 10.3390/plants8110468 [ Links ]
Sandhya, S., Balasundaram, A., & Arunkumar, S. (2022). Deep learning and computer vision based model for detection of diseased mango leaves. International Journal on Recent and Innovation Trends in Computing and Communication, 10(6), 70-79. doi: 10.17762/ijritcc.v10i6.5555 [ Links ]
Servicio de Información Agroalimentaria y Pesquera (SIAP) (2022). Aguacate. Panorama Agroalimentario. México: Servicio de Información Agroalimentaria y Pesquera. [ Links ]
Suresh, S., & Mohan, S. (2020). ROI-based feature learning for efficient true positive prediction using convolutional neural network for lung cancer diagnosis. Neural Computing and Applications, 32(20), 15989-16009. doi: 10.1007/s00521-020-04787-w [ Links ]
Srinivasa, G. N., Venkata, R. P., Anusha, T. M., Sai, H. V., & Bhanu, P. B. (2022). Detection of plant leaf diseases using random forest classifier. International Journal of Innovative Research in Technology, 9(1), 1300-1302. Retrieved from https://ijirt.org/master/publishedpaper/IJIRT155613_PAPER.pdf [ Links ]
Téliz, D., & Mora, A. (2019). Enfermedades. In: Téliz, D., & Mora, A. (Eds.), El aguacate y su manejo integrado (pp. 171-173). Texcoco: Biblioteca Básica de Agricultura. [ Links ]
Wardhani, N. W., Rochayani, M. Y, Iriany, A., Sulistyono, A. D., & Lestantyo, P. (2019). Cross-validation metrics for evaluating classification performance on imbalanced data. International Conference on Computer, Control, Informatics and its Applications, 2019, 14-18. doi: 10.1109/IC3INA48034.2019.8949568 [ Links ]
Yuvali, M., Yaman, B., & Tosun, O. (2022). Classification comparison of machine learning algorithms using two independent CAD datasets. Mathematics, 10(3), 1-15. doi: 10.3390/math10030311 [ Links ]
Recibido: 24 de Marzo de 2023; Aprobado: 24 de Julio de 2023











 texto en
texto en 




