Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista Chapingo. Serie horticultura
versión On-line ISSN 2007-4034versión impresa ISSN 1027-152X
Rev. Chapingo Ser.Hortic vol.25 no.2 Chapingo may./ago. 2019
https://doi.org/10.5154/r.rchsh.2018.06.012
Articles
Diversidad genética de nochebuena de sol (Euphorbia spp.) en Morelos, México, con marcadores moleculares RAPD
1Universidad Autónoma del Estado de Morelos, Facultad de Ciencias Agropecuarias. Av. Universidad núm. 1001, Cuernavaca, Morelos, C. P. 62209, MÉXICO.
2Universidad de Guanajuato, Programa de Biotecnología. Mutualismo núm. 303, Celaya, Guanajuato, C. P. 38060, MÉXICO.
3Instituto Nacional de Investigaciones Forestales, Agrícolas y Pecuarias, Campo Experimental Zacatepec. Carretera Zacatepec-Galeana km 0.5, Zacatepec de Hidalgo, Morelos, C. P. 62780, MÉXICO.
4Universidad Autónoma Chapingo. Departamento de Fitotecnia. Carretera México-Texcoco km 35.6, Chapingo, Estado de México, C. P. 56230, MÉXICO.
La nochebuena es una especie ornamental nativa de México conocida y distribuida mundialmente, que ha sido mejorada para obtener los cultivares que adornan actualmente en fiestas decembrinas. El objetivo de este estudio fue determinar la diversidad genética de Euphorbia spp., mediante marcadores moleculares RAPD, en 35 genotipos colectados en el estado de Morelos, México. En el estudio se incluyeron cuatro variedades de nochebuena de sol registradas en México y una especie de E. leucocephala Lotsy. Se utilizaron 21 iniciadores de 10 nucleótidos aleatorios que produjeron 91.82 % de polimorfismo, es decir, 203 fragmentos de ADN polimórficos de 222 generados. A partir de un análisis clúster se realizó una prueba de remuestreo por el método Jackknifing con 1,000 repeticiones y se formaron siete grupos consistentes representados en un dendrograma. Las accesiones que presentaron fragmentos genéticos distintos fueron PASCUA, ROSA, AMA 2, TVOL 2, JIU 1 y JIU 2, procedentes de Cuernavaca, Tetela del Volcán y Jiutepec, diferenciadas principalmente por su hábitat y color de brácteas. Los análisis realizados coincidieron en la agrupación de accesiones, separando a E. leucocephala de E. pulcherrima por ser una especie diferente. Los resultados obtenidos indicaron amplia diversidad genética de los materiales colectados en Morelos, con accesiones de importancia para próximos estudios de mejoramiento genético.
Palabras clave: bráctea; color; PCR; agrupación; diversidad
The poinsettia is an ornamental species native to Mexico, known and distributed worldwide, which has been bred to obtain the cultivars that currently adorn homes at Christmas. The objective of this study was to determine the genetic diversity of Euphorbia spp., through RAPD molecular markers, in 35 genotypes collected in the state of Morelos, Mexico. The study included four sun poinsettia varieties registered in Mexico and one species of E. leucocephala Lotsy. Twenty-one primers of 10 random nucleotides were used, producing 91.82 % polymorphism, i.e. 203 polymorphic DNA fragments of 222 generated. From a cluster analysis a resampling test was carried out using the Jackknifing method with 1,000 replicates, forming seven consistent groups represented in a dendrogram. The accessions that presented different genetic fragments were PASCUA, ROSA, AMA 2, TVOL 2, JIU 1 and JIU 2, from Cuernavaca, Tetela del Volcán and Jiutepec, differentiated mainly by their habitat and bract color. The analyses conducted coincided in the clustering of accessions, separating E. leucocephala from E. pulcherrima because it is a different species. The results showed a wide genetic diversity of the materials collected in Morelos, with accessions of importance for future breeding studies.
Keywords: bract; color; PCR; clustering; diversity
Introducción
La nochebuena (Euphorbia pulcherrima Willd. ex Klotzsch.) en su hábitat natural es un arbusto que florece en invierno y crece hasta 3 m. Las partes estructurales más importantes de esta especie son las brácteas que rodean a las flores, debido a sus tamaños, formas y colores diferentes (Canul-Ku et al., 2012a), y por su belleza se utiliza frecuentemente en paisajismo (Taylor, López, Currey, & Janick, 2011). En México, se puede encontrar de forma silvestre en su hábitat natural, establecida en traspatios y jardines, y cultivada como variedades mejoradas para decorar interiores (Canul-Ku et al., 2014).
Esta planta es originaria del norte de Guerrero, México (Trejo-Hernández et al., 2012), aunque crece de manera natural en los bosques tropicales subcaducifolios de la costa del pacífico, desde Sinaloa, México, hasta Guatemala, y en bosques tropicales secos de la Cuenca del Balsas, particularmente, en el norte de Guerrero y en Morelos (Trejo-Hernández, Olson-Zúnica, & Bye-Boettler, 2015). En 2017, se establecieron en México 18.05 millones de plantas con un valor de producción de 576.08 millones de pesos (Servicio de Información Agroalimentaria y Pesquera [SIAP], 2018), en donde Morelos participó con 33.12 % del total de la superficie establecida a nivel nacional, seguido de Michoacán, Ciudad de México y Puebla (SIAP, 2018). No obstante, las variedades disponibles en el mercado nacional se producen en el extranjero, lo que implica costos altos de adquisición de esqueje y problemas de manejo agronómico por la falta de adaptación (Canul-Ku et al., 2018a).
En 1950, inició en Morelos el cultivo de algunos materiales silvestres de nochebuena que se diferenciaban por la forma y color de brácteas (Colinas et al., 2015), los cuales se conocen como nochebuena de sol, de traspatio, eliofitica o semicultivados, y en los últimos años ha incrementado el interés por estos materiales (Galindo-García et al., 2012) cultivados comercialmente en viveros a campo abierto con escasa tecnología, aunque recientemente se ha generado información sobre su manejo agronómico (Galindo-García, Alia-Tejacal, Colinas-León, & Valdez-Aguilar, 2015a; Galindo-García et al., 2015b; García-Pérez et al., 2015). Por su parte, las variedades mejoradas o de sombra se cultivan en contenedores de diferente tamaño y presentación para su comercialización en temporada navideña (Canul-Ku et al., 2014).
En la presente década, diversos grupos de investigación en México han realizado colectas de nochebuena silvestre y de sol en varios estados del país (Canul-Ku, García-Pérez, Osuna-Canizales, & Ramírez-Rojas, 2012b) con la finalidad de conocer, conservar, proteger y utilizar este recurso fitogenético de México (Galindo-García et al., 2015a), ya que las áreas de distribución de esta especie se redujeron de un año de colecta a otro (Canul-Ku et al., 2014). Estos grupos han caracterizado, morfológicamente, 11 variedades de nochebuena de sol, las cuales están registradas en el Catalogo Nacional de Variedades Vegetales de México (Colinas et al., 2015; Galindo-García et al., 2015a); además, se han generado variedades mexicanas derivadas del mejoramiento por hibridación de accesiones de nochebuena silvestre (Canul-Ku et al., 2017, 2018a; Canul-Ku, García-Pérez, Barrios-Gómez, & Rangel-Estrada, 2018b). Sin embargo, dichos materiales no se han caracterizado molecularmente, lo cual podría servir para conocer su variación y diversidad genética para su uso en programas de mejoramiento genético u otras actividades (Canul-Ku et al., 2014).
En nochebuenas mejorada o de sombra se han realizado caracterizaciones con marcadores RAPD (Ling, Sauve, & Gawe, 1997), DAF (Starman, Duan, & Abbit, 1999) y AFLP (Parks & Moyer, 2004), lo cual ha resultado eficiente para identificar cultivares de nochebuena, determinar las relaciones entre los cultivares y desarrollar marcadores para características importantes en las variedades estrechamente relacionadas. En nochebuenas de sol y silvestre existen pocos trabajos con marcadores moleculares (Trejo-Hernández et al. 2012, 2015; Trejo-Hernández, Briones-Dumas, Gómez-Bermejo, & Olson, 2018); no obstante, estos se realizaron con fines de filogenia e historia evolutiva, y no de conocer su diversidad.
Es importante conocer la variabilidad y diversidad genética de las nochebuenas de sol en Morelos para su uso y conservación, además de identificarlas, estudiarlas y emplearlas en futuras investigaciones y programas de mejoramiento genético. Por ello, el presente estudio se llevó a cabo con el fin de determinar la diversidad genética, mediante marcadores RAPD, de 35 accesiones de Euphorbia spp. colectadas en el estado de Morelos, México.
Materiales y métodos
El ensayo experimental se realizó en el laboratorio de Producción Agrícola del Campo Experimental de la Universidad Autónoma del Estado de Morelos, localizada en Cuernavaca, Morelos, México. Se empleó la técnica de marcadores moleculares RAPD (Williams, Kubelik, Levak, & Tingey, 1990) y el protocolo de Doyle y Doyle (1987) para el aislamiento de ADN, con algunas modificaciones recomendadas por Andrade-Rodríguez, Villegas-Monter, Gutiérrez-Espinosa, Carrillo-Castañeda, y García-Velázquez (2005). Se estudiaron 35 accesiones colectadas en 19 municipios de Morelos, 34 de la especie E. pulcherrima (en su mayoría de brácteas de color rojo, una rosa y dos amarillas) y una de E. leucocephala (con brácteas de color blanco) (Cuadro 1). Entre las colectas se incluyeron cuatro variedades de nochebuena de sol registradas en el Catálogo Nacional de Variedades Vegetales del Servicio Nacional de Inspección y Certificación de Semillas (SNICS, 2018): ‘Valenciana’ (VAL), ‘Rehilete’ (REH 1), ‘Juan Pablo’ (ROSA) y ‘Amanecer Navideño’ (A. NAV) (Cuadro 1).
Cuadro 1 Accesiones colectadas en diferentes municipios de Morelos.
| Accesión | Clave | Localidad | Municipio | Color de brácteas | Desarrollo de nectarios |
|---|---|---|---|---|---|
| 1 | CUE 1 | Ocotepec | Cuernavaca | Rojo | Presente |
| 2 | REH 1a | Santa María Ahuacatitlán | Cuernavaca | Rojo | Ausente |
| 3 | REH 2 | Ocotepec | Cuernavaca | Rojo | Presente |
| 4 | CUE 4 | Tetela del Monte | Cuernavaca | Rojo | Presente |
| 5 | VALa | Tetela del Monte | Cuernavaca | Rojo | Ausente |
| 6 | CUE 5 | Tetela del Monte | Cuernavaca | Rojo | Ausente |
| 7 | ROSAa | Tetela del Monte | Cuernavaca | Rosado | Presente |
| 8 | A. NAVa | Tetela del Monte | Cuernavaca | Amarillo | Presente |
| 9 | PASCUA | Tetela del Monte | Cuernavaca | Blanco | Presente |
| 10 | BM 1 | Buenavista del Monte | Cuernavaca | Rojo | Presente |
| 11 | BM 2 | Buenavista del Monte | Cuernavaca | Rojo | Ausente |
| 12 | AMA 2 | Ocotepec | Cuernavaca | Amarillo | Presente |
| 13 | CUE 13 | Ocotepec | Cuernavaca | Rojo | Ausente |
| 14 | TEP 1 | Tepoztlán | Tepoztlán | Rojo | Ausente |
| 15 | TEP 2 | Tepoztlán | Tepoztlán | Rojo | Presente |
| 16 | YAU | Yautepec | Yautepec | Rojo | Presente |
| 17 | TLALT | San Miguel 30 | Tlaltizapán | Rojo | Presente |
| 18 | PIXTL | Puente de Ixtla | Puente de Ixtla | Rojo | Ausente |
| 19 | MIAC | Miacatlán | Miacatlán | Rojo | Ausente |
| 20 | TET | Tetecala | Tetecala | Rojo | Ausente |
| 21 | COATLAN | Coatlán del Rio | Coatlán del Rio | Rojo | Ausente |
| 22 | TYC 1 | Tlayacapan | Tlayacapan | Rojo | Ausente |
| 23 | TYC 2 | San Andrés | Tlayacapan | Rojo | Ausente |
| 24 | TLALN | Tlalnepantla | Tlalnepantla | Rojo | Ausente |
| 25 | TOTO | Totolapan | Totolapan | Rojo | Presente |
| 26 | OCU | Santa Mónica | Ocuituco | Rojo | Ausente |
| 27 | TVOL 1 | Tetela del Volcán | Tetela del Volcán | Rojo | Presente |
| 28 | TVOL 2 | San Miguel | Tetela del Volcán | Rojo | Presente |
| 29 | ZACU | Tlacotepec | Zacualpan | Rojo | Ausente |
| 30 | CUA | Gabriel Tetepa | Cuautla | Rojo | Ausente |
| 31 | JIU 1 | El Texcal | Jiutepec | Rojo | Presente |
| 32 | JIU 2 | El Texcal | Jiutepec | Rojo | Presente |
| 33 | TEPAL | Atotonilco | Tepalcingo | Rojo | Ausente |
| 34 | AXO | Axochiapan | Axochiapan | Rojo | Ausente |
| 35 | TLAQ | Las Garzas | Tlaquiltenango | Rojo | Ausente |
a = Variedades registradas.
El ADN se aisló a partir de 75 mg de tejido de hojas jóvenes frescas de cinco plantas clonales de las 35 accesiones para cuantificarlo, amplificarlo y evaluar su calidad. La cuantificación se realizó en un espectrofotómetro (DR5000, HACH®, Alemania) mediante lecturas en dos densidades ópticas (DO). Con la relación de la lectura de absorbancia a 260 y 280 nm (DO260/280) se estimó la pureza de los ácidos nucleícos y se calculó la concentración de ADN con la siguiente fórmula:
El grado de integridad del ADN se determinó por electroforesis a partir de una alícuota de 2 μL de la muestra de ADN en gel de agarosa ultrapura (Invitrogen®) al 1 % (Andrade-Rodríguez et al., 2005). La cámara de electroforesis (75-2321, Apollo®, EUA) trabajó a 80 V durante 1 h; posteriormente, el gel obtenido se colocó en un fotodocumentador (GVM20, Syngene®, Reino Unido) para fotografiarse en computadora (MT-M8131-35U, IBM®, EUA) mediante el editor de fotografías Zoombrowser de Cannon®.
La amplificación aleatoria de ADN polimórfico (RAPD, por sus siglas en inglés) se realizó con 21 iniciadores aleatorios decámeros (Cuadro 2), seleccionados de 60 indicadores probados previamente. La mezcla de reacción contenía 10 μL de dNTPs, 2 μL de amortiguador de carga (buffer PCR), 1.5 μL de cloruro de magnesio (MgCl), 4.7 μL de agua destilada desionizada estéril (adda) y 0.3 μL (1.5 U) de enzima Taq® ADN polimerasa nativa (Invitrogen®). La amplificación se realizó con la técnica de reacción en cadena de la polimerasa (PCR, por sus siglas en inglés), para la cual se utilizó un termociclador (TC-412, Techne®, Reino Unido) con el siguiente programa: un ciclo de predesnaturalización a 94 °C por 4 min, 36 ciclos de desnaturalización a 94 °C por 1 min, alineación a 36 °C por 1 min y polimerización a 72 °C por 2 min, y una extensión a 72 °C por 10 min (Andrade-Rodríguez et al., 2005).
Cuadro 2 Iniciadores utilizados en la amplificación de ADN.
| Iniciador | Secuencia de bases |
|---|---|
| 1 | 5’ AGG GGT CTT G 3’ |
| 2 | 5’ GAA ACG GGT G 3’ |
| 3 | 5’ GGG TAA CGC C 3’ |
| 4 | 5’ CAA TCG CCG T 3’ |
| 5 | 5’ AGG TGA CCG T 3’ |
| 6 | 5’ CAA ACG TCG G 3’ |
| 7 | 5’ GTT GCG ATC C 3’ |
| 8 | 5’ GTT TCG CTC C 3’ |
| 9 | 5’ TGC TCT GCC C 3’ |
| 10 | 5’ GGT GAC GCA G 3’ |
| 11 | 5’ CTG CTG GGA C 3’ |
| 12 | 5’ GTA GAC CCG T 3’ |
| 13 | 5’ AGG GAA CGA G 3’ |
| 14 | 5’ CCA CAG CAG T 3’ |
| 15 | 5’ GTG AGG CGT C 3’ |
| 16 | 5’ CCG CAT CTA C 3’ |
| 17 | 5’ GAA CGG ACT C 3’ |
| 18 | 5’ CTC ACC GTC C 3’ |
| 19 | 5’ AAG CCT CGT C 3’ |
| 20 | 5’ CAC ACT CCA G 3’ |
| 21 | 5’ ACT TCG CCA G 3’ |
A = adenina; G = guanina; T = timina; C = citosina.
La separación de fragmentos amplificados se realizó por electroforesis, con las mismas condiciones de la cámara y fotografía descritas previamente, a partir de 24 μL de la mezcla de reacción de PCR en gel de agarosa ultrapura (Invitrogen®) al 1.5 % (Andrade-Rodríguez et al., 2005). El tamaño de los fragmentos producidos por los marcadores RAPD se obtuvieron con el programa Genetools®.
Los porcentajes de polimorfismo se obtuvieron dividiendo el número de bandas generadas entre las bandas polimórficas. El contenido de información polimórfica (PIC, por sus siglas en ingles) se obtuvo con la formula PICi = 2fi (1-fi) (Fernández, Soto, Salazar, & Betancourt, 2010; Roldán-Ruiz, Dendauw, Van Bockstaele, Depicker, & de Loose, 2000), donde fi es la frecuencia de las bandas donde está presente el indicador i y el PICi es el promedio de PIC de todas las bandas de dicho iniciador i. El PIC se utilizó para determinar los iniciadores que generaron más información polimórfica en las accesiones evaluadas.
Se aplicó un análisis clúster mediante el coeficiente de similitud de Dice (Nei & Li, 1979) y el método de ligamiento promedio aritmético de grupos de pares no ponderados (UPGMA, por sus siglas en ingles), que es la combinación más adecuada para este tipo de estudios de acuerdo con Núñez-Colín y Escobedo-López (2011). Adicionalmente, se realizó una prueba de remuestreo por el método Jackknifing (Efron, 1979), con 1,000 repeticiones, mediante el programa Free Tree® ver. 0.9.1.50 (Pavlíček, Hrdá, & Flerg, 1999). Los métodos de remuestreo se emplean con el fin de obtener datos numéricos de consistencia del árbol generado, y han sido utilizados comúnmente para disminuir los errores estándar e intervalos de confianza (Mohammadi & Prassana, 2003). Posteriormente, se dibujó un dendrograma con el programa Tree View® ver. 1.6.6 (Page, 1996) y se partió en grupos de acuerdo con los valores de remuestreo, tomando como referencia el punto donde se tuviera el 100 % de certeza.
Finalmente, se hizo un análisis de coordenadas principales (Gower, 2005) con el programa NTSYS® ver. 2.1p. (Rohlf, 2000) para tener una representación tridimensional de las accesiones y poder corroborar los grupos del análisis clúster.
Resultados y discusión
Se revelaron en total 222 bandas, de las cuales 203 fueron polimórficas en la amplificación del ADN de las 35 accesiones estudiadas (Cuadro 3); además, se obtuvo 100 % de polimorfismo en 10 iniciadores utilizados (1, 2, 5, 7, 11, 14, 15, 18, 20 y 21), con un promedio de 91.82 % (Cuadro 3). En variedades comerciales de nochebuena, Ling et al. (1997) obtuvieron 69 % de polimorfismo con 33 bandas polimórficas de 48 registradas, muy por debajo del obtenido en el presente estudio, esto quizás porque únicamente evaluaron variedades comerciales; además, reportaron que 42 indicadores, de 57, fueron totalmente monomórficos. En este caso, los iniciadores 8, 12 y 19 produjeron un mayor número de fragmentos de los genotipos de Euphorbia y más de 80 % de polimorfismo.
Cuadro 3 Número de fragmentos encontrados, porcentaje de polimorfismo y contenido de información polimórfica.
| Iniciador | Total de bandas | Bandas polimórficas | Bandas únicas | Bandas monomórficas | Polimorfismo (%) | PIC1 |
|---|---|---|---|---|---|---|
| 1 | 8 | 8 | 2 | 0 | 100 | 0.18 |
| 2 | 12 | 12 | 5 | 0 | 100 | 0.15 |
| 3 | 12 | 9 | 4 | 3 | 75 | 0.06 |
| 4 | 10 | 9 | 2 | 1 | 90 | 0.11 |
| 5 | 9 | 9 | 3 | 0 | 100 | 0.13 |
| 6 | 10 | 8 | 2 | 2 | 80 | 0.14 |
| 7 | 11 | 11 | 1 | 0 | 100 | 0.24 |
| 8 | 15 | 12 | 4 | 3 | 80 | 0.09 |
| 9 | 9 | 7 | 2 | 2 | 77.78 | 0.06 |
| 10 | 7 | 6 | 3 | 1 | 85.71 | 0.16 |
| 11 | 9 | 9 | 0 | 0 | 100 | 0.20 |
| 12 | 14 | 12 | 3 | 2 | 85.71 | 0.18 |
| 13 | 11 | 9 | 2 | 2 | 81.82 | 0.20 |
| 14 | 10 | 10 | 4 | 0 | 100 | 0.18 |
| 15 | 11 | 11 | 2 | 0 | 100 | 0.19 |
| 16 | 9 | 8 | 2 | 1 | 88.89 | 0.20 |
| 17 | 11 | 10 | 1 | 1 | 90.91 | 0.11 |
| 18 | 12 | 12 | 4 | 0 | 100 | 0.12 |
| 19 | 13 | 12 | 2 | 1 | 92.31 | 0.21 |
| 20 | 8 | 8 | 0 | 0 | 100 | 0.30 |
| 21 | 11 | 11 | 4 | 0 | 100 | 0.10 |
| Total | 222 | 203 | 52 | 19 | - | - |
| Media | 10.57 | 9.67 | 2.48 | 0.90 | 91.82 | 0.16 |
1PIC = contenido de información polimórfica.
El número total de bandas amplificadas variaron entre 7 y 15, con un promedio de 10.5 bandas por iniciador (Cuadro 3). Este valor fue superior a las 5.4 bandas, en promedio, reportadas en nochebuenas de sombra (Ling et al., 1997). El promedio del PIC obtenido fue de 0.16, en donde el iniciador 20 obtuvo el mayor valor, seguido del 6 y 19 (Cuadro 3), lo que indica el poder discriminatorio de estos marcadores al diferenciar los genotipos estudiados, por lo que se recomienda su uso en futuros estudios de Euphorbia. Asimismo, se identificaron 52 bandas únicas, de las cuales PASCUA presentó 46, ROSA y JIUT 1 presentaron dos, y TVOL 1 y TVOL 2 sólo uno (Cuadro 2).
Se detectó alta variabilidad genética entre y dentro de las especies estudiadas de acuerdo con sus porcentajes de polimorfismo y sus valores de PIC. Además, las accesiones PASCUA, TVOL 2, ROSA, AMA 2, JIU 1 y JIU 2 presentaron mayor polimorfismo en sus fragmentos de ADN obtenidos, así como mayor número de fragmentos únicos (Figura 1).

Figura 1 Amplificación de ADN de accesiones colectadas en Morelos, México, en gel de agarosa al 1.5 % teñido con bromuro de etidio. Sólo se muestran tres iniciadores de los 21 empleados: a) 8, b) 13 y c) 19.
A partir del análisis clúster se formaron siete grupos con 100 % de consistencia y con diferencias genéticas entre ellos, lo que indica amplia diversidad genética de las accesiones colectadas en Morelos (Figura 2). El grupo con mayor número de accesiones integradas fue el 2 con 21, seguido del 1 con ocho, el grupo 5 con dos y el resto de los grupos con una sola accesión, lo que concuerda con las diferencias presentadas (Figura 2).
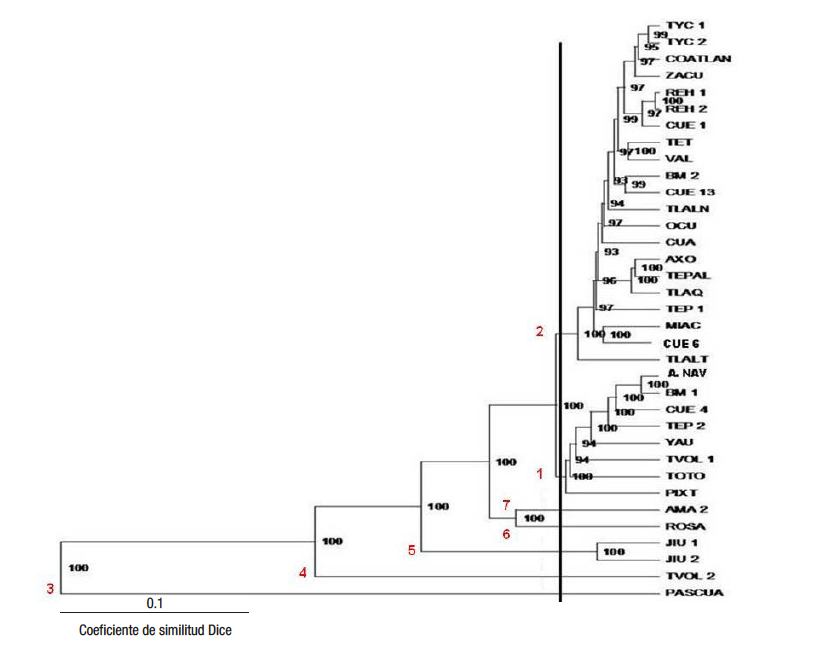
Figura 2 Dendrograma de caracterización molecular, mediante el coeficiente de similitud de Dice, de 35 accesiones colectadas en Morelos, México.
De acuerdo con Mohammadi y Prasanna (2003), los porcentajes mayores a 70 en las ramificaciones internas del dendrograma son aceptables. En este caso se obtuvieron valores superiores a 93 %, y en 18 ramificaciones se logró 100 %, valores adecuados para la formación y consistencia de los grupos creados (Tapia-Pérez & Legaria-Solano, 2007) (Figura 2). Las proporciones de las ramificaciones internas del dendrograma pueden servir para demostrar la consistencia de los grupos, como se ha utilizado en algunos cultivos: Jatropha curcas L. (Pecina-Quintero et al., 2011), ajonjolí (Sesamum indicum L.) (Salazar, Laurentin, Dávila, & Castillo, 2006) y guayaba (Psidium guajava L.) (Tapia-Pérez & Legaria-Solano, 2007), para caracterizar molecularmente o identificar variabilidad genética. Además, dichos valores son útiles para determinar el corte del dendrograma, tomando en cuenta las ramificaciones que presenten el 100 %.
El grupo 1 se caracterizó por presentar plantas de nochebuena de sol que desarrollaron nectarios, excepto la accesión de PIXT (Figura 2 y 3a). Todas las accesiones de este grupo presentaron brácteas de color rojo, menos A. NAV (brácteas color rosa) que es una variedad registrada en México, lo que se sugiere que el contenido genético es similar en las demás (Figura 2).
Las plantas del grupo 2 presentaron brácteas de color rojo, además de agrupar dos accesiones registradas (REH 1 y VAL). Las accesiones REH 1 y REH 2 tienen brácteas deformadas, por lo que probablemente surgieron como una modificación de la nochebuena ‘Valenciana’ (Figura 3b). Las variedades de sol ‘Valenciana’ y ‘Rehilete’ son las más cultivadas en Morelos (Galindo-García et al., 2012), por ello este grupo es el más numerosos.

Figura 3 Ejemplos de accesiones que presentaron mayor diversidad genética de cada grupo: a) grupo 1 (TEP 2), b) grupo 2 (VAL), c) grupo 3 (PASCUA), d) grupo 4 (TVOL 2), e) grupo 5 (JIU 1), f) grupo 6 (ROSA) y g) grupo 7 (AMA 2).
Los grupos 1 y 2 presentaron materiales recolectados y variedades registradas de nochebuena de sol, por lo que se puede inferir que estas últimas son el resultado de la domesticación de especies debido al transporte de plantas de un lugar a otro, ya que los primeros pobladores de las grandes ciudades se dispersaron hacia otros municipios y se llevaron este material vegetal (Trejo-Hernández et al., 2015). Aunque otra opción es que los habitantes hayan obtenido el material vegetal de poblaciones silvestres para adornar sus jardines (Trejo-Hernández et al., 2018).
Seis pares de accesiones en el grupo 2 fueron muy similares genéticamente entre sí: TYC 1 y 2, REH 1 y 2, TET y VAL, BM 2 y CUE 13, AXO y TEP, MIAC y CUE 6. La accesión de CUE 1 está cerca de REH 1 y 2, y TLAQ está cerca de AXO y TEP 1, posiblemente debido al flujo genético reportado por Tapia-Pérez y Legaria-Solano (2007). Estos autores mencionan que la similitud entre accesiones puede ser originada por dos factores: transporte de semilla por aves y transporte semillas o esquejes de genotipos por los productores para propagarlos. En este trabajo se sugiere que habitantes de otros municipios compraron plantas de nochebuena de sol en Cuernavaca y se las llevaron a sus lugares de origen. Trejo-Hernández et al. (2015) mencionan que no se conocen híbridos naturales de esta planta, por lo que se sugiere que la dispersión de la nochebuena de sol ha sido por esquejes.
El grupo 3 se integró únicamente por la accesión PASCUA (E. leucocephala) colectada en Cuernavaca, la cual presentó desarrollo de nectarios y brácteas muy pequeñas de color blanco (Figura 3c). Esta accesión pertenece a otra especie, lo que coincide con que tuvo el mayor número de fragmentos de ADN diferentes, alejándola de las demás en el dendrograma (Figura 2). Asimismo, el grupo 4 también se formó por una accesión (TVOL 2) (Figura 3d), la cual presenta brácteas color rojo, desarrollo de nectarios y una ligera deformación de brácteas, por lo que se infiere que puede ser un material silvestre, ya que se colectó en un hábitat natural en Tétela del Volcán. Lo anterior corrobora el resultado obtenido con TVOL 2, que fue genéticamente diferente a las demás, y presentó un fragmento único y un fragmento que sólo coincidió con uno de PASCUA.
El grupo 5 se integró por dos accesiones: JIU 1 y JIU 2 (Figura 3e), con brácteas rojas y desarrollo de nectarios, ambas originarias de Texcal, Jiutepec, Morelos, y de hábitat silvestre. En Jiutepec existe una población importante de Euphorbia pulcherrima en un área de conservación ecológica decretada en 1992, actualmente declarada como área natural protegida (Comisión Estatal del Agua y Medio Ambiente [CEAMA], 2018). Trejo-Hernández et al. (2015) indican que las plantas de apariencia silvestre desarrollan inflorescencias simples y con variantes genéticas. En JIU 1 y JIU 2 se identificaron tres fragmentos únicos, es decir, que no se presentaron en otras accesiones, lo que las colocó en un grupo diferente.
El grupo 6 se formó por el genotipo ROSA (Figura 3f), el cual presenta brácteas de color rosa y desarrollo de nectarios, lo que da lugar a inflorescencias simples. Finalmente, el grupo 7 se integró por la accesión AMA 2 (Figura 3e) que desarrolla nectarios y brácteas delgadas de color amarillo con ligera deformación. Se sugiere que su hábitat es natural por localizarse en los límites de la ciudad de Cuernavaca y presentar similitud con ROSA, ya que coinciden en la presencia de dos fragmentos que únicamente presentan ambos genotipos.
La variabilidad genética de las accesiones originarias de Morelos se atribuye principalmente al lugar de colecta, ya que las plantas silvestres mostraron diferencias en su genotipo, lo que implica que se conserva la pureza varietal (Fernández et al., 2010). Otra variable de correlación fue el color de bráctea, en donde la mayoría de las accesiones de diferentes municipios de Morelos son de color rojo y las de diferente color se concentraron en Cuernavaca: PASCUA (blancas), AMA 2 y A. NAV (amarillas), y ROSA (rosas); además, las variedades registradas se agruparon de acuerdo con el color de brácteas: ‘Amanecer Navideño’ (A. NAV-amarillas) en grupo 1, ‘Valenciana’ y ‘Rehilete’ (VAL y REH-rojas) en grupo 2, y ‘Juan Pablo’ (ROSA-rosas) en grupo 6. Esto coincide con Prasad (2014), quien reportó que el color de las flores en Hibicus rosasinensis determina la agrupación de accesiones.
La agrupación de las 35 accesiones colectadas en Morelos se mostró consistente en los siete grupos bien definidos en el análisis clúster y representados en la gráfica del análisis de coordenadas principales (Figura 4), los cuales se diferenciaron por su hábitat y color de brácteas. En esta gráfica se puede notar la distancia de PASCUA (grupo 3) con las demás, al ser una especie diferente.
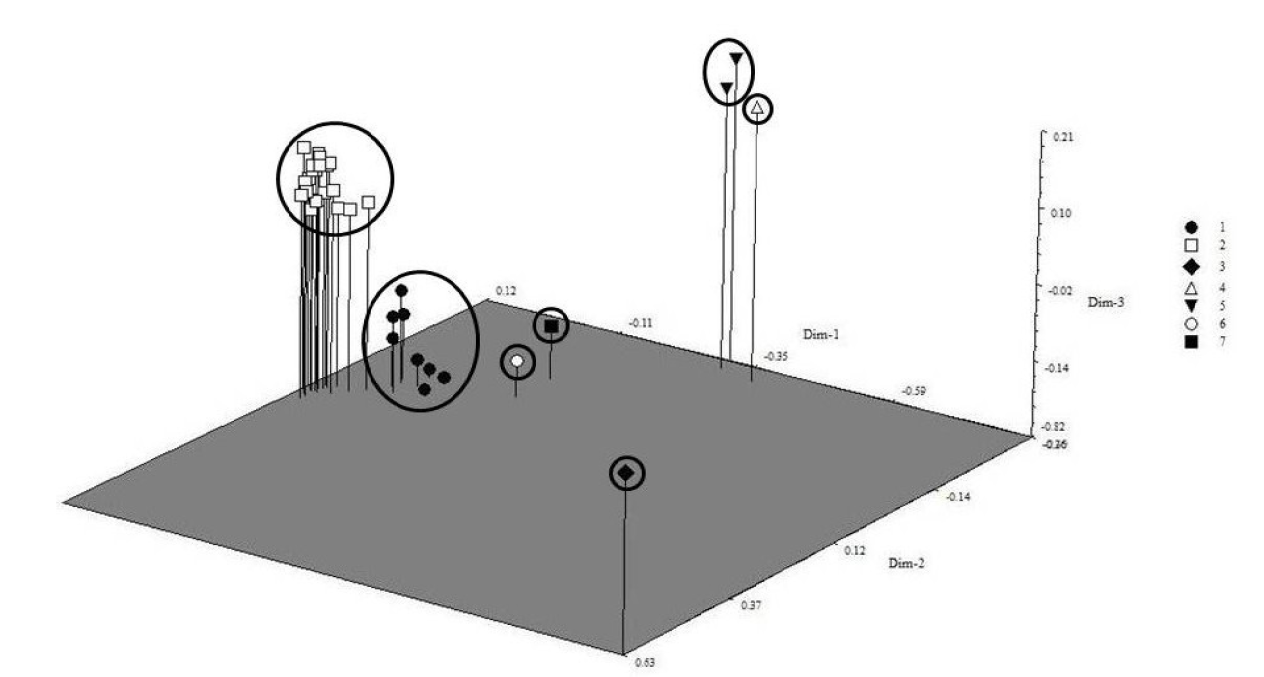
Figura 4 Gráfica tridimensional de dispersión, obtenida mediante el análisis de coordenadas principales, de las 35 accesiones colectadas en Morelos, México.
El desarrollo de nectarios se presentó en 16 accesiones (CUE 1, REH 2, CUE 4, ROSA, A. NAV, AMA 2, PASCUA, BVM 1, TEP 2, YAU, TLALTI, TOTO, TVOL 1, TVOL 2, JIU 1 y JIU 2), lo que es de suma importancia debido al posible cruzamiento entre plantas. El resto de las accesiones no presentaron nectarios, por lo que no desarrollan semilla y en su lugar brotaron unas estructuras conocidas como bractéolas (Colinas et al., 2015). Estas accesiones no pueden ser utilizadas en cruzas debido a que están presentes en todos los grupos aquí descritos. El identificar y cruzar genotipos distantes genéticamente permite incrementar la probabilidad de tener heterosis en el sistema (Wright, 1978).
El análisis de diversidad genética permitió obtener una agrupación confiable de las accesiones colectadas en Morelos. Además, se identificaron grupos de accesiones núcleo con la posibilidad de utilizarlos en próximos estudios de mejoramiento genético (Mohammadi & Prasanna, 2003; Valera-Montero, Hernández-Dávila, Silos-Espino, & Flores-Benítez, 2017).
Conclusiones
La caracterización molecular permitió detectar amplia diversidad genética de accesiones colectadas en Morelos, México, con características de importancia para utilizarse en próximos programas de mejoramiento genético. Con los marcadores moleculares RAPD y los análisis estadísticos se diferenciaron y agruparon las accesiones colectadas, identificándose 203 fragmentos polimórficos (91.82 %) que determinaron el grado de diversidad genética en la población de estudio. Se formaron siete grupos consistentes, los cuales se asociaron con el color de brácteas y su hábitat. Las accesiones que presentaron mayor variabilidad en sus fragmentos de ADN fueron PASCUA, TVOL 2, JIU 1, JIU 2, ROSA y AMA 2. La especie E. leucocephala fue diferente a E. pulcherrima en su genotipo.
References
Andrade-Rodríguez, M., Villegas-Monter, A., Gutiérrez-Espinosa, M. A., Carrillo-Castañeda, G. & García-Velázquez, A. (2005). Poliembrionía y marcadores RAPD para la identificación de plántulas cigóticas y nucelares en Citrus. Agrociencia, 39(4), 371-383. Retrieved from https://www.colpos.mx/agrocien/Bimestral/2005/jul-ago/art-2.pdf [ Links ]
Canul-Ku, J., García-Pérez, F., Campos-Bravo, E., Barrios-Gómez, E., Cruz-Torres, E., García-Andrade, J. M., Osuna-Canizalez, F. J., & Ramírez-Rojas, S. (2012a). Efecto de la irradiación sobre la nochebuena silvestre (Euphorbia pulcherrima Willd. ex Klotszch) en Morelos. Revista Mexicana de Ciencias Agrícolas, 31(8), 1495-1507. doi: 10.29312/remexca.v3i8.1316 [ Links ]
Canul-Ku, J., García-Pérez, F., Osuna-Canizales, F. J., & Ramírez-Rojas, S. (2012b). Metodologías de mejoramiento genético aplicables en nochebuena. México: Instituto Nacional de Investigaciones Forestales, Agrícolas y Pecuarias. [ Links ]
Canul-Ku, J., García-Pérez, F., Barrios-Gómez, E., Osuna-Canizales, F. J., Ramírez-Rojas, S., Alia-Tejacal, I., & Montoya-Contreras, R. E. (2014). Caracterización morfológica de nochebuena (Euphorbia pulcherrima Willd. ex Klotzsch). Ciencia y Tecnología Agropecuaria, 2(2), 16-23. Retrieved from http://www.somecta.org.mx/Revistas/CYTAM2.2/CYTAM-2014-2-12.pdf [ Links ]
Canul-Ku, J., García-Pérez, F., Barrios-Gómez, E., Rangel-Estrada, S. E., Ramírez-Rojas, S., & Osuna-Canizales, F. J. (2017). Alondra: nuevo híbrido de nochebuena para interiores. Revista Mexicana de Ciencias Agrícolas , 8(5), 1203-1208. doi: 10.29312/remexca.v8i5.119 [ Links ]
Canul-Ku, J., García-Pérez, F., Barrios-Gómez, E. J., Osuna-Canizalez, F. J., Rangel-Estrada, S. E., & Ramírez-Rojas, S. G. (2018a). Rubí, nueva variedad Mexicana de nochebuena para interior. Revista Fitotecnia Mexicana, 41(1), 91-92. Retrieved from https://www.revistafitotecniamexicana.org/documentos/41-1/12a.pdf [ Links ]
Canul-Ku, J., García-Pérez, F., Barrios-Gómez, E. J., & Rangel-Estrada, S. E. (2018b). Formación de híbridos clonales en nochebuena (Euphorbia pulcherrima Willd. ex Klotzsch). Revista Fitotecnia Mexicana, 41(3), 311-316. Retrieved from https://www.revistafitotecniamexicana.org/documentos/41-3/11a.pdf [ Links ]
Colinas, L. M. T., Espinosa, A., Mejía, M. J., Rodríguez, M. A., Pérez, M. L., & Alia, T. I. (2015). Cultivars of Euphorbia pulcherrima from Mexico. Acta Horticulturae, 1104, 487-490. doi: 10.17660/ActaHortic.2015.1104.70 [ Links ]
Comisión Estatal del Agua y Medio Ambiente (CEAMA). (2018). Sistema estatal de áreas naturales protegidas. Retrieved from http://www.ceamamorelos.gob.mx [ Links ]
Doyle, J. J., & Doyle, J. L. (1987) A rapid DNA isolation procedure for small quantities of fresh leaf tissue. Phytochemical Bulletin, 19, 11-15. [ Links ]
Efron, B. (1979). Bootstrap methods: another look at the jackknife. The Annals of Statistics, 7(1), 1-26. Retreived from http://www.jstor.org/stable/2958830 [ Links ]
Fernández, H., Soto, E., Salazar, E., & Betancourt, M. (2010). Análisis RAPD entre y dentro de poblaciones derivadas de girasol silvestre x cultivado. Agronomía Tropical, 60(3), 263-269. Retrieved from http://sian.inia.gob.ve/revistas_ci/Agronomia%20Tropical/at6003/pdf/at6003_fernandez_h.pdf [ Links ]
Galindo-García, D. V., Alia-Tejacal, I., Andrade-Rodríguez, M., Colinas-León, M. T., Canul-Ku, J., & Sainz-Aispuro, M. J. (2012). Producción de nochebuena de sol (Euphorbia pulcherrima Willd. ex Klotzsch) en Morelos, México. Revista Mexicana de Ciencias Agrícolas , 3(4), 751-763. doi: 10.29312/remexca.v3i4.1428 [ Links ]
Galindo-García, D. V., Alia-Tejacal, I., Colinas-León, M. T. B., & Valdez-Aguilar, L. A. (2015a). Caracterización agronómica de la nochebuena de sol (Euphorbia pulcherrima Willd. ex Klotzsch). México: Universidad Autónoma del Estado de Morelos. [ Links ]
Galindo-García, D. V., Alia-Tejacal, I., Valdez-Aguilar, L. A., Colinas-León, M. T., Villegas-Torres, O. G., López-Martínez, V., Sainz-Aispuro, M. J., & Guillén-Sánchez, D. (2015b). Extracción de macronutrimentos y crecimiento em variedades de nochebuena de sol nativas de México. Revista Fitotecnia Mexicana , 38(3), 305-312. Retrieved from https://www.revistafitotecniamexicana.org/documentos/38-3/8a.pdf [ Links ]
García-Pérez, F., Canul-Ku, J., Osuna-Canizalez, F. J., Ramírez-Rojas, S. G., Rangel-Estrada, S. E., & Portas-Fernández, B. (2015). Propagación de nochebuena de sol (Euphorbia pulcherrima Willd. ex Klotzsch). México: Instituto Nacional de Investigaciones Forestales, Agrícolas y Pecuarias . [ Links ]
Gower, J. C. (2005). Principal coordinates analysis. In: Armitage, P., & Colton, T. (Ed.), Encyclopedia of biostatistics (pp. 3514-3518). Chichester, UK: John Wiley & Sons. [ Links ]
Ling, J. T., Sauve, R., & Gawe, N. (1997). Identification of poinsettia cultivars using RAPD markers. Hortscience, 32(1), 122-124. doi: 10.21273/HORTSCI.32.1.122 [ Links ]
Mohammadi, S. A., & Prasanna, B. M. (2003). Analysis of genetic diversity in crop plants-salient statistical tools and considerations. Crop Science, 43(4), 1235-1248. doi: 10.2135/cropsci2003.1235 [ Links ]
Nei, M., & Li, W. H. (1979). Mathematical model for studying genetic variation in terms of restriction endonucleases. Proceedings of the National Academy of Science, 76, 5269-5273. doi: 10.1073/pnas.76.10.5269 [ Links ]
Núñez-Colín, C. A., & Escobedo-López, D. (2011). Uso correcto del análisis clúster en la caracterización de germoplasma vegetal. Agronomía Mesoamericana, 22(2), 415-427. doi: 10.15517/am.v22i2.8746 [ Links ]
Page, R. D. M. (1996). Tree view: An application to display phylogenetic trees on personal computers. Bioinformatics, 12(4), 357-358. doi: 10.1093/bioinformatics/12.4.357 [ Links ]
Parks, E. J., & Moyer, J. W. (2004). Evaluation of AFLP in poinsettia: polymorphism, selection, analysis, and cultivar identification. Journal of American Society for Horticultural Science, 129(6), 863-869. doi: 10.21273/JASHS.129.6.0863 [ Links ]
Pavlíček, A., Hrdá, S., & Flegr, J. (1999). Free Tree--freeware program for construction of phylogenetic trees on the basis of distance data and bootstrap/jackknife analysis of the tree robustness. Application in the RAPD analysis of genus Frenkelia. Folia Biologica (Praha), 45(3), 97-99. Retrieved from http://www.flegr.com/wp-content/uploads/2017/05/FL14-freetree.pdf [ Links ]
Pecina-Quintero, V., Anaya-López, J. L., Zamarripa-Colmenero, A., Montes-García, N., Núñez-Colín, C. A., Solís-Bonilla, J. L., & Mejía-Bustamante, D. J. (2011). Molecular characterization of Jatropha curcas L. genetic resources from Chiapas, México through AFLP markers. Biomass and Bioenergy, 35(5), 1897-1905. doi: 10.1016/j.biombioe.2011.01.027 [ Links ]
Prasad, M. P. (2014). Molecular characterization and genetic diversity determination of Hibiscus species using RAPD molecular markers. Asian Journal of Plant Science and Research, 4(3), 50-56. [ Links ]
Rohlf, F. J. (2000). Numerical taxonomy and multivariate analysis system version 2.1 . User Guide. New York, USA: Exeter Software. [ Links ]
Roldán-Ruiz, I., Dendauw, J., Van Bockstaele, E., Depicker, A., & de Loose, M. (2000) AFLP markers reveal high polymorphic rates in ryegrass (Lolium spp.). Molecular Breeding, 6(2), 125-134. doi: 10.1023/A:1009680614564 [ Links ]
Salazar, B., Laurentin, H., Dávila, M., & Castillo, M. A. (2006). Rehability of the RAPD technique for germplasm analysis of sesame (Sesamum indicum L.) from Venezuela. Interciencia, 31(6), 456-460. Retrieved from http://www.redalyc.org/pdf/339/33911712.pdf [ Links ]
Servicio Nacional de Inspección y Certificación de Semillas (SNICS). (2018). Catálogo Nacional de Variedades Vegetales. México: Secretaria de Agricultura, Ganadería, Desarrollo Rural, Pesca y Alimentación. Retrieved from http://snics.sagarpa.gob.mx [ Links ]
Servicio de Información Agroalimentaria y Pesquera (SIAP). (2018). Cierre de la producción (1980-2017). Retrieved from https://www.gob.mx/siap/acciones-y-programas/produccion-agricola-33119 [ Links ]
Starman, T. W., Duan, X., & Abbitt, S. (1999). Nucleic acids scanning techniques distinguish closely related cultivars of poinsettia. HortScience, 34(6), 1119-1122. Retrieved from http://hortsci.ashspublications.org/content/34/6/1119.full.pdf+html [ Links ]
Tapia-Pérez, D., & Legaria-Solano, J. P. (2007). Variabilidad genética en cultivares de guayabo (Psidium guajava L.). Revista Fitotecnia Mexicana , 30(4), 391-401. Retrieved from https://www.revistafitotecniamexicana.org/documentos/30-4/6a.pdf [ Links ]
Taylor, J. M., López, R. G., Currey, C. J., & Janick, J. (2011). The poinsettia: history and transformation. Chronica Horticulturae, 51, 23-28. Retrieved from https://www.ishs.org/system/files/chronica-documents/ch5103.pdf [ Links ]
Trejo-Hernández, L., Feria-Arroyo, T., Olsen, K. M., Eguiarte, L. E., Arroyo, B., Gruhn, J. A., & Olson, M. E. (2012). Poinsettia’s wild ancestor in the Mexican dry tropics: historical, genetic, and environmental evidence. American Journal of Botany, 99(7), 1146-1157. doi: 10.3732/ajb.1200072 [ Links ]
Trejo-Hernández, L., Olson-Zúnica, M. E., & Bye-Boettler, R. A. (2015). Datos históricos y diversidad genética de las nochebuenas (Euphorbia pulcherrima) del Distrito Federal, México. Revista Mexicana de Biodiversidad, 86(2), 478-485. doi: 10.1016/j.rmb.2015.04.033 1870-3453 [ Links ]
Trejo-Hernández, L., Briones-Dumas, E., Gómez-Bermejo, R., & Olson, M. E. (2018). Molecular evidence for repeated recruitment of wild christmas poinsettia (Euphorbia pulcherrima) intro traditional horticulture in Mexico. Genetic Resources and Crop Evolution, 66(2). doi: 10.1007/s10722-018-0727-1 [ Links ]
Valera-Montero, L. L., Hernández-Dávila, A., Silos-Espino, H., & Flores-Benítez, S. (2017). Variación genética en guayaba mediante RAPDs y descriptores morfológicos en Calvillo, Aguascalientes. Revista Mexicana de Ciencias Agrícolas , 8(1), 65-75. doi: 10.29312/remexca.v8i1.72 [ Links ]
Williams, J. G., Kubelik, A. R., Levak, K. J., & Tingey, S. V. (1990). DNA polymorphisms amplified by arbitrary primers are useful as genetic markers. Nucleic Acids Research, 18(22), 6531-6535. doi: 10.1093/nar/18.22.6531 [ Links ]
Wright, S. (1978). Evolution and the genetics of populations. Variability within and among natural populations. Chicago: University of Chicago. [ Links ]
Recibido: 24 de Junio de 2018; Aprobado: 07 de Marzo de 2019











 texto en
texto en 


