Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Similars in
SciELO
Similars in
SciELO
Share
Revista Chapingo. Serie horticultura
On-line version ISSN 2007-4034Print version ISSN 1027-152X
Rev. Chapingo Ser.Hortic vol.24 n.2 Chapingo May./Aug. 2018
https://doi.org/10.5154/r.rchsh.2017.12.038
Análisis filogenético de algunos miembros del subgénero Persea (Persea, Lauraceae)
1Universidad Autónoma Chapingo, Departamento de Fitotecnia. Carretera México-Texcoco km 38.5, Texcoco, Estado de México, C. P. 56230, MÉXICO.
2Instituto Nacional de Investigaciones Forestales, Agrícolas y Pecuarias, Centro Nacional de Recursos Genéticos. Av. Biodiversidad núm. 2498, col. Centro, Tepatitlán de Morelos, Jalisco, C. P. 47600, MÉXICO.
3Universidad Autónoma del Estado de México. Carretera Tenancingo-Villa Guerrero km 1.5, Tenancingo, Estado de México, C. P. 52400, MÉXICO.
El aguacate pertenece al género Persea, el cual es uno de los más controversiales de la familia Lauraceae debido a que las relaciones entre el subgénero Persea no están claras, y solo se reconocen dos especies, Persea americana y Persea schiedeana. Su relación con el subgénero Eriodaphne también es compleja, y existe un debate sobre si este es un género independiente. Por ello, el objetivo de esta investigación fue analizar las relaciones filogenéticas dentro del género Persea, con énfasis en el subgénero Persea, utilizando la máxima parsimonia e inferencia bayesiana con la secuencia de ocho fragmentos diferentes de ADN nuclear, cloroplástico y mitocondrial. Se emplearon secuencias del cloroplasto ndhF, rbcL, matK, rpoC, trnH-psbA, mitocondrias atp4 y cox3, y 18S rARN nuclear. Se encontraron 14 mutaciones fijas en especies del subgénero Eriodaphne. La máxima parsimonia, los análisis filogenéticos bayesianos de las supermatrices de las cinco secuencias de cloroplastos y las ocho concatenadas separaron a los miembros de ambos subgéneros en dos clados diferentes con un alto bootstrap y soporte de probabilidad posterior. Lo anterior sugiere que el origen de Persea no es monofilético y, por lo tanto, ambos subgéneros, Persea y Eriodaphne, podrían ser reconocidos como géneros filogenéticamente independientes.
Palabras clave: “aguacatillo”; aguacate; filogenia; ADN de cloroplastos; ADN mitocondrial
The avocado belongs to the genus Persea, which is one of the most controversial genera of the Lauraceae family, since the relationships within the subgenus Persea are not clear and only recognized two species, Persea americana and Persea schiedeana. Its relationship with the subgenus Eriodaphne is also complex and there is a debate as to whether it is an independent genus. For this reason, the study aims to analyze the phylogenetic relationships within the genus Persea, with an emphasis on the subgenus Persea, using maximum parsimony and bayesian inference with the sequence of eight different fragments from nuclear, chloroplast and mitochondrial DNA. Sequences of the chloroplast ndhF, rbcL, matK, rpoC, trnH-psbA; mitochondria atp4 and cox3 and nuclear 18S rRNA were used. Fourteen fixed mutations were found in species of the subgenus Eriodaphne. The maximum parsimony and bayesian phylogenetic analyses of the super-matrices of the five chloroplast sequences and the eight concatenated ones, separated the members of both subgenera into two different clades with high bootstrap and posterior probability support, suggesting that the origin of Persea is not monophyletic and therefore both subgenera, Persea and Eriodaphne, could be recognized as phylogenetically independent genera.
Keywords: “aguacatillo”; avocado; phylogeny; chloroplast DNA; mitochondrial DNA
Introducción
El aguacate (Persea americana Mill.) se encuentra entre los cultivos de frutos subtropicales/tropicales más importantes del mundo (Bost, Smith, & Crane, 2013), con una producción que supera las 3.5 millones de toneladas, de las cuales aproximadamente 20 % se comercializan internacionalmente (Schaffer, Wolstenholme, & Whiley, 2013). Chanderbali et al. (2008) consideraron al aguacate como el producto más importante de las Lauráceas.
La conservación de los recursos genéticos del aguacate y sus parientes es importante para poder enfrentar los problemas potenciales de la industria del aguacate. Recientemente, han surgido amenazas en la industria del aguacate, como el marchitamiento del laurel, causado por el hongo Raffaelea lauricola simbionte del escarabajo ambrosial (Xyleborus glabratus). Este hongo es responsable de la muerte extensiva de Lauráceas nativas de Estados Unidos desde el 2000, cuando fue detectado por primera vez (Fraedrich et al., 2008). En agosto de 2011, cerca del centro de producción de aguacate en Florida, Estados Unidos, un árbol de aguacate de un jardín familiar se vio afectado por la marchitez del laurel (Ploetz et al., 2015), por lo que la resistencia a esta enfermedad es de alta prioridad, y el acervo para buscar esta resistencia se encuentra en los recursos genéticos del género Persea.
Los bancos de germoplasma han tratado de conservar la diversidad existente de aguacate y sus parientes (Barrientos, 2010). Uno de estos bancos está ubicado en la Fundación Salvador Sánchez Colín-CICTAMEX, S.C., el cual comenzó a concentrar más diversidad en 1988 (Barrientos, 1999) y es considerado el más rico en cuanto a diversidad y variabilidad. Se ha reportado la variabilidad de este banco de germoplasma (López-López, Barrientos-Priego, & Ben-Ya’acov, 1999), así como su potencial (Ben-Ya’acov & Barrientos, 2003) y la caracterización molecular de algunas accesiones con amplificación aleatoria de ADN polimórfico (RAPD, por sus siglas en inglés) (Reyes-Alemán, Valadez-Moctezuma, Simuta-Velázco, Barrientos-Priego, & Gallegos-Vázquez, 2013), marcadores moleculares ISSR (Reyes-Alemán, Valadez-Moctezuma, & Barrientos-Priego, 2016) y SSR (Gutiérrez-Díez, Barrientos-Priego, & Campos-Rojas, 2015), y con la secuencia de trnL-trnF de cpADN (Cabrera-Hernández et al., 2017). En estos estudios, la gran variabilidad existente en ese banco fue evidente, donde las accesiones representan sobre todo la diversidad que existe en el subgénero Persea.
El conocimiento de las relaciones filogenéticas del subgénero Persea con el Eriodaphne es importante para tomar decisiones en relación al manejo y organización de bancos de germoplasma, guiar futuras colectas y para definir acciones con respecto al mejoramiento genético.
El género Persea L. (Lauraceae) está formado por cerca de 85 especies distribuidas en América (Barrientos-Priego, Muñoz-Pérez, Borys, & Martínez-Damián, 2015), aunque se han descrito algunas especies nuevas (Lorea-Hernández, 2002; van der Werff, 2002) y probablemente ya haya más de 100. El género está distribuido desde el sur de los Estados Unidos (Persea borbonia [L.] Spreng) hasta Chile (Persea lingue Ruiz & Pavon), una especie en las Islas Canarias (P. indica [L.] Spreng.) y probablemente algunos representantes en el sur de Asia (Barrientos-Priego et al., 2015). No obstante, es controvertido si se debe considerar que Persea incluye especies de Asia, ya que los resultados sugieren que Persea es estrictamente americano (Li et al., 2011). El género se divide en los subgéneros Persea y Eriodaphne (Kopp, 1966); el primero tiene frutos conocidos como aguacates reales (~ 5 a 20 cm) y el segundo aguacates muy pequeños conocidos como “aguacatillos” (< 5 cm).
Dentro del subgénero Persea, P. americana Mill. es la especie más estudiada, principalmente por su importancia como recurso alimenticio humano y su alto contenido de aceite. Por estas razones, y considerando la compatibilidad del injerto entre las especies, se ha explorado la posibilidad de utilizar especies del subgénero Eriodaphne como portainjerto de P. americana para mejorar la resistencia a Phytophthora cinnamomi Rands.; sin embargo, resultados infructuosos revelaron incompatibilidad vegetativa entre las especies de ambos subgéneros (Frolich, Schroeder, & Zentmyer, 1958).
Existe gran controversia sobre el origen monofilético del género Persea, lo cual indica que los estudios filogenéticos basados en caracteres morfológicos no son concluyentes (Rohwer et al., 2009), y los subgéneros Persea y Eriodaphne podrían ser reconocidos como géneros independientes. No obstante, un estudio reciente de Li et al. (2011) muestra nuevamente a Persea como monofilético, si se incluye a Apollonias y se excluyen algunas especies aberrantes. Varios estudios de la familia Lauraceae, basados en datos moleculares, brindan información sobre la filogenia de Persea (Chanderbali, van der Werff, & Renner, 2001); sin embargo, la inclusión de pocas especies y especímenes hizo que los resultados no fueran informativos para el clado Persea-Eriodaphne. El subgénero Eriodaphne se ha estudiado secuenciando fragmentos de ADN nuclear y cloroplástico de manera más extensa por algunos autores (Chanderbali et al., 2001; Li et al., 2011; Rohwer et al., 2009), mientras que para el subgénero Persea no se ha hecho. Cabrera-Hernández et al. (2017) indicaron que otras secuencias (cloroplastos, mitocondrias y núcleos) deben estudiarse de forma concatenada para obtener una mejor resolución del subgénero Persea.
Específicamente, dentro de Persea, el análisis cladístico de Campos-Rojas, Terraza, y López-Mata (2007), el estudio filogenético ITS de Rohwer et al. (2009) y el estudio trnL-trnF de cpADN de Cabrera-Hernández et al. (2017) podrían separar en diferentes clados a las especies del subgénero Persea y Eriodaphne, apoyando la hipótesis de un origen polifilético del género Persea, y proporcionar una explicación de la incompatibilidad vegetativa (Frolich et al., 1958) y gamética (Lahav & Lavi, 2013) entre los dos subgéneros. Sin embargo, aún existe polémica sobre este tema, debido a que las relaciones filogenéticas entre los dos subgéneros son muy complejas (Kopp, 1966), y hasta ahora no hay suficiente evidencia de datos de ADN molecular para la separación de los dos subgéneros de Persea.
En varias familias de angiospermas, las secuencias de ADN de regiones codificantes, espaciadores intergénicos y espaciadores transcritos internos del cloroplasto, las mitocondrias y el núcleo se han utilizado en forma concatenada para obtener un mejor entendimiento de las relaciones filogenéticas de los taxa analizados. Entre los genes más utilizados, a partir de ADN de cloroplastos, se encuentran: rbcL (Kress & Erickson, 2007), ndhF (Beilstein Nagalingum, Clements, Manchester, & Mathews, 2010), matK, rpoC1 (Chase et al., 2007) y la región espaciadora intergénica trnH- psbA (Dong, Liu, Yu, Wang, & Zhou, 2012). Además, se han considerado fragmentos de ADN mitocondrial como el gen atp4 (Duminil, Pemonge, & Petit, 2002) y el gen nuclear 18S rARN. Con estos análisis, es evidente que se requiere la información de diferentes genes de ADN de varias especies de Persea para reconstruir la historia filogenética de este género. Por esta razón, el presente estudio tiene como objetivo analizar las relaciones filogenéticas dentro del género Persea, con énfasis en el subgénero Persea, utilizando la máxima parsimonia e inferencia bayesiana con la secuencia de ocho fragmentos diferentes de ADN nuclear, cloroplástico y mitocondrial.
Materiales y métodos
Material vegetal
Se obtuvo material vegetal de 35 especímenes del género Persea, 29 de subgénero Persea, cinco de subgénero Eriodaphne y uno de Beilschmiedia anay (Blake) Kosterm, del banco de germoplasma de la Fundación Salvador Sánchez Colín-CICTAMEX, S.C. (Coatepec Harinas, México) y de especímenes depositados en el herbario de la División de Ciencias Forestales de la Universidad Autónoma Chapingo, México (CHAP). Los especímenes son de México y otros países (Cuadro 1). Las accesiones incluidas en este estudio representan prácticamente toda la diversidad (siete especies) del subgénero Persea, según la clasificación de Kopp (1966); aunque la especie Persea zentmayerii, no reconocida, no está incluida (Schieber & Bergh, 1987). En el caso de Persea americana, se incluyeron todas las razas o variedades botánicas, así como la propuesta de la cuarta raza Persea americana var. costaricensis. Además, se consideraron algunos híbridos (Cuadro 1), así como Beilschmiedia anay que se utilizó como grupo externo.
Cuadro 1 Número de accesión de la colección de la Fundación Salvador Sánchez Colín-CICTAMEX, lugar de origen y número de accesión de GenBank de las especies utilizadas en el análisis.
| Nombre de la especie | Número de accesión | Lugar de origen | Número de accesión del GenBank | |||||||
| trnH-psbA | matK | rpoC1 | cox3 | 18S rARN | atp4 | rbcL | ndh | |||
| Género Beilschmiedia | ||||||||||
| Beilschmiedia anay | CG-Hu-56 | Puebla, México. | JF966434 | JF966448 | JF966482 | JF966516 | JF966550 | JF966584 | JF966618 | JF966644 |
| Género Persea | ||||||||||
| Subgénero Eriodaphne | ||||||||||
| P. chamissonis | CHAP 37473Z | Hidalgo, México | JF966426 | JF966466 | JF966500 | JF966534 | JF966568 | JF966602 | JF966636 | JF966661 |
| P. cinerascens | CH-C-30 | Michoacán, México | JF966431 | JF966452 | JF966486 | JF966520 | JF966554 | JF966588 | JF966622 | JF966670 |
| P. lingue | CH-Pl-1 | Chile | JF966423 | JF966445 | JF966479 | JF966513 | JF966547 | JF966581 | JF966615 | JF966641 |
| P. longipes | CH-G-36 | Veracruz, México | JF966424 | JF966456 | JF966490 | JF966524 | JF966558 | JF966592 | JF966626 | JF966652 |
| P. sp. ‘PR’ | CH-PR-1 | Veracruz, México | JF966432 | JF966457 | JF966491 | JF966525 | JF966559 | JF966593 | JF966627 | JF966671 |
| Subgénero Persea | ||||||||||
| Persea americana (P.a.) | ||||||||||
| P. a. var. americana | CH -CR- 28 | Costa Rica | JF966410 | JF966454 | JF966488 | JF966522 | JF966556 | JF966590 | JF966624 | JF966650 |
| P. a. var. americana | CH-G-48 | Yucatán, México | JF966396 | JF966442 | JF966476 | JF966510 | JF966544 | JF966578 | JF966612 | JF966669 |
| P. a. var. americana | CH-G-45 | Yucatán, México | JF966416 | JF966450 | JF966484 | JF966518 | JF966552 | JF966586 | JF966620 | JF966646 |
| P. a. var. americana | CH-I-6 | Veracruz, México | JF966403 | JF966458 | JF966492 | JF966526 | JF966560 | JF966594 | JF966628 | JF966653 |
| P. a. var. drymifolia x P. a. var. guatemalensis | ‘Hass’ | California, Estados Unidos | JF966409 | JF966447 | JF966481 | JF966515 | JF966549 | JF966583 | JF966617 | JF966643 |
| P. a. var. costaricensis | CH-CR-25 | Costa Rica | JF966430 | JF966438 | JF966472 | JF966506 | JF966540 | JF966574 | JF966608 | JF966665 |
| P. a. var. costaricensis | CH-CR-44 | Costa Rica | JF966407 | JF966437 | JF966471 | JF966505 | JF966539 | JF966573 | JF966607 | JF966664 |
| P. a. var. drymifolia | CH-C-10 | Puebla, México | JF966395 | JF966441 | JF966475 | JF966509 | JF966543 | JF966577 | JF966611 | JF966668 |
| P. a. var. drymifolia | CH-C-47 | Michoacán, México | JF966411 | JF966462 | JF966496 | JF966530 | JF966564 | JF966598 | JF966632 | JF966657 |
| P. a. var. drymifolia | CH-C-57 | México, México | JF966397 | JF966443 | JF966477 | JF966511 | JF966545 | JF966579 | JF966613 | JF966639 |
| P. a. var. drymifolia | CH-C-63 | México, México | JF966402 | JF966453 | JF966487 | JF966521 | JF966555 | JF966589 | JF966623 | JF966649 |
| P. a. var. drymifolia | CH-Der-2 | México, México | JF966401 | JF966451 | JF966485 | JF966519 | JF966553 | JF966587 | JF966621 | JF966648 |
| P. a.var. guatemalensis | CH-G-7 S2 | Chiapas, México | JF966413 | JF966464 | JF966498 | JF966532 | JF966566 | JF966600 | JF966634 | JF966659 |
| P. a. var. guatemalensis | CH-G-11 S1 | Chiapas, México | JF966412 | JF966463 | JF966497 | JF966531 | JF966565 | JF966599 | JF966633 | JF966658 |
| P. a. var. guatemalensis | CH-GU-5 | Guatemala | JF966417 | JF966455 | JF966489 | JF966523 | JF966557 | JF966591 | JF966625 | JF966651 |
| P. a. var. guatemalensis | CH-GU-6 | Guatemala | JF966399 | JF966449 | JF966483 | JF966517 | JF966551 | JF966585 | JF966619 | JF966645 |
| P. floccosa | CH-I-3 | Veracruz, México | JF966406 | JF966435 | JF966469 | JF966503 | JF966537 | JF966571 | JF966605 | JF966647 |
| P. a. var. drymifolia | CH-I-2 | México, México | JF966398 | JF966444 | JF966478 | JF966512 | JF966546 | JF966580 | JF966614 | JF966640 |
| P. nubigena | CH-G-76 | Chiapas, México | JF966414 | JF966467 | JF966501 | JF966535 | JF966569 | JF966603 | JF966637 | JF966662 |
| P. nubigena | CH-I-4 | Israel | JF966425 | JF966459 | JF966493 | JF966527 | JF966561 | JF966595 | JF966629 | JF966654 |
| P. parvifolia | CH-Ve-2 | Veracruz, México | JF966408 | JF966446 | JF966480 | JF966514 | JF966548 | JF966582 | JF966616 | JF966642 |
| P. schiedeana | CH-Der-1 | Veracruz, México | - | JQ352803 | - | - | - | - | - | - |
| P. schiedeana | CH-Gu-1 | Guatemala | JF966420 | JF966440 | JF966474 | JF966508 | JF966542 | JF966576 | JF966610 | JF966667 |
| P. schiedeana | CH-H-5 | Honduras | JF966404 | JF966460 | JF966494 | JF966528 | JF966562 | JF966596 | JF966630 | JF966655 |
| P. schiedeana | CH-H-7 | Honduras | JF966418 | JF966465 | JF966499 | JF966533 | JF966567 | JF966601 | JF966635 | JF966660 |
| P. schiedeana x P. a. var. guatemalensis | CH-C-62 | Guatemala | JF966405 | JF966461 | JF966495 | JF966529 | JF966563 | JF966597 | JF966631 | JF966656 |
| P. steyermarkii | CH-G-Ch1 | Chiapas, México | JF966429 | JF966439 | JF966473 | JF966507 | JF966541 | JF966575 | JF966609 | JF966666 |
| P. tolimanensis | Mv1 | Chiapas, México | JF966433 | JF966468 | JF966502 | JF966536 | JF966570 | JF966604 | JF966638 | JF966663 |
| P. sp. ‘Freddy 4’ | CH-CR-29 | Costa Rica | JF966428 | JF966436 | JF966470 | JF966504 | JF966538 | JF966572 | JF966606 | JF966672 |
ZMaterial vegetal tomado de especímenes del herbario de la División de Ciencias Forestales de la Universidad Autónoma Chapingo (CHAP).
Extracción, amplificación y secuenciación de ADN
El ADN se extrajo de ~ 50 a 100 mg de hojas previamente secadas en sílica gel. En algunos casos, se usaron hojas de especímenes del herbario. El ADN genómico se extrajo por el método basado en bromuro de hexadeciltrimetilamonio (CTAB, por sus siglas en inglés) (Gambino, Perrone, & Gribaudo, 2008). Al final del procedimiento, el ADN se purificó con columnas Qiaquick (Qiagen®, E.U.A), siguiendo las instrucciones del fabricante. La calidad y la cantidad del ADN se evaluaron con un espectrofotómetro NanoDrop® ND-1000. La amplificación de cada uno de los ocho fragmentos se realizó en un volumen total de 25 μL que contenía: 50 a 100 ng de ADN, 200 μM de mezcla dNTPs, 1X GoTaq® Flexi Reaction Buffer incoloro (Promega, E.U.A.), 20 pM de iniciadores específicos (Cuadro 2), 2.5 mM de MgCl2 y 2 U de ADN GoTaq® Flexi Polymerasa (Promega, E.U.A.). Los programas de amplificación consistieron en un ciclo de desnaturalización inicial de 4 min a 94 °C, seguido de 35 ciclos de 45 s a 94 °C, 1 min a temperatura de fusión específica (Cuadro 2), 1 min a 72 °C y una extensión de 5 min a 72 °C. Las reacciones de amplificación se realizaron en un termociclador GeneAmp® PCR System 9700 (Applied Biosystems, E.U.A.).
Cuadro 2 Iniciadores utilizados en la amplificación y secuenciación de ADN mitocondrial, nuclear y cloroplástico.
| Locus / Segmento | Nombre | Secuencia 5’-3’ | Tf (°C) | Referencia |
|---|---|---|---|---|
| nz 18S rARN | NS1 | GTAGTCATATGCTTGTCTC | 56 | White, Bruns, Lee, & Taylor (1990) |
| NS4 | CTTCCGTCAATTCCTTTAAG | 56 | White et al. (1990) | |
| NS5 | AACTTAAAGGAATTGACGGAAG | 56 | White et al. (1990) | |
| NS8 | TCCGCAGGTTCACCTACGGA | 56 | White et al. (1990) | |
| cp rpoC1 | 1f | GTGGATACACTTCTTGATAATGG | 56 | Ford et al. (2009) |
| 4r | TGAGAAAACATAAGTAAACGGGC | 56 | Ford et al. (2009) | |
| cp trnH-psbA | trnH2 | CGCGCATGGTGGATTCACAATCC | 51 | Tate & Simpson (2003) |
| psbAF | GTTATGCATGAACGTAATGCTC | 51 | Tate & Simpson (2003) | |
| cp rbcL | 1f | ATGTCACCACAAACAGAAAC | 56 | Olmstead, Michaels, Scott, & Palmer (1992) |
| 724r | TCGCATGTACCTGCAGTAGC | 56 | Fay, Swensen, & Chase (1997) | |
| cp ndhF | 389f | CTGCBACCATAGTMGCAGCA | 59 | Este estudio |
| 461r | GATTRGGACTTCTRSTTGTTCCGA | 59 | Este estudio | |
| cp matK | 1326R | TCTAGCACACGAAAGTCGAAGT | 48 | Schmitz-Linneweber et al. (2001) |
| 390F | CGATCTATTCATTCAATATTTC | 48 | Schmitz-Linneweber et al. (2001) | |
| mt atp4 | Orf1 | AAGACCRCCAAGCYYTCTCG | 50 | Duminil et al. (2002) |
| Orf2 | TTGCTGCTATTCTATCTATT | 50 | Duminil et al. (2002) | |
| mt cox3 | Cox3r | CTCCCCACCAATAGATAGAG | 51 | Duminil et al. (2002) |
| Cox3f | CCGTAGGAGGTGTGATGT | 51 | Duminil et al. (2002) |
zn: ADN del genoma nuclear; cp: ADN del genoma del cloroplasto; mt: ADN del genoma mitocondrial; Tf: temperatura de fusión.
Los fragmentos de ADN amplificados se visualizaron en un gel de agarosa al 1.2 % teñido con bromuro de etidio. Los productos de la reacción en cadena de la polimerasa (PCR, por sus siglas en inglés) se limpiaron usando el kit de purificación de PCR en columnas Qiaquick® (Qiagen, E.U.A), siguiendo las instrucciones proporcionadas por el fabricante. Los productos de PCR se secuenciaron directamente usando los mismos iniciadores (Cuadro 2) en un sistema de secuenciación automatizada en Macrogen Inc., Corea del Sur. Las secuencias se editaron y ensamblaron con el programa BioEdit versión 7.0.9.0 (http://www.mbio.ncsu.edu/BioEdit/bioedit.html).
Alineación de secuencias
Las 34 secuencias obtenidas de los espaciadores intergénicos trnH-psbA, ndhF, rbcL, rpoC1, 18S rARN, cox3 y genes atp4, y 35 del gen matK (Cuadro 2), se alinearon con MUSCLE versión 3.8 (Edgar, 2004). Adicionalmente, se alinearon 16 secuencias de matK con 36 secuencias descargadas del GeneBank (http://ncbi.nlm.nih.gov): dos de Persea y 18 de los géneros estrechamente relacionados (Sassafras, Litsea, Lindera, Ocotea, Cinnamomum, Nectandra, Actinodaphne, Parasassafras, Sinosassafras, Neolitsea, Iteadaphne, Endlicheria, Aniba, Laurus, Umbellularia, Alseodaphne, Phoebe y Machilus). Posteriormente, se construyeron manualmente dos supermatrices, la primera con las secuencias de ADN del cloroplasto (ndhF + rbcL + matK + rpoC1 + trnH-psbA) y la segunda con las ocho.
Análisis filógenetico
Las 52 secuencias alineadas de matK y las dos supermatrices mencionadas anteriormente se analizaron con los métodos de máxima parsimonia (MP), utilizando el programa PAUP ver. 4.0b10 (Swofford, 2001), e inferencia bayesiana (IB), usando MrBayes ver. 3.1.2 (Ronquist & Huelsenbeck, 2003). Los genes mitocondriales y los datos de rADN nuclear no se analizaron por separado, ya que no mostraron suficientes caracteres informativos. En cada análisis de MP, todos los caracteres se ponderaron por igual y los vacíos se consideraron como datos faltantes. Se obtuvo un conjunto de árboles más parsimoniosos de los diferentes conjuntos de datos mediante búsquedas heurísticas de 1,000 repeticiones con adición de secuencias escalonadas aleatorias, intercambio de ramas con bisección-reconexión de árbol (BRA), opción “MulTrees” activada en “effect” y guardando 10 árboles de cada adición de secuencia aleatoria. La robustez de los clados se estimó mediante un análisis de bootstrap (BS) con 1,000 repeticiones, adición de secuencia simple, intercambio de BRA y guardando solo 10 árboles por réplica para reducir el tiempo empleado en el intercambio de un gran número de árboles subóptimos. La IB se realizó con el modelo GTR + G y dos réplicas independientes de cuatro cadenas, con un máximo de 10 millones de generaciones y árboles muestreados cada 100 generaciones.
Resultados
Características de las alineaciones de secuencia
Se obtuvieron en total 273 secuencias de ndhF, rbcL, matK, rpoC1, trnH-psbA, 18S rARN, atp4 y cox3, y se depositaron en el GenBank con los números de accesión JF966395-JF966399, JF966401-JF966414, JF966416-JF966418, JF966420, JF966423-JF966426, JF966428-JF966672 y JQ352803 (Cuadro 1). La alineación trnH-psbA tuvo la variación más alta, con 32 sitios informativos de parsimonia (IP, 6.44 %) y 67 sitios variables (SV, 13.48 %) (Cuadro 3). Los genes mitocondriales atp4 y cox3 tuvieron la menor variación, con 0 a 1 sitios IP, y 0.18 y 0.43 % de SV, respectivamente (Cuadro 3); a pesar de los pocos sitios informativos obtenidos, se decidió incluirlos. Beilschmiedia anay CG-Hu-56 tuvo la secuencia más divergente de los ocho segmentos, con variación de 0 a 4 % en comparación con las secuencias de P. americana. B. anay CG-Hu-56 se utilizó como grupo externo en el análisis filogenético.
Cuadro 3 Descripción de las alineaciones de secuencia de 34 materiales del género Persea y uno de Beilschmiedia anay.
| Locus / Segmento | Longitud de alineación (bp) | RC z | RNC | IP (%) | SC (%) | SV (%) | S | MFE |
|---|---|---|---|---|---|---|---|---|
| n 18S rARN | 1748 | 0 | 1748 | 6 (0.34) | 1719 (98.34) | 29 (1.69) | 23 | 2 |
| cp rpoC1 | 599 | 599 | 0 | 2 (0.33) | 577 (96.33) | 22 (3.67) | 20 | 2 |
| cp trnH-psbA | 497 | 98 | 399 | 32 (6.44) | 428 (86.12) | 67 (13.48) | 41 | 5 |
| cp rbcL | 1481 | 1428 | 53 | 10 (0.67) | 1390 (93.86) | 91 (6.14) | 81 | 4 |
| cp ndhF | 739 | 739 | 0 | 4 (0.54) | 707 (95.67) | 32 (4.33) | 28 | 0 |
| cp matK | 909 | 909 | 0 | 7 (0.77) | 866 (95.27) | 43 (4.73) | 36 | 1 |
| mt atp4 | 507 | 507 | 0 | 1 (0.20) | 501 (99.82) | 6 (1.18) | 5 | 0 |
| mt cox3 | 695 | 695 | 0 | 0 (0.00) | 692 (99.57) | 3 (0.43) | 3 | 0 |
| matK+rbcL+ndhF+rpoC1+ trnH-psbA | 4236 | 3773 | 463 | 55 (1.30) | 3965 (93.60) | 261 (6.16) | 206 | 12 |
| 18S rARN +cox3+atp4+matK+ rbcL+ ndhF+rpoC1+trnH-psbA | 7183 | 4983 | 2200 | 62 (0.86) | 6874 (95.69) | 299 (4.16) | 237 | 14 |
zRC: región codificada; RNC: región no codificante; IP: sitios informativos de parsimonia; SC: sitios conservados; SV: sitios variables; S: sitios tipo singleton; MFE: mutaciones fijas exclusivas de Eriodaphne.
Análisis filógenetico de matK
Se realizó un gran análisis filogenético con matK. Para colocar los subgéneros Persea y Eriodaphne dentro de la familia Lauraceae, en el análisis se incluyeron representantes de 18 géneros estrechamente relacionados. Tanto los enfoques IB como los de MP resultaron en topologías bastante congruentes con respecto al subgénero Eriodaphne y el clado Litsea-Ocotea. Aunque las especies del subgénero Persea se agruparon con una probabilidad posterior (PP) débil en IB, el árbol de consenso de la regla de mayoría del BS de MP no es compatible con este clado (Figura 1). La MP y IB recuperaron el subgénero Eriodaphne y el clado Litsea-Ocotea con BS débil y valores fuertes de PP; los BS para estos clados fueron 52 y 66 %, y los valores de IB fueron 86 y 96 %, respectivamente. Dentro del clado Eriodaphne, ambos análisis soportaron el subclado P. lingue-P. longipes, con 63 y 100 % de BS y PP, respectivamente. Al igual que el anterior, en Litsea-Ocotea ambos análisis respaldaron la formación de ocho subclados diferentes, principalmente con especies del mismo género, con valores de 63 a 98 % en BS y de 71 a 100 % en PP (Figura 1). Adicionalmente, se observa que Beilschmiedia anay JF966448 y Machilus rimosa AB259098 están separadas del núcleo principal (100 % PP).

Figura 1 Filograma de consenso de la regla de mayoría bayesiana del 50 % resultante del análisis de secuencias parciales del gen matK de Persea y otros géneros de Lauraceae. Las probabilidades posteriores se indican arriba de los nodos, los valores de apoyo de bootstrap de máxima parsimonia (donde 50 %) aparecen debajo de los nodos. En el análisis de parsimonia se obtuvieron 133 árboles igualmente parsimoniosos con una longitud de 121 pasos, y un índice de consistencia de 0.88, índice de homoplasias de 0.12 e índice de retención de 0.88.
Análisis de las secuencias de cloroplastos concatenados
El análisis filogenético de las cinco secuencias de cloroplastos se realizó con secuencias de 34 accesiones de plantas diferentes, con miembros de los subgéneros Persea y Eriodaphne, más uno de Beilschmedia anay. Los análisis de IB y MP resultaron en topologías bastante congruentes (Figura 2). Los análisis recuperaron dos clados principales, subgéneros Eriodaphne y Persea, con valores de BS/PP bien soportados (88/100 %) y valores moderados (82/84 %), respectivamente. Lo anterior indica que los caracteres informativos de parsimonia adicional, de las otras secuencias de cloroplastos, pueden haber mejorado la señal filogenética.

Figura 2 Árbol de consenso de la regla de mayoría bayesiana del 50 % resultante del análisis de la concatenación de las cinco secuencias de cloroplastos matK+rbcL+ndhF+rpoC1+trnH-psbA de Persea y Beilschmiedea anay (Lauraceae). Las probabilidades posteriores se indican arriba de los nodos, los valores de apoyo de bootstrap de máxima parsimonia (donde 50 %) aparecen debajo de los nodos. En el análisis de parsimonia se obtuvieron 160 árboles igualmente parsimoniosos con una longitud de 311 pasos, y un índice de consistencia de 0.87, índice de homoplasias de 0.13 e índice de retención de 0.82.
Por otro lado, los cinco segmentos de cloroplastos presentaron 261 SV, con 22 de rpoC1 y 91 de rbcL; del total, 55 son sitios IP, de los cuales 32 corresponden a trnH-psbA (Cuadro 3). Es importante notar la presencia de 12 mutaciones fijas en los cinco segmentos del subgénero Eriodaphne, lo que ha llevado a la formación de un clado muy sólido (Cuadro 3).
Dentro de las cinco accesiones del clado del subgénero Eriodaphne, la IB soporta dos grupos en el árbol de consenso de la regla de mayoría MP-BS, aunque solo el clado de Persea chamissonis-Persea sp. ‘PR’ tiene un soporte débil de 61 %. Este clado también se apoyó en el análisis matK. Dentro del clado Persea, hubo politomía basal de dos accesiones de Persea americana (var. americana, CH-G-45 de Yucatán, México y var. guatemalensis CH-G-11 S1 de Chiapas, México), de Persea parvifolia (CH-Ve-2 de Veracruz, México) y un clado que comprende el resto de las accesiones (Figura 2). En este subclado, el árbol IB mostró cinco clados; dos de ellos apoyados fuertemente, uno con todas las accesiones de Persea americana var. drymifolia y otro con Persea nubigena CH-I-4, Persea steyermarkii CH-G-Ch1 y P. tolimanensis Mv1; uno con apoyo débil, otro insignificante y uno que contiene el único Persea floccosa CH-I-3 (Figura 2).
Análisis de las ocho secuencias concatenadas
El análisis filogenético de las ocho secuencias se realizó con accesiones de plantas de 29 miembros del subgénero Persea, cinco del subgénero Eriodaphne y una de Beilschmiedia anay. Los análisis IB y MP resultaron en topologías bastante congruentes (Figura 3); en general, muy similares al árbol IB y MP de las secuencias de cloroplastos concatenados. Los valores obtenidos de los subgéneros Eriodaphne y Persea en BS/PP fueron ligeramente superiores a los anteriores (94/100 % y 84/86 %, respectivamente; Figura 3). La adición de los genes 18S rARN, cox3 y atp4 proporcionó 38 SV más, de los cuales siete son sitios IP (Cuadro 3); sin embargo, esta información no fue capaz de mejorar significativamente la señal filogenética. Las mutaciones fijas de Eriodaphne aumentaron de 12 a 14 mediante la adición del gen 18S rARN (Cuadro 3).
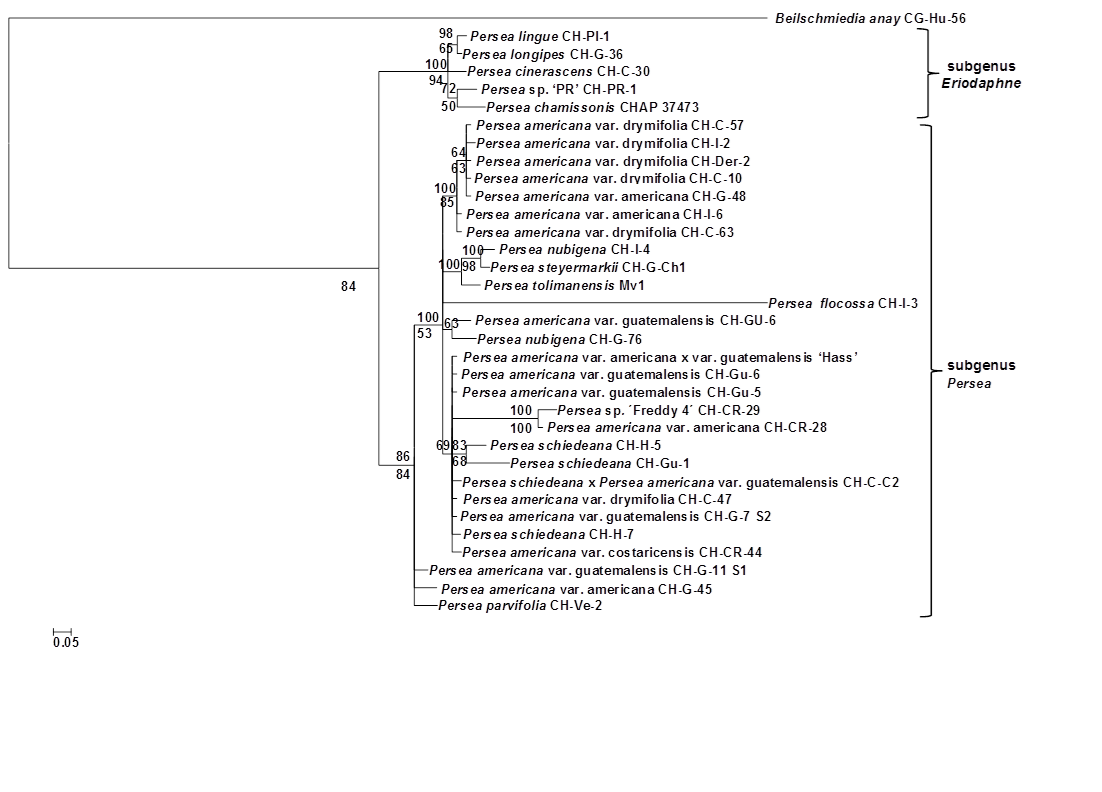
Figura 3 Filograma de consenso de la regla de mayoría bayesiana del 50 % resultante del análisis de la concatenación de 18S rARN + cox3+atp4+matK+rbcL+ndhF+rpoC1+trnH-psbA de Persea y Beilschmiedea anay (Lauraceae). Las probabilidades posteriores se indican arriba de los nodos, los valores de apoyo de bootstrap de máxima parsimonia (donde 50 %) aparecen debajo de los nodos. En el análisis de parsimonia se obtuvieron 264 árboles igualmente parsimoniosos con una longitud de 355 pasos, y un índice de consistencia de 0.87, índice de homoplasias de 0.13 e índice de retención de 0.81.
Discusión
Persea es uno de los géneros más complejos de las Lauraceae. Análisis filogenéticos anteriores al gen matK (Chanderbali et al., 2001; Rohwer, 2000; Rohwer et al., 2009) han demostrado que Persea es un grupo monofilético profundamente anidado dentro de las Lauraceae, cerca de los complejos Litsea y Ocotea. En análisis previos, como el árbol filogenético trnL-trnF / trnH-psbA de Chanderbali et al. (2001), ambos subgéneros de Persea se agrupan en el mismo clado relacionado con Machilus thunbergii y Alseodaphne semecarpifolia. En la filogenia ITS de Chanderbali et al. (2001), las tres especies del subgénero Eriodaphne formaron un pequeño clado (97 % BS), con Persea americana como su inmediato grupo hermano y varias otras especies, principalmente asiáticas del grupo Persea. Sin embargo, los pocos especímenes analizados de los dos subgéneros no permitieron resolver las relaciones dentro del grupo Persea.
Por su parte, Rohwer et al. (2009) utilizaron secuencias ITS de varios géneros de la familia y encontraron que las especies de Machilus y los subgéneros Persea y Eriodaphne se agrupan por separado entre sí. En el presente estudio, aunque el gen matK mostró un grado bajo de divergencia en las secuencias analizadas, las filogenias IB y MP podrían establecer al subgénero Eriodaphne en un clado independiente, separado de especies del subgénero Persea y de los otros géneros analizados. En este sentido, Rohwer (2000) también encontró niveles bajos de divergencia dentro de las secuencias de matK en Lauraceae (9.7 %) y menos de 1 % dentro del género Persea.
Aunque las regiones trnH-psbA y rbcL son más variables que matK (Cuadro 3), no se seleccionaron para investigar la posición de Persea dentro de las Lauraceae debido a que trnH-psbA tiene dos áreas sujetas a inversiones frecuentes que no se analizaron en este estudio. Además, los árboles filogenéticos de la rbcL (datos no mostrados) tuvieron las mismas topologías que los árboles de matK.
Los árboles obtenidos del análisis de secuencias de cloroplastos y las ocho concatenadas son muy similares debido a los 55 sitios de IP de las secuencias de cloroplastos, por lo que fueron los más útiles para la reconstrucción filogenética de los clados, especialmente para el subgénero Persea. Por otro lado, los genes mitocondriales y nuclear 18S rARN solo contribuyeron a la separación de dos accesiones de Persea schiedeana (CH-H-5 y CH-Gu-1), aunque con un apoyo moderado.
En Eriodaphne todas las especies consideradas se resolvieron completamente, pero en el subgénero Persea el análisis no logró separar Persea americana de todas las especies, especialmente de Persea schiedeana. Esta última ha sido encontrada en un estudio de germoplasma de aguacate y especies adicionales de subgénero Persea con marcadores ISSR (Reyes-Alemán et al., 2016). El nivel de variabilidad genética del aguacate, a pesar de tener un sistema de polinización cruzada, no es considerado excepcionalmente alto en comparación con las estimaciones que se han hecho con especies de frutos de clima templado (Chen, Morrel, de la Cruz, & Clegg, 2008); lo cual parece haber sido encontrado en parte en el presente estudio.
Persea parvifolia L. O. Williams (Persea pallescens [Mez] Loera-Hernández) es un arbusto con brotes delgados, hojas pequeñas y estrechas de forma obovada a elíptica, y frutos pequeños (Figura 4) que fue descrito por primera vez por L. O. Williams (1977) y no fue considerado por van der Werff (2002) como un subgénero de la especie Persea. P. parvifolia es una de las especies más ancestrales del clado del subgénero Persea, por lo que podría considerarse un buen candidato para ser la especie que dio origen al aguacate; sin embargo, no se ha resuelto con otros dos individuos de P. americana, que también tienen una secuencia conservada, por lo que podrían ser formas primitivas de aquellas razas. Para poder sustentar esto, se necesitan más individuos de esta especie para un análisis posterior, así como otras fuentes de P. americana y de P. parvifolia.

Figura 4 Rama y fruto de Persea parvifolia L. O. Williams (Persea pallescens [Mez] Loera-Hernández).
Se ha indicado que P. nubigena, P. steyermarkii y P. floccosa, aunque podrían estar separadas de P. americana por polimorfismos en la longitud de los fragmentos de restricción (RFLP, por sus siglas en inglés) se consideran solo variantes de P. americana (Furnier, Cummings, & Clegg, 1990). No obstante, los resultados muestran que algunas de estas especies se agrupan, es el caso de P. nubigena, P. steyemarkii y P. tolimanensis, las cuales se considera que contribuyen en la ascendencia de P. americana var. guatemalensis (Schieber & Bergh, 1987), aunque esto parece no corresponder con los hallazgos de este trabajo.
Con respecto a P. americana, un clado bien apoyado que incluye cinco accesiones de la raza Mexicana (P. americana var. drymifolia) junto con dos Antillanos (P. americana var. americana) indica que están relacionadas de manera muy cercana. Se puede suponer que las dos últimas accesiones no son completamente puras y que pueden tener características genéticas de la raza Mexicana. Por el contrario, un aparente conflicto entre los datos fenotípicos y genotípicos puede ayudar a ajustar la información de pedigrí (Ashworth & Clegg, 2003), y ser utilizado para reclasificar las accesiones en el banco de germoplasma como posibles híbridos. Lo anterior, también se aplica a otro clado que agrupó accesiones de la raza Guatemalteca, posiblemente híbrida (P. americana var. costaricensis) y una P. schiedeana de Honduras. Esta última también se ha analizado con marcadores DFP y SSR, y no se encontraron patrones únicos de ADN que pudieran ayudar a caracterizar las tres razas de P. americana y las tres accesiones de P. schiedeana (Mhameed et al., 1997); lo cual concuerda con el subclado que agrupa a dos P. schiedeana, una de Honduras y otra de Guatemala. Mientras que en el otro subclado se juntaron dos accesiones de Costa Rica, una no clasificada (‘Freddy 4’) y una P. americana var. americana (CH-CR-28), que probablemente sea el subclado de la raza Antillana.
El complejo legado de mejora del aguacate antiguo y reciente ha dejado una profusión de afinidades inciertas de genotipos con límites raciales difusos (Ashworth & Clegg, 2003). En este sentido, otros factores pueden tener un papel, incluyendo la posibilidad de eventos de hibridación remota (Buffer & Ben- Ya'acov, 1992) o una fecha más reciente para la diferenciación racial de lo que se pensaba anteriormente (Ashworth & Clegg, 2003).
Se debe considerar que, aunque los análisis de las ocho secuencias concatenadas separan a ambos subgéneros de Persea, la variación de las ocho secuencias es baja, 4.16 % de los SV y 0.86 % de los sitios IP (Cuadro 3). Esto se reportó para trnH-psbA (Chanderbali et al., 2001) y matK (Rohwer, 2000) de la familia Lauraceae, pero no para las otras secuencias. Por lo tanto, es necesario encontrar secuencias que muestren mayor variación, que permita mejorar la resolución de las relaciones filogenéticas dentro del subgénero Persea. Un candidato adecuado puede ser la región nuclear ITS, que tiene 33 % de sitios IP para muchas accesiones de Lauraceae (Rohwer et al., 2009); sin embargo, algunas accesiones de Persea tienen la desventaja de ser difícil de amplificar, secuenciar y alinear debido a que presentan demasiados indeles. Liu, Chen, Song, Zhang, y Chen (2012) encontraron que la región ITS2 produjo una tasa baja de éxito en la amplificación y secuenciación directas por PCR en especies de Lauraceae, además, tampoco es adecuada para ser el código de barras de ADN de la familia.
Con base en la hipótesis de un origen monofilético del género Persea, los resultados de este trabajo sugieren parcialmente que este género no es un grupo monofilético; por lo que, se podría pensar que los subgéneros Persea y Eriodaphne deberían reconocerse como géneros independientes. Esto concuerda con el análisis de Rohwer et al. (2009), donde Persea no parece ser monofilético, pues parece estar relacionado más cercanamente con Phoebe y Alseodaphne que con el subgénero Eriodaphne.
Conclusiones
Las ocho secuencias concatenadas separaron a ambos subgéneros (Persea y Eriodaphne) en dos clados diferentes, donde se encontraron 14 mutaciones fijas en las especies estudiadas del subgénero Eriodaphne, lo cual respalda la hipótesis de géneros independientes. En el subgénero Persea, las secuencias concatenadas utilizadas no lograron separar a Persea americana de todas las especies, especialmente de Persea schiedeana, la más distinta de este subgénero. La secuencia de la región espaciadora intergénica trnH-psbA del cloroplasto mostró la mayor variación y contenido de sitios informativos, mientras que las secuencias de rADN mitocondrial y nuclear no fueron informativas.
Agradecimientos
Al Consejo Nacional de Ciencia y Tecnología (CONACYT-México) por la beca de maestría otorgada a la primera autora. Este estudio fue financiado por el proyecto FRU-AGU-10-01 del Sistema Nacional de Recursos Fitogenéticos para la Alimentación y la Agricultura (SINAREFI) y por la Fundación Salvador Sánchez Colín-CICTAMEX, S.C.
REFERENCIAS
Ashworth, V. E. T. M., & Clegg, M. T. (2003). Microsatellite markers in avocado (Persea americana Mill.): Genealogical relationships among cultivated avocado genotypes. Journal of Heredity, 94(5), 407-415. doi: 10.1093/jhered/esg076 [ Links ]
Barrientos-Priego, A. F. (1999). Conservation of avocado genetic resources in Mexico. Subtropical Fruit News, 7(1), 1-2. [ Links ]
Barrientos Priego, A. F. (2010). El aguacate. Biodiversitas, 88(1), 1-7. Retrieved from http://www.biodiversidad.gob.mx/Biodiversitas/Articulos/biodiv88art1.pdf [ Links ]
Barrientos-Priego, A. F., Muñoz-Pérez, R., Borys, M. W., & Martínez-Damián, M. T. (2015). Taxonomía, cultivares y portainjertos. In: Téliz, D. & Mora, A. (Eds.), El aguacate y su manejo integrado (pp. 31-62). Montecillos, México: Biblioteca Básica de Agricultura, Colegio de Postgraduados. [ Links ]
Beilstein, M. A., Nagalingum, N. S., Clements, M. D., Manchester, S. R., & Mathews, S. (2010). Dated molecular phylogenies indicate a Miocene origin for Arabidopsis thaliana. Proceedings of the National Academy of Sciences, 107(43), 18724-18728. doi: 10.1073/pnas.0909766107 [ Links ]
Ben-Ya’acov, A., & Barrientos-Priego, A. (2003). The Persea germplasm resources potential as discovered during an international collection project (pp. 21-26). Proceedings of The V World Avocado Congress. Malaga, Spain. [ Links ]
Bost, J. B., Smith, N. J. H., & Crane, J. H. (2013). History, distribution and uses. In: Schaffer, B. A., Whiley, A. W., & Wolstenholme, B. N. (Eds.), The avocado, botany and uses (pp. 10-30). Oxfordshire, UK: CAB International Publishing. doi: 10.1079/9781845937010.0010 [ Links ]
Bufler, G., & Ben-Ya’acov, A. (1992). A study of the avocado germplasm resources, 1988-1990. 3 Ribosomal DNA repeat unit polymorphism in avocado (pp. 545-550). Proceedings of the Second World Avocado Congress, University of California, Riverside, California. [ Links ]
Cabrera-Hernández, C., Valadez-Moctezuma E., Cruz-Maya, M. E., Zelaya-Molina, L. X., Barrientos-Priego, A. F., & Reyes-Alemán, J. C. (2017). EL trnL-trnF de cpADN contribuye a la separación de los subgéneros Persea y Eriodaphne (Lauraceae; Persea) como géneros independientes. Chilean Journal of Agricultural & Animal Sciences, 33(3), 231-240. doi: 10.4067/S0719-38902017005000701 [ Links ]
Campos-Rojas, E., Terrazas, T., & López-Mata, L. (2007). Persea (avocados) phylogenetic analysis based on morphological characters: hypothesis of species relationships. Genetic Resources and Crop Evolution, 54(2), 249-258. doi: 10.1007/s10722-005-3808-x [ Links ]
Chanderbali, A. S., van der Werff, H., & Renner, S. S. (2001). Phylogeny and historical biogeography of Lauraceae: Evidence from the chloroplast and nuclear genomes. Annals of the Missouri Botanical Garden, 88(1), 104-134. doi: 10.2307/2666133 [ Links ]
Chanderbali, A. S., Albert, V. A., Ashworth, V. E., Clegg, M. T., Litz, R. E., Soltis, D. E., & Soltis, P. S. (2008). Persea americana (avocado): bringing ancient flowers to fruit in the genomics era. BioEssays, 30(4), 386-396. doi: 10.1002/bies.20721 [ Links ]
Chase, M. W., Cowan, R. S., Hollingsworth, P. M., van den Berg, C., Madriñán, S., Petersen, G., Seberg, O., Jørgsensen, T., Cameron, K. M., Carine, M., & Pedersen, N. (2007). A proposal for a standardized protocol to barcode all land plants. Taxon, 56(2), 295-299. Retrieved from https://botanica.uniandes.edu.co/investigacion/pdfs/Chase-Plant%20Barcodes.pdf [ Links ]
Chen, H., Morrel, P. L., de la Cruz, M., & Clegg, M. T. (2008). Nucleotide diversity and linkage disequilibrium in wild avocado (Persea americana Mill.). Journal of Heredity, 99(4), 382-389. doi: 10.1093/jhered/esn016 [ Links ]
Dong, W., Liu, J., Yu, J., Wang, L., & Zhou, S. (2012). Highly variable chloroplast markers for evaluating plant phylogeny at low taxonomic levels and for DNA barcoding. PloS One, 7(4), 35071. doi: 10.1371/journal.pone.0035071 [ Links ]
Duminil, J., Pemonge, M. H., & Petit, R. J. (2002). A set of 35 consensus primer pairs amplifying genes and introns of plant mitochondrial DNA. Molecular Ecology Notes, 2(4), 428-430. doi: 10.1046/j.1471-8286.2002.00263.x [ Links ]
Edgar, R. C. (2004). MUSCLE: multiple sequence alignment with high accuracy and high throughput. Nucleic Acids Research, 32(5), 1792-1797. doi: 10.1093/nar/gkh340 [ Links ]
Fay, M. F., Swensen, S. M., & Chase, M.W. (1997). Taxonomic affinities of Medusagyne oppositifolia (Medusagynaceae). Kew Bulletin, 52(1), 111-120. doi: 10.2307/4117844 [ Links ]
Ford, C. S., Ayres, K. L., Toomey, N., Haider, N., Van Alphen, S. J., Kelly, L. J., Wikström, N., Hollingsworth, P. M., Duff, R. J., Hoot, S. B., Cowan, R. S., Chase, M. W., & Wilkinson, M. J. (2009). Selection of candidate coding DNA barcoding regions for use on land plants. Botanical Journal of the Linnean Society, 159(1), 1-11. doi: 10.1111/j.1095-8339.2008.00938.x [ Links ]
Fraedrich, S. W., Harrington, T. C., Rabaglia, R. J., Ulyshen, M. D., Mayfield, A. E., Hanula, J. L., Eickwort, J. M., & Miller, D. R. (2008). A fungal symbiont of the redbay ambrosia beetle causes a lethal wilt in redbay and other Lauraceae in the southeastern United States. Plant Disease, 92(2), 215-224. doi: 10.1094/PDIS-92-2-0215 [ Links ]
Frolich, E. F., Schroeder, C. A., & Zentmyer, G. A. (1958). Graft compatibility in the genus Persea. California Avocado Society, 42, 102-105. Retrieved from http://www.avocadosource.com/CAS_Yearbooks/CAS_42_1958/CAS_1958_PG_102-105.pdf [ Links ]
Furnier, G. R., Cummings, M. P., & Clegg, M. T. (1990). Evolution of the avocados as related by DNA restriction fragment variation. Journal of Heredity, 81(3), 183-188. doi: 10.1093/oxfordjournals.jhered.a110963 [ Links ]
Gambino, G., Perrone, I., & Gribaudo, I. (2008). A rapid and effective method for RNA extraction from different tissues of grapevine and other woody plants. Phytochemical Analysis, 19(6), 520-525. doi: 10.1002/pca.1078 [ Links ]
Gutiérrez-Díez, A., Barrientos-Priego, A. F., & Campos-Rojas, E. (2015). Caracterización molecular y análisis filogenético de los subgéneros Persea y Eriodaphne (Lauraceae) (pp. 88-94). Recursos genéticos y manejo de viveros. Lima, Perú. [ Links ]
Kopp, L. E. (1966). A taxonomic revision of the genus Persea in the Western Hemisphere (Persea-Lauraceae). Memoirs of the New York Botanical Garden, 14(1), 1-120. [ Links ]
Kress, W. J., & Erickson, D. L. (2007). A two-locus global DNA barcode for land plants: the coding rbcL gene complements the non-coding trnH-psbA spacer region. PLoS One, 2(6), 508. doi: 10.1371/journal.pone.0000508 [ Links ]
Lahav, E., & Lavi, U. (2013). 4 Genetics and breeding. In: Schaffer, B. A., Whiley, A. W., & Wolstenholme, B. N. (Eds.), The avocado botany, and uses (pp. 51-85). Oxfordshire, UK.: CAB International Publishing. [ Links ]
Li, L., Li, J., Rohwer, J. G., van der Werff, H., Wang, Z. H., & Li, H. W. (2011). Molecular phylogenetic analysis of the Persea group (Lauraceae) and its biogeographic implications on the evolution of tropical and subtropical amphi-pacific disjunctions. American Journal of Botany, 98(9), 1520-1536. doi: 10.3732/ajb.1100006 [ Links ]
Liu, Z., Chen, S. L., Song, J. Y., Zhang, S. J., & Chen, K. L. (2012). Application of deoxyribonucleic acid barcoding in Lauraceae plants. Pharmacognosy Magazine, 8(29), 4-11. doi: 10.4103/0973-1296.93301 [ Links ]
López-López, L., Barrientos-Priego, A. F., & Ben-Ya’acov, A. D. (1999). Variabilidad genética de los bancos de germoplasma de aguacate preservados en el Estado de México. Revista Chapingo Serie Horticultura, 5, 19-23. doi: 10.5154/r.rchsh.1999.02.012 [ Links ]
Lorea-Hernández, F. G. (2002). La familia Lauraceae en el sur de México: diversidad, distribución y estado de conservación. Boletín de la Sociedad Botánica de México, 71, 59-70. Retrieved from http://www.redalyc.org/articulo.oa?id=57707104 [ Links ]
Mhameed, S., Sharon, D., Kaufman, D., Lahav, E., Hillel, J., Degani, C., & Lavi, U. (1997). Genetic relationships within avocado (Persea americana Mill.) cultivars and between Persea species. Theoretical and Applied Genetics, 94(2), 279-286. doi: 10.1007/s001220050411 [ Links ]
Olmstead, R., Michaels, H., Scott, K., & Palmer, J. (1992). Monophyly of the Asteridae and identification of their major lineages inferred from DNA sequences of rbcL. Annals of the Missouri Botanical Garden, 79(2), 249-265. doi: 10.2307/2399768 [ Links ]
Ploetz, R. C., Peña, J. E., Smith, J. A., Dreaden, T. J., Crane, J. H., Schubert, T., & Dixon, W. (2015). Laurel Wilt, caused by Raffaelea lauricola, is confirmed in Miami-Dade County, center of Florida's commercial avocado production. Plant Disease, 95(12), 33-44. doi: 10.1094/PDIS-08-11-0633 [ Links ]
Reyes-Alemán, J. C., Valadez-Moctezuma, E., Simuta-Velázco, L., Barrientos-Priego, A. F., & Gallegos-Vázquez, C. (2013). Distinción de especies del género Persea mediante RAPD e ISSR de ADN. Revista Mexicana de Ciencias Agrícolas, 4(4), 517-529. Retrieved from http://scielo.unam.mx/pdf/remexca/v4n4/v4n4a3.pdf [ Links ]
Reyes-Alemán, J. C., Valadez-Moctezuma, E., & Barrientos-Priego, A. F. (2016). Assessment of genetic relationship in Persea spp. by traditional molecular markers. Genetics and Molecular Research, 15(2), 1-11. doi: 10.4238/gmr.15027359 [ Links ]
Rohwer, J. G. (2000). Toward a phylogenetic classification of the Lauraceae: evidence from matK sequences. Systematic Botany, 25(1), 60-71. doi: 10.2307/2666673 [ Links ]
Rohwer, J. G., Li, J., Rudolph, B., Schmidt, S. A., van der Werff, H., & Li, H. W. (2009). Is Persea (Lauraceae) monophyletic? Evidence from nuclear ribosomal ITS sequences. Taxon , 58(4), 1153-1167. Retrieved from http://www.jstor.org/stable/27757009 [ Links ]
Ronquist, F., & Huelsenbeck, J. P. (2003). MrBayes 3: Bayesian phylogenetic inference under mixed models. Bioinformatics, 19(12), 1572-1574. doi: 10.1093/bioinformatics/btg180 [ Links ]
Schaffer, B., Wolstenholme, B. N., & Whiley, A. W. (2013). Introduction. In: Schaffer, B. A., Whiley, A. W., & Wolstenholme, B. N. (Eds.), The avocado, botany and uses (pp. 1-9). Oxfordshire, UK: CAB International Publishing . doi: 10.1079/9781845937010.0001 [ Links ]
Schieber, E., & Bergh, B. O. (1987). Persea zentmyerii: a new species from Guatemala. California Avocado Society, 71, 199-203. Retrieved from http://avocadosource.com/CAS_Yearbooks/CAS_71_1987/CAS_1987_PG_199-203.pdf [ Links ]
Schimitz-Linneweber, C., Maier, R. M., Alcaraz, J. P., Cottet, A., Herrmann, R. G., & Mache, D. R. (2001). The plastid chromosome of spinach (Spinacia oleracea): complete nucleotide sequence and gene organization. Plant Molecular Biology, 45(3), 307-315. doi: 10.1023/A:1006478403810 [ Links ]
Swofford, D. L. (2001). PAUP* Phylogenetic analysis using parsimony (*and other methods), 4.0 B5. Sunderland, USA: Sinauer Associates. Retrieved from http://citeseerx.ist.psu.edu/viewdoc/summary?doi=10.1.1.458.6867 [ Links ]
Tate, J. A., & Simpson, B. B. (2003). Paraphyly of Tarasa (Malvaceae) and diverse origins of the polyploid species. Systematic Botany, 28(4), 723-737. doi: 10.1043/02-64.1 [ Links ]
Van der Werff, H. (2002). A synopsis of Persea (Lauraceae) in Central America. Novon, 12(4), 575-586. doi: 10.2307/3393142 [ Links ]
White, T. J., Bruns, T., Lee, S., & Taylor, J. W. (1990). Amplification and direct sequencing of fungal ribosomal RNA genes for phylogenetics, In: Innis, M. A., Gelfand, D. H., Sninsky, J. J., & White T. J. (Eds.), PCR Protocols: A guide to methods and applications (pp. 315-322). New York, USA: Academic Press, Inc. [ Links ]
Williams, L. O. (1977). The avocado, a synopsis of the genus Persea, subg. Persea. Economic Botany, 31(3), 315-320. Retrieved from http://www.jstor.org/stable/4253853 [ Links ]
Recibido: 23 de Noviembre de 2017; Aprobado: 20 de Marzo de 2018











 text in
text in 


