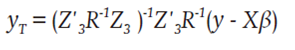Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Acessos
Acessos
Links relacionados
-
 Similares em
SciELO
Similares em
SciELO
Compartilhar
Revista Chapingo. Serie horticultura
versão On-line ISSN 2007-4034versão impressa ISSN 1027-152X
Rev. Chapingo Ser.Hortic vol.23 no.1 Chapingo Jan./Abr. 2017
https://doi.org/10.5154/r.rchsh.2016.06.021
Articles
Predicción de rendimiento y firmeza de fruto de híbridos de tomate con BLUP y RR-BLUP mediante marcadores moleculares ISSR
1Universidad Autónoma Chapingo, Instituto de Horticultura, Departamento de Fitotecnia. Carretera México-Texcoco km 38.5, Chapingo, Estado de México, C. P. 56230, MÉXICO.
En el desarrollo de híbridos de tomate, un número grande de líneas progenitoras implica un número de híbridos posibles demasiado alto, y su formación y evaluación se vuelve un problema. La magnitud de éste puede reducirse a niveles manejables con el uso de métodos de predicción del desempeño de los híbridos. En el presente estudio se compararon dos métodos basados en huellas genómicas, teoría de los modelos mixtos y en la evaluación experimental de una muestra de híbridos: 1) el mejor predictor lineal insesgado o BLUP y 2) la regresión ridge BLUP. Se realizaron 36 cruzas simples con el objetivo de determinar el número, firmeza y rendimiento total y comercial de frutos. Con 1,000 muestras aleatorias independientes de tamaños, n = 6, 12, 18, 24 y 30, se predijo el comportamiento de los 36-n híbridos restantes. Los coeficientes de correlación entre valores predichos y observados fluctuaron entre 0.25 y 0.83. El BLUP de manera consistente registró los valores más altos. En ambos métodos de predicción cuando n aumentó la magnitud de las correlaciones también se incrementó.
Palabras clave: Solanum lycopersicum L.; mejor predictor lineal e insesgado; regresión ridge BLUP; componentes de rendimiento
In the development of tomato hybrids, a large number of parental lines involves an excessively high number of possible hybrids, making their formation and evaluation problematic. The magnitude of this can be reduced to manageable levels with the use of hybrid performance prediction methods. In this study we compared two methods based on genomic fingerprints, mixed model theory and the experimental evaluation of a sample of hybrids: 1) best linear unbiased prediction or BLUP and 2) ridge regression BLUP. Thirty-six single crosses were performed with the objective of determining the number, firmness and total and commercial yield of fruits. With 1,000 size-independent random samples, n = 6, 12, 18, 24 and 30, the behavior of the remaining 36-n hybrids was predicted. The correlation coefficients between predicted and observed values ranged between 0.25 and 0.83. BLUP consistently recorded the highest values. In both prediction methods when n increased the magnitude of the correlations also increased.
Keywords: Solanum lycopersicum L.; best linear unbiased prediction; ridge regression BLUP; fruit yield components
Introducción
El manejo de la heterosis que permite la producción de variedades híbridas sobresalientes es probablemente el mayor éxito del mejoramiento genético de las plantas. El desarrollo de variedades híbridas sobresalientes, sin embargo, no es fácil: menos del 1 % de los híbridos de cruza simple de maíz (Zea mays L.) llegan a ser variedades comerciales (Bernardo, 1996), y algo similar debe suceder en otros cultivos, particularmente entre algunos que corresponden a especies autógamas, como el tomate (Solanum lycopersicum L.). Entre los obstáculos que encuentra el fitomejorador destaca la necesidad de evaluar en el campo una cantidad elevada de híbridos experimentales, que usualmente rebasa las capacidades de los programas de mejoramiento. Para enfrentar esta dificultad se han desarrollado teoría y métodos de predicción del valor genotípico de variedades que no existen físicamente. Estos métodos se basan en la formación y evaluación experimental de una muestra pequeña de cruzas, y en las coancestrías de las líneas progenitoras, estimadas con base en sus registros de pedigrí o en sus huellas genómicas (Henderson, 1985; Bernardo, 1994; Gbur et al., 2012).
En maíz se ha predicho el rendimiento de grano de cruzas simples mediante una ecuación de predicción que produce “los mejores predictores lineales insesgados” (BLUP, por sus siglas en inglés: best linear unbiased prediction) (Bernardo, 1993, 1995). Estas predicciones se basaron en marcadores moleculares RFLP´s (Bernardo, 1994, 1996) y en marcadores microsatélites (Balestre, Von Pinho, & Souza, 2010). Los coeficientes de correlación (r) entre rendimientos observados y pronosticados fluctuaron entre 0.43 y 0.76, y entre 0.55 y 0.70, respectivamente. También en maíz, Massman, Gordillo, Lorenzana, y Bernardo (2013) realizaron predicción genómica mediante regresión ridge BLUP (RR-BLUP) basada en marcadores SNP (polimorfismo de nucleótido único), con niveles de predicción similares a los de BLUP. Una característica atractiva del método RR-BLUP es que utiliza todos los marcadores moleculares y asume que cada uno de éstos representa la misma proporción de la varianza genética (Meuwissen, Hayes, & Goddard, 2001).
Aunque el tomate es la segunda especie hortícola en importancia a nivel mundial, y su mejoramiento genético en las últimas décadas se ha orientado a la producción y liberación de variedades híbridas F1, los reportes sobre la predicción de rendimiento de híbridos son pocos. Lo anterior posiblemente se deba a que el fitomejoramiento es realizado por el sector privado y en forma limitada por programas públicos. El costo de producción del tomate es muy elevado, sobre todo si se hace en invernadero. La evaluación de campo de nuevos cultivares implica inversión financiera alta. Para hacerla más productiva se requiere implementar estrategias, como la predicción de rendimiento de fruto, que permitan hacer y evaluar sólo las cruzas con mayor probabilidad de éxito.
Mirshamsi, Farsi, Shahriari, y Nemati (2008) utilizaron marcadores RAPD para estimar distancias genéticas entre progenitores y determinar la correlación entre éstas y los rendimientos de fruto de los híbridos producidos por sus cruzas. Sin embargo, los coeficientes de correlación obtenidos, por su reducido valor (r < 0.40), sugieren que este método es de poca utilidad práctica.
En mejoramiento genético de hortalizas, y en especial en tomate (pese a su gran valor metodológico), son escasos los antecedentes de predicción de caracteres de rendimiento. Por esta razón, se planeó una investigación con el propósito de predecir la firmeza, el número y el rendimiento total y comercial de frutos de cruzas simples de tomate mediante el empleo de los métodos BLUP y regresión ridge BLUP.
Materiales y métodos
La presente investigación se realizó de febrero de 2013 a noviembre de 2015 y consistió en tres etapas: 1) análisis de líneas élite de tomate con marcadores moleculares (ISSR), selección de líneas progenitoras y realización de cruzamientos dirigidos, 2) evaluación de firmeza, número y rendimiento de fruto de las cruzas simples y 3) predicción de rendimiento de cruzas mediante BLUP y RR-BLUP y validación de los métodos.
Selección de líneas progenitoras
En 2013 se sembraron 39 líneas élite F8 de tomate en charolas de poliestireno de 200 cavidades con turba como sustrato. Entre 30 y 40 días después de la siembra (dds), de 10 plántulas se obtuvieron muestras de tejido de hojas tiernas, verdes y sin daño aparente. Las hojas se lavaron con alcohol al 70 % y se secaron con toallas de papel. Se colocó 0.3 g de hoja en un mortero de porcelana, se agregó nitrógeno líquido y se molió hasta obtener un polvo fino; el cual se transfirió a un microtubo de 1.5 mL con 600 μL de amortiguador de extracción (Tris HCl 100 mM, EDTA 20 mM, NaCl 1.4 M, CTAB 2.0 % y 2-Mercaptoetanol al 0.2 %), se precalentó a 65 °C en termoblock y se agitó hasta homogenizar. Nuevamente se calentó a 65 °C durante 20 min. En seguida, se retiró del calor, dejó enfriar y centrifugó a 21,000 x g durante 20 min. La solución se transfirió a otro microtubo, se agregaron 600 μL de cloroformo: alcohol isoamílico (24:1) y se agitó durante 20 min. Posteriormente, se centrifugó a 12,000 x g por 5 min y la fase acuosa (superior) se colocó en otro tubo con 500 μL de isopropanol frío, se agitó suavemente por inversión, se mantuvo a -20 °C por 60 min para precipitar el ADN, se centrifugó a 12,000 x g durante 5 min y decantó el sobrenadante. A la pastilla se le agregaron 700 μL de solución para disolver (Tris HCl 10 mM, EDTA 1 mM, NaCl 10 mM, pH 8.0) y se dejó en refrigeración (4 °C) durante 12 h.
Una vez resuspendido el ADN, se agregaron 4 μL de ARNasa e incubó a 37 °C por 1 h. El ADN se reprecipitó con 70 μL de acetato de sodio 3 M y 700 μL de isopropanol frío, se agitó por inversión e introdujo al congelador por 2 h. Se centrifugó a 8,000 x g durante 10 min, se eliminó el sobrenadante y se lavó la pastilla con 500 μL de etanol al 70 % (grado reactivo). Nuevamente se centrifugó, se eliminó el sobrenadante y secó la pastilla a temperatura ambiente. Finalmente, ésta se disolvió con 50 μL de TE y se almacenó a 4 °C.
Para cuantificar la pureza del ADN, se empleó un espectrofotómetro modelo NanoDrop Lite (Thermo Scientific). Se prepararon soluciones de trabajo con 10 μg·μL-1 de ADN. La calidad del ADN se verificó en un gel de agarosa al 0.8 % de 4 mm de grosor y TAE al 0.5 %. En cada pozo se depositaron 10 μL de la solución de trabajo con ADN. La electroforesis se realizó por 2 h en una cámara modelo Owl A3-1 (Thermo Scientific). El gel se tiñó con solución de Bromuro de etidio en concentración de 0.5 μg·mL-1 durante 20 min y se fotografió con fotodocumentador modelo TFM-26 transilluminator (UVP).
En la amplificación y separación de fragmentos mediante la reacción en cadena de la polimerasa (PCR, por sus siglas en inglés) se emplearon 29 iniciadores ISSR (Inter Secuencias Simples Repetidas), las temperaturas de alineamiento aparecen en el Cuadro 1. La reacción de amplificación se realizó con: 5.2 μL de H2O, 10.0 μL de dNTP’s [500 μM], 2.5 μL de amortiguador [10 x], 1.5 μL de MgCl2 [50 mM], 3.0 μL de iniciador ISSR [10 ng·μL-1], 0.3 μL de enzima Taq ADN polimerasa [5 u·μL-1] y 2.5 μL de ADN [10 ng·μL-1], dado un volumen total de 25 μL.
Cuadro 1 Secuencia de iniciadores ISSR utilizados en la caracterización de la divergencia genética de 39 líneas élite de tomate, temperatura de alineamiento (Ta), número total de bandas amplificadas (TB) y polimórficas (BP) y porcentaje de polimorfismo (PP).
| Iniciador | Secuencia 5’-3’ | Ta (ºC) | TB | PB | PP (%) | Iniciador | Secuencia 5’-3’ | Ta (ºC) | TB | PB | PP (%) |
|---|---|---|---|---|---|---|---|---|---|---|---|
| 17898 B | (CA)6GT | 42 | 7 | 4 | 57.1 | PHV7 | (GTG)2(TG)6T | 58 | 2 | 1 | 50.0 |
| 17899 A | (CA)6AG | 42 | 9 | 4 | 44.4 | UBC807 | (AG)8T | 50 | 9 | 7 | 77.8 |
| Echt 5 | (AGAC)2GC | 42 | 6 | 3 | 50.0 | UBC811 | (GA)8C | 52 | 5 | 2 | 40.0 |
| ISSR01 | (CA)8AAGG | 62 | 15 | 15 | 100.0 | UBC815 | (CT)8G | 52 | 9 | 2 | 22.2 |
| ISSR02 | (CA)8AAGCT | 62 | 15 | 15 | 100.0 | UBC822 | (TC)8 ª | 50 | 3 | 0 | 0.0 |
| ISSR03 | (GA)8CTC | 58 | 17 | 17 | 100.0 | UBC827 | (AC)8G | 52 | 8 | 5 | 62.5 |
| ISSR04 | (AG)8CTC | 58 | 18 | 17 | 94.4 | UBC836 | (AG)8CTA | 52 | 11 | 9 | 81.8 |
| ISSR05 | (AC)8CTA | 56 | 10 | 8 | 80.0 | UBC844 | (CT)8AC | 54 | 9 | 8 | 88.9 |
| ISSR06 | (AC)8CTG | 58 | 12 | 11 | 91.7 | UBC845 | (CT)8AGG | 54 | 13 | 12 | 92.3 |
| ISSR07 | (AG)8CTG | 53 | 10 | 10 | 100.0 | UBC847 | (CA)8AGC | 52 | 7 | 3 | 42.9 |
| ISSR08 | (AC)8CTT | 56 | 9 | 9 | 100.0 | UBC848 | (CA)8AGG | 56 | 8 | 8 | 100.0 |
| ISSR10 | (GA)8T | 50 | 9 | 8 | 88.9 | UBC862 | (AGC)6 | 68 | 9 | 6 | 66.7 |
| LOL2 | (CT)8GC | 56 | 8 | 6 | 75.0 | UBC873 | (GACA)4 | 48 | 11 | 6 | 54.5 |
| LOL9 | (CAC)3GC | 38 | 9 | 8 | 88.9 | UBC886 | YXY(CT)7 | 52 | 10 | 8 | 80.0 |
| PHV6 | CCA(CT)3 | 57 | 8 | 3 | 37.5 | Total | 276 | 215 | 77.9 |
Y = GAC, X = GAT.
La PCR se llevó a cabo en un termociclador modelo FTC41H2D marca TECHNE, con el siguiente programa: desnaturalización inicial a 93 °C por 1 min; seguido de 40 ciclos, cada uno con 20 s a 93 °C, 60 s a la temperatura de alineación del iniciador probado y 20 s a 72 °C, y una extensión final a 72 °C por 6 min. La muestra se enfrió a 10 °C. Los productos finales se llevaron a electroforesis en gel de agarosa al 2 % con TAE 1x; posteriormente se realizó la tinción con bromuro de etidio y fotodocumentación.
A partir de la matriz de datos binarios obtenida de las huellas genómicas, se construyó el coeficiente de similitud de Jaccard (S rs ) para cada par de genotipos (Khattree & Naik, 2000). Con la matriz de disimilitud (d = 1 - S rs ), se realizó el agrupamiento mediante el método de varianza mínima de Ward con el programa Infostat. El resultado fue la formación de tres grupos de los que se eligieron dos, considerados genéticamente diferentes. A estos dos se les denominó X y Y.
Evaluación de rendimiento de fruto de cruzas intergrupales
Se seleccionaron 12 líneas élite F8 de alto rendimiento de fruto; seis pertenecientes al grupo X (líneas L5BI, L37SI, L59BI, L61BI, L65BI y L76BI) y seis al Y (líneas L6BI, L45BI, L68BI, L69BD, L80BI y L88BI). Éstas se sembraron en febrero de 2013 y se realizaron las 36 (6x6) cruzas simples (CS) directas intergrupales; debido a que no se han reportado efectos maternos en tomate, las líneas de los grupos X y Y se usaron como hembras y machos, respectivamente. Al alcanzar la madurez de consumo, los frutos se cosecharon y la semilla de cada cruza se separó y benefició.
Con el propósito de determinar el rendimiento de fruto de las CS, éstas se sembraron en febrero de 2014 y 2015, en charolas de poliestireno de 200 cavidades con turba como sustrato. El trasplante se hizo 35 dds en invernadero bajo un sistema hidropónico. Se usaron bolsas de polietileno negro con capacidad de 18 L y sustrato de espuma volcánica. La solución nutritiva utilizada fue la propuesta por Cadahia (2000) para tomate; la cantidad aplicada varió de acuerdo con la etapa fenológica y las condiciones climáticas. La densidad de plantación fue de 3.3 plantas∙m-2.
La unidad experimental consistió en dos macetas, cada una con dos plantas. Se empleó un diseño experimental de bloques completos al azar con tres repeticiones. El rendimiento de fruto se evaluó en los primeros cuatro racimos. Las variables respuesta registradas fueron: número de frutos (NF) por metro cuadrado, rendimiento de fruto total (RFT) en kg∙m-2, rendimiento de fruto comercial (RFC) en kg∙m-2 y firmeza del fruto (FF) registrada en kgf∙cm-2. Un fruto alcanzó la categoría de fruto comercial si su peso fue mayor a 100 g y no presentaba daño físico o mecánico, desorden fisiológico o afectación por plagas y enfermedades que demeritaran su apariencia. La FF se cuantificó en cuatro frutos por unidad experimental, doce días después de haber sido cosechados en la etapa de maduración rompiente y haber permanecido a temperatura ambiente. Se empleó un penetrómetro de fruta manual modelo GY-1 (Sundoo Instruments).
Predicción de rendimiento de cruzas mediante BLUP y RR-BLUP
BLUP
Se calcularon los coeficientes de coancestría (f ij ) con base en ISSR (Bernardo, 1993), de acuerdo con la expresión
donde S ij es la similaridad de Jaccard estimada con marcadores moleculares entre las líneas i y j del grupo X, Si- proporción promedio de variantes ISSR que comparte i con todas las líneas del grupo heterótico opuesto (Y) y S j- proporción promedio de variantes ISSR que comparte la línea j con todas las líneas del grupo heterótico opuesto (X). Como por definición los coeficientes de coancestría no son negativos, aquéllos que así resultaron se igualaron a cero.
El modelo para el rendimiento de fruto (y) fue (Bernardo, 1994; Massman et al., 2013):
en donde y es un vector p×1 de p rendimientos observados de las N cruzas simples, β es un vector c×1 de efectos fijos de los c ambientes de evaluación, g1 es un vector n 1×1 de efectos aleatorios de aptitud combinatoria general (ACG) de las líneas del grupo X, g 2 es un vector n 2×1 de efectos aleatorios de ACG de las líneas del grupo Y, s es un vector N×1 de los efectos aleatorios de aptitud combinatoria específica (ACE) de las N cruzas simples, e es el vector p×1 de residuos y X, Z 1, Z 2, y Z 3 son matrices cuyos elementos son 0 o 1 para denotar ausencia o presencia de efectos que relacionan a y con β, g 1, g 2 y s, respectivamente.
La covarianza (Cov) entre las CS intergrupales ij e i'j' se expresó con base en un modelo de k loci y en la simplificación que resulta de la derivación de Melchinger (1988):
donde V A(X/Y) es la varianza aditiva de los alelos i y j provenientes del grupo X o Y a través de los k loci de i’ y j’ involucrados y V D(XY) es la varianza de dominancia a través de los k loci de los pares alélicos formados cada uno por un alelo de X y uno de Y. Las estimaciones de máxima verosimilitud restringida fueron obtenidas mediante iteración a partir de sendas ecuaciones para la V E (varianza no genética), V A(X/Y) , V A(Y/X) y V D(XY) (Henderson, 1985; Bernardo, 1994; Gbur et al., 2012). Además de la predicción, las estimaciones de estas varianzas se utilizaron para obtener heredabilidades.
Los rendimientos de fruto de los híbridos se basó en la teoría de los modelos mixtos que produce predictores lineales, insesgados y de varianza mínima (BLUP). La predicción del rendimiento de híbridos se hizo con base en los rendimientos observados de los híbridos restantes, llamados híbridos predictores. De acuerdo con Bernardo (1994), el rendimiento ajustado por efectos fijos de estos híbridos (y T ) se expresó como:
donde R es una matriz diagonal y los elementos son el recíproco del número de ambientes del experimento.
Por otra parte, el vector formado con los rendimientos predichos de los híbridos desconocidos (y U ) fue calculado según la expresión:
en donde C UT es una matriz N U ×N T cuyo ij-ésimo elemento es la covarianza genética entre el i-ésimo híbrido sujeto a predicción y el j-ésimo híbrido predictor, y C TT es la matriz N T ×N T de las covarianzas de los n híbridos predictores. Los elementos de C UT y los que van fuera de la diagonal de C TT se calcularon según la expresión de la covarianza entre un híbrido desconocido y uno predictor, en términos de coancestría y varianzas genéticas.
RR-BLUP
La predicción del rendimiento de CS mediante la utilización del genoma completo (genomewide), se hizo por medio de RR-BLUP con el modelo lineal propuesto por Massman et al. (2013) para N M marcadores:
donde y, X, β y e fueron ya definidos, m
1 es un vector N
M
×1 de efectos aleatorios de ACG de los marcadores de las líneas maternas, m
2 es un vector N
M
×1 de efectos aleatorios de ACG de los marcadores de las líneas paternas y m
3 es un vector N
M
×1 de efectos aleatorios de ACE asociados a los marcadores alélicos de ambos grupos heteróticos. W
1 y W
2 son matrices cuyos elementos fueron igual a 1 o -1. Los elementos de W
3 en un locus dado fueron iguales al producto de los elementos de W
1 y W
2 en el mismo locus, según lo propuesto por Massman et al. (2013). En teoría  ,
,  ,
,  , además m
1, m
2 y m
3 son independientes; e es un vector de errores aleatorios de dimensión p×1 y
, además m
1, m
2 y m
3 son independientes; e es un vector de errores aleatorios de dimensión p×1 y  .
.
Para evaluar el método de predicción BLUP en términos de tamaño de muestra, se trabajó con conjuntos de n híbridos predictores (n = 6, 12, 18, 24 y 30), seleccionados al azar, de los 36 posibles. Para verificar la estabilidad de las predicciones, en cada número de híbridos predictores (n) se realizaron 1,000 muestreos aleatorios y se predijo el rendimiento de fruto de los 36-n híbridos restantes en cada uno de los casos. Posteriormente, para cada valor de n se calcularon las correlaciones entre los rendimientos observados y los predichos correspondientes. En el caso de RR-BLUP, n correspondió al conjunto de entrenamiento y 36-n al conjunto de prueba. El procedimiento descrito se aplicó en las cuatro variables.
Para predecir mediante el BLUP, se construyó un programa en el módulo IML del paquete Statistical Analysis System (SAS). La predicción con RR-BLUP se hizo con base en la librería de funciones BGLR (Pérez & de los Campos, 2014) del paquete estadístico R.
Resultados y discusión
Selección de líneas progenitoras mediante ISSR
Se amplificaron 276 bandas, de las cuales 215 fueron polimórficas. El número de bandas por iniciador ISSR varió desde 2 hasta 18, con promedio de 9.52 (Cuadro 1). Estos valores son bajos comparados con los de Aguilera et al. (2011), que muestran la amplificación de 9 a 22 bandas por iniciador, con promedio de 14.4, aunque reportaron un nivel reducido de polimorfismo (34 %), con promedio de 5.3 bandas polimórficas por iniciador. En el presente trabajo se amplificaron 7.4 bandas polimórficas por ISSR con 77.9 % de polimorfismo general, con mínimo de cero y máximo de 100 %.
El método de agrupamiento de Ward definió tres conjuntos de líneas (Figura 1). El primer grupo estuvo constituido por 17 líneas (color verde a amarillo), el segundo con cuatro (tonos amarillos) y el tercero con 18 (naranja a rojo intenso). En dicha figura, los tonos de azul más claros indican distancia genética menor; por lo anterior, dentro de cada grupo predominan tonos azul claro y entre grupos tonalidades más obscuras. No se detectó asociación de los grupos generados con marcadores moleculares con alguna característica fenotípica. En cada grupo hay diversidad en el tipo de crecimiento (determinado e indeterminado), tipo de fruto (saladette y bola) y color de fruto (amarillo, naranja y rojo).
Evaluación de rendimiento y firmeza de fruto de cruzas intergrupales
De las 36 cruzas intergrupales evaluadas (Cuadro 2), en 2014 los valores de las cuatro variables fueron superiores, mientras que en 2015 se produjeron en promedio menos frutos (7) y kilogramos de fruto total (0.94) y comercial (1.3). Sin embargo, las cruzas se comportaron de manera similar dentro de cada ambiente. La misma cruza registró el valor más alto (o más bajo) para el NF, RFC y RFT en ambas evaluaciones. En el caso de la FF, la cruza con el mayor registro no fue la misma en los dos años evaluados; no obstante, la mayor FF estuvo asociada a cruzas que involucraron a la línea L68BI y la menor FF correspondió a cruzas con la línea L59BI (Cuadro 2).
Cuadro 2 Máximos, mínimos, medias y estimaciones de varianzas y heredabilidades de cuatro caracteres de 36 cruzas simples de tomate en dos ciclos de producción (2014 y 2015), cultivados en condiciones de hidroponía e invernadero.
| Max, Min estimaciones de varianzas y h 2 | NF | RFT (kg∙m -2 ) | RFC (kg∙m -2 ) | FF (kgf∙cm -2) |
|---|---|---|---|---|
| Evaluación 2014 | ||||
| Max(cruza) | 117.0 (L37SIxL80BI) | 13.723 (L76BIxL69BD) | 11.958 (L76BIxL68BI) | 13.83 (L5BIxL68BI) |
| Min(cruza) | 51.7 (L76BIxL45BI) | 8.153 (L59BIxL88BI) | 4.373 (L37SIxL80BI) | 5.98 (L59BIxL88BI) |
| Media | 77.3 | 10.488 | 8.630 | 9.44 |
| Evaluación 2015 | ||||
| Max (cruza) | 102.0 (L37SIxL80BI) | 12.048 (L76BIxL69BD) | 10.736 (L76BIxL68BI) | 11.04 (L61BIxL68BI) |
| Min (cruza) | 49.2 (L76BIxL45BI) | 7.358 (L65BIxL6BI) | 2.658 (L37SIxL80BI) | 6.81 (L59BIxL45BI) |
| Media | 70.7 | 9.553 | 7.332 | 8.94 |
| Varianzas | ||||
| VACG1 | 184.85 | 2.837 | 4.753 | 6.714 |
| VACG2 | 147.53 | 2.083 | 4.120 | 5.881 |
| VACE | 39.67 | 0.586 | 1.390 | 1.662 |
| VE | 10.77 | 0.192 | 0.371 | 0.491 |
| h2 | 0.986 | 0.983 | 0.982 | 0.983 |
Max: máximos, Min: mínimos, NF: número de frutos, RFT: rendimiento de fruto total, RFC: rendimiento de fruto comercial, FF: firmeza de fruto, VACG1: varianza de los efectos de aptitud combinatoria general de las líneas del grupo X, VACG2: varianza de los efectos de aptitud combinatoria general de las líneas del grupo Y, VACE: varianza de los efectos de aptitud combinatoria específica de las cruzas, VE: varianza del error y h2: heredabilidad. La estimación se hizo mediante marcadores moleculares y BLUP.
Predicción de rendimiento de cruzas mediante BLUP y RR-BLUP
Los coeficientes de coancestría estimados mediante marcadores moleculares, en general, fueron bajos. En las líneas del grupo X, fluctuaron desde 0.01 entre el par de líneas L5BI y L65BI, hasta 0.327 entre L59BI y L65BI, con coancestría media de 0.108. En el grupo de líneas Y se presentaron valores inferiores, desde 0 (en seis combinaciones) hasta 0.175 entre el par L45BI y L80BI, con promedio de 0.06.
Las estimaciones de las varianzas genética y no genética de las cuatro variables estudiadas, con marcadores ISSR mediante BLUP (Cuadro 2), permitieron calcular la heredabilidad (h 2) de cada carácter (Bernardo, 1996). De esta manera, para RFT, RFC, y FF, la heredabilidad fue muy alta (0.98); el NF registró h 2 = 0.99. En el caso del RFT, el valor estimado de h 2 en este estudio dista del publicado por Dordevic, Zecevic, Zdravkovic, Zivanovic, y Todorovic (2010) de 0.451; además de las variaciones aleatorias del error, esta discrepancia se puede atribuir a diferencias entre materiales genéticos y ambientes. Wessel-Beaver y Scott (1992) obtuvieron estimaciones de h2 de 0.65 y 0.81 para el rendimiento de la misma población de tomate cultivada en Puerto Rico y Florida, respectivamente.
Los valores de heredabilidad en este estudio permiten predecir éxito en la selección de los mejores híbridos, así como en un programa de mejoramiento basado en selección. La proporción de la varianza de la aptitud combinatoria específica (V ACE) respecto de la varianza genética total entre las CS fue 0.107, 0.106, 0.135 y 0.117, para NF, RFT, RFC y FF, respectivamente. Valores similares fueron reportados por Massman et al. (2013) en maíz al emplear el BLUP (desde 0.10 para humedad hasta 0.18 para rendimiento de grano). Por lo anterior, se puede inferir que en esta investigación el BLUP explotó efectos aditivos en mayor medida que de dominancia.
Correlaciones entre rendimientos observados y predichos
El promedio de 1,000 correlaciones entre el valor predicho y el registrado en campo para cada conjunto de n híbridos predictores se resume en el Cuadro 3. Tanto el BLUP como el RR-BLUP pronosticaron adecuadamente en las cuatro variables analizadas, y en todos los casos el BLUP estimó valores más cercanos a los reales que el RR-BLUP. Como era de esperarse, cuando se aumentó n se incrementaron también los valores de las correlaciones, aunque a partir de n = 24 estos incrementos fueron de poca magnitud e incluso se registró reducción. En este estudio, con 18 híbridos predictores (n = 18), se obtuvo la mayor estabilidad en las predicciones y sería el número de predictores ideal en un escenario de costos donde un incremento en n implica mayor inversión.
Cuadro 3 Correlaciones promedio de la validación cruzada para dos métodos (BLUP y RR-BLUP) de predicción de híbridos de tomate en cuatro variables. Los datos corresponden a 1,000 muestras aleatorias independientes de 36 híbridos predictores.
| N | FN | RFT (kg∙m -2 ) | RFC (kg∙m -2 ) | FF (kgf∙cm -2 ) | ||||
|---|---|---|---|---|---|---|---|---|
| BLUP | RR-BLUP | BLUP | RR-BLUP | BLUP | RR-BLUP | BLUP | RR-BLUP | |
| 6 | 0.53 | 0.45 | 0.52 | 0.36 | 0.36 | 0.25 | 0.41 | 0.32 |
| 12 | 0.72 | 0.66 | 0.69 | 0.58 | 0.48 | 0.40 | 0.57 | 0.55 |
| 18 | 0.80 | 0.75 | 0.76 | 0.68 | 0.55 | 0.49 | 0.65 | 0.63 |
| 24 | 0.83 | 0.80 | 0.79 | 0.73 | 0.58 | 0.54 | 0.69 | 0.68 |
| 30 | 0.83 | 0.83 | 0.75 | 0.69 | 0.57 | 0.53 | 0.71 | 0.67 |
| Media | 0.74 | 0.70 | 0.70 | 0.60 | 0.51 | 0.44 | 0.61 | 0.57 |
N: número de híbridos predictores, BLUP: mejor predictor lineal e insesgado, RR-BLUP: regresión ridge BLUP, NF: número de frutos, RFT: rendimiento de fruto total, RFC: rendimiento de fruto comercial y FF: firmeza de fruto.
Al considerar cada variable por separado, el NF registró los coeficientes de correlación más altos (de 0.45 a 0.83), seguido del RFT (0.36 a 0.73), la FF (0.32 a 0.68) y los resultados más bajas fueron del RFC (0.25 a 0.54), con RR-BLUP. Los valores de correlación entre observados y predichos con BLUP mantuvieron la misma tendencia, el NF (0.53 a 0.83), el RFT (0.52 a 0.79), la FF (0.41 a 0.71) y por último el RFC (0.36 a 0.58).
Las correlaciones obtenidas para el RFT son, en general, similares a las reportadas por Hernández-Ibáñez, Sahagún-Castellanos, Rodríguez-Pérez, y Peña-Ortega (2014) en tomate (entre 0.45 y 0.79), y por Bernardo (1994) en rendimiento de grano de maíz. Este último utilizó RFLP’s y las correlaciones más altas fueron de 0.80. Las correlaciones altas obtenidas sugieren que el método utilizado para predecir el rendimiento de los híbridos en tomate es prometedor. Sin embargo, la extrapolación debe hacerse con reservas, ya que la correlación entre híbridos observados y predichos aumenta conforme incrementa el número de híbridos predictores, pero hasta cierto nivel, después de éste se corre el riesgo de tener correlaciones más pequeñas e incluso negativas. Además, los resultados expuestos corresponden a un conjunto relativamente pequeño de CS. Bernardo (1996) aplicó esta metodología a una escala mayor (16 combinaciones de nueve grupos heteróticos en maíz), y las correlaciones entre rendimientos predichos y observados que obtuvo fueron ligeramente más bajas (de 0.426 a 0.762), muy parecidas a las obtenidas en el presente trabajo.
Massman et al. (2013) realizaron una comparación entre BLUP y RR-BLUP en cuatro variables en maíz y produjo correlaciones altas (0.87, 0.90, 0.69 y 0.84, para rendimiento de grano, humedad del grano, materia seca del tallo y raíz, respectivamente), sin diferencias significativas entre metodologías; por lo que afirmaron que RR-BLUP no superó los resultados obtenidos con BLUP. Estos resultados anteriores sugieren que el factor genético puede modificar la habilidad predictiva de los modelos.
Los resultados obtenidos en el presente trabajo en tomate se pueden considerar acordes con los reportados en maíz, a pesar de que se trata de especies contrastantes. Mientras que el tomate es una especie autógama con nivel reducido de heterosis, el maíz es alógama con expresiones de heterosis altas. En tomate, además del RFT, es muy importante el RFC y la FF después de la cosecha, por lo que se ven involucrados otros genes además de los que controlan estrictamente el rendimiento; los cuales afectan negativamente las correlaciones entre los diversos componentes del rendimiento y la producción de fruto de tomate. Los efectos epistáticos y de ligamiento, entre otros factores genéticos, además del ambiente, también pueden afectar la correlación. Lo anterior explica la dificultad en predecir estos caracteres, y por ello los niveles bajos de predicción obtenidos.
Conclusiones
Los coeficientes de correlación entre valores observados y predichos para NF, RFT, RFC y FF, estimados mediante BLUP y RR-BLUP en CS de tomate, fluctuaron entre 0.25 y 0.83, valores que sugieren que estos métodos pueden tener utilidad en el mejoramiento genético.
El BLUP registró consistentemente resultados más altos de correlación entre valores observados y predichos, comparado con RR-BLUP. En ambos casos, la magnitud de las correlaciones siempre aumentó cuando incrementó n. Por lo tanto, para alcanzar mayor precisión y confiabilidad de las predicciones con BLUP o RR-BLUP se deben evaluar en campo la mayor cantidad de cruzas predictoras, en tantos ambientes como el presupuesto lo permita.
Agradecimientos
Al Concejo Nacional de Ciencia y Tecnología (CONACYT) de México, por la beca doctoral otorgada al primer autor. Al Q.F.B. Ricardo Gaspar Hernández por el soporte técnico en marcadores moleculares de ADN. A los revisores anónimos por sus valiosas contribuciones que permitieron mejorar sustancialmente el manuscrito.
REFERENCIAS
Aguilera, J. G., Pessoni, L. A., Rodrigues, G. B., Elsayed, A. Y., da Silva, D. J. H., & de Barros, E. G. (2011). Genetic variability by ISSR markers in tomato (Solanum lycopersicon Mill.). Revista Brasileira de Ciências Agrárias, 6(2), 243-252. doi: 10.5039/agraria.v6i2a998 [ Links ]
Balestre, M., Von Pinho, R. G., & Souza, J. C. (2010). Prediction of maize single-cross performance by mixed linear models with microsatellite marker information. Genetics and Molecular Research, 9(2), 1054-1068. doi: 10.4238/vol9-2gmr791 [ Links ]
Bernardo, R. (1993). Estimation of coefficient of coancestry using molecular markers in maize. Theoretical and Applied Genetics, 85, 1055-1062. doi: 10.1007/BF00215047 [ Links ]
Bernardo, R. (1994). Prediction of maize single-cross performance using RFLPs and information from related hybrids. Crop Science, 34(1), 20-25. doi: 10.2135/cropsci1994.0011183X003400010003x [ Links ]
Bernardo, R. (1995). Genetic models for predicting maize single-cross performance in unbalanced yield trial data. Crop Science, 35(1), 141-147. doi: 10.2135/cropsci1995.0011183X003500010026x [ Links ]
Bernardo, R.(1996). Best linear unbiased prediction of maize single-cross performance. Crop Science, 36(1), 50-56. doi: 10.2135/cropsci1996.0011183X003600010009x [ Links ]
Cadahia, C. (2000). Fertirrigación, cultivos hortícolas y ornamentales (2da. Ed.). Madrid, España: Mundi Prensa. [ Links ]
Dordevic, R., Zecevic, B., Zdravkovic, J., Zivanovic, T., & Todorovic, G. (2010). Inheritance of yield components in tomato. Genetika, 42(3), 575-583. doi: 10.2298/GENSR1003575D [ Links ]
Gbur, E. E., Stroup, W. W., Mc Carter, K. S., Durham, S., Young, L. J., Christman, M., West, M., & Kramer, M. (2012). Analysis of generalized linear mixed model in the agricultural and natural resources sciences. Madison, Wisconsin, USA.: ASA, SSSA, CSSA, Inc. [ Links ]
Henderson, C. R. (1985). Best linear unbiased prediction of nonadditive genetic merits in noninbred populations. Journal of Animal Science, 60(1), 111-117. doi: 10.2134/jas1985.601111x [ Links ]
Hernández-Ibáñez, L., Sahagún-Castellanos, J., Rodríguez- Pérez, J. E., & Peña-Ortega, M. G. (2014). Predicción de rendimiento de fruto de híbridos de tomate con huellas genómicas. Phyton Revista Internacional de Botánica Experimental, 83, 311-318. Retrieved from http://www.revistaphyton.fund-romuloraggio.org.ar/vol83/HernandezIbanez.pdf [ Links ]
Khattree, R., & Naik, D. N. (2000). Multivariate data reduction and discrimination with SAS software. Cary, N.C., U.S.A: SAS Institute Inc. [ Links ]
Massman, J. M., Gordillo, A., Lorenzana, R. E., & Bernardo, R. (2013). Genomewide predictions from maize single-cross data. Theoretical and Applied Genetics, 126, 13-22. doi: 10.1007/s00122-012-1955-y [ Links ]
Melchinger, A. E. (1988). Means, variances, and covariances between relatives in hybrid populations with disequilibrium in the parent populations. In: Wei, B. S., & Eisen, E. J. (Eds.), Proc. 2nd. int. conf. quantit. genet. (pp. 400-405). Raleigh, NC.: Sinnauer Asoc. [ Links ]
Meuwissen, T. H. E., Hayes, B. J., & Goddard, M. E. (2001). Prediction of total genetic value using genome-wide dense marker maps. Genetics, 157(4), 1819-1829. Retrieved from http://www.genetics.org/content/genetics/157/4/1819.full.pdf [ Links ]
Mirshamsi, A., Farsi, M., Shahriari, F., & Nemati, H. (2008). Use of random amplified polymorphic DNA markers to estimate heterosis and combining ability in tomato hybrids. Pakistan Journal of Biological Sciences, 11(4), 499- 507. doi: 10.3923/pjbs.2008.499.507 [ Links ]
Pérez, P., & de los Campos, G. (2014). Genome-wide regression and prediction with the BGLR statistical package. Genetics, 198(2), 483-495. doi: 10.1534/genetics.114.164442 [ Links ]
Wessel-Beaver, L., & Scott, J. W. (1992). Genetic variability of fruit set, fruit weight, and yield in a tomato population grown in two high-temperature environments. Journal of the American Society of Horticultural Science, 117(5), 867- 870. Retrieved from http://journal.ashspublications.org/content/117/5/867.full.pdf+html [ Links ]
Recibido: 22 de Junio de 2016; Aprobado: 11 de Octubre de 2016











 texto em
texto em