Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista Chapingo. Serie horticultura
versión On-line ISSN 2007-4034versión impresa ISSN 1027-152X
Rev. Chapingo Ser.Hortic vol.19 no.2 Chapingo may./ago. 2013
https://doi.org/10.5154/r.rchsh.2012.09.047
Erosión genética en las cruzas simples progenitoras de una variedad sintética
Genetic erosion in single-cross progenitors of a synthetic variety
José Luis Escalante-González; Jaime Sahagún-Castellanos*; Juan Enrique Rodríguez-Pérez; Aureliano Peña-Lomelí
Universidad Autónoma Chapingo. Departamento de Fitotecnia km 38.5 Carretera México-Texcoco. Chapingo, Estado de México. MÉXICO. C.P. 56230. Correo-e: jsahagunc@yahoo.com.mx Tel.: 9521500 Ext. 6185 (*Autor para correspondencia).
Recibido: 30 de septiembre, 2012.
Aceptado: 8 de abril, 2013.
Resumen
El azar, el número finito de individuos y la variabilidad genética entre las plantas que representan cada progenitor de un sintético de cultivos como la cebolla (Allium cepa L.) y el maíz (Zea mays L.) pueden ocasionar erosión genética. Esto puede ocurrir en el desarrollo de una variedad sintética cuyos progenitores son L/2 cruzas simples (CSs) formadas con L líneas. Si el coeficiente de endogamia de éstas (FL) es menor a uno, y el número de plantas de cada CS (m) es pequeño, el número de genes no idénticos por descendencia (NIPD) de cada CS se convierte en una variable aleatoria (Xm). Los objetivos de este trabajo fueron determinar: 1) la media de Xm [E(Xm)], 2) la varianza de Xm [Var(Xm)], y 3) la pérdida promedio de genes NIPD que ocurre en la transmisión de genes de dos líneas a su CS. Se encontró que E(Xm) = 2 + [2 - (1/2)m-2 ](1 - FL). Asimismo, se encontró que la pérdida promedio de estos genes es (1/2)m-2(1-FL) y que Var(Xm) = [2m+2 - 8](1-FL)2/4m . Estos dos resultados implican que con líneas puras (FL = 1) la pérdida y la inestabilidad del número de genes NIPD se reducen a su mínimo (cero), y se aproximan a éste a partir de m = 8, más rápidamente cuando FL es más grande.
Palabras clave: Allium cepa L., Zea mays L., variedad sintética, genes no idénticos por descendencia, apareamiento aleatorio.
Abstract
Randomness, finite number of individuals and genetic variability among those that represent each parent of a synthetic of crop species such as onion (Allium cepa L.) and maize (Zea mays L.) can cause genetic erosion during the development of a synthetic variety. This may happen when the parents are L/2 single crosses (SCs) derived from L lines. If the inbreeding coefficient of the lines (FL) is less than 1, and the number (m) of plants that represent each SC is small, the number of non-identical by descent (NIBD) genes of each SC is a random variable (Xm). The objectives of this study were to determine: 1) the mean of Xm [E(Xm)], 2) the variance of Xm [Var(Xm)], and 3) the average loss of NIBD genes which are contributed by two lines to their SC. It was found that E(Xm) = 2 + [2 - (1/2)m-2 ](1 - FL). In addition, the average loss of NIBD genes was (1/2)m-2(1-FL) and Var(Xm) = [2m+2 - 8](1-FL)2/4m. These two results imply that when lines are pure (FL=1), the loss of NIBD genes and the instability of the number of these genes reach their minimum (zero), and approach to it from m = 8 on, more rapidly as FL becomes larger.
Keywords: Allium cepa L., Zea mays L., synthetic variety, non-identical by descent genes, random mating.
INTRODUCCIÓN
Las variedades sintéticas (VSs) de especies cultivadas como la cebolla (Allium cepa L.), el maíz (Zea mays L.), etc., tienen como origen el apareamiento aleatorio de varios progenitores, usualmente entre 6 y 12 (Márquez-Sánchez, 1992; Kutka y Smith, 2007; Sahagún, 2011). Si el coeficiente de endogamia de las líneas es uno y cada una se representa por m plantas, de acuerdo con el modelo de un locus de una especie diploide, los 2m genes de las m plantas de cada línea deben ser idénticos por descendencia (Falconer y Mackay, 2007). Sin embargo, si las líneas no son puras, cada una podrá contener genes que entre sí son idénticos por descendencia, así como genes que no lo son.
Por otra parte, la formación de VSs tiene variantes que no han sido estudiadas en forma completa. En particular, varios autores han propuesto su formación mediante el apareamiento aleatorio de varios híbridos (Villanueva et al., 1994; Sahagún-Castellanos et al., 2005; Márquez-Sánchez, 2008); sin embargo, este procedimiento todavía tiene temas interesantes inéditos. Al respecto, una característica que se supone inseparable de una variedad sintética (VS) es la estabilidad de su arreglo genotípico. De acuerdo con este supuesto, la VS debe reproducirse a sí misma año tras año, lo que significa que el agricultor puede producir la semilla de su VS en su propio campo de producción. Desafortunadamente, hay casos en que la ocurrencia de esta característica puede cuestionarse aun cuando la liberación de la variedad no ha ocurrido. Por ejemplo, supóngase el caso de una VS generada por el apareamiento aleatorio de L/2 cruzas simples (CSs), formadas con L líneas (Sahagún-Castellanos y Villanueva, 1997); cada gameto que emita una planta que representa una CS para formar la VS (SinCS) puede portar un gen que es copia del gen que le transmitió una u otra de sus dos líneas progenitoras. Si la progenie de esta CS es pequeña, el número de genes no idénticos por descendencia (NIPD) que aporta al SinCS por azar puede ser menor que lo esperado en términos de las frecuencias teóricas. Más aún, sólo si las líneas son puras, la aportación de cada una de las dos a su cruza simple (CS) será de m genes idénticos por descendencia. Cuando las líneas no son puras, pueden aportar un número mayor que uno de genes NIPD, lo que a su vez generaría variabilidad aleatoria que puede afectar la estabilidad genética de la variedad.
Una medida de la estabilidad y heterogeneidad genética de una VS puede expresarse en términos del número de genes NIPD que contiene. Como ya se mencionó, este número, puede comportarse como una variable aleatoria de un ciclo a otro; y la magnitud de la variabilidad que muestre debe reflejar el grado de incertidumbre del proceso de formación de la VS en términos de los genes que debe contener según la constitución genética de las líneas progenitoras iniciales. Evidentemente, la inestabilidad en este caso significa pérdida de genes. Esto es de importancia singular para el fitomejorador en primera instancia, y para los agricultores posteriormente.
La inestabilidad en el número de genes NIPD repercute en la composición genotípica, el coeficiente de endogamia, y en las medias genotípica y fenotípica de la VS. Generar información relativa a la magnitud de la inestabilidad y de la pérdida de los genes NIPD que ocurren en el proceso de desarrollo de una VS debe contribuir al mejor diseño de las VS. El objetivo de este estudio fue determinar la media, la varianza y la reducción del número de genes no idénticos por descendencia en las cruzas simples que generarán un sintético.
MÉTODOS Y MARCO TEÓRICO
En este estudio se utilizaron dos conceptos para una población en que el gen Ai y el genotipo AiAj tienen frecuencias pi y pij, respectivamente; los arreglos gamético (AGA) y genotípico (AGE) se definen en la forma:

Además, si los arreglos gaméticos de las poblaciones P1 y P2 son 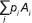 y
y 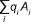 , la cruza de estas dos poblaciones produce una progenie cuyo arreglo genotípico [AGE(P1xP2)] es el producto de sus arreglos gaméticos; es decir,
, la cruza de estas dos poblaciones produce una progenie cuyo arreglo genotípico [AGE(P1xP2)] es el producto de sus arreglos gaméticos; es decir,

En particular, si como sucede en los sintéticos, la reproducción de una población que tiene el arreglo gamético 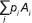 es por apareamiento aleatorio, el arreglo genotípico de su progenie es (Sahagún-Castellanos, 1994):
es por apareamiento aleatorio, el arreglo genotípico de su progenie es (Sahagún-Castellanos, 1994):
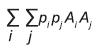
Por otra parte, respecto a las L líneas iniciales, para la formación de una variedad sintética se consideró que no tenían parentesco, y la línea i (i = 1, 2, ..., L) se visualizó como una población formada por individuos que se clasificaron en dos grupos: los portadores de genotipos formados por dos genes idénticos por descendencia (Ai Ai) y los formados por dos genes no idénticos por descendencia (Ai Aci, i ≠ ci) con frecuencias FL y 1−FL, respectivamente. Además, se consideró que los genes no idénticos por descendencia de una línea son: 2, cuando FL < 1; 1, cuando FL = 1; y, en general, si 0 ≤ FL ≤ 1 el número de genes no idénticos por individuo en promedio debe ser 1(FL) + 2(1-FL) = 2-FL.
DERIVACIÓN DE RESULTADOS
Número de genes no idénticos por descendencia
Si Bip1 Bip2 es el genotipo del p−ésimo individuo (p = 1, 2,..., m) de la línea i (i = 1, 2, ..., L; L es un número par) la frecuencia del gameto Bipk (k = 1, 2) es (2mL)−1 y el arreglo genotípico del sintético formado por el apareamiento aleatorio de los mL individuos (AGESL) es:
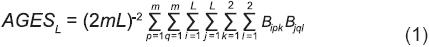
Para investigar qué es AGESL (Ecuación 1) en términos del sintético que genera el apareamiento aleatorio de L/2 cruzas simples formadas con la participación de cada una de las L líneas, se consideró que la cruza simple h (h = 2, 4, 6,..., L), representada por m plantas, está formada por las líneas h y h−1. El arreglo genotípico de la población que producen las L/2 cruzas (AGESCS) es de la forma:

Evidentemente, si las L líneas son puras (FL = 1) no habrá pérdida de genes no idénticos por descendencia (NIPD) en la formación de los m genotipos que resultarán del apareamiento de las dos líneas de cada una de las L/2 cruzas simples. Sin embargo, esto puede ser diferente cuando las L líneas no son puras (FL < 1) y m es pequeño. Por ejemplo, si m = 1 y FL < 1, como cada línea inicial, en promedio, es portadora de 2 – FL genes NIPD, las 2 líneas de una cruza simple portan en promedio 4 – 2FL genes NIPD, y a una planta (m = 1) que produzca su cruza le aportarán exactamente 2 de estos genes porque las líneas no están emparentadas. Esto implica una pérdida promedio de 2 −2FL genes NIPD por cada uno de los L/2 individuos que representan sendas cruzas simples.
Para m = 2, supóngase que los genotipos de las 2 líneas progenitoras de una cruza simple se representan como A1A2 y B1B2. Las probabilidades (P) de que los genes de cada genotipo sean idénticos por descendencia (≡) son P(A1 ≡ A2) = P(B1 ≡ B2) = FL y de que no lo sean (≠) es P(A1 ≠ A2) = P(B1 ≠ B2) = 1 – FL. Además, el arreglo genotípico de la cruza entre las dos líneas (AGECL) es:
AGECL = (1/4)A1B1 + (1/4)A1B2 + (1/4)A2B1 + (1/4) A2B2
Las dos plantas de la cruza que participarán en el SinCS de este ejemplo (m = 2) se pueden visualizar como el resultado de un muestreo aleatorio de tamaño 2 con reemplazo del conjunto de los 4 genotipos diferentes del AGECL. Los tipos de resultados posibles son:
a) el mismo genotipo las dos veces. Por ejemplo, A1B1 y A1B1 (2 genes NIPD);
b) dos genotipos diferentes de la forma: A1B1 y A1B2 (3 – FL genes NIPD), y
c) dos genotipos diferentes de la forma: A1B1 y A2B2 (4 – 2 FL genes NIPD).
Las probabilidades de ocurrencia de estos tres tipos de resultados son, en el mismo orden: 4/16, 8/16 y 4/16. Y el número de genes NIPD que se espera que aporte esta cruza simple al arreglo genotípico del SinCS en promedio [E(X2)] se deriva a continuación:
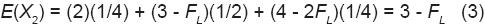
Según la Ecuación 3, con m = 2 la cantidad esperada de genes NIPD en una cruza simple representada por dos plantas cuando las líneas son puras (FL = 1) es igual a 2; en este caso con un individuo por cada línea progenitora se aportarían estos 2 genes. Sin embargo, cuando FL < 1, cada individuo de una línea aportaría, en promedio, 2 – FL genes NIPD; y los dos individuos aportarían, en promedio, 4 – 2FL genes NIPD. Esto significa que con m = 2 en la cruza simple y m = 1 en sus dos líneas progenitoras, a pesar de la compensación del número de individuos (2 en la cruza simple y 1 en cada línea progenitora), cada cruza simple pierde 1 – FL genes NIPD ya que sólo aporta 3 – FL de estos 4 – 2FL genes NIPD (Ecuación 3).
Un caso más para entender mejor los detalles de la derivación general se hará a continuación. Si m = 3 y la variable aleatoria X3 representa el número de genes no idénticos por descendencia (NIPD) que se aporta a una cruza simple, E(X3) se puede derivar según la expresión
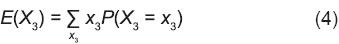
En la Ecuación 4, x3 representa los números de genes NIPD que puede aportar una cruza simple al arreglo genotípico del SinCS, y P(X3 = x3) es la probabilidad de que ocurra el valor x3 cuando se efectúa el fenómeno visualizable como la toma de una muestra aleatoria con reemplazo de tamaño 3 del conjunto que forman los 4 genotipos que produce la cruza simple A1A2 x B1B2. El muestreo de tamaño 3 puede producir los tipos de resultados siguientes:
a) el mismo genotipo las 3 veces (por ejemplo A1B1, A1B1 y A1B1). Esto puede ocurrir de 4 formas (los cuatro genotipos posibles); y x3 =2.
b) 2 genotipos iguales y uno diferente: i) ejemplo 1: A1B1, A1B1 y A1B2; ii) ejemplo 2: A1B1, A1B1 y A2B2. En los casos i) y ii), x3 = 3 – FL y x3 = 4 – 2FL, respectivamente. Además, los números de casos del tipo i) son 24 [4 combinaciones de 2 genotipos, 2 posibilidades respecto al que ocurre 2 veces, y 3 órdenes de ocurrencia en cada caso, que hacen un total de 4×2×3 = 24 ] y del tipo ii) son 12 [2 combinaciones de 2 genotipos, 2 posibilidades respecto al que ocurre 2 veces, y 3 órdenes de ocurrencia en cada caso].
c) 3 genotipos diferentes. Por ejemplo: A1B1, A1B2 y A2B1. En esto casos, x3 = 4 – 2FL. El número de casos es 24 [4 combinaciones de 3 genotipos, y 3! = 6 órdenes de ocurrencia posibles en cada caso].
De acuerdo con la información generada para m = 3 en a), b) y c) y la consideración de que el muestreo produce 64 (43) resultados posibles:
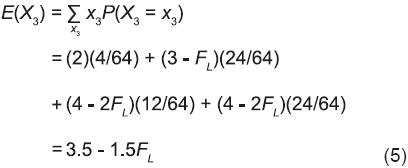
En general, cuando cada cruza simple se representa por m plantas, los datos requeridos para calcular E(Xm) se consignan en el Cuadro 1. De acuerdo con éstos:

Según la Ecuación 6, el número total de genes NIPD que se espera que contengan todas las cruzas simples (NTGNIPD) debe ser (L/2)[E(Xm)]; es decir:

Variabilidad del número de genes NIPD
La varianza (Var) del número de genes (Xm) no idénticos por descendencia (NIPD) para cada cruza simple, con ayuda de la Ecuación 6 y del Cuadro 1 puede derivarse con base en la expresión inicial siguiente:

El desarrollo de la Ecuación 8, se hará agrupando términos de acuerdo con la inclusión o ausencia de FL y FL2. La contribución a Var(Xm) de los términos que no contienen el coeficiente de endogamia FL ni FL2 (NCE) es:

Similarmente, las contribuciones de los términos que contienen FL (CCE) y FL2 [C(CE)2] a Var (Xm) corresponden a las expresiones siguientes:

y
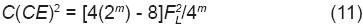
De acuerdo con las Ecuaciones 8 a 11, la expresión de la varianza del número de genes NIPD que recibe una cruza simple [Var (Xm)] es:
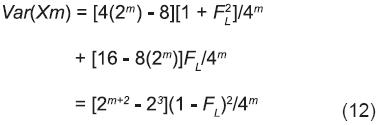
La Ecuación 12 implica que la varianza del número de genes NIPD es mayor cuando el coeficiente de endogamia de las líneas iniciales (FL) y el número de individuos que representan cada línea (m) son menores. De hecho, cuando las líneas progenitoras iniciales son puras (FL=1), independientemente del tamaño de m, Var(Xm) es igual a cero; y la contribución esperada a cada cruza simple es de 2 genes NIPD (Ecuación 6). Notablemente, las Ecuaciones 6 y 12 implican que los incrementos de m redundan en la reducción de la pérdida de genes NIPD y en la estabilidad del arreglo genotípico del SinCS (Ecuación 2). En este contexto es de particular interés la selección recurrente en que la prueba temprana de aptitud combinatoria general se aplica a líneas de una sola autofecundación (FL=1/2) y la formación de sintéticos con estas líneas. En ambos casos, con m ≥ 8 la pérdida esperada y la varianza de genes NIPD de las cruzas simples se acercan considerablemente a cero (Cuadro 2, Ecuaciones 6 y 12).
DISCUSIÓN
Si el coeficiente de endogamia de las L líneas con que se genera las L/2 cruzas simples (CS) es menor que 1 (FL < 1), y cada CS se representa por más de 1 planta (m > 1), en los m genotipos de las m plantas de cada cruza simple debe haber de 2 a 4 genes no idénticos por descendencia (NIPD). Según la Ecuación 6, el número esperado de genes NIPD [E(Xm)] que tiene una cruza simple es 2 + [2 - (1/2)m-2](1 - FL) . Esta expresión implica que E(Xm) aumenta cuando m es mayor. Esto se debe a que cada gen adicional que contribuye a la formación de un genotipo de una cruza simple es una oportunidad de que sea un gen NIPD que no se hubiera contribuido previamente. Sin embargo, de acuerdo con la Ecuación 6, estos incrementos del número de genes NIPD aportados son cada vez más pequeños. El Cuadro 2 refleja claramente esta tendencia. En el extremo opuesto, cuando cada cruza simple se representa por una planta (m = 1) su genotipo siempre tiene que estar formado por 2 genes NIPD debido a que las líneas no están emparentadas; y la media y la varianza del número de genes NIPD deben ser 2 (Ecuación 6) y 0 (Ecuación 12), respectivamente, para cualquier valor de FL (Cuadro 2).
La Ecuación 6 también refleja que E(Xm) y FL tienen una relación inversa, lo que era de esperarse porque FL es una medida del grado de identidad por descendencia de los dos genes que integran cada genotipo de las líneas iniciales. Además, la relación entre E(Xm) y FL es lineal; es decir, cada que FL aumenta (o disminuye) una misma cantidad, E(Xm) disminuye (o aumenta) una cantidad constante también.
Respecto a la varianza de Xm, las diferencias en sus magnitudes debidas a cambios en FL y a cambios en m son predecibles según la Ecuación 12. En cambio, la variación que se consigna para cada combinación de valores de m y FL en el Cuadro 2 es variación que se debe a la naturaleza aleatoria del mecanismo genético. Esta variabilidad se intensifica cuando las L líneas iniciales no son puras (FL < 1) y m es pequeña pero mayor que 1. Esta variación aleatoria es importante porque impacta la constitución genotípica de las cruzas simples en primera instancia, y posteriormente la del sintético que se forme con esas L/2 cruzas simples. La Ecuación 12 implica que si m > 1 la magnitud de Var(Xm) se relaciona inversamente con FL y con m. En un extremo, cuando las líneas iniciales son puras (FL = 1), las cruzas simples son genotípicamente uniformes y portan sólo 2 genes NIPD, independientemente del tamaño de m, en congruencia con la Ecuación 12.
Por sus características (coeficiente de endogamia FL y ausencia de parentesco entre ellas), las L líneas iniciales contienen un número esperado de L(2−FL) genes NIPD, sea cual fuere la magnitud de m. Además, según la Ecuación 6, se espera que las L/2 cruzas simples sean portadoras de L + L[1 - (1/2)m-1](1 - FL) genes NIPD. La diferencia entre el primero y el segundo de estos resultados, (1/2)m-1](1 - FL), es el número promedio de genes NIPD que se pierde en la formación de las L/2 cruzas simples. Evidentemente, la pérdida de genes es mayor a medida que FL y m son más pequeños. Y esta pérdida influye en que el arreglo genotípico del SinCS (Ecuación 2) sea diferente del SinL (Ecuación 1). En cambio, cuando se parte de líneas puras (FL = 1) no se pierde ningún gen NIPD hasta la formación de las cruzas simples.
La reducción del número de genes NIPD debe repercutir en las frecuencias génicas y genotípicas y, consecuentemente, fenotípicas. Por esta razón, si m y/o FL son pequeñas la representación del SinL mediante el SinCS no será fidedigna. Sin embargo, si las líneas no son puras, según los datos del Cuadro 2 y la Ecuación 6, a partir de m = 8, aproximadamente, la pérdida de genes se reduce prácticamente a 0, y el número de genes NIPD se estabiliza en 2(2 – FL) en cada cruza simple.
En el apareamiento aleatorio de las CSs para producir el SinCS, sin embargo, nuevamente el azar y el tamaño de m actúan como factores que pueden causar una pérdida adicional de genes NIPD, y, en consecuencia, que el AGESCS (Ecuación 2) difiera del AGESL (Ecuación 1).
CONCLUSIONES
Con la participación de cada una de L líneas no emparentadas y un coeficiente de endogamia FL se consideró la formación de las L/2 cruzas simples (CSs) representadas por sendos conjuntos de m plantas. La media y la varianza del número de genes no idénticos por descendencia (NIPD) que transmiten cada dos líneas progenitoras a su CS fueron 2 + [2 - (1/2)m-2](1-FL) y [2m+2 -8](1-FL)2/4m, respectivamente. Como cada par de líneas progenitoras de una CS porta en promedio 2(2 – FL) genes NIPD, por cada CS se pierden en promedio (1/2)m-2(1 – FL). Estos resultados sugieren la conveniencia del uso de líneas puras (FL = 1), para evitar pérdida de genes NIPD y aumentar la estabilidad genotípica de los progenitores del sintético. Además, aún con FL < 1 a partir de m = 8, la pérdida de genes NIPD prácticamente se reduce a 0 y E(Xm) se estabiliza en 2(2 – FL).
LITERATURA CITADA
FALCONER, D. S.; MACKAY, T. F. C. 2007. Introducción a la Genética Cuantitativa. 4ª Edición. Editorial Acribia. S. A. Zaragoza, España. 469 p. [ Links ]
KUTKA, F.J.; SMITH., M. E. 2007. How many parents give the highest yield in predicted synthetic and composite populations of maize? Crop Sci 47: 1905-1913. doi: 10.2135/cropsci2006.12.0802sc. [ Links ]
MÁRQUEZ-SÁNCHEZ, F. 1992. Inbreeding and yield prediction in synthetic maize cultivars made with parental lines: Basic methods. Crop Sci. 32: 345-349. doi: 10.2135/cropsci1992.0011183X003200020013x. [ Links ]
MÁRQUEZ-SÁNCHEZ, F. 2008. Endogamia y predicción de sintéticos de maíz de cruzas dobles. Revista Fitotecnia Mexicana 31(Especial 3): 1-4. http://www.revistafitotecniamexicana.org/documentos/31-1%20Especial%203/1a.pdf. [ Links ]
SAHAGÚN-CASTELLANOS, J. 1994. Sobre el cálculo del coeficiente de endogamia de variedades sintéticas. Agrociencia Serie Fitociencia 5: 67-78. [ Links ]
SAHAGÚN-CASTELLANOS, J., RODRÍGUEZ-PÉREZ, J. E.; PEÑA-LOMELÍ, A. 2005. Predicting yield of synthetics derived from double crosses. Maydica 50: 129-136. http://www.maydica.org/articles/50_129.pdf. [ Links ]
SAHAGÚN C., J. 2011. Inbreeding and yield of synthetic varieties derived from single and double cross hybrids. Maydica 56: 265-271. http://www.maydica.org/articles/56_265.pdf. [ Links ]
SAHAGÚN C., J.; VILLANUEVA V., C. 1997. Teoría de las variedades sintéticas formadas con híbridos de cruza simple. Revista Fitotecnia Mexicana 20: 69-72. http://www.redalyc.org/articulo.oa?id=61020106. [ Links ]
VILLANUEVA V., C.; CASTILLO G., F.; MOLINA G., J. D.1994. Aprovechamiento de cruzamientos dialélicos entre híbridos comerciales de maíz: análisis de progenitores y cruzas. Revista Fitotecnia Mexicana 17: 175-185. [ Links ]




![Comportamiento productivo del lisianthus (Eustoma grandiflorum [Raf.] Shinn) en cultivo sin suelo](/img/es/prev.gif)









