Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista Chapingo. Serie horticultura
versión On-line ISSN 2007-4034versión impresa ISSN 1027-152X
Rev. Chapingo Ser.Hortic vol.17 no.3 Chapingo sep./dic. 2011
Endogamia de sintéticos formados con líneas y cruzas simples
Inbreeding of synthetic varieties derived from lines and single crosses
Jaime Sahagún–Castellanos*; Juan Enrique Rodríguez–Pérez
Instituto de Horticultura, Departamento de Fitotecnia, Universidad Autónoma Chapingo, km 38.5 Carretera México–Texcoco, C. P. 56230 Chapingo, Estado de México. C. P. 56230. MÉXICO. Correo–e: jsahagunc@yahoo.com.mx (*Autor para correspondencia).
Recibido: 9 de febrero, 2011.
Aceptado:12 de septiembre, 2011.
Resumen
La explotación de la heterosis en cultivos como el jitomate (Licopersicon esculentum Mill.), la cebolla (Allium cepa L.), el maíz (Zea mays L.), etc., ha sido limitada por el alto costo de la semilla Fr Para evitar éste, los productores han recurrido al uso de semilla F2, F3,... Sin embargo, la depresión endogámica que esta práctica produce ha hecho que los fitomejoradores consideren las variedades sintéticas, particularmente para los cultivos alógamos, las cuales, de todos modos, no están exentas de problemas. En este estudio se propone la formación de una variedad sintética (VS) con L líneas y s cruzas simples (SinL'CS) como una forma de reducir el número de progenitores (originalmente L+2s líneas) y con esto el costo y trabajo para predecir los VSs que se pueden formar. Sin embargo, los indicadores de calidad más importantes del SinL'CS deben conocerse para hacerles una valoración objetiva. Para el SinL'CS los objetivos fueron determinar: 1) su coeficiente de endogamia (FSinL'CS) y 2) la mejor combinación de valores de L y s. La derivación de fórmulas se basó en el arreglo genotipico del SinL'CS (AGESinL'CS) y en la sustitución del genotipo general de este arreglo por la coancestría de los individuos que aportan los genes que lo forman (para FsinL'CS). Y la mejor combinación de valores de L y s fue la que produjo el menor coeficiente de endogamia. Para un número par fijo de L + 2s líneas homocigóticas no emparentadas, se hicieron dos descomposiciones del AGESinL'CS y se encontró que FSinL,CS ={1+Ls/[2(L+s)2]}/(L+2s), que alcanza su mínimo cuando los progenitores son sólo líneas (s = 0) o sólo cruzas simples (L = 0). Sin embargo, con sólo cruzas simples la predicción demanda menos recursos que la basada en todas las líneas.
Palabras clave: Allium cepa L., Zea mays L., coancestría, predicción, arreglo genotípico.
Abstract
The exploitation of heterosis in crops such as tomato (Licopersicon esculentum Mill.), onion (Allium cepa L.) and corn (Zea mays L.) has been limited by the high cost of F1 seed. To avoid this, producers have resorted to using F2, F3 ... seed. However, the inbreeding depression that this practice produces has led plant breeders to consider the synthetic varieties (SVs), particularly for cross–pollinated crops, which are not, however, without problems. In this work, the derivation of a SV whose parents are L lines and s single crosses (SynL'SC) is proposed as a way of decreasing the parent number (originally L+2s lines) and thereby also the costs and work involved in prediction. However, the most important SynL'SC quality indicators must be known in order to make an objective assessment. Thus, the study objectives related to SynL'SC were to determine: 1) the inbreeding coefficient (FSynL'SC) and 2) the best combination of L and s values. The derivation of FSynL'SCwas based on the replacement of the general genotype of the genotypic array of SynL'SC(GASynL'SC) by the coancestry between the individuals that contribute the genes that make up this genotype, and the best combination of L and s values was the one that produced the lowest inbreeding coefficient. From a fixed even number of L + 2s fully inbred and unrelated lines and from two decompositions of GASynL'SC, it was found that FSynL,SC={1+Ls/[2(L+s)2]}/(L+2s). By inspection of the formula, FSynL'SC reaches its minimum in two cases: when L = 0 and when s = 0. However, the use of (L+2s)/2 single crosses requires fewer resources for prediction.
Key words: Allium cepa L., Zea mays L., coancestry, prediction, genotypic array.
INTRODUCCIÓN
El origen de las variedades sintéticas tiene que ver con la depresión endogámica que se observó en las generaciones avanzadas de los primeros híbridos de cruza simple de maíz (Zea mays L.) cuando éstos irrumpieron en el escenario agrícola y, posteriormente, en los híbridos de cruza doble y en los trilineales (Márquez, 1992). La observación de este fenómeno fue posible en México debido a que algunos agricultores, para evitar el alto costo de la semilla de las variedades híbridas en cada ciclo agrícola, sembraban la semilla que cosechaban de un híbrido, primero, y después de sus generaciones avanzadas (Villanueva et al., 1994). La depresión endogámica también ha sido observada en cultivos hortícolas como el jitomate (Lycopersicon esculentum Mill.), según reportan Mendoza et al. (2010) y Martínez et al. (2005), y en cebolla (Allium cepa L.). Si bien las cruzas simples, híbridos trilineales y cruzas dobles se construyen con dos, tres y cuatro líneas, respectivamente, la pregunta natural a ser contestada fue la relativa al comportamiento general de las variedades formadas por más líneas. Wright (1922) se refirió a la predicción de la media genotípica de las poblaciones que pueden construirse por el apareamiento aleatorio de las líneas de cada uno de los subconjuntos de dos o más elementos de un conjunto de n líneas homocigóticas. A cualquier población así formada y a cualquiera de sus sucesivas generaciones producidas por apareamiento aleatorio se le denomina variedad sintética. Posteriormente, Busbice (1970) desarrolló una fórmula de predicción más general que la de Wright (1922) para este tipo de variedades. Según Busbice (1970), la media genotípica del sintético en la generación t es una función lineal del coeficiente de endogamia de la variedad en esa generación.
Respecto a la forma de desarrollar una variedad sintética, Sahagún y Villanueva (1997) propusieron el uso de híbridos de cruza simple como progenitores, en lugar de las líneas involucradas. Con esto, se pensó, se reduce en un 50 % el número de progenitores, lo que a su vez reduce el número de cruzas entre ellos que, una vez evaluadas experimentalmente junto con los progenitores, permiten predecir las medias genotípicas de las variedades que se pueden formar con las cruzas simples. En efecto, si con un número par p de líneas se pueden construir 2p –(p+1) sintéticos de dos o más progenitores, con p/2 cruzas simples formadas con las p líneas el número de sintéticos que se puede formar con uno o más de este tipo de progenitores es 2 p/2 – p/2 . En el primer caso el número de cruzas (directas y recíprocas) que hay que hacer y después evaluar experimentalmente es p(p –1) y en el segundo es (p/2)[p/2) – 1] ; con p= 12 , por ejemplo, estos números son 132 y 30, respectivamente. Sin embargo, con el uso de híbridos de cruza simple o doble como progenitores de sintéticos no es posible la predicción de sintéticos formados con un número non de líneas ni de sintéticos que prescindan de ninguna de las líneas progenitoras de esos híbridos.
En maíz, por ejemplo, una variedad sintética alcanza su rendimiento óptimo con no más de 12 líneas (Kutka y Smith, 2007). De acuerdo con esto, en la búsqueda de la mejor variedad sintética de maíz que se puede formar si se parte de 12 líneas habría que investigar el comportamiento de las variedades sintéticas que se pueden construir con dos o más de estas líneas. Una solución intermedia al problema del número elevado de sintéticos que se puede construir (4,083) se ejemplifica para este caso particular de 12 líneas. Con s = 4 y L = 4 las VSs posibles de ser construidas con dos o más líneas son 252, y con s = 5 y L = 2 este número se reduce a 126. Sin embargo, habrá que considerar que en una variedad sintética formada con L líneas y s cruzas simples (SinL'CS) se genera un desbalance en las frecuencias de los genes de las líneas progenitoras con relación a las frecuencias de los genes de las líneas que forman las cruzas simples. Este desbalance puede tener efecto en el coeficiente de endogamia y en la media genotípica de la variedad sintética; además, habrá que afrontar el problema de determinar la mejor combinación de valores de L y s. Con el fin de generar información que aporte al desvanecimiento de estas dudas, se planeó este trabajo téorico en torno a un sintético formado con s cruzas simples y L líneas (SinL'CS). Los objetivos fueron: 1) derivar fórmulas generales para el coeficiente de endogamia de SinL'CS, y 2) determinar el mejor diseño de progenitores para SinL'CS (la mejor combinación de valores para s y L).
MÉTODOS Y MARCO TEÓRICO
Sea Api1Api2 el genotipo del p – ésimo (p= 1, 2, ...,m) individuo que representa al progenitor i. Si i= 1, 2, ...,L el progenitor es una línea, y si i = L+1, L+2, ...,L+s, el progenitor es una cruza simple. En lo sucesivo se usará esta notación y los conceptos y enfoque metodológico utilizados por Sahagún (1994). El arreglo genotípico de los progenitores (AGEP) es

De acuerdo con Busbice (1970), esta población es el "sintético 0".
Similarmente, el arreglo gamético de estos L + s progenitores (AGAP) es
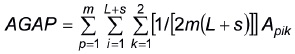
Con este AGAP, el arreglo genotípico de las cruzas (directas y recíprocas) entre los L + s progenitores (AGCP) es
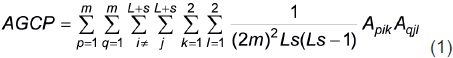
La población representada por AGCP se suele llamar "sintético 1" (Busbice, 1970).
Como el arreglo genotipico del sintético (AGESinL'CS) es el de la población que resulta del apareamiento aleatorio de los progenitores o de sus cruzas, resulta que
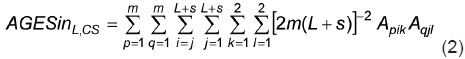
De la Ecuación 2 se pueden generar diferentes expresiones para el coeficiente de endogamia de SinL'CS (FSinL'CS) mediante descomposiciones de su miembro derecho en partes que faciliten el cálculo, la interpretación y/o el manejo de este coeficiente de endogamia. Por ejemplo, la descomposición se puede hacer en partes que incluyan: 1) subpoblaciones derivadas de un mismo tipo de progenitores, y 2) subpoblaciones generadas por un mismo tipo de evento reproductivo (por ejemplo, por autofecundación, cruzas intrapaternales y cruzas interpaternales). Cada uno de estos procedimientos para determinar el coeficiente de endogamia del sintético debe producir el mismo resultado, pero las diferencias permiten observar y estudiar las contribuciones que hacen a este coeficiente los diferentes componentes del sintético.
La determinación de FSinL'CS, de acuerdo con Sahagún et al. (2005), se basó en la expresión que resulta de la sustitución del genotipo ApikAqjl del arreglo genotípico del sintético (Ecuación 2) por la coancestría entre los dos individuos que aportan sendos genes, Apik y Aqjl. Esta coancestría es la probabilidad (P) de que Apik y Aqjl sean idénticos por descendencia (≡) [P(Apik = Aqj)].
Se consideró que la mejor combinación de valores de L y s es la que produce el SinL'CS cuyo FSinL'CS es el menor porque, de acuerdo con Busbice (1970), hay una relación inversa entre FSinL'CS y la media genotípica de la variedad sintética.
DERIVACIÓN DE RESULTADOS Y DISCUSIÓN
El coeficiente de endogamia de una variedad sintética derivada de L lineas y s híbridos de cruza simple (FSinL'CS) puede ser expresado como una combinación lineal de los coeficientes de endogamia de dos variedades sintéticas, una construida con las L lineas (FSinL) y la otra con las s cruzas simples (FSincs), v el de la coancestría promedio entre líneas e híbridos (ΓLxCS). En efecto, de la descomposición del arreglo genotipico (Ecuación 2) acorde con esta combinación lineal y de su adecuación al coeficiente de endogamia, resulta que

Lo que se ha hecho para llegar a la Ecuación 3 también se puede interpretar como una descomposición de la coancestría promedio de los individuos que por apareamiento aleatorio producen la VS. Además, de la identificación de FSinL y FSinSC en la Ecuación 3, resulta la fórmula

Similarmente, FSinL'SC puede expresarse en términos del coeficiente de endogamia promedio de las poblaciones que resultan del apareamiento aleatorio de los m individuos de cada progenitor (FPAA) y de la coancestría de las (L+s) (L + s – 1) cruzas directas y recíprocas entre los L + s progenitores Γcp . Así, de la Ecuación 2 y de su adecuación al FSinL'CS, expresado como una combinación lineal de FPAA y de Γcp resulta que

Además, el coeficiente de endogamia de cada variedad sintética en la Ecuación 4 se puede descomponer en las partes que involucran las progenies producidas por: 1) autofecundación, 2) cruzas entre individuos de cada progenitor, y 3) cruzas entre individuos de diferentes progenitores (Busbice, 1970; Márquez–Sánchez, 1992; Sahagún–Castellanos, 1994). Con base en esta descomposición adicional, FSinL'CS, además de (coancestría promedio entre líneas y cruzas), incluye tres Γcp términos relacionados con las líneas:
FLA: Coeficiente de endogamia promedio de la población producida por autofecundación de los Lm individuos que representan a las líneas.
ΓWL: Coancestría promedio entre dos individuos que pertenecen a una misma línea.
ΓBL: Coancestría promedio entre dos individuos que pertenecen a líneas diferentes.
Otros tres términos que incluye FSinL'CS son los que involucran a los individuos de las cruzas simples (CSs) y son análogos a FLA, ΓWL y ΓBL; es decir, son: FCSA, ΓWCS y ΓBCS.
De acuerdo con las dos descomposiciones de los primeros dos términos de la Ecuación 4, FSinL'CS también puede expresarse como
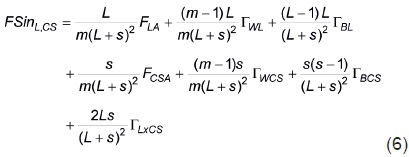
En el caso particular de importancia práctica en que los L + 2s líneas que forman los L + s progenitores son líneas homocigóticas no emparentadas, los términos de la Ecuación 6 toman los valores numéricos siguientes:

De acuerdo con estos resultados (Ecuaciones 6 y 7), FSinL'CS se puede expresar como
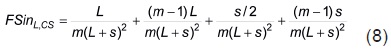
Si en la Ecuación 8 se suman el primer y segundo términos y, por separado, los dos restantes se genera la expresión siguiente:

En la Ecuación 9, [L/(L + s)]2 y [s/(L + s)]2 son las proporciones con que SinL y SinCS participan en SinL'CS (Ecuación 4), y 1/L y 1/(2s) son sus coeficientes de endogamia (Busbice, 1970; Sahagún y Villanueva, 1997).
Respecto al coeficiente de endogamia de las L + s poblaciones producidas por el apareamiento aleatorio de los m individuos de cada progenitor (Ecuación 5), la coancestría entre dos individuos es 1 si el progenitor es una línea y 1/2 si es una cruza simple. por estos resultados,

Por la Ecuación 10 y porque la coancestría entre progenitores, el otro componente de FSinL,CS según la Ecuación 5, es cero (ΓCP = 0), la Ecuación 5, para las condiciones que abarca la Ecuación 7, se reduce a la forma

Las Ecuaciones 9 y 11 describen el coeficiente de endogamia de una misma variedad sintética (formada por L líneas y s cruzas simples) y, evidentemente, producen el mismo resultado. Interesantemente, con líneas homocigóticas no emparentadas FSinL'CS no depende de m (Ecuaciones 9 y 11). Esto desde luego representa una ventaja, pues se pueden hacer las adecuaciones en términos del tamaño de m que la disponibilidad de recursos del programa de mejoramiento y objetivos requieran.
De la expresión FSinL'cs= (2L + s)/[2(L + s)2] (Ecuación 11) se desprende que el número de líneas (L) es más importante que el número de híbridos de cruza simple (s) en la determinación del valor del coeficiente de endogamia del sintético. Una línea contribuye a FSinL'CS el doble de lo que contribuye una cruza simple de la misma variedad sintética. Una explicación de la contribución relativa de las líneas y las cruzas, que involucra argumentos genéticos, se basa en que cada progenitor (línea o cruza simple) está representado por un mismo número de individuos (m) y que el apareamiento es aleatorio. De esto se debe esperar que los números de autofecundaciones y cruzas entre individuos de un mismo progenitor sean iguales, independientemente de que éste sea línea o cruza simple. Además, como las L + 2s líneas originales son homocigóticas, la coancestría entre dos individuos de una misma línea (o el coeficiente de endogamia de su progenie) es 1 y la de dos individuos de una misma cruza simple es 1/2. Similarmente, la probabilidad de identidad por descendencia en todos los genotipos producidos por autofecundación (que es la coancestría de un individuo consigo mismo) es 1 si los individuos representan a una línea y es 1/2 si los individuos representan a una cruza simple. Resumiendo, como las autofecundaciones y las cruzas entre individuos de una misma línea son iguales en número a las que ocurren en una cruza simple, pero el coeficiente de endogamia de las progenies producidas en las líneas (1) es el doble que el de las progenies producidas en las cruzas (1/2), la contribución de las líneas al coeficiente de endogamia de una misma variedad sintética es el doble de la de las cruzas simples. Esto es importante porque los sintéticos más rendidores, según la ecuación de predicción de Busbice (1970), y el conocimiento empírico, suelen ser los que tienen los coeficientes de endogamia más pequeños.
Respecto a las magnitudes del coeficiente de endogamia, la Ecuación 11 implica que los dos sintéticos extremos [los dos formados por progenitores de un mismo tipo; sólo líneas (s = 0) o sólo cruzas simples (L = 0)] tienen coeficientes de endogamia que son iguales [FSinL = 1/L and FSinsc = 1/(2s)]. Esto se debe a que en estos dos casos las frecuencias de genes son iguales, y el apareamiento aleatorio, en consecuencia, produce el mismo arreglo genotípico en los dos sintéticos. Por estos resultados, para un número par de L + 2s líneas las dos VSs extremas deben, ambas, tener un coeficiente de endogamia igual a 1/ (L + 2s) . En cambio, para una VS formada por L líneas y s cruzas simples, el coeficiente de endogamia (Ecuación 11) es (2L + s)/[2(L + s)2]. También se puede encontrar que:
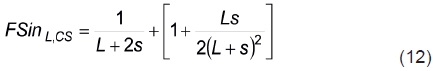
De acuerdo con la Ecuación 12 y los resultados relativos a las dos VSs extremas, es claro que para un número fijo de líneas FSinL'CS es mayor cuando L > 0 y s > 0 que en los casos de las VSs extremas. Esto significa que las VSs extremas, cuyos coeficientes de endogamia son iguales, son las de menor coeficiente de endogamia y presumiblemente las de mayor media genotípica. Por ejemplo, con ocho líneas y con base en los coeficientes de endogamia, los dos mejores diseños de progenitores son: 1) L = 8 y s = 0, y 2) L = 0 y s = 4. Con éstos, según la Ecuación 11, FSinL'SC = 0.125 (o bien FSinL = 1 / L = 0.125. Para el resto de diseños de progenitores que incluyan las ocho líneas, los coeficientes de endogamia son mayores y diferentes. Por ejemplo: 1) L = 6 y s = 1 (0.1327), 2) L = 4 y s = 2 (0.1378), y 3) L = 2 y s = 3 (0.1400).
Respecto a la predicción, si bien la fórmula de Busbice (1970) permite predecir y comparar las medias genotípicas de sintéticos formados con diferentes diseños de progenitores basados en un mismo conjunto de líneas, su valor predictivo depende del grado de realismo de la suposición de que la relación entre la media genotípica y FSinL'CS es lineal.
CONCLUSIONES
Para variedades sintéticas formadas con el apareamiento aleatorio de L líneas y s cruzas simples se derivaron dos fórmulas para su coeficiente de endogamia (FSinL'CS) basada en la descomposición de la coancestría promedio de los individuos que por apareamiento aleatorio producen el sintético en partes de facilidad interpretativa y/o manejo. Se derivó la fórmula
FSinL,CS= [L(L + s)]2FSinL + [s (L + s)]2FSinCS + [2Ls (L + s)/(2s)2]ΓLxCS
; en donde FSinL y FSinCS son el coeficiente de endogamia del sintético formado con las L líneas y el del formado con las s cruzas, respectivamente, y ΓLXCS es el coeficiente de coancestría promedio entre líneas y cruzas simples. Para líneas puras no emparentadas, se llegó a la expresión FSinL,CS = {1 + Ls [2(L + s)2]} (L + 2s). Esta fórmula implica que FSinL'SC alcanza su mínimo cuando el sintético es formado con sólo líneas (s = 0) o con sólo cruzas simples (L = 0), que es cuando las frecuencias génicas de los progenitores son iguales. Sin embargo, cuando los progenitores son sólo cruzas simples la predicción demanda menos recursos y constituye el mejor diseño de progenitores.
LITERATURA CITADA
BUSBICE, T. H. 1970. Predicting yields of synthetic varieties. Crop Science 10: 260–269. [ Links ]
KUTKA, F. J.; SMITH, M. E. 2007. How many parents give the highest yield in predicted synthetic and composite populations of maize? Crop Science 47: 1905–1913. [ Links ]
MÁRQUEZ–SÁNCHEZ, F. 1992. Inbreeding and yield prediction in synthetic maize cultivars made with parental lines: Basic methods. Crop Science 32: 271–274. [ Links ]
MARTÍNEZ–SOLÍS, J.; PEÑA–LOMELÍ, A.; RODRÍGUEZ–PÉREZ, J. E.; VILLANUEVA–VERDUZCO, C.; SAHAGÚN–CASTELLANOS, J. 2005. Comportamiento productivo en híbridos y en sus respectivas poblaciones F2. Revista Chapingo Serie Horticultura 11(2): 299–307. [ Links ]
MENDOZA DE JESÚS, V.; SAHAGÚN–CASTELLANOS, J.; RODRÍGUEZ–PÉREZ, J. E.; LEGARIA–SOLANO, J. P.; PEÑA–LOMELÍ, A.; PÉREZ–GRAJALES, M. 2010. Heterosis intervarietal de jitomate (Lycopersicon esculentum Mill.) tipo saladete indeterminado. Revista Chapingo Serie Horticultura 16(1): 57–66. [ Links ]
SAHAGÚN–CASTELLANOS, J. 1994. Sobre el cálculo de coeficientes de endogamia de variedades sintéticas. Agrociencia serie Fitociencia 5(2): 67–78. [ Links ]
SAHAGÚN C., J.; VILLANUEVA V., C. 1997. Teoría de las variedades sintéticas formadas con híbridos de cruza simple. Revista Fitotecnia Mexicana 20: 60–80. [ Links ]
SAHAGÚN CASTELLANOS, J.; RODRÍGUEZ PÉREZ, J. E.; PEÑA LOMELÍ, A. 2005. Predicting yield of synthetics derived from double crosses. Maydica 50(2): 129–136. [ Links ]
VILLANUEVA V., C.; SÁNCHEZ DEL CASTILLO, F. G.; MOLINA G., J. D. 1994. Aprovechamiento de cruzamientos dialélicos entre híbridos comerciales de maíz: Análisis de progenitores y cruzas. Revista Fitotecnia Mexicana 17: 175–185. [ Links ]
WRIGHT, S. 1922. The effects of inbreeding and cross–breeding of guinea pigs. Tech. Bull. U.S. Dep. Agric. 1112. Washington D.C. [ Links ]




![Cultivo in vitro de pitayo (Stenocereus stellatus [Pfeiffer] Riccobono)](/img/es/prev.gif)









