Servicios Personalizados
Revista
Articulo
Indicadores
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista Chapingo. Serie horticultura
versión On-line ISSN 2007-4034versión impresa ISSN 1027-152X
Rev. Chapingo Ser.Hortic vol.14 no.3 Chapingo sep./dic. 2008
Relación genética de materiales experimentales de chile tipo chilaca con variedades comerciales
Genetic relationship between experimental long chilaca–type chili pepper materials and commercial varieties
M. A. Hermosillo–Cereceres1, J. González–García1*, S. J. Romero–Gómez2, M. Luján–Favela3; A. Hernández–Martínez1 y S. Arévalo–Gallegos4
1 Facultad de Ciencias Agrícolas y Forestales. Universidad Autónoma de Chihuahua. Km. 2.5 Carretera Delicias–Rosales. Cd. Delicias, Chihuahua. C. P. 33000. México. Correo–e: juvgonza@uach.mx (*Autor responsable).
2 Médica Fértil. Constituyentes # 218 Col. El Jacal, Querétaro, Querétaro. C. P. 76180 México. Correo–e: serrom@mac.com
3 Instituto Nacional de Investigación Forestal, Agrícola y Pecuaria. Km. 2 Carretera Delicias–Rosales. Cd. Delicias, Chihuahua. C. P. 33000. México. Correo–e: lujanfm@yahoo.com.mx
4 Facultad de Ciencias Químicas. Universidad Autónoma de Chihuahua, Chihuahua. México. Correo–e: sareval@uach.mx
Recibido: 14 de febrero, 2006
Aceptado: 5 de junio, 2008
Resumen
Utilizar variedades extranjeras de chile tipo chilaca ha traído como consecuencia altos costos en la producción y limitaciones para su aceptación, debido entre otras cosas, a la carencia de sabor y picor agradable. Buscando mejorar esas características, se desarrolló un programa de mejoramiento genético a partir de tres variedades comerciales (Colegio 64, Negro y Sandía), donde se obtuvieron 11 materiales que reúnen las características que exigen los consumidores. Para conocer la relación genética de estos materiales con sus progenitores, se utilizó la técnica denominada PCR–RAPDs. Se probaron 20 oligonucleótidos y se encontró que cinco de éstos presentaron polimorfismos entre las tres variedades comerciales. Posteriormente, estas variedades se compararon con los 11 genotipos utilizando los oligonucleótidos seleccionados. La relación genética de los once materiales con las tres variedades comerciales se determinó mediante el índice de Jacard, encontrando un alto grado de similaridad (86%), mostrando distancias genéticas que varían entre 0.825 y 1 para el grupo uno (dos genotipos) y entre 0.083 y 0.101 para el grupo dos (12 genotipos).
Palabras clave: Chilaca, RAPD–PCR, oligonucleótido, polimorfismo, distancia genética.
Abstract
The use of foreign varieties of chilaca–type chili peppers has had negative consequences: high production costs and limited acceptance primarily because of a lack of a pleasant spicy flavor. To improve these characteristics, a plant breeding program was developed with 3 commercial varieties (Colegio 64, Negro and Sandía), from which 11 materials were obtained with characteristics accepted by consumers. To determine the genetic relationship of these materials with their parents, the PCR–RAPDs technique was used. Twenty oligonucleotides were tested and five of these exhibited polymorphism among the three commercial varieties. Later, these varieties were compared with 11 genotypes using the selected oligonucleotides. The genetic relationship of the 11 materials to the three commercial varieties was determined by the Jacard index. Results showed that 86% of the studied materials had a high degree of similarity, with genetic distances that varied from 0.825 to 1 for group one (two genotypes) and from 0.083 to 0.101 for group two (twelve genotypes).
Key words: Chilaca, RAPD–PCR, oligonucleotide, polymorphism, genetic distance.
INTRODUCCIÓN
Entre los diversos géneros existentes de la familia de las solanáceas, el de Capsicum tiene gran importancia. Se produce tanto en regiones tropicales como subtropicales y templadas, encontrándose desde 0 a 2,500 m. Dado que se adapta a una gran diversidad de áreas agroecológicas, se le puede encontrar en el mercado durante todo el año (Pozo et al., 1991). México es el segundo productor de chile (Capsicum annuum L.). El estado de Chihuahua en los últimos años ha incrementado su producción, aportando el 32% del volumen total nacional. Específicamente, la producción de chile chilaca equivale al 31.4% a nivel estatal, y de ésta, la variedad Negro representa 0.1% (SAGARPA, 2002). En Chihuahua se distinguen tres zonas productoras de chilaca: el norte donde se siembran 4,050 ha con una producción de 80,520 t, de las cuales el 30% son para mercado de exportación; el centro con 400 ha, produce 1,200 t, y; el sur, con 2,140 ha, aporta 4,680 t. Estas dos últimas áreas productoras destinan la producción al mercado nacional. La producción de chile en la zona norte, presenta una diversificación de tipos, donde el 20% es de jalapeño y 67% de chilaca (SAGARPA, 2002). Se sabe que la mayoría de los materiales genéticos de chile chilaca que actualmente se utilizan en Chihuahua, son procedentes del extranjero (González et al., 2005). Lo anterior origina un incremento del costo de producción, y la aparición en el mercado de materiales con características diferentes a las preferidas por los consumidores. En el noroeste de Chihuahua, los productores introdujeron del extranjero algunos genotipos de chile tipo chilaca. Estos se produjeron por veinte años, y al obtenerse la semilla año tras año, se lograron nuevas variantes con mejor adaptación a la región. En general, para esta región se pueden identificar tres tipos: a) tipo chilaca Colegio 64, usado para mercado en fresco; b) tipo Sandía, empleado para mercado fresco y para deshidratado, y; c) el tipo Negro (criollo no importado), que se utiliza para deshidratado. Estos tipos de chile son muy demandados por productores y consumidores regionales, aunque presentan problemas de heterogeneidad, bajos rendimientos y susceptibilidad a enfermedades, sobre todo radicales. Lo anterior condujo a establecer un programa de mejoramiento genético de chilaca. Se partió de los materiales extranjeros y criollos de la región de Buenaventura, Chih., a los cuales se les aplicó selección familial de medios hermanos para formar varios materiales genéticos. Estos han mostrado características sobresalientes en rendimiento y otras que son aceptadas por el productor y el consumidor. De ahí que es importante asegurar la identidad genética de dichos materiales para conservar su autenticidad. Los marcadores genéticos moleculares, como los RAPD (Amplificación al azar de ADN Polimórfico), se han utilizado para la identificación de variedades cultivadas (Koller et al., 1993; Warburton y Bliss, 1996), como en soya para conocer la relación genética entre cultivares (Thompson et al., 1998); Martínez et al. (2004) y Tapia y Legaria (2007) también utilizaron esta técnica para determinar la diversidad y variabilidad genética en árboles de guayabo, y Sánchez et al. (2003) la emplearon para diferenciar líneas y su híbrido en tomate. En el caso particular de las variedades experimentales de chilaca evaluadas, el uso de marcadores moleculares permite conocer qué características comparten éstas con sus progenitores y entre ellas mismas. A partir de ahí se puede programar un plan de cruzamientos para incorporar aquellos caracteres de interés económico, adaptabilidad, calidad y sanidad demandante por el consumidor. A este nivel, es muy importante determinar la relación genética entre todos estos materiales para que dicho programa de cruzas sea de impacto. Con base en lo anterior, el objetivo de este trabajo fue obtener la relación genética de las 11 variedades experimentales y sus progenitores mediante el uso de marcadores moleculares conocidos como RAPD.
MATERIALES Y MÉTODOS
Material vegetal
Se emplearon catorce materiales genéticos de chilaca, de los cuales 11 fueron derivados de una serie de cruzamientos entre las variedades de chile tipo chilaca: Colegio 64 y Sandía de origen estadounidense y Negro, material criollo de Buenaventura, Chih. A partir de los cruzamientos, los 11 materiales genéticos se mejoraron genéticamente a través de un proceso de selección familial de medios hermanos durante tres ciclos que se llevaron a cabo entre 1999 y 2003. Estos materiales fueron seleccionados básicamente por tamaño y forma de fruto, así como por su grado de picosidad (Cuadro 1).
Extracción y cuantificación de ADN
Para extraer ADN se utilizó el método CTAB (bromuro de cetil–trimetil amonio). Por cada muestra de material vegetativo se tomaron 10 hojas con un peso promedio de 0.10 g. Posteriormente se depositaron en un tubo Eppendorf de 1.5 ml y se maceraron con nitrógeno líquido hasta obtener un polvo fino, que se suspendió en 500 ml de búfer CTAB nuclear (50 mM EDTA, pH 8.0; 200 mM Tris–HCl, pH 8.0; 2.0 M NaCl, 2% (w/v); bromuro de cetil–trimetil amonio, 0.5% (w/v); PVP, Polyvinilpyrrolidona) y se mezcló con un micropistilo. Las muestras se incubaron a 60 °C por 30 minutos con inversión lenta cada 7 min. Concluido este paso se dejaron reposar a temperatura ambiente por 10 min. Se agregaron 250 μl de cloroformo alcohol: isoamílico (24:1). Se mezcló por inversión lenta y constante durante 10 min y se centrifugó por 5 min a 10,000 rpm en una microcentrífuga BIO–RAD modelo 14K. Se transfirió el sobrenadante a un tubo nuevo y se agregaron 50 μl al 10% (w/v) de CTAB 0.7 M NaCl. Se hizo por segunda vez la extracción de cloroformo alcohol:isoamílico, seguida por centrifugación a10 000 rpm por 5 min e inversión lenta y constante durante 10 min. La fase acuosa se transfirió a un tubo nuevo y se mezcló con 1 ml de etanol al 100%. El ADN se precipitó a 4 °C por 30 min y después se lavó con 1 ml de etanol al 70% y se reposó por una hora a 4 °C. Se centrífugo el ADN para recuperarlo (5 min a 10,000 rpm) y se lavó por segunda vez en 1 ml de etanol frío al 70%, se mezcló por inversión lenta y constante por 5 min. Posteriormente se centrifugó por 2 min a 10,000 rpm. El pelet de ADN se secó al aire y se disolvió en 50 μl de búfer 1X TE (10 mM Tris–HCl, pH 8.0; 1 mM EDTA, pH 8.0) y 1 μl de ARNasa a una concentración de 100 μg·ml–1. La solución de ADN se diluyó en búfer 1X TE hasta alcanzar la concentración de trabajo de 10 ng·μl–1 y almacenado a 4 °C. El ADN se cuantificó con un espectrofotómetro ultravioleta UV– visible (Jenway 6405 uv/vis), utilizando una celda de cuarzo donde se colocó TE como diluyente, involucrando dos lecturas: una a 260 nm y otra a 280 nm para determinar la concentración y pureza (lectura a 260 nm/lectura a 280 nm).
Análisis de los RAPDs
La amplificación del ADN se realizó en un termociclador Gene AmpR PCR System 9700 (PE Biosystems; Foster City, California) con el siguiente programa: 1 ciclo de 5 min a 94 °C (desnaturalización); 45 ciclos de 1 min a 94 °C (desnaturalización), 2 min a 37 °C (anillamiento de los oligonucleótidos) y 2 min a 72 °C (extensión), y; 7 min a 72 °C para asegurar el proceso de extensión. Al finalizar el protocolo, las muestras se mantuvieron a 4 °C. Por cada reacción de PCR, se obtuvo un volumen de 25 ml conteniendo: 2.5 μl de Buffer Stoffel de Polimerasa Amplitaq [1x], 3.3 μl de MgCl2 [4 mM], 2.5 μl de dNTPs [0.2 mM], 0.2 μl de Taq polymerasa [1u], 2.5 μl de oligonucleótido simple [0.2 μM], 3 μl de ADN [10 ng·μl–1]. Los productos de amplificación se sometieron a electroforesis de agarosa al 1% (peso/volumen) a 150 voltios por 75 min en una cámara de electroforesis BIORAD Sub–Cell Modelo 192. El amortiguador utilizado fue 1X TBE (0.090 M Tris–borate, 0.01 M EDTA). Previo a la visualización de los fragmentos, los geles se tiñeron con una solución de bromuro de etidio (0.5 mg·ml–1) y visualizados en un transiluminador de luz UV (BIO RAD 2000) y fotografiado con cámara Canon y películas Kodak. Se probaron 20 oligonucleótidos (decámeros, Invitrogen Custom Primers) con una concentración mínima de 60% G+C. Toda reacción se corrió por duplicado para asegurar reproducibilidad.
Construcción de la matriz de datos
Las bandas polimórficas fueron consideradas como presentes o ausentes en una matriz de similaridad con los datos obtenidos de la técnica de Jacard (Swofford y Olson, 1990).
Desarrollo del Dendrograma
Se utilizó el método de agrupación promedio UPGMA (Unweighted Pair–Group Method Arithmetic average, por sus siglas en inglés) determinado mediante análisis Cluster con el Software StatistiXL versión Excel, obtenido a prueba por 30 días para análisis de los datos.
RESULTADOS Y DISCUSIÓN
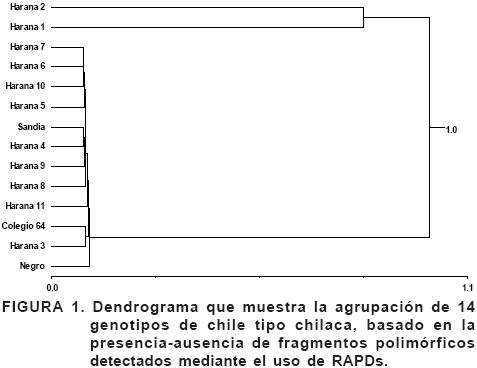
De los 20 oligonucleótidos decámeros probados, cinco detectaron polimorfismos entre Colegio 64, Negro y Sandía. En total se generaron 47 bandas polimórficas, distribuidas según los oligonucleotidos: MFG–04 (5 bandas), MFG–06 (8 bandas), MFG–11 (16 bandas), MFG–17 (12 bandas), MFG–18 (6 bandas). Estas bandas se compararon con el resto de los genotipos involucrados en el estudio por medio de la técnica de Jacard, a partir de ésta, se hizo la matriz de similaridad que se ajustó al método de agrupación promedio UPGMA. De la matriz de distancias genéticas en la que se encontró 86% de similaridad en los genotipos evaluados se elaboró un dendrograma (Figura 1) donde se observa la formación de dos grandes grupos. En el grupo 1 se ubican los materiales Harana 1 y Harana 2 con distancias genéticas de 0.825 a 1, mismos que resultaron los más distantes del grupo 2, pero muy parecidos entre sí. Esto último se atribuye básicamente a que H1 y H2 se derivaron de una cruza entre Negro y Colegio 64, porque el productor procuró reunir las características de estas dos variedades en un lote aislado. En el segundo grupo aparecen los Haranas 3, 4, 5, 6, 7, 8, 9, 10, 11 y las variedades Colegio 64, Negro y Sandía con distancias genéticas entre 0.083 y 0.101, indicando que son los que tienen mayor similaridad entre ellos. En este grupo se localizan los materiales genéticos que aparentemente poseen características de las tres variedades comerciales en igual proporción de cruzamiento, y derivados en comunidades muy cercanas entre ellos. Al hacer el análisis de los 14 genotipos con base en su distancia genética, se generaron 13 grupos (Cuadro 2) con distancias genéticas que variaron de 0.083 a 1. En un estudio de diversidad genética de Pitahaya, las poblaciones que tenían un origen común presentaron un menor grado de divergencia genética (Legaria y Alvarado, 2005). En el caso de las variedades comerciales de chilaca, en el campo se siembran muy cercanas una de la otra, provocando mezcla de genes, consecuentemente se genera gran similitud entre su progenie. La información correspondiente a los 20 oligonucleótidos utilizados en el estudio se muestra en el Cuadro 3. Sólo se menciona lo obtenido con los iniciadores MFG–17 y MFG–18 para diferenciar genes de las tres variedades comerciales. Sin embargo, se anexan las Figuras 4, 5 y 6 correspondiente a los iniciadores MFG–04, MFG–06 y MFG–11 que mostraron la misma tendencia de MFG–17 y MFG–18. Los cinco oligonucleótidos en conjunto fueron la base de este trabajo de investigación, por ser los únicos que mostraron polimorfismo en al menos un par de fragmentos de ADN de genotipos diferentes.



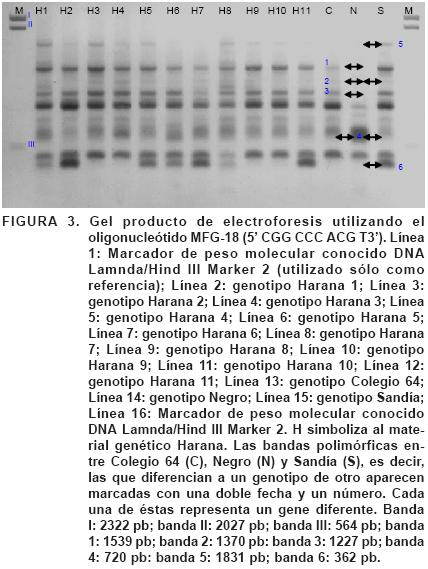

El análisis de los polimorfismos entre las tres variedades comerciales (Colegio 64, Negro y Sandía) con el oligonucleótido MFG–17 (Figura 2) detectó 12 fragmentos polimórficos: los fragmentos 1 y 2 permiten diferenciar a Colegio 64 de Negro; el fragmento 3 hace la diferencia entre Colegio 64 con Negro y Sandía; los fragmentos 4, 5, 6 y 7 diferencian a Negro de Colegio 64 y de Sandía; el fragmento 8 permite diferenciar a Sandía tanto de Negro como de Colegio 64, y; los fragmentos 9, 10, 11 y 12 hacen la diferencia entre Sandía y Negro. Los fragmentos indicativos son: el 3 para Colegio 64, ya que sólo se encuentra en H1, H4, H8 y H11; el fragmento 5, que se encuentra en Negro, se localiza en H2, H3, H4, H7 y H8, por lo que este oligonucleótido ayuda a identificar los genotipos que presentan más genes de Negro; el fragmento 6 es indicativo para Negro, ya que está presente en Negro y en H2, H3 y H5; el fragmento 8 permite identificar aquellos genotipos donde hay genes de Sandía como lo son H1, H2 y H8. En la Figura 3 se presenta el análisis del oligonucleótido MFG–18. Se encontraron fragmentos polimórficos entre las tres variedades comerciales (Colegio 64, Negro y Sandía). Los fragmentos 1, 2 y 3 permiten diferenciar a Colegio 64 de Negro. El fragmento 4 diferencia a Negro de Colegio 64 y de Sandía. Los fragmentos 5 y 6 permiten diferenciar a Sandía de Colegio 64 y Negro. El fragmento 5 localizado en Sandía, también está presente en H1, H3, H4, H5, H8, H9 y H10. Esto refleja que dichos genotipos tienen genes distintivos de Sandía. Por otro lado, el fragmento 6 indicativo también para Sandía, se encuentra en H1, H2, H5, H6, H7, H8 y H11, lo que permite confirmar que los 11 genotipos poseen genes de Sandía. Este oligonucleótido [MFG–18 (5' ..... CGGCCC ACG T ..... 3')] es útil para determinar la relación filogenética entre los últimos materiales experimentales con Sandía. Los resultados obtenidos son útiles para relacionar a líneas progenitoras con su progenie (Figuras 3, 4, 5 y 6). Trabajos similares que muestran las bondades del uso de marcadores moleculares tipo RAPD, han sido reportados por Tapia et al. (2005) quienes realizaron una caracterización genética de piña en Veracruz, Vidal et al. (2006) al determinar la variabilidad genética de siete cultivares criollos y 14 cultivares mejorados de frijol común (Phaseolus vulgaris L.) de la Chontalpa, Tab., Alfaro et al. (2007) quienes determinaron la diversidad genética entre y dentro de poblaciones de maguey pulquero del Nororiente del Estado de México, García et al. (2007) en sus estudios de variación patogénica y molecular de 16 aislamientos de Puccinia horiana colectadas en los estados de México, Morelos y Puebla, entre otros.


CONCLUSIONES
Los marcadores moleculares utilizados que arrojaron bandas polimórficas, permitieron diferenciar a los 11 genotipos experimentales entre sí e identificar la participación de genes de las tres variedades comerciales progenitoras en los materiales experimentales. Se construyeron dos grupos de genotipos. Su relación genética posiblemente está asociada con el grado de aislamiento manipulado por parte de los productores.
LITERATURA CITADA
ALFARO R., G.; LEGARIA S., J. P.; RODRÍGUEZ P, J. E. 2007. Diversidad genética en poblaciones de agaves pulqueros (Agave spp.) del nororiente del Estado de México. Rev. Fitotecnia Mexicana. 30(1): 1–12. [ Links ]
GARCÍA V., R.; ZAVALETA M., E.; ROJAS M., R. I.; LEYVA M., S. G.; FUENTES A., G.; SIMPSON, J. 2007. Variación patogénica y molecular de aislamientos de Puccinia horiana Henn. Rev. Fitotecnia Mexicana. 30(3): 223–234. [ Links ]
GONZÁLEZ G., J.; ACOSTA R., G. F.; LUJAN F., M. 2005. Selección de genotipos para la zona árida y semiárida de México: II chilaca, pp.106–110. XI Congreso Nacional de la Sociedad Mexicana de Ciencias Hortícolas. Chihuahua, Chih., México. [ Links ]
KOLLER, B.; LEMÁN, A.; MCDERMOTT, J. M.; GESSLER, C. 1993. Identification of apple cultivars using RAPD markers. Theor. Appl. Genet. 85: 901–904. [ Links ]
LEGARIA S., J. P.; ALVARADO, C. M. E. 2005. Diversidad genética en pitahaya (Hylocereus undatus Haworth. Britton y Rose). Rev. Fitotecnia. Mexicana. 28(3):179–185. [ Links ]
MARTÍNEZ, D. J.; BARRIENTOS L., M. C.; REYES D., A. C.; HERNÁNDEZ D., S.; PADILLA R., J. S.; MAYEC P, N. 2004. Diversidad fenotípica y genética en huertas de guayabo de Calvillo, Aguascalientes. Rev. Fitotecnia Mexicana. 27(3): 243–249. [ Links ]
POZO C., O.; MONTES, S.; RENDÓN, E. 1991. Chile (Capsicum spp.), pp. 217–238. In: Ortega, R.; Palomino, G.; Castillo, F.; González, V.A.; Livera M., M. (eds.). Avances en el estudio de los recursos genéticos de México, Chapingo, Edo. de México. México. Sociedad Mexicana de Fitogenética. [ Links ]
SAGARPA. 2002. Anuario estadístico. Sistema de información de estadísticas agropecuarias. http.www.sagarpa.mx. (Revisado en Junio del 2005). [ Links ]
SÁNCHEZ A., C.; VALADEZ M., E.; CARBALLO C., A.; CASTILLO, F. G. 2003. Diferenciación de dos líneas de tomate y su híbrido con marcadores moleculares Rev. Fitotecnia Mexicana. 26(1): 67–72. [ Links ]
SNEATH, P. H. A.; SOKAL, R. R. 1973. Numerical Taxonomy. The Principles and Practice of Numerical Classification. Freeman & Co. San Francisco. 573 p. [ Links ]
SWOFFORD, D. L.; OLSON, G. J. 1990. Phylogeny reconstruction pp. 411–501 In: Molecular systematics. HILLIS, D. M.; MORTIZ, C. (eds). Sinauer Associates, Sunderland, Massachusetts, USA. [ Links ]
TAPIA C., E.; ANDRADE, H. G.; GUTIÉRREZ E., M. A. 2005. Caracterización genética de piña (Ananas spp.) mediante RAPD e ISSR. Rev. Fitotecnia Mexicana. 28(3): 187–194. [ Links ]
TAPIA, P. D.; LEGARIA S, J. P. 2007. Variabilidad genética en cultivares de guayabo (Psidium guajava L.). Rev. Fitotecnia Mexicana. 30(4): 391–401. [ Links ]
THOMPSON, J. A.; NELSON, R. L.; VODKIN, L. 1998. Identification of diverse soybean germplasm using RAPD markers. Crop Sci. 38(5): 1350. [ Links ]
VIDAL–BARAHONA, A.; LAGUNES–ESPINOZA, L. C.; VALADEZ M., E.; ORTÍZ–GARCÍA, C. F. 2006. Variabilidad morfológica y molecular de cultivares criollos y mejorados de frijol común en Tabasco, México. Rev. Fitotecnia Mex. 29(4): 273–281. [ Links ]
WARBURTON, M. L.; BLISS, F. A. 1996. Genetic diversity in peach (Prunus persica L. Batch) revealed by RAPD markers and compared to inbreeding coefficients. J. Amer. Soc. Hortic. Sci. 121: 1012–1019. [ Links ]














