Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Similars in
SciELO
Similars in
SciELO
Share
Revista de la Sociedad Química de México
Print version ISSN 0583-7693
Rev. Soc. Quím. Méx vol.48 n.4 Ciudad de México Oct./Dec. 2004
Investigación
Síntesis en paralelo de decapéptidos en fase sólida y sus afinidades a cationes de interés biológico
Ignacio A. Rivero,* Tania González y Mario Basterrechea
Centro de Graduados e Investigación. Instituto Tecnológico de Tijuana. Apartado Postal 1166. 22000. Tijuana, Baja California, México. Tel: (664) 6233772, Fax: (664) 6234043. E-mail: irivero@tectijuana.mx
Recibido el 10 de noviembre del 2004.
Aceptado el 10 de diciembre de 2004.
Resumen
El diseño de los péptidos soportados en fase sólida es importante en los campos de la química médica y de reconocimiento molecular, por ejemplo, para determinar el impacto de péptidos en el transporte activo de iones a través de membranas. En el presente trabajo se realizó la síntesis de 20 decapéptidos en paralelo y su análisis para la formación de aductos con iones de interés biológico. Se utilizaron sales de litio, sodio y potasio (A). La caracterización de los aductos formados [M+A]+n se evaluaron utilizando un equipo de espectrometría de masas con ionización por electrospray con un analizador de trampa de iones (EM-IES). Se obtuvieron las señales m/z correspondientes al aducto péptido-metal y se analizaron en un grupo de metales alcalinos. Se reporta la evaluación de la selectividad o afinidad del péptido en las mezclas de multicomponentes metálicos.
Palabras clave: síntesis de péptidos, fase sólida, cationes, afinidad.
Abstract
Peptide design supported on solid phase is important in the fields of medical chemistry and molecular recognition, for example, to determinate de impact of such peptides in the active transport of ions through membranes. In this work were synthesized 20 decapeptides with paralel chemistry and was carried out the analysis of the formation of adducts with litium, sodium and potassium salts (A). The characterization of the formed adducts [M+A]+n was carried out using an equipment of electrospray ionization mass spectrometry (ESI-MS). The method was established to afford the equilibrium with the adducts and alkali metal ions. Signals m/z were obtained corresponding to the adduct metal-peptide. Selectivity of the peptide was evaluated in the mix of metallic multi-components.
Key words: Peptide synthesis, solid phase, cations, affinity.
Dedicado a la memoria del Dr. Raymundo Cruz Almanza, por su gran profesionalismo y amistad.
Introducción
Sintetizar péptidos de secuencia definida es de gran interés por varias razones: los péptidos sintéticos pueden revelar cuando una secuencia provoca la conformación tridimensional de las proteínas, una gran variedad de hormonas de los seres vivos son proteínas, los péptidos pueden utilizarse como fármacos, y sirven como antígenos para la formación de anticuerpos específicos [1].
El avance significativo en la síntesis de péptidos se produjo cuando Merrifield desarrolló el procedimiento para la síntesis en fase sólida [2]. El método de Merrifield se basa en el uso de una resina de poliestireno. Esta resina se emplea en forma de pequeñas esferas y es insoluble en casi todos los disolventes. A diferencia de la síntesis orgánica en solución, la síntesis orgánica en fase sólida (SOFS), permite que las reacciones sean prácticamente cuantitativas en cada etapa y requiere procesos simples de purificación como lavado y filtración [3].
Los aminoácidos se unen escalonadamente a la cadena peptídica creciente, fijada a la matriz polimérica, evitando la necesidad de eliminar intermediarios no deseados. Todas las reacciones se llevan a cabo en el mismo recipiente, lo que elimina pérdidas por transvasado de reactivos. Por lo anterior, el diseño de los péptidos soportados en fase sólida se ha vuelto extremadamente importante en los campos de la química médica y de reconocimiento molecular [4].
Los péptidos tiene la propiedad de transportar metales a través de las paredes celulares, por lo tanto, la evaluación de afinidad a los metales es importante ya que una de las funciones de las proteínas es el transporte así como su activación en procesos de catálisis. Se ha establecido que la adición de iones metálicos puede cambiar la carga en la superficie del amino ácido de la cadena peptídica y en consecuencia, modificar las propiedades de las proteínas y péptidos [5-7]. Los efectos más importantes que causan los iones metálicos en estas biomoléculas son: la estabilización a través de interacciones electrostáticas y el cambio en su solubilidad [6,7].
La técnica recientemente desarrollada de espectrometría de masas con ionización electrospray (EM-IES) ha permitido su uso en el estudio de interacciones moleculares dirigidas por fuerzas no covalentes. La técnica de EM-IES ha sido utilizada para detectar complejos no covalentes que involucran ADN, ARN, oligonucleótidos, medicamentos e interacciones entre proteínas y metales, ligandos y péptidos [8].
Para entender las interacciones célula-célula y las interacciones de células con materiales no biológicos, existen estudios de fuerzas de enlace entre proteínas celulares y entre proteínas y metales, que demuestran que las proteínas tienen un tipo de interacción específica con metales que probablemente es un paso intermediario en el proceso de absorción [9].
La técnica de ionización EM-IES es más sensible para detectar pequeñas cantidades de compuestos. Con el surgimiento de esta técnica, pueden ser estudiados tanto grandes biomoléculas como complejos no covalentes [10]. La función de una biomolécula casi siempre depende de sus interacciones no covalentes específicas con otra molécula. Por ejemplo, una proteína interactúa con otras proteínas, péptidos, pequeñas moléculas, iones metálicos, lípidos, polisacáridos, ácidos nucléicos y oligonucleótidos. Estas interacciones llevan a cabo procesos celulares de gran importancia tales como la división celular, transporte de iones, homeostasis, transcripción y traducción [11]. Además, hay reportes que sugieren que la técnica de EM-IES es capaz de detectar cambios conformacionales [12,13] y medir constantes de ionización [14]. Por otra parte, el método de elección para determinar las secuencias de péptidos es la espectrometría de masas en tandem (EM-IES/EM) [14].
La suavidad de este proceso no causa ninguna fragmentación molecular no deseada y permite que interacciones débiles de tipo no covalente queden intactas durante del proceso de ionización [15]. La capacidad de EM-IES para estudiar complejos no covalentes específicos fue sugerido por primera vez basado en la observación de interacciones entre la mioglobina y el grupo hemo [16], así como un complejo receptor-ligando [17].
La detección de interacciones no covalentes en una disolución involucra la formación de complejos que se encuentran en un equilibrio dinámico. Estas interacciones generan moléculas asociadas que son determinadas por esta técnica, se presentan como la suma [S1+S2+M]+ de sus masas (S) y de un catión (M+n). Cuando el sustrato presenta protones ácidos, entonces se tendrá una preferencia por iones metálicos no monovalentes y la disociación de protones, para que finalmente la carga total del complejo sea de +1 [S1+S2+M+n-Hn-1]+. Además, la medición de la masa permite la identificación del compuesto y la determinación de la estequiometría de los componentes del complejo. Por ejemplo, los aductos de iones metálicos como Ca+2 y Zn+2 con metaloproteínas pueden detectarse por esta técnica [18,19].
Estudios recientes demuestran que la técnica de EM-IES muestra la capacidad de llegar a niveles de concentraciones pico- (10-12 mol) y femtomolar (10-15 mol) [20,21]. Estos niveles de concentración son importantes dado que una gran cantidad de procesos biológicos son requeridos de una evaluación de los iones metálicos como cocatalizadores de procesos fisiológicos y patológicos. Además de que se ha incrementado la evidencia de que los complejos observados utilizando EM-IES son prácticamente iguales a los encontrados en solución.
En este sentido, EM-IES ha desarrollado una herramienta para la evaluación de selectividades de complejos huésped-anfitrión [22-37]. Esta técnica ha sido empleada para evaluar las propiedades como complejantes de éteres Lariat con metales [27-35], y otros éteres corona, así como para determinar las habilidades de éteres aza-corona como complejantes con metales alcalinos y metales pesados mediante la EM-IES [29, 31-33, 35]. Además, esta técnica se ha utilizado para determinar constantes de enlace y la estereoquímica de ensamblajes que involucran proteínas [37].
Discusión de resultados
Así, en el presente trabajo se realizó la síntesis de 20 decapéptidos en paralelo y su análisis para la formación de aductos con sales de litio, sodio y potasio (A); así como la evaluación de la selectividad del péptido en la mezcla de multicomponentes metálicos. La caracterización de los aductos formados [M+A]+n se evaluó utilizando un equipo EM-IES y fueron obtenidos los picos correspondientes al aducto péptido-metal. La respuesta de las intensidad es proporcional a la contribución de cada uno de los aductos formados ya que pueden interaccionar con más de un ion metálico, y se mantiene la concentración constante durante el análisis. Esto es determinante ya que al desolvatar la muestra, los complejos en la fase gaseosa salen del equilibrio dinámico manteniendo su concentración constante [22-35]. Se utilizaron sales en forma de cloruros: litio, sodio y potasio (alcalinos).
Diseño y síntesis de la biblioteca de decapéptidos
La secuencia 1 se tomó como base para el diseño de la biblioteca, para describir la presencia de un aminoácido se puede utilizar el nombre reconocido por la IUPAC (ácido 2-amino acético), el nombre genérico (glicina), tres letras (Gly) o con una letra mayúscula (G) como se muestra en la tabla 1.

Para el diseño de la biblioteca se tomo como base a la estructura original del acarreador de iones (1, VQAAID-YING), se conservó a la glicina (G) en posición inicial y el aminoácido de la posición 2 (N) fue colocado al final de la secuencia base respetando el orden del resto de aminoácidos (Fig. 1)

El tamaño de la biblioteca fue de 20 decapéptidos como se describe en la Tabla 2. De esta manera, conservando el mismo número y tipo de aminoácidos en el péptido original (1) se han modificado las posiciones. Así, el análisis de afinidad a metales y sus posibles variaciones dependerán de la posición de los aminoácidos en cada péptido sintetizado.

Para la síntesis de dichos péptidos se utilizó una resina Wang cargada comercialmente con glicina. Primeramente, se llevó a cabo la expansión de la resina en una mezcla de CH2Cl2/NMP para después adicionar una solución de Fmoc-aminoácido/HOBt, DMAP y DIC en N-metilpirrolidona. La posterior desprotección se realizó con piperidina al 20% en NMP y se hicieron acoplamientos-desprotecciones sucesivas hasta lograr la cadena deseada (Fig. 2). Las reacciones se llevaron a cabo en un equipo de química combinatoria.

Resultados
Evaluación por espectrometría de masas con ionización electrospray
Se muestran los casos representativos seleccionados que ilustran los resultados obtenidos. En los experimentos de afinidad se prepararon dos soluciones salinas con mezclas de LiCl-NaCl-KCl y con una concentración de 1.5 × 10-5 M en metanol. Posteriormente se realizaron las mezclas 1:1 del péptido con cada una de las soluciones salinas y se analizaron después de 30 min.
En el caso del péptido 3, es más favorable la formación de un aducto con Na+ (m/z 1085), que con K+ (m/e 1101), mientras que no existe afinidad alguna para Li+ (Tabla 3). Para el péptido 5, solamente se observa afinidad para Na+.

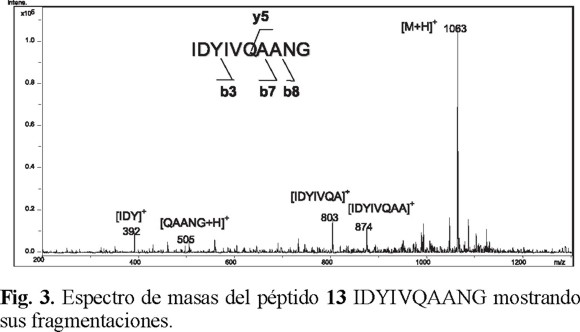
En el caso del péptido 13 (Figs. 3 y 4), el pico base es para el aducto formado con K+, el cual es 10 veces mayor que el ión cuasi-molecular [M+H]+. Asimismo, se presenta la formación de un aducto con dos iones metálicos diferentes de la mezcla (Na+ y K+), m/z 1123, en el que existe el desplazamiento de un protón, formándose el aducto [M-H+Na+K]+ con una sola carga. No se observó la formación de m/z 562, correspondiente a [(M+Na+K)/2]+. Además se observó la formación de un aducto con dos iones K+, [M-H+2K]+. Las relaciones de afinidad para el péptido 13 se presentan en la Tabla 4, observándose que existe una preferencia de más del doble para la formación del aducto con K+, que para la formación de aductos con dos iones metálicos. En este caso no se presentó afinidad alguna para Li+.


En el experimento realizado con el péptido 14, se presenta una mayor afinidad para la formación del aducto con Na+, además de que también se observa la formación de un aducto con dos iones Na+, m/z 1107, en el que se desplaza un protón formándose el aducto [M-H+2Na]+ con una sola carga. No se observó la formación de m/z 554, correspondiente a [(M+2Na)/2]+. También se observó la formación de un aducto con los tres metales de la mezcla, desplazándose dos protones para obtener la formación de [M-2H+Li+Na+K]+ con una sola carga.
En el caso del péptido 18, el pico base se presenta para el aducto formado con Na+, seguido en abundancia del ión cuasi-molecular [M+H]+. También se observa la formación de aductos con dos iones Na+, m/z 1107, con el desplazamiento de un protón. Asimismo, se presenta la formación de un aducto con los tres metales de la mezcla, m/z 1129, desplazándose dos protones para obtener la formación de [M-2H+Li+Na+K]+ con una sola carga.
Conclusión
De acuerdo con los resultados obtenidos en las evaluaciones de afinidades en mezcla de multicomponentes metálicos, se confirmó que el método de EM-IES permite la evaluación de la formación de aductos péptido-metal, el equilibrio dinámico, la afinidad a los metales y el transporte de los iones metálicos estudiados. La secuencia de los aminoácidos también tiene una repercusión con respecto a las afinidades. Se encontró que la estabilización del equilibrio dinámico de estos péptidos se daban en 30 min.
Parte Experimental
Los espectros de infrarrojo (IR) fueron realizados en un espectrofotómetro Perkin Elmer FT-IR 1600. La espectroscopia de resonancia magnética nuclear de protón (1H) y de carbono (13C) fue realizada en un espectrómetro Varian Mercury 200 en CDCl3 con TMS como estándar interno a 200 MHz y 50.289 MHz, respectivamente. La espectrometría de masas y la pureza fueron obtenidas en un equipo acoplado Agilent Series 1100 LC/MSD Trap, columna C-18. La química combinatoria se llevó a cabo en un reactor Advanced ChemTech modelo Vantage.
Procedimiento general de síntesis de decapéptidos en fase sólida.
Wang-decapéptido-OH. En 20 reactores de 3 mL de capacidad se colocó la resina Wang-OH (50 mg, 0.03 mmol), se suspendió en 950 µL de DCM y 100 µL de NMP y se agitó durante tres min a 600 rpm. Luego se agregó 750 µL de una solución con el correspondiente Fmoc-AA-OH (0.036 mmol)/HOBt (0.036 mmol), en NMP. La mezcla se agitó durante 2 min. Finalmente, se adicionaron 100 µL de DMAP (0.003 mmol) y DIC (0.036 mmol) respectivamente, en NMP. La reacción se dejó en agitación durante 3 h. La resina fue filtrada y se realizaron lavados con NMP (1 × 2 mL), MeOH (1 × 2 mL) y DCM (1 × 2 mL). El grupo Fmoc del primer amino ácido unido a la resina se removió usando 20 % de piperidina en NMP (2 mL), y se dejó agitar durante 40 min. Posteriormente se realizaron lavados con DCM (2 × 2 mL). Las adiciones de los aminoácidos subsecuentes se realizaron utilizando las mismas técnicas de acoplamiento y desprotección; las reacciones se llevaron a cabo hasta lograr la cadena decapeptídica.
Liberación del H2N-decapéptido-OH: Para la liberación del producto se utilizó un volumen total de 2 mL de una mezcla de liberación conteniendo TFA-triisopropilsilano-H2O 95:2.5:2.5. Se agitó durante 1.5 h y una vez finalizada la reacción se filtró la resina y el producto liberado se recibió en viales de 3 mL. La mezcla fue transferida a tubos de ensaye de 10 mL y se adicionó éter anhidro frío para precipitar el producto y se dejó por 12 h a 0-4 °C. Finalmente, el sobrenadante fue descartado por decantación y el producto se secó por paso de argón. La identificación de los 20 decapéptidos sintetizados se llevó a cabo por espectrometría de masas.
Preparación de las muestras para estudio de afinidad: Se prepararon dos soluciones salinas con una mezcla de LiCl-NaCl-KCl y una solución del péptido con una concentración de 1.5 × 10-5 M en metanol. Se realizaron las mezclas (1:1) de cada uno de los péptidos con la solución salina y se dejaron reposar por 30 min. Las soluciones fueron analizadas mediante inserción directa en el espectrómetro de masas con ionización electrospray, utilizando una bomba de jeringa (KD Scientific), con una velocidad de 240 µL/h.
Agradecimientos
Agradecemos el apoyo financiero de este proyecto al Consejo Nacional de Ciencia y Tecnología, México (CONACyT, GRANT No. 28488-E) y al Consejo Nacional de Educación Tecnológica, México (COSNET. Apoyo No. 623.97-P). Tania González agradece a CONACyT por su beca.
Referencias
1. Wakselman, M. J. Med. Chem. 1998, 41, 2100-2110. [ Links ]
2. Merrifield, R. B. J. Am. Chem. Soc. 1963, 85, 2149-2154. [ Links ]
3. Rivero, I. A. Avance y Perspectiva 1997, 16, 375-383. [ Links ]
4. Wakselman, M. J. Med. Chem. 1998, 41, 2100-2110. [ Links ]
5. a) Busch, M. R.; Ho, C. E. Biophys. Chemist. 1990, 37, 313-322. [ Links ] b) Chothia, C.; Lesk, A. M.; Dodson, G. G.; Hodgkin, D. C. Nature 1983, 302, 500-505. [ Links ]
6. Cerasoli, E.; Kelly, S. M.; Coggins, J. R.; Lapthorn, A. J.; Clarke, D. T.; Price, N. C. Biochim. Biophys. Acta 2003, 1648, 43-54. [ Links ]
7. a) Goto, Y.; Takahashi, N.; Fink, A. L. Biochemistry 1990, 29, 3480-3488. [ Links ] b) Baldwin, R. L.; Biophys. J. 1996, 71, 2056-2063. [ Links ] c) Imai, T.; Kinoshita, M.; Hirata, F. Bull. Chem. Soc. Jpn. 2000, 73, 1113-1122. [ Links ]
8. Sarni-Manchado, P.; Cheynier, V. J. Mass Spectrom. 2002, 37, 609-616. [ Links ]
9. Eckert, R.; Jeney, S.; Höber, J. K. H. Cell Biology International 1997, 11, 707-713. [ Links ]
10. (a) Merrifield, R. B. Angew Chem. 1985, 97, 801-812; [ Links ] Angew, Chem. Int. Ed. Engl. 1985, 24, 799. [ Links ] (b) Merrifield, R. B. Advan. Enzymol. 1969, 32, 221. [ Links ] (c) Barany G.; Merrifield R.B. The peptides, Vol 2, 3, Gross E.; Meienhofer J. Ed. (Academic Press, Nueva York, 1979). [ Links ]
11. Veenstra, T.D. Biophysical Chemistry 1999, 79, 63-79. [ Links ]
12. Veenstra, T. D.; Johnson, K. L.; Tomlinson, A. J.; Craig, T. A.; Kumar, R.; Naylor, S. J. Am. Soc Mass Spectrom. 1998, 9, 8-14. [ Links ]
13. Veenstra, T. D.; Johnson, K. L.; Tomlinson, A. J.; Kumar, R.; Taylor, S. Rapid Commun. Mass Spectrom. 1998, 12, 613-619. [ Links ]
14. a) Bruce, J. E.; Anderson, G. A.; Chen, R.; Cheng, X.; Gule, D. C.; Hofstadler, S. A.; Schwartz, B. L.; Smith, R. D. Rapid Commun. Mass Spectrom. 1995, 9, 644. [ Links ] b) Ross, A. R.; Ikonomou, M. G.; Orians, K. J. Anal. Chim. Acta 2000, 91-102. [ Links ]
15. Siuzdak, G. Proc. Natl. Acad. Sci. U.S.A. 1994, 91, 11290-11297. [ Links ]
16. Katta, V.; Chait, B. T. J. Am. Chem. Soc. 1991, 113, 8534-8535. [ Links ]
17. Ganem, B.; Li, Y. T.; Henion, J. D. J. Am. Chem. Soc. 1991, 113, 6294-6296. [ Links ]
18. Feng, R.; Castelhano, A. L.; Billedeau, R.; Yuan, Z. J. Am. Soc. Mass. Spec. 1995, 6, 1105-1111. [ Links ]
19. Veenstra, T. D.; Johnson, K. L.; Tomlinson, A. J.; Taylor, S.; Kumar, R. Eur. Mass. Spectrom. 1998, 3, 1105-1112. [ Links ]
20. Fitzgerald, M. C.; Chernushevich, I.; Standing, K. G.; Whitman, C. P.; Kent, S. B. H. Proc. Natl. Acad. Sci. U. S. A. 1996, 93, 6851-6856. [ Links ]
21. Schwartz, B. L.; Bruce, J. E.; Anderson, G. A. J. Am. Soc. Mass Spectrom. 1995, 6, 459-465. [ Links ]
22. Leize, E.; Jaffrezic, A.; Van Dorsselaer, A. J. Mass Spectrom. 1996, 31, 537. [ Links ]
23. Wang, K.; Gokel, G. W. J. Org. Chem. 1996, 61, 4693-4697. [ Links ]
24. Young, D. S.; Hung, H.-Y.; Liu, L. K. J. Mass Spectrom. 1997, 32, 432-437. [ Links ]
25. Blair, S. M.; Kempen, E. C.; Brodbelt, J. S. J. Am. Soc. Mass Spectrom. 1998, 9, 1049-1059. [ Links ]
26. Brodbelt, J. S.; Kempen, E.; Reyzer, M. Struct. Chem. 1999, 10, 213-215. [ Links ]
27. Kempen, E. C.; Brodbelt, J. S.; Bartsch, R. A.; Jang, Y.; Kim, J. S. Anal. Chem. 1999, 71, 5493-5500. [ Links ]
28. Blair, S. M.; Brodbelt, J. S.; Reddy, G. M.; Marchand, A. P. J. Mass Spectrom. 1998, 33, 721-728. [ Links ]
29. Blair, S. M.; Brodbelt, J. S.; Marchand, A. P.; Kumar, K. A.; Chong, H.-S. Anal. Chem. 2000, 72, 2433-2445. [ Links ]
30. Kempen, E.; Brodbelt, J. S. Anal. Chem. 2000, 72, 5411-5416. [ Links ]
31. Blair, S.; Brodbelt, J. S.; Marchand, A. P.; Chong, H.-S.; Alihodzic, S. J. Am. Soc. Mass Spectrom. 2000, 11, 884-891. [ Links ]
32. Reyzer, M. L.; Brodbelt, J. S.; Marchand, A. P.; Chen, Z.; Huang, Z.; Namboothiri, I. N. N. Int. J. Mass Spectrom. 2001, 204, 133-142. [ Links ]
33. Marchand, A. P.; Huang, Z.; Chen, Z.; Hariprakasha, H. K. Namboothiri, I. N. N.; Brodbelt, J. S.; Reyzer, M. L. J. Heterocycl. Chem. 2001, 38, 1361-1368. [ Links ]
34. Williams, S.; Blair, S. M.; Brodbelt, J. S.; Huang, X.; Bartsch, R. A. Int. J. Mass Spectrom. 2001, 212, 389-401. [ Links ]
35. Williams, S. M.; Brodbelt, J. S.; Cal, D.; Marchand, A. P. Anal. Chem. 2002, 74, 4423-4433.
36. Marchand, A. P.; Huang, Z.; Lai, H.; McKim, A. S.; Brodbelt, J. S.; Williams, S. Heterocycles 2004, 62, 279 - 296. [ Links ]
37. Van Dongen, W. D.; Heck, A. J. R. Analyst 2000, 583-589. [ Links ]














