Servicios Personalizados
Revista
Articulo
Indicadores
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Agricultura técnica en México
versión impresa ISSN 0568-2517
Agric. Téc. Méx vol.34 no.1 México ene./mar. 2008
Artículos
Detección de Salmonella y coliformes fecales en agua de uso agrícola para la producción de melón "Cantaloupe"*
Detection of Salmonella and fecal coliforms in water for agricultural use destined to melon"Cantaloupe"
Carmela Hernández–Domínguez1, Ana María Hernández–Anguiano1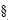 , Cristóbal Cháidez–Quiroz2, Gilberto Rendón–Sánchez3 y Trevor Suslow3
, Cristóbal Cháidez–Quiroz2, Gilberto Rendón–Sánchez3 y Trevor Suslow3
1 Especialidad de Fitopatología, Colegio de Postgraduados. km 36.5 carretera México–Texcoco. Montecillo, Estado de México. C. P. 56230.
2 Área de Microbiología, ISEI. Especialidad de Estadística y Cálculo, Centro de Investigación en Alimentación y Desarrollo.
3 Mann Laboratory, Department of Plant Science, UC Davis.
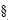 Autora para correspondencia:
Autora para correspondencia:
ahernandez@colpos.mx
* Recibido: Marzo de 2006
Aceptado: Febrero de 2008
RESUMEN
El agua que se utiliza en la producción de cultivos hortofrutícolas representa una fuente potencial de microorganismos que ocasionan enfermedades de transmisión alimentaria. Con el objetivo de evaluar la calidad sanitaria de diferentes fuentes de agua empleadas en la producción de melón Cantaloupe (Cucumis melo L. [grupo reticulatus] cv. Ovación y Caminos), en Zirándaro de los Chávez, Guerrero, se analizaron 71 muestras de agua provenientes de dos unidades de campo (23) y de una unidad de empaque (48) mediante métodos bacteriológicos convencionales para la detección deSalmonella spp, y por el método de filtración en membrana y el crecimiento en medios selectivos, para la detección de coliformes fecales, como indicadores de contaminación fecal. Del total de muestras de agua analizadas sólo tres muestras de campo resultaron positivas a la presencia de Salmonella spp. y nueve muestras, siete de campo y dos de la unidad de empaque, resultaron positivas a coliformes fecales. Salmonella spp. y coliformes fecales se detectaron principalmente en muestras de agua no clorada a 29 °C y 7.5 de pH, en promedio. En pruebas de reacción en cadena de la polimerasa (PCR), dos de cuatro cepas presuntivas de Salmonella ssp. dieron amplificaciones positivas con los iniciadores Sal–3 y Sal–4, y invA–1 e invA–2; de las otras dos, sólo dieron amplificación positiva con Sal–3 y Sal–4. Aparentemente se tiene más de una raza o serovar de Salmonella en la región. Estos resultados sugieren que algunas de las fuentes de agua empleadas en la producción de melón Cantaloupe en Zirándaro de los Chávez, Guerrero, no cumplen con la normatividad sanitaria por lo que estas fuentes deben establecerse como puntos prerequisitos de control para evitar la contaminación de melones frescos con patógenos de humanos.
Palabras clave: Melón Cantaloupe, Salmonella spp., coliformes fecales, agua.
SUMMARY
Water used in the production of horticultural crops represents a potential source of microorganisms that cause food–transmitted diseases. In order to evaluate the sanitary quality of different agricultural water sources used in the production of Cantaloupe melon (Cucumis melo L. [group reticulatus] cv. Ovacion and Caminos), in Zirandaro Chavez, Guerrero, 71 water samples were analyzed from two field units (23) and one packaging house unit (48) through traditional bacteriological methods, to detect Salmonella spp, and the filtering membrane method and growth selective media, to detect fecal coliforms, as fecal contamination indicators. Of the total analyzed water samples only three field samples were positive to Salmonella spp. and nine samples, seven coming from the field and two from the packaging house, were positive to fecal coliforms. Salmonella spp. and fecal coliforms were detected mainly in non–chlorinated water samples at 29 °C and pH of 7.5, on average. Two out of four presumptive Salmonella spp. isolates were confirmed by the polymerase chain reaction (PCR) using primers Sal–3 and Sal–4, and invA–1 and invA–2; the other two were only confirmed with Sal–3 y Sal–4. Apparently more than one race or serovar of Salmonella spp. are present in this region. Results suggest that some water sources used in the Cantaloupe melon production in Zirandaro Chavez, Guerrero, do not meet sanitary standards therefore these sources should be considered as critical control points to prevent fresh melon contamination with human pathogens.
Key words: Cantaloupe melon, Salmonella ssp., fecal coliforms, water.
INTRODUCCIÓN
En los últimos años las frutas y hortalizas frescas han llamado la atención de los investigadores no solamente por los beneficios que su consumo brinda a la salud de las personas sino también por su asociación con diversos brotes de enfermedades gastrointestinales registrados en la ultima década (Xuan et al., 2000; FDA, 2004).
Específicamente, el melón Cantaloupe se ha relacionado con seis brotes de salmonelosis y la muerte de al menos dos personas en Estados Unidos de América y Canadá (Economic Research Service, 2001). Las fuentes de contaminación por patógenos de humanos de las frutas y hortalizas frescas son diversas. Entre ellas se encuentran las manos de los trabajadores y el agua de uso agrícola (Castillo et al. , 2003). Al respecto, en 1990 y 1993 se registraron dos brotes de enfermedades uno por Salmonella y otro por Escherichia coli O157:H7 en EE. UU. Estos brotes se asociaron con el consumo de tomates y lechugas frescas. En ambos casos, los estudios epidemiológicos realizados señalaron que el agua con la que se regaron estos productos estaba contaminada con las bacterias mencionadas (Buck et al., 2003). Todo lo anterior señala que el agua puede ser un vehículo importante de contaminación por microorganismos patógenos de humanos en productos hortofrutícolas por lo que este trabajo tuvo como objetivo determinar la calidad sanitaria del agua de uso agrícola en dos unidades de producción y una de empaque de melón Cantaloupe (Cucumis melo L. [grupo reticulatus] cv. Ovación y Caminos), en Zirándaro de los Chávez, Guerrero.
MATERIALES Y MÉTODOS
Muestreo y análisis de agua
Previo al muestreo de agua, se hizo una inspección y evaluación de las características de producción y de las actividades de dos unidades de producción y una unidad de empaque de melón Cantaloupe para determinar su influencia en la calidad sanitaria del agua. Se realizaron dos muestreos de agua de acuerdo al protocolo de la norma Oficial NOM–014–SSA1–1993: uno del 6 al 8 de enero (primer muestreo), y otro del 18 al 20 de abril (segundo muestreo) del 2005. En campo los puntos de muestreo se ubicaron en la orilla de una sección del río Balsas y dentro de los lotes de producción. En la unidad de empaque las muestras se tomaron de espreas (tubos perforados de cobre), cisternas, lavabos, pilas (1.70 x 1.30 m con paredes y piso de mosaico) y tinacos que abastecen para las diversas actividades que ahí se realizan. El agua de los lavabos proviene de la red municipal y la del resto de un pozo, que se encuentra dentro de la unidad, y el cual también se muestreo. Para el muestreo se utilizó el método sistemático con iniciación aleatoria y una tabla de números aleatorios para determinar el tiempo de toma de la muestra (Scheaffer et al., 1987). Para esto, la hora de colecta de la toma de la primera muestra se definió al azar y a partir de ese tiempo los siguientes muestreos fueron sistemáticos con intervalos definidos de tiempo según el caso. Después de cada colecta, tanto en campo como en la unidad de empaque, se determinó la temperatura del agua con un termómetro de mercurio, se etiquetaron las muestras con datos de fecha, lugar y hora de muestreo, y se colocaron en una hielera térmica que contenía hielo y un registrador automático de temperatura HOBO® H8 (Onset Computer Corporation, USA) adherido a la tapa. Al iniciar el procesamiento de las muestras se registró el pH con un potenciómetro (HANNA INSTRUMENTS modelo PH210).
Primer muestreo
Campo. Se estableció en una de las dos unidades de producción donde el agua de la presa "La Calera" se bombea, filtra (filtros de arena) y pasa a una tubería principal de la cual parten cintas de goteo (CG). En bolsas Whirl–PakTM de 3 00 mL con pastillas de tiosulfato se colectaron cinco muestras de 200 mL de agua, con su correspondiente duplicado, por 2 h con intervalos de 30 min iniciando a las 13:00 h.
Unidad de empaque. En bolsas Whirl–PakTM de 300 mL con pastillas de tiosulfato se colectaron muestras de 200 mL de agua de espreas y pila, utilizada para el lavado y desinfección de frutos. En total se colectaron seis muestras, cinco de espreas y una de pila por 2.25 h con intervalos de 20 min, iniciando a las 15:30 h. Cada muestra contó con un duplicado.
A. Salmonella
El análisis se realizó de acuerdo al protocolo de la norma Oficial NOM–114–SSA1–1994. En general el protocolo empleado constó de las siguientes etapas: preenriquecimiento en Agua Peptonada (AP, 0.1 %), para restaurar las células de Salmonella, que pudieran estar dañadas, a una condición fisiológica estable; enriquecimiento, en caldo base de tetrationato (CBT) (MCD Lab S. A. de C. V) para incrementar las poblaciones de Salmonella e inhibir otros organismos presentes en la muestra; aislamiento, en agar entérico hektoen (AEH) (BD BioxonMR) y agar xilosa lisina desoxicolato (XLD) (BD BioxonMR), que restringen el crecimiento de otros géneros diferentes a Salmonella y permite el reconocimiento visual de colonias sospechosas; e identificación bioquímica en agar Urea (BD BioxonMR), agar hierro lisina (LIA, por sus siglas en inglés) (BD BioxonMR) y agar hierro triple azúcar (TSI por sus siglas en inglés) (BD BioxonMR). Además se inocularon tubos con medio indol ácido sulfídrico para sulfuro, indol y movilidad (SIM por sus siglas en inglés). Para confirmar la identificación de la bacteria se establecieron pruebas por PCR y como testigo positivo se incluyó una cepa de S. typhimurium ATCC23564.
En el embudo de un sistema de filtración reutilizable se colocó un filtro de membrana de 47 mm de diámetro, con tamaño de diámetro nominal de poro de 0,45 um (Milipore Mixed Cellulose Ester, Fisher Scientific) y se vaciaron 100 mL de muestra. Finalizada la filtración al vació se colocó la membrana en un tubo de ensaye con 7.0 mL de AP a 0.1 % e incubó a 3 7 °C por 2 h. De la suspensión en AP se tomó una alícuota de 200 uL y se vació en tubos con 5 mL de CBT e incubó por 7 h a 37 °C. Con un asa estéril se tomó una muestra de la suspensión en CBT y se estrió en AEH. Después de 24 h a 37 °C se resembraron en agar XLD aquellas colonias que presentaron pigmentación negra con halo transparente, para confirmar su identificación; y se preservaron en glicerol a 50% (P/V) a –20 °C para pruebas posteriores.
B. Coliformes fecales
Los análisis se realizaron por el método de filtración en membrana de acuerdo a la norma NMX–AA–102–1987. Se filtraron muestras de 100 mL de agua a través de membranas de 47 mm de diámetro, con tamaño de diámetro nominal de poro de 0.45 um (Milipore Mixed Cellulose Ester, Fisher Scientific). Después de la filtración la membrana se colocó en medio Agar m Endo Les (BD DifcoTM) e incubó de manera invertida a 45.5 °C por 24 h. Para el cálculo del número de unidades formadoras de colonias (UFC) de coliformes fecales se registraron únicamente las colonias con brillo metálico verde–dorado, características del grupo de coliformes fecales, en los filtros que contenían de 20 a 80 colonias presuntivas. El cálculo se estimó con la fórmula siguiente:

El número promedio de coliformes fecales se obtuvo del número de colonias registradas en la muestra (R1) y el de su correspondiente duplicado (R2).
Segundo muestreo
Campo. Se estableció en la segunda unidad de producción donde el agua del río Balsas se bombea, filtra (filtros de arena) y clora (200 ppm de cloro gas (Superior Gas Clorinator, Chemical Injection. Technologies Ft. Pierce, Florida)). Se tomaron 6 muestras por duplicado de 300 mL en frascos de plástico de 500 mL, adicionadas con 1 mL de tiosulfato de sodio a 10%, de cada una de las siguientes fuentes: muestra directa de agua de río, muestra de agua de río filtrada y clorada, y muestra de agua de cintas de goteo, a intervalos de 1 h, iniciado a las 9:00 h.
Empaque. Se tomaron siete muestras de 300 mL en frascos de plástico de 500 mL, adicionadas con 1 mL de tiosulfato de sodio a 10%, de cada una de las siguientes fuentes: de la pileta de agua (la misma que se muestreo en el primer muestreo), de la pileta de agua con fungicida (Captan 5 0 PH, y de características similares a la anterior), de las espreas, cisterna, lavabos, tinaco y del pozo. En total se colectaron seis muestras de agua por fuente con intervalos de 2 h cada una, iniciando a las 9:00 h.
A. Salmonella
El análisis de las muestras fue de manera similar al primer muestreo pero con las siguientes modificaciones: entre el preenriquecimiento en 0.1% AP y el enriquecimiento en CBT, las mue stras se agitaron en un vortex para incrementar el desprendimiento de las células adheridas a la membrana. Posteriormente, se transfirió 1 mL de la suspensión de CBT a un tubo con 10 mL de caldo M (BD Difco®) y se incubó a 37 °C sin agitación por 24 h. Después de la incubación, se tomaron muestras de la suspensión bacterial para pruebas de detección e identificación. Para la detección se estriaron muestras de 100 µL de la suspensión en caldo M en AEH y se incubaron a 37 °C por 24 h; las colonias presuntivas a Salmonella se resembraron en agar XLD para confirmar su identidad. De las colonias purificadas se tomaron porciones con asa estéril para inocular por punción tubos con los siguientes medios: Agar TSI, LIA y agar urea; además se inocularon tubos con medio indol ácido sulfídrico para la prueba SIM. Los medios se incubaron a 3 7 °C, registrándose cambios en color a las 24 h. Las colonias presuntivas a Salmonella se preservaron en glicerol (50% p/v).
B. Coliformes fecales
El análisis de cada muestra de agua se realizó por el método de filtración de membrana de acuerdo a la norma NMX–AA–102–1987 y al manual APHA (1998). Los análisis se realizaron por duplicado a las 24 h después de haberse colectado las muestras. Las muestras se procesaron de manera similar a las del primer muestreo; pero en este caso, se filtraron muestras de 10 y 100 mL. La detección se realizó en Agar m FC (EM®) y se incubaron de manera invertida a 45.5 °C por 24 h. Posteriormente se contaron las colonias azul–grisáceas y se obtuvieron promedios entre el número de colonias en la muestra de 10 y en la de 100 mL (R1) y el de su correspondiente duplicado (R2). Las UFC en el agua filtrada se obtuvieron con la fórmula siguiente para dos volúmenes de una misma muestra (10 y 100 mL):

donde:
Promedio= promedio de 2 repeticiones
CF= coliformes fecales
A= 10 mL de agua filtrada
B= 100 mL de agua filtrada
Identificación de Salmonella por PCR
Se utilizaron los iniciadores Sal–3 y Sal–4 y invA–1 e invA–2 sintetizados de acuerdo a la secuencia publicada del DNA para la amplificación de una región especifica del ADN para el gen InvA de Salmonella (Rhan et al., 1992). El gen InvA le confiere a la bacteria la habilidad de invadir las células epiteliales de los portadores. Los iniciadores tienen las siguientes secuencias: Sal–3, 5'–TATCGCCACGTTCGGGCAA–3' y Sal–4, 5' TCGCACCGTCAAAGGAACC–3'; invA–1, 5 '–ACA GTG CTC GTT TAC GAC CTG AAT–3' y invA–2, 5' –AGA CGA CTG GTA CTG ATC GAT AAT–3', y generan un producto de amplificación de 275 y de 244 pares de bases (pb), respectivamente. Para las reacciones de PCR se utilizó el sistema PCR Core System I (Promega) y muestras de lisados celulares (DNA crudo). Las muestras de lisados celulares se prepararon a partir de 1.5 mL de cultivo (1X107 UFC/mL) en caldo M o CST de 18 h de crecimiento el cual se centrifugó (Centrifuge 5415D, Eppendorf, Hamburg, Germany) a 14 000 RPM por 5 min; el sobrenadante se decantó y la pastilla (masa bacteriana) se resuspendió en 1 mL de agua nanopura estéril. Después de dos lavados y centrifugados, la pastilla se resuspendió en 200 uL de agua nanopura estéril, se colocó a 100 °C por 5 min y se centrifugó como se describió anteriormente. Las muestras lisadas se almacenaron a –20 °C previo a la utilización.
Identificación con Sal–3 y Sal–4
Por cada 11.5 µL de mezcla de reacción se adicionaron: 8.48 µL H2O destilada estéril, 0.75 µL MgCl2 25 Mm (Promega), 0.66 µL dNTPs (PCR nucleotide Mix 10 mM), 1.25 µL amortiguador (Thermophilic DNA Polymerase 10x) (Promega), 0.13 µL iniciador Sal–3 (0.25 ug/µ),0.13 µL iniciador Sal–4 (0.25 ug/µL), 1 UTaq DNA Polymerase (Promega) y 1 µL de muestra lisada (107 ufc/mL). Como control positivo y negativo se incluyó material lisado de la cepa de S. typhimurium ATCC235 64 y agua destilada estéril, respectivamente. La reacción se realizó en un termociclador (Mastercycler gradient, eppendorf TM) con el siguiente programa: preincubación, 94 °C por 1 min; 35 ciclos con las siguientes condiciones por ciclo: desnaturalización a 94 °C por 1 min, alineación de los iniciadores a 50 °C por 1 min y extensión del DNA a 72 °C por 2 min y extensión final a 72 °C por 5 min. El producto de PCR se analizó por electroforesis en un gel de agarosa (Promega) a 1% con bromuro de etidio. Las bandas se visualizaron en un transiluminador (Transilluminator SelectTM Series Spectroline) y se documentó con un equipo digital (hp Photosmart 620®).
Identificación con invA–1 e invA–2
La mezcla constó de la siguiente formulación: por cada 25µl de la mezcla de reacción se adicionaron, 13.875 µL H20,1.5 µlMgCl225mM(Promega), 1.0µL dNTPs (40mM),2.5µl amortiguador 10X, 1.0 µL oligonucleótido InvA–1 o invA–2 (10 mM), 5 U de Taq. DNA polimerasa B, y 2 µL muestra lisada (107 ufc/mL). Como controles positivo y negativo se incluyó una cepa de S. typhimurium ATCC23564 y agua destilada estéril, respectivamente. La reacción de PCR se realizó en un termociclador (Perkin Gene Amp PCR System 2400) con la siguiente programación: preincubación, 94 °C por 10 min; 30 ciclos con las siguientes condiciones por ciclo: desnaturalización a 94 °C por 30s, alineación de los iniciadores a 56 °C por 30s y extensión del DNA a 72 °C por 2 min y, extensión final a 72 °C por 10 min. El producto de PCR se analizó en un gel de agarosa (Promega) a 1.5% en un transiluminador (Gel Doc 2000 Bio Rad) y se documentó con el programa Gel Doc 2000.
RESULTADOS Y DISCUSIÓN
Inspección de unidades de producción y empaque
En las dos unidades de producción se encontró que las principales fuentes potenciales de contaminación por microorganismos patógenos para el agua de uso agrícola fueron las áreas adyacentes de pastoreo. En la unidad de producción, donde se realizó el primer muestreo, se encontró que el agua de riego se pasa a través de un sistema de bombeo y filtración (filtros de arena) pero no se clora; en contraste, a lo registrado en la unidad del segundo muestreo, donde el agua se clora (200 ppm) después del filtrado. En la unidad de empaque, el pozo estaba cerrado y rodeado por malla de alambre para restringir el acceso a personas y animales. Las acciones anteriores como el filtrar y clorar el agua así como el proteger las principales fuente de abastecimiento de agua constituyen algunas de las acciones preliminares para la implementación de buenas prácticas agrícolas (BPA) y buenas prácticas de manejo (BPM). Sin embargo, en ambas unidades de producción y unidad de empaque se encontraron algunos sitios que pueden ser considerados puntos críticos de control. En la unidad del primer muestreo se carece de un sistema de inyección de cloro gas para desinfección de agua; y en la unidad de empaque, pilas y cisterna de agua están no cubiertas y a las que pueden llegar polvo y materia orgánica del ambiente.
Temperatura y pH
Durante el primer muestreo la temperatura promedio dentro de la hielera fue de 2.25 °C (de 0.7 a 3.8 °C). En campo y en la unidad de empaque, las muestras registraron temperatura promedio de 29 °C y 27 °C, respectivamente y pH promedio de 7.7 y 7.35 (de 7.4 a 7.3), respectivamente, al momento de la colecta. En tanto que durante el segundo muestreo, la temperatura promedio dentro de la hielera fue de 9.1 °C (de 6.8 a 11.4 °C); la de las muestras no se determinó. En campo y en la unidad de empaque el pH promedio de las muestras fue de 8.3 (de 7.9 a 8.7) y 9.4 (de 8.0 a 10.8), respectivamente. La diferencia de pH entre los valores del agua de campo y de la unidad de empaque se debe, probablemente, a que al agua de la unidad de empaque se le adiciona fungicida (Captan 50 PH) y cal para el control de enfermedades.
Primer muestreo
A. Salmonella
De la siembra del CBT a los medios XLD y AEH, cuatro de cinco muestras de agua (CG1:00, CG1:30, CG2:00, CG2:30 y CG3:00), de cintas de goteo en campo, resultaron con colonias presuntivas (sospechosas) de Salmonella spp. (Cuadro 1). En XLD se obtuvieron colonias negras sin halo, blancas de tamaño pequeño o rosas con centro negro; y en AEH, negras con halo claro. Estas características coinciden con las reportadas para Salmonella por la norma NOM 114–SSA1–1994 y la FDA (2004). Los resultados de las pruebas por PCR, para la identificación de las únicas cuatro muestras sospechosas, mostraron que con los iniciadores Sal–3 y Sal–4 sólo las muestras CG2:00 y CG2:30 generaron resultados de amplificación positivos mientras que con invA–1 e invA–2, sólo CG2:00 dio amplificación, para el gen InvA (Cuadro 1, Figura 1). El peso molecular de las bandas amplificadas fue igual al registrado para S. tiphymurium ATCC23564. Las muestras CG2:00 y CG2:30 se colectaron a las 14:00 h de las cintas de goteo después de que el agua se bombeo del río y se filtró. Las condiciones de temperatura (29 °C) y pH (7.7) promedio registradas en estas muestras de agua, al momento de la colecta, coinciden con las condiciones adecuadas para el desarrollo de Salmonella (Fernández, 2000) las cuales son temperatura entre 5 °C y 47 °C con un óptimo de 37 °C; y pH de 3.8 a 9.5 con un óptimo de 7.7. Lo anterior indica que si el agua de las cintas de goteo estaba contaminada con Salmonella, ésta encontró las condiciones ambientales adecuadas para su desarrollo y sobrevivencia en campo.

B. Coliformes fecales
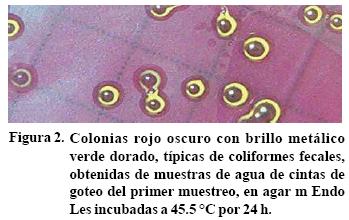
En campo en las cinco muestras de agua tomadas de las cintas de goteo se detectaron coliformes fecales; en promedio, se contaron 15.5 unidades formadoras de colonias (UFC) por 100 mL de agua filtrada (Cuadro 2). En contraste, en la unidad de empaque no se registraron muestras con estas bacterias. En las membranas se desarrollaron colonias pequeñas, de amarillo a verde metálico y con bordes lisos (Figura 2). Estas características coinciden con las reportadas para coliformes fecales en agar m Endo Les, en la norma NMX–AA–102–1987, el APHA (1998), y la FDA (2002).
En la unidad de producción donde se llevó a cabo el primer muestreo, aunque el agua del río se filtra, antes de su distribución a través de las cintas de goteo para el riego, esto no es suficiente para eliminar la presencia de microorganismos patógenos como se evidencia en este estudio. Por otra parte, es probable que en la unidad de empaque, la ausencia de Salmonella y coliformes fecales en el agua de las espreas y en la de la pila se deba a que el agua se clora (3 60 ppm) previo al uso para el lavado y desinfección de frutos. Lo anterior destaca la importancia de clorar el agua cuando esta carece de la calidad sanitaria requerida para uso y consumo humano (NOM–127–SSA1–1994). Según estudios realizados por Costilow et al. (1984) y Park et al. (1991), el cloro actúa como desinfectante reduciendo significativamente la presencia de patógenos en el agua al ejercer un efecto microbicida en los microorganismos. Por su parte, Tortora (1995) menciona que el cloro al agregarse en el agua se separa en dos formas: la activa o ácido hipocloroso (HOCL) con carga eléctrica neutra, e inactiva, o ion hipoclorito (OCI–) carga negativa y que es el HOCL el que tiene el efecto microbicida ya que se difunde rápidamente a través de las paredes celulares.
Segundo muestreo
A. Salmonella
Del muestreo en campo, de 18 muestras analizadas, sólo en tres de ellas, R12:05 y R14:05, de río, y CG11:05 de cintas de goteo se obtuvieron colonias presuntivas de Salmonella en los agares selectivos XLD y AEH. En agar XLD las colonias se observaron negras sin halo, blancas o rosas con centro negro; y en AEH negras con halo transparente. Sin embargo, únicamente la muestra R14:05 fue positiva a todas las reacciones bioquímicas, TSI, LIA, Urea y SIM (Cuadro 3). En la prueba de PCR el material genético de la cepa R14 generó resultados de amplificación positivos con productos de 275 pb (Sal–3 y Sal–4) y de 244 pb (invA–1 e invA–2) para el gen InvA de Salmonella (Figura 3). Por otra parte en la unidad de empaque, de 42 muestras de agua analizadas en los agares XLD y AEH, 10 muestras resultaron con colonias presuntivas de Salmonella. Cuando se analizaron estas muestras por pruebas bioquímicas solo la muestra L13:05 de lavabo dio reacción positiva a las pruebas LIA, Urea y SIM pero reacción negativa a TSI. Al analizarse por PCR, la cepa L13 proveniente de la muestra L13:05 no generó resultados de amplificación con ninguno de los pares de iniciadores probados (Cuadro 3, Figura 3).
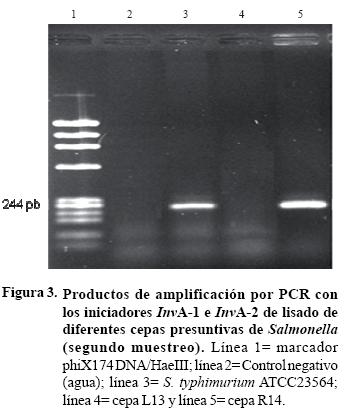
Los agares selectivos tienen buena sensibilidad pero su especificidad es pobre y pueden conducir a resultados falsos positivos. Particularmente especies de Proteus que son lactosa negativa y positiva para la producción de ácido sulfhídrico no pueden ser diferenciadas de Salmonella spp. (Difco Manual, 1998). Por lo anterior está la importancia de establecer pruebas bioquímicas y moleculares para la identificación y confirmación. Por otra parte los materiales genéticos de las cepas CG201 y R14 generaron resultados de amplificaron positivos con los iniciadores Sal–3 y Sal–4 y invA–1 e invA–2; en contraste, al de las cepas CG202 y CG203 que sólo generaron amplificación positiva con Sal–3 y Sal–4 (Figuras 1, 3). Aunque ambos pares de iniciadores sintetizan para el gen invA de Salmonella (Rahn et al., 1992) estos difieren en las secuencias y en el sitio de anillamiento sobre la secuencia de nucleótidos de dicho gen. Es probable que el contraste en los resultados de amplificación aquí obtenidos se deba a que las cepas tengan diferencias en la secuencia de nucleótidos del gen InvA y formen parte de grupos diferentes o que correspondan a diferentes serotipos de Salmonella (Way et al., 1993). Los métodos moleculares para evaluar la diversidad genética de los serotipos de Salmonella, Pulsed–Field Gel Electroforesis (PFGR) (Ebner Mat y Hew. 2001) y Restriction Fragment Length Polymorphism PCR (RFLP–PC) (Hong et al. , 2003), que han demostrado ser una alternativa rápida, exacta y económica, podrían utilizarse para definir el grupo o serotipo de las cepas CG201 CG202, CG230 y R14 de Salmonella de este estudio.
B. Coliformes fecales
De 18 muestras de agua colectadas en campo, solamente en dos muestras de río (R10:05 y R13:05), se detectaron colonias típicas de coliformes fecales en medio m FC (Cuadro 2). Las colonias se observaron azul grisáseo, de forma redonda, menores a 0.5 cm de diámetro (Figura 4); características que concuerdan con las reportadas en norma NMM–AA–102–1987, APHA (1998, 2001) y FDA (2002) para colonias de coliformes fecales. La presencia de coliformes fecales en las muestras R10:05 y R13:05 indica que el agua de río estaba contaminada con heces fecales y de la importancia de filtrar y clorar el agua previo a su uso en la producción de melón.

En la unidad de empaque de 42 muestras de agua analizadas, únicamente en dos muestras, PO 17:05 de pozo y P 17:05 de pila 1, se detectaron coliformes fecales en el agua filtrada. Cuadro 2. Cabe señalar que la muestra P17:07 se colectó al final de la jornada de trabajo (P 17:05 h), lo que sugiere que a este tiempo la efectividad del agua clorada era poca. Lo anterior esta relacionado con el hecho de que la pila 1 con agua clorada (360 ppm) está al descubierto lo cual favorece la pérdida del cloro por volatilización. Al respecto, Suslow (2001) sugiere prestar especial atención en la calidad del agua clorada ya que la presencia de materia orgánica, entre otros factores también reduce su poder microbicida.
CONCLUSIONES
Los resultados de este estudio demuestran que algunas de las fuentes de agua no garantizan una calidad sanitaria adecuada en la producción y manejo de melón Cantaloupe, por lo que deben establecerse programas preventivos como las buenas prácticas agrícolas (BPA y BPM) en las unidades de campo y unidad de empaque en Zirándaro de los Chávez, Guerrero.
El análisis microbiológico así como las pruebas bioquímica y de PCR permitieron la detección e identificación de Salmonella spp. en muestras de agua de río (R14:05) y de agua filtrada pero no clorada de cintas de goteo (CG2:00 y CG2:30). Debido a que en las pruebas de PCR las cepas CG201 y R14 dieron resultados de amplificación positivos con ambos pares de iniciadores, Sal–3 y Sal–4 y invA–1 e invA–2, y a que las cepas CG202 y CG203 sólo amplificaron con Sal–3 y Sal–4, se infiere que en esta región se tiene más de un serovar de Salmonella.
Los análisis microbiológicos también indicaron que en una de las unidades campo, el agua de río y el agua filtrada de cintas de goteo, y en unidad de empaque, el agua de pozo y el agua clorada de pila no cumplen con los criterios de calidad por la presencia de coliformes fecales por lo que estas fuentes deben establecerse como puntos prerequisitos de control para evitar la contaminación del melón con patógenos de humanos. Medidas preventivas como el uso de agua clorada, y el monitoreo frecuente de las concentraciones de cloro activo son recomendables para asegurar la calidad sanitaria del agua de uso agrícola en la producción de melón Cantaloupe.
AGRADECIMIENTO
Al Consejo Nacional de Ciencia y Tecnología (CONACyT) y al Colegio de Postgraduados, por la beca otorgada al primer autor durante sus estudios de maestría; al Programa de Extensión "National Integrated Food Safety Initiative", a través del subcontrato del Dr. Trevor V. Suslow, Department of Plant Science, University of California, Davis, Ca., por los recursos económicos recibidos; y al Centro de Investigación en Alimentación y Desarrollo (CIAD), Culiacán, Sinaloa por permitir el acceso a sus laboratorios.
LITERATURA CITADA
American Public Health Association (APHA). 1998. Stanford methods for analysis of water. Washington, DC. American Public Health Association. p. 28–95. [ Links ]
American Public Health Association (APHA). 2001. Compendium of methods for the microbiological examination of foods, 4th edition. Washington, DC, American Public Health Association. p. 70–80. [ Links ]
Castillo, A., Mercado, I., Martinez, R. Y., Ponce de León J, Murano, E. A. and Acuff, G. R. 2003. Salmonella Contamination during Production of Cantaloupe: A Binational study. Journal food protection 67: 713–720. [ Links ]
Buck, J. W., Walcott, R. R. and Beuchat, L. R. 2003. Recent trends in microbiological safety of fruits and vegetables. Plant health progress 10:10–94. [ Links ]
Costilow, R. N., Uebersax, M. A. and Ward, P. J. 1984. Use of Chlorine dioxide for controlling microorganisms during the handling and storage of fresh cucumber. Journal food Science 49:396– 40. [ Links ]
Difco Manual. 1998. 11th Ed. Difco Laboratories. Maryland, USA. 862 p. [ Links ]
Ebner, P. D. and Mathew, A. G. 2001. Three molecular methods to identify Salmonella enteric serotype Typhimurium DT104: PCRfingerprinting, multiplex PCR and rapid PFGE. FEMS Microbiol. Lett. 205:25–29. [ Links ]
Economic Research Service. 2001. Product Liability and Microbial Foodborne Illness. www.ers.usda.gov. [ Links ]
Food and Drug Administration (FDA). 2002. Enumeration of Escherichia coli and the coliform bacterial. Bacteriological analytical manual. Center for Safety and Applied. Documents and Settings\Propietario\ Misdocumentos\Otros\salmonella\FDA–CFSAN Detection of Salmonella in Environmental Samples from Poultry Houses.htm.
Food And Drug Administration (FDA). 2004. Detection of Salmonella in environmental samples from poultry houses, new microbiological methods. Bacteriological analytical manual, en :\Documents and Settings\Propietario\Misdocumentos\Otros\salmonella\FDA–CFSAN Detection of Salmonella in Environmental Samples from Poultry Houses.htm. [ Links ]
Fernández, E. E. 2000. Microbiología e inocuidad de los alimentos. Primera adición. Universidad Autónoma de Querétaro. México. 56 p. [ Links ]
Hong, Y. L., Tongrui, H. C., Maier, M., White, D. G., Ayers, S., Wang L. and Maurer, J. J. 2003. A Restriction fragment length polymorphism–based polymerase chain reaction as an alternative to serotyping for identifying Salmonella serotypes. J. Avian. Deseases. 47:387–395. [ Links ]
Norma Mexicana. NMX–AA–102–1987. Calidad de agua, detección enumeración de organismos coliformes, termotolerantes y Escherichia coli presuntiva. Método de filtración por membrana. [ Links ]
Norma Oficial Mexicana. NOM–114–SSA1–1993, bienes y servicios. Método para la determinación de Salmonella en alimentos. [ Links ]
Norma Oficial Mexicana. NOM–014–SSA1–1994. Procedimientos sanitarios para el muestreo de agua para uso y consumo humano en sistemas de abastecimiento de agua públicos y privados. [ Links ]
Norma Oficial Mexicana. NOM–127–SSA1–1994, salud ambiental. Agua para uso y consumo humano. Límites permisibles de calidad y tratamientos a que debe someterse el agua para su potabilización. [ Links ]
Park, D. L., Rua, S. M. and Acker, R. F., 1991. Direct application of a new hypochlorite sanitizer for reducing bacterial contamination on foods. Journal Food Protection 54:960–965. [ Links ]
Rahn, K., De Grandis, S. A. and Clarke, R. 1992. Amplification of invA gene sequence of Salmonella typhimurium by polymerase chain reaction as a specific method of detection of Salmonella. Molecular and Cellular Probes 6:271–279. [ Links ]
Scheaffer, R. L, Mendehhall and W, Ott, L. 1987. Elementos de muestreo. Tercera edición, Editorial Iberoamericana, México. 321 p. [ Links ]
Suslow, 2001. Water Disinfection. University of California, Division of agriculture and Natural resources. http://anrcatalog.Ucdavis.edu. [ Links ]
Tortora, J. G., Funke, R. B. and Case, L. C., 1995. Microbiology. Fifth edition, Cummings Publishing Company, INC. p 158–170. [ Links ]
Way, S. J.; Josephson, K. L.; Pillai, S. D.; Abbaszadegan, M.; Gerbera, C. P. and Pepper, I. L. 1993. Specific detection of Salmonella spp. by multiplex polymerase chain reaction. Applied and Environmental Microbiology 59:1473–1479. [ Links ]
Xuan, G., Chen, J., Beuchat, L. R. and Brackett, R. E. 2000. PCR Detection of Salmonella enterica serotype Montevideo in and on raw tomatoes using primers derived from hilA. American Society for Microbiology 12:5248–5252. [ Links ]














