Serviços Personalizados
Journal
Artigo
Indicadores
Links relacionados
-
 Similares em
SciELO
Similares em
SciELO
Compartilhar
Agricultura técnica en México
versão impressa ISSN 0568-2517
Agric. Téc. Méx vol.33 no.3 México Set./Dez. 2007
Artículos
MARCADORES GENÉTICOS DE RESISTENCIA A ROYA DE TALLO (Puccinia graminis Persoon f. sp. avenae) EN AVENA (Avena sativa L.)*
GENETIC MARKERS OF RESISTANCE TO RUST STEM (Puccinia graminis Persoon f. sp. avenae) IN OAT (Avena sativa L.)
Irineo Torres Pacheco1, Mario Martín González Chavira1 , Héctor Eduardo Villaseñor Mir2, Julio Huerta Espino2, Emiliano Villordo Pineda1, Eduardo Espitia Rangel1, Ramón Guevara González3 y Lorenzo Guevara Olvera3
, Héctor Eduardo Villaseñor Mir2, Julio Huerta Espino2, Emiliano Villordo Pineda1, Eduardo Espitia Rangel1, Ramón Guevara González3 y Lorenzo Guevara Olvera3
1 Unidad de Biotecnología, Campo Experimental Bajío, INIFAP. Km. 6.5 carretera Celaya–San Miguel de Allende, Celaya, Guanajuato.
2 Programa de Trigo, Campo Experimental Valle de México, INIFAP.
3Departamento de Bioquímica, Instituto Tecnológico de Celaya.
 Autor para correspondencia:
Autor para correspondencia:
gonzalez.mario@inifap.gob.mx
* Recibido: Noviembre de 2005
Aceptado: Octubre de 2007
RESUMEN
La roya del tallo (Puccinia graminis Persoon f. sp. avenae) disminuye la productividad de avena en México. Las variedades que actualmente se siembran son susceptibles a esta enfermedad. El objetivo de este estudio fue buscar e identificar marcadores moleculares ligados a genes de resistencia a la roya del tallo en cuatro cultivares de avena. En primavera–verano de 2004 se evaluó en campo, en siete localidades, la reacción a la roya del tallo, y en 2005 en invernadero, la reacción a ocho aislados de roya del tallo colectados en diversas localidades del territorio nacional, de las variedades Chihuahua, Obsidiana, Cevamex y Karma. Se utilizaron 30 iniciadores con regiones NBS–LRR en el genoma de la avena y 12 ligados a genes de resistencia a roya en gramíneas. La identificación de los marcadores se realizó con base en el concepto de genes análogos de resistencia (RGAs) y el mapa comparativo de la especie. Los resultados de la evaluación mostraron la susceptibilidad de la variedad Chihuahua; Obsidiana y Cevamex fueron moderadamente resistentes y Karma resistente; esta última mostró el mayor número (17) de marcadores polimórficos ligados a genes de resistencia. Los iniciadores: PIC11–K2/PIC11–WEL, Kinase2 D–E/ KQCFA 3–4, PLOOP1–4/WMA1–4, Kinase2 D–E/WMA1–4, NBS B, F2/R2 y Hv3Lrk, pueden ser indicadores de los genes que confieren resistencia a la roya del tallo.
Palabras clave: Avena, marcadores genéticos, resistencia, roya de tallo.
ABSTRACT
Stem rust (Puccinia graminis Persoon f. sp. avenae) reduces oat productivity in Mexico. Present day oat cultivars are susceptible to this disease. The objective of this study was to search and identify molecular markers for genes linked to stem rust resistance in four oat cultivars. During the spring–summer season of 2004, cultivars Chihuahua, Obsidiana, Cevamex and Karma were sown under rainfed conditions in seven locations and evaluated for stem rust reaction and in 2005 were inoculated under greenhouse conditions in order to assess the reaction of the cultivars to eight different isolates of oat stem rust collected from different locations in Mexico. Thirty primers with regions NBS–LRR in the oat genome and twelve additional primers selected on the basis of its background as being linked to stem rust resistance genes in gramineous crops were used. The identification of the markers was made following the concept of analogous resistance genes and the comparative map of the species. Evaluations at field and greenhouse conditions showed that cultivar Chihuahua was susceptible, Obsidiana and Cevamex were moderately resistant and Karma was resistant. Concurrently, Karma showed the highest number of polymorphic markers (17) linked to stem rust resistance genes. Primers: PIC 11–K2/PIC11–WEL, Kinase2 D–E/ KQCFA 3–4, PLOOP1–4/WMA1–4, Kinase2 D–E/WMA1–4, NBS B, F2/R2 y Hv3Lrk, might be markers associated to stem rust resistance in oats.
Key words: Oats, genetic markers, resistance, stem rust.
INTRODUCCIÓN
La avena es el cultivo con mayor tasa de crecimiento en la superficie sembrada en México. En el período de 1980–1995 se sembraron entre 300 y 400 000 ha por año; a partir de 1996 la siembra se extendió hasta alcanzar las 700 000 ha en 2003 (SIAP, 2003), debido, que es un cultivo emergente con calidad forrajera (Villaseñor–Mir et al, 2003). El 90% de la superficie con avena se siembra bajo condiciones de temporal durante el verano, lo que favorece el desarrollo de la roya o chahuistle (Moreno, 1968). La roya de tallo (Puccinia graminis Persoon f. sp. avenae) disminuye el rendimiento de grano hasta en 70% (Epstein et al., 1988; Archila y Hernández, 2002) y el forraje cosechado es de bajo valor nutrimental (Espitia–Rangel et al., 2002).
La resistencia genética es la mejor defensa para evitar el daño de la roya de tallo (Villaseñor–Mir et al., 2001). Las regiones productoras de avena con pocas variedades en las siembras comerciales son ideales para el desarrollo de nuevas razas fisiológicas de roya, por lo que en ocasiones se presentan epifitias (Singh et al., 2004); esta situación ha prevalecido en México durante los últimos años, lo que ha propiciado la pérdida de resistencia de las variedades. Ante una amplia gama de razas fisiológicas del patógeno, es necesario implementar estrategias de mejoramiento genético orientadas al desarrollo de variedades con resistencia horizontal o duradera (Huerta–Espino y Singh, 2000); para lo cual se deben identificar genotipos de avena que posean genes con efectos de resistencia.
Hasta la fecha no se ha logrado obtener variedades con resistencia amplia y duradera contra la roya de tallo, debido, entre otras causas, al desconocimiento de los genes que confieren la resistencia a la avena. Sin embargo, la selección recurrente se dificulta, yaque este método requiere la presencia del patógeno y las condiciones ambientales adecuadas para que se desarrolle la enfermedad, lo cual no siempre sucede. Con el uso de marcadores moleculares de ácido desoxirribonucleico (ADN) es posible realizar un inventario de los genes de resistencia presentes en el material elite de avena, que permita la selección asistida por marcadores moleculares (SAM). El mejoramiento genético por selección recurrente puede efectuarse por medio de estos marcadores sin necesidad de la patogénesis.
La base del proceso de mejoramiento genético por selección asistida es la identificación de marcadores ligados a los genes de resistencia al patógeno, lo cual puede hacerse por medio de diferentes métodos (Beckman y Osborn, 1992; Vanderbeek y Vanarendonk, 1993; Maliepaard et al, 1997; Nuez y Carrillo, 2000). La identificación de los marcadores se realiza mediante dos estrategias. La primera es la búsqueda de marcadores por reacción en cadena de la polimerasa (PCR) con iniciadores para genes de resistencia análogos (RGAs). Para ello, se usan iniciadores diseñados para amplificar dominios conservados de genes de resistencia (previamente clonados y publicados), de importancia en el cultivo o en especies relacionadas. La base teórica para identificar RGAs, surge del conocimiento generado en los últimos años de la secuencia nucleotídica de varios genes R de resistencia a enfermedades. En estos genes R, se han encontrado homologías significativas en sus secuencias de ADN (motivos) y de aminoácidos. Las clases más abundantes de genes R codifican las proteínas que contienen en la parte central un sitio conformado con secuencias que unen nucleótidos (nucleotide binding site: NBS) y en el extremo carboxilo, un sitio de secuencias repetidas que codifican para varias leucinas (leucine rich repeats: LRR), formando un gen R del tipo NBS–LRR (Grant et al., 1995). También pueden encontrarse secuencias similares que codifican a las estructuras de la familia de receptores de respuesta inmune de Drosophila, llamados receptores tipo Toll (Toll like receptors –TLR–). Este grupo de receptores pertenece a la gran familia de los llamados TIR (receptores Toll/IL–1 –interleukina), ya que todos ellos tienen un dominio de gran similitud estructural y poseen mecanismos de transducción similares con mediadores secundarios comunes (O'Neill, 2000). Otras secuencias que producen dominios alfa helicoidales en las proteínas conocidos como "coiled–coil" (CC), y otros como cierres de leucina (leucine zipper: LZ), pueden también estar presentes, lo que da como resultado genes del tipo TIR–NBS–LRR, CC–NBS–LRR o de LZ–NBS–LRR. (Staskawicz et al., 1995; Hammond–Kosack y Jones, 1997; McDowell y Woffenden, 2003). Esta estrategia ha sido utilizada con éxito en trigo (Spielmeyer et al., 1998), soya (Kanazin et al., 1996), papa (Leister et al., 1996), lechuga (Shen et al. 1998) y maíz (Collins et al., 1998) entre otros.
La segunda estrategia se basa en la similitud de los mapas genéticos comparativos entre especies que pertenecen a una misma familia (Paterson et al., 2000). Por ejemplo, para las especies de la familia de las gramíneas se han elaborado mapas comparativos detallados con los que es posible predecir el contenido y orden de los genes para cada una de ellas (Bennetzen et al., 1998; Gale y Devos, 1998; Wilson et al., 1999). Las especies dentro de una familia han sido ligadas por un sistema común de genes ortólogos detectados por medio de hibridación del ADN. Una aplicación práctica de este conocimiento es el diseño de iniciadores degenerados, los cuales se utilizan para identificar un gen común entre especies de una misma familia (Fulton et al., 2002).
El objetivo de este estudio fue la búsqueda e identificación de marcadores moleculares ligados a genes de resistencia a roya de tallo (Puccinia graminis Persoon f. sp. avenae) en cuatro variedades comerciales de avena.
MATERIALES Y MÉTODOS
La búsqueda de los genes se realizó por medio de PCR en variedades de avena previamente clasificadas por su resistencia a roya. Se utilizaron dos metodologías: a) iniciadores para amplificar dominios conservados y b) iniciadores degenerados para amplificar genes de resistencia aislados y secuenciados en otras especies de la misma familia. La investigación se realizó en el Laboratorio de Marcadores Moleculares del Instituto Nacional de Investigaciones Forestales, Agrícolas y Pecuarias (INIFAP) ubicado en el Campo Experimental Bajío, en 2005.
Patógeno y variedades de avena
Los aislados de roya se colectaron de tallos de plántulas infectadas de manera natural en 25 sitios ubicados desde la Mixteca Oaxaqueña hasta Ciudad Cuauhtémoc, Chihuahua, durante la evaluación del cuarto ensayo uniforme nacional de avena del INIFAP en el verano de 2004. Las muestras se colocaron en bolsas de glacine, se secaron de uno a dos días en oscuridad a condiciones ambientales y se depositaron en un refrigerador para su conservación; en un invernadero del Centro Internacional para el Mejoramiento de Maíz y Trigo (CIMMYT) se inocularon plántulas de la variedad Ópalo con esporas aisladas de cadamuestracon el propósito de purificar las razas (técnica de mono postulares). Una vez purificados los aislados, se probaron en diferenciales para definir cuáles eran similares. Se determinó que ocho de ellos representaban razas diferentes. Con estos aislados se procedió a realizar la evaluación de resistencia a roya de tallo en estado de plántula. Se utilizaron las variedades comerciales de avena: Chihuahua, Karma, Cevamex y Obsidiana.
Evaluaciones en campo y en invernadero
La evaluación en campo se realizó en siete localidades: Yacuita y Yanhuitlán, Oaxaca; Cuyuaco y Mazapiltepec, Puebla; Calera, Zacatecas, y Pabellón y Sandovales, Aguascalientes, bajo condiciones de temporal en el ciclo P–V 2004. Se evaluó la severidad de la enfermedad con base en la escala modificada de Cobb de 0 a 100% de infección (Peterson et al. , 1948) y la reacción a la infección (resistencia) con la siguiente escala: 0= Inmune; 1= Muy resistente; 2= Resistente; 3= Susceptible; 4= Muy susceptible (Roelfs, 1984).
La evaluación bajo condiciones de invernadero se realizó en el CIMMYT, en 2005. Las cuatro variedades se sembraron por separado en ocho charolas de plástico cada una y se mantuvieron a 70–80 de HR, 15–17 °C y 16 h de luz diarias. Doce días después de la siembra las plántulas fueron inoculadas, en charolas individuales, con las ocho razas colectadas.
Reacción en cadena de la polimerasa
Se utilizaron 30 iniciadores para la detección de genes de resistencia a enfermedades con regiones NBS–LRR en el genoma de la avena (Cuadro 1) y 12 iniciadores seleccionados por sus antecedentes de estar ligados a genes de resistencia a roya en gramíneas (Cuadro 2), todos ellos sintetizados por la empresa Invitrogen Custom Primers, Carlsbad California, EE. UU. Para todas las pruebas de amplificación se prepararon a 25 (iL de reacción a una concentración final de: 1X de buffer PCR Taq libre de Mg, 1.0 (iM de cada iniciador o marcador específico, 200 (iM de dNTPs, 2 mM de MgCl2, 1 unidad (U) de la enzima ADN Taq Polimerasa y 100 ng de ADN genómico. Las reacciones de amplificación fueron corridas en un termociclador Px2 Thermo Electron Corporation Milford MA, USA., con el siguiente programa de amplificación: 1 min a 94 °C, 2 min a la temperatura específica de cada iniciador y 2 min a 72 °C.


Electroforesis
Se verificaron los fragmentos amplificados en un gel de agarosa al 3% con una corrida de 4 h a 80 V y se usó ADN de Φ174 digerido con Hae III como marcador de peso molecular. La visualización de los fragmentos se logró tiñéndolos con bromuro de etidio a una concentración de 10 mg mL–1 y se documentó con equipo ChemiDoc XRS de Bio–Rad Hércules California, EE.UU.
RESULTADOS Y DISCUSIÓN
Evaluación de la resistencia
La variedad Chihuahua fue la más susceptible a roya de tallo, ya que mostró los niveles más altos de infección en los siete sitios de evaluación. Las variedades Karma, Cevamex y Obsidiana presentaron diferentes niveles de infección y reacción a la enfermedad. La variedad Karma tuvo mayor estabilidad, ya que los porcentajes de infección observados fueron los de menor variación (Cuadro 3).
Los resultados de la evaluación en plántula bajo condiciones de invernadero, al igual que en campo, muestran que la variedad Chihuahua fue susceptible a todos los aislados; Cevamex lo fue a cuatro, Obsidiana fue susceptible a dos y moderadamente resistente a tres y Karma manifestó el mayor nivel de resistencia, razón por la cual se puede inferir que esta variedad posee más genes de resistencia a las razas fisiológicas de royade tallo. Los aislados 3.1 y 4.1 fueron, en promedio, los más agresivos con las variedades evaluadas, en tanto que el aislado 14.2 fue el más atenuado (Cuadro 4). La variabilidad de la interacción entre los aislados y las variedades demuestra que existe interacción específica patógeno–hospedero y explica, en parte, la inestabilidad de resistencia a roya de tallo en las variedades que se cultivan en México (Villaseñor–Mir et al. , 2003). Lo anterior permite inferir que en el germoplasma de avena que se cultiva en México existe una amplia diversidad de genes de resistencia (Cuadro 4).
Marcadores moleculares ligados a genes de resistencia
Se detectaron 38 bandas polimórficas: 13 en la variedad Karma, 9 en Cevamex, 9 en Obsidiana y 7 en Chihuahua (Figura 1 y Cuadro 5). El polimorfismo probablemente representa la secuencia, o parte de ella, del gen que codifica el motivo de aminoácidos del tipo NBS–LRR en los genes de resistencia a enfermedades (Grant et al., 1995). El marcador PIC13–GL/PIC13–K2 relacionado con resistencia a roya de tallo en cebada (Collins et al., 2001) sólo se detectó en la variedad Chihuahua (Cuadro 5, Figura 1), por lo que es probable que esta variedad sea resistente a razas fisiológicas del patógeno bajo condiciones ambientales específicas que no se presentaron en los sitios de evaluación.
Es notable la coincidencia entre la cantidad de polimorfismos observados y la reacción de tolerancia a roya previamente detectada en campo y en invernadero. También sobresale el hecho de que en la variedad Karma (clasificada en este trabajo como resistente a roya de tallo) se detectaron sólo tres polimorfismos de manera exclusiva con los iniciadores PIC11–K2/PIC11 –WEL y Kinase2 D–E/KQCFA 3 –4.
Los iniciadores de tipo PIC11–K2/PIC11–WEL se han relacionado con genes de resistencia a Pseudomonas siryngae, "downy mildew" y roya en Arabidopsis thaliana y a marchitez de origen vascular en tomate (Grant et al. 1995; Lawrence et al., 1995; Ori et al., 1997; Parker et al. 1997). La relación entre este tipo de iniciadores y los genes de resistencia a alguna forma de roya permite inferir que las bandas polimórficas detectadas en la variedad Karma pueden ser marcadores relacionados con genes de resistencia a roya de tallo.
El iniciador Kinase2 D–E/KQCFA 3–4 se relaciona con genes involucrados en la resistencia a virus, bacterias y hongos; entre ellos, se han reportado dos especies de roya en sorgo: Puccinia sorghi y P. polysora (Collins et al., 1998). Igualmente, este tipo de iniciadores se relacionan con genes de resistencia a roya. Aunque en otra especie, en este caso se trató de gramíneas, por lo que existe alta posibilidad de que el polimorfismo observado en la variedad Karma esté asociado a genes de resistencia a la roya de tallo.
De los 12 iniciadores ligados a genes de resistencia a roya en gramíneas, que no codifican motivos NBS–RRL probados, sólo se detectó polimorfismo con F2/R2, Hv3Lrk y URIC/LN2. En los amplificados con los marcadores se identificaron dos fragmentos polimórficos que posiblemente estén relacionados con genes de resistencia: el primero fue el marcador Hv3Lrk (Brunner et al., 2000) de 260 pb que se observó en las variedades Cevamex y Karma; el segundo de 260 pb correspondió a los marcadores URIC/LN2 (Helguera et al., 2003) en la variedad Obsidiana (Figura 2).
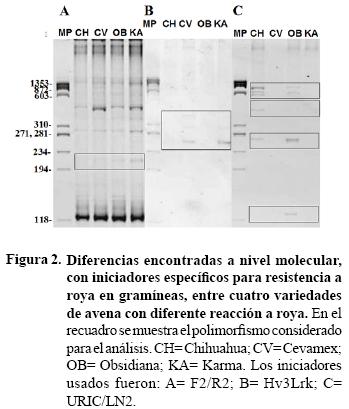
Las variedades Karma y Obsidiana mostraron una banda amplificada polimórfica común con el iniciador F2/R2 y Cevamex mostró 3 bandas polimórficas amplificadas con el iniciador Hv3Lrk (Cuadro 6). Las variedades Karma y Cevamex no compartieron polimorfismo con la variedad Chihuahua; mientras que Obsidiana y Chihuahua compartieron tres bandas polimórficas con el iniciador URIC/LN2. En otros trabajos se reportó que el polimorfismo detectado con el iniciador Hv3Lrk se ubica a 3.2 cM del gen de resistencia a roya Rhp7.g en una población F2 de 112 individuos derivados de la cruza entre "Cebada Capa" y "Bowman" (Brunner et al., 2000). Por lo tanto, Hv3Lrk se puede usar como marcador para la selección asistida con cierto nivel de seguridad.
Se detectó mayor presencia de fragmentos polimórficos de marcadores ligados a genes de resistencia en las variedades con mayor tolerancia a la enfermedad (Cuadros 5 y 6). Sin embargo, con los iniciadores PLOOP1 –4/WMA1 –4, Kinase2 D–E/KQCFA 3–4, Kinase2 D–E/WMA1–4, NBS B, F2/R2 y Hv3Lrk no se detectaron fragmentos polimórficos en la variedad susceptible; lo que sugiere que los polimorfismos detectados en las variedades resistentes pueden ser marcadores ligados a genes que confieren resistencia a las razas fisiológicas de la roya de tallo utilizadas.
La mayoría de los fragmentos polimórficos detectados fueron amplificados con iniciadores de marcadores ligados a genes de resistencia a roya; sin embargo, las variedades resistentes también mostraron marcadores de resistencia a otros factores de estrés que, aunque bióticos, son distantes como virus y bacterias. Lo anterior, sugiere que puede tratarse de genes conservados de resistencia genérica al estrés biótico (Tabaeizadeh et al., 1999).
CONCLUSIONES
La metodología aplicada a nivel de marcadores moleculares permitió detectar diferencias entre variedades de avena respecto a su reacción al ataque de roya de tallo (Puccinia graminis Persoon f. sp. avenae).
En la variedad Karma se observaron la mayoría de los marcadores de genes de resistencia que poseen Obsidiana y Cevamex.
LITERATURA CITADA
Aghaee–Sarbarzeh, M.; Harjit–Singh and Dhaliwal, H. S. 2001. A microsatellite marker linked to leaf rust resistance transferred from Aegilops triuncialis into hexaploid wheat. Plant Breed. 20:259–261. [ Links ]
Archila M, A. B. y Hernández, M. 2002. Efecto de la roya del tallo (Puccinia graminis f. sp. avenae Eriks. y Henn.) sobre el rendimiento y sus componentes en avena (Avena sativa L.). Tesis de Licenciatura. Departamento de Parasitología Agrícola, Universidad Autónoma Chapingo. Chapingo, Estado de México, México. 50 p. [ Links ]
Beckmann, J. S. and Osborn T. C. 1992. Plant Genomes: Methods for Genetic and Physical Mapping. Kluwer Academic Publishers, Dordrecht, The Netherlands. [ Links ]
Bennetzen, J. L.; Sanmiguel, P.; Chen, M.; Tikhonov, A.; Francki, M. and Avramova, Z. 1998. Grass genomes. Proc. Natl. Acad. Sci. USA 95:1975–1978. [ Links ]
Brunner, S.; Séller, B. and Feuillet, C. 2000. Molecular mapping of the Rph7.g leaf rust resistance gene in barley (Hordeum vulgare L.). Theor. Appl. Genet. 101:783–788. [ Links ]
Chen, X.; Soria, M. A.; Yan, G.; Sun, J. and Dubcovsky, J. 2003. Development of sequence tagged site and cleaved amplified polymorphic sequence markers for wheat stripe rust resistance gene Yr5. Crop Sci. 43:2058–2064. [ Links ]
Cheng, D. W.; Ken, C. A.; Tinker, N.; Wight, C. P.; He, S.; Lybaert, A.; Fedak, G. and Molnar, S. J. 2002. Genetic and physical mapping of Lrk10–like receptor kinase sequences in hexaploid oat (Avena sativa L.). Genome 45:100–109. [ Links ]
Collins, N. C.; Webb, C. A.; Seah, S.; Ellis, J. G.; Hulbert, S. H. and Pryor, A. 1998. The isolation and mapping of disease resistance gene analogs in maize. Mol. Plant Microbe Interact. 11:968–978. [ Links ]
Collins, N.; Park, R.; Spielmeyer, W.; Ellis, J. and Pryor, A. J. 2001. Resistance gene analogs in barley and their relationship to rust resistance genes. Genome 44:375–381. [ Links ]
Epstein, A. H.; Simons, M. D.; Frey, K. J. and Rothman P. G. 1988. Field resistance of oats to Puccinia graminis f. sp. avenae measured by yield and seed weight reduction. Plant Dis. 72:154–1546. [ Links ]
Espitia–Rangel, E.; Villaseñor–Mir, H. E.; Tovar M., R.; Pérez H., P. y Limón O., A. 2002. Momento óptimo de corte y comparación de genotipos de avena forrajera. In: Memoria del XIX Congreso Nacional de Fitogenética. Saltillo, Coahuila, México. 282 p. [ Links ]
Fulton, T. M.; van der Hoeven, R.; Eannetta, N. T. and Tanksley, S. D. 2002. Identification, analysis, and utilization of conserved ortholog set markers for comparative genomics in higher plants. Plant Cell 14:1457–1467. [ Links ]
Gale, M. and Devos, K. 1998. Comparative genetics in the grasses. Proc. Natl. Acad. Sci. USA 95:1971–1974. [ Links ]
Graner, A.; Streng, S.; Drescher, A.; Jin, Y.; Borovkova, I. and Steffenson, B. J. 2000. Molecular mapping of the leaf rust resistance gene Rph7 in barley. Plant Breed. 119:389–392. [ Links ]
Grant, M. R.; Godiard, L.; Straube, E.; Ashfield, T.; Lewald, J.; Sattler, A.; Innes, R.W. and Dangl, J. L. 1995. Structure of the Arabidopsis RPM1 gene enabling dual specificity disease resistance. Science 269:843–846. [ Links ]
Hammond–Kosack, K. E. and Jones, J. D. G. 1997. Plant disease resistance genes. Annu. Rev. Plant Physiol. Plant Mol. Biol. 48:575–607. [ Links ]
Helguera, M.; Kha, I. A. and Dubcovsky, J. 2000. Development of PCR markers for the wheat leaf rust resistance gene Lr47. Theor. Appl. Genet. 100:1137–1143. [ Links ]
Helguera, M.; Khan, I. A.; Kolmer, J.; Lijavetzky, D.; Zhong Q, L. and Dubcovsky, J. 2003. PCR assays for the LR37–YR17–SR38 cluster of rust resistance genes and their use to develop isogenic hard red spring wheat lines. Crop Sci. 43:1839–1847. [ Links ]
Huerta–Espino, J. y Singh, R. P. 2000. Las royas del trigo. In: El trigo de temporal en México. Villaseñor–Mir, H. E. y Espitia–Rangel, E. (Eds.) Secretaría de Agricultura (SAGAR), Instituto Nacional de Investigaciones Forestales, Agrícolas y Pecuarias, Centro de Investigación de la Región Centro, Campo Experimental Valle de México. México. p. 231–251. [ Links ]
Kanazin, V.; Marek, L. F. and Shoemaker, R. C. 1996. Resistance gene analogs are conserved and clustered in soybean. Proc. Natl. Acad. Sci. USA 93:11746–11750. [ Links ]
Lawrence, G. J.; Finnegan, E. J.; AyliffeIb, M. A. and Ellisai, J. G. 1995. The L6 gene for flax rust resistance is related to the Arabidopsis bacterial resistance gene RPSP and the tobacco vira1 resistance gene N. Plant Cell 7:1195–1206. [ Links ]
Leister, D.; Ballvors, A.; Slamini, F. and Gebhardt, C. 1996. A PCR–based approach for isolating pathogen resistance genes from potato with potential for wide application in plants. Nature Genet. 14:421–429. [ Links ]
Maliepaard, C.; Janse, J. and van Ooijen, J. W. 1997. Linkage analysis in a fullsib family of an outbreeding plant spacies: overview and consequences for applications. Genet Res. 70:237–350. [ Links ]
McDowell, J. M. and Woffenden, B. J. 2003. Plant disease resistance genes: recent insights and potential applications. Trends Biotechnol. 21:178–183. [ Links ]
Moreno G, R. 1968. Aspectos del mejoramiento de los cereales en México. In: Memoria del Tercer Congreso Nacional de Fitogenética. Chapingo, Estado de México, México. 16 p. [ Links ]
Nuez, F. y Carrillo, J. M. 2000. Los marcadores genéticos en la mejora vegetal. Editorial de la UPV, Valencia, España. [ Links ]
O'Neill, L. 2000. The toll/interleukin–Ireceptor domain: a molecular switch for inflammation and host defence. Biochem. Soc. Trans. 28:557–63. [ Links ]
Ori, N.; Eshed, Y.; Paran, I.; Presting, G.; Aviv, D.; Tanksley, S.; Zamir, D. and Fluhr, R. 1997. The 12C family from the wilt disease resistance locus 12 belongs to the nucleotide binding, leucine–rich repeat superfamily of plant resistance genes. Plant Cell 9:521–532. [ Links ]
Parker, J. E.; Coleman, M. J.; Szabò, V.; Frost, L. N.; Schmidt, R.; van der Biezen, E. A.; Moores, T.; Dean, C.; Daniels, M. J. and Jones, J. D. G. 1997. The Arabidopsis downy mildew resistance gene RPP5 shares similarity to the toll and interleukin–i receptors with N and L6. Plant Cell 9:879–894. [ Links ]
Paterson, A. H.; Bowers, J. E.; Burow, M. D.; Draye, X.; Elsik, C. G.; Jiang, C. X.; Katsar, C. S.; Lan, T. H.; Lin, Y. R.; Ming, R. and Wright, R. J. 2000. Comparative genomics of plant chromosomes. Plant Cell 12:1523–1540. [ Links ]
Peterson, R. F.; Campbell, A. B. and Hannah, A. E. 1948. A diagramatic scale for estimating rust intensity of leaves and stem of cereals. Can. J. Res. Sect. C. 26:496–500. [ Links ]
Pflieger, S.; Lefebvre, V.; Caranta, C.; Blattes, A.; Goffinet, B. and Palloix, A. 1999. Disease resistance gene analogs as candidates for QTLs involved in pepper–pathogen interactions. Genome 42:1100–1110. [ Links ]
Roelfs, A. P. 1984. Race specificity and methods of study. In: Bushnell W. R.. and A. P. Roelfs (eds). The Cereal Rust. Vol. 1. Origins, Specificity Structure and Physiology. Academic Press, Orlando, FL. USA. p. 132–164. [ Links ]
Shen, K. A.; Meyers, B. C.; Islam–Faridi, M. N.; Chin, D. B.; Stelly, D. M. and Michelmore, R. W. 1998. Resistance gene candidates identified by PCR with degenerate oligonucleotide primers map to clusters of resistance genes in lettuce. Mol. Plant Microbe Interact. 11:815–823. [ Links ]
Servicio de Información y Estadística Agroalimentaria y Pesquera (SIAP) Secretaría de Agricultura, Ganadería, Pesca y Alimentación (SAGARPA) 2003. México, D. F. [ Links ]
Singh, R. P.; Huerta–Espino, J.; Pfeiffer, W. and Figueroa–Lopez, P. 2004. Ocurrence and impact of a new leaf rust race of durum wheat in the nothwe stern Mexico during 2001–2203. Plant Dis. 88:703–708. [ Links ]
Spielmeyer, W.; Robertson, M.; Collins, N.; Leister, D.; Schulze–Lefert, P.; Seah, S.; Moullet, O. and Lagudah, E. S. 1998. A superfamily of disease resistance gene analogs is located on all homoeologous chromosome groups of wheat (Triticum aestivum). Genome 41:782–788. [ Links ]
Staskawicz, B. J.; Ausubel, F. M.; Baker, B. J.; Ellis, J. G. and Jones, J. D. G. 1995. Molecular genetics of plant disease resistance. Science 268:661–667. [ Links ]
Sun, Q.; Wei, Y.; Ni, Z.; Xie, C. and Yang, T. 2002. Microsatellite marker for yellow rust resistance gene Yr5 in wheat introgressed from spelt wheat. Plant Breed. 121:539–541. [ Links ]
Tabaeizadeh, Z.; Agharbaoui, Z.; Harrak, H. and Poysa, V. 1999. Transgenic tomato plants expressing a Lycopersicon chilense chitinase gene demonstrate improved resistance to Verticillum dahliae race 2. Plant Cell Rep. 19:197–202. [ Links ]
van der Linden, C.; Doret, G.; Wouters, C. A. E.; Mihalka, V.; Kochieva, E. Z.; Smulders, M. J. M. and Vosman, B. 2004. Efficient targeting of plant disease resistance loci using NBS profiling. Theor. Appl. Genet. 109:384–393. [ Links ]
Vanderbeek, S. and Vanarendonk, J. A. M. 1993. Criteria to optimize designs for detection and estimation of linkage between marker loci from segregating populations containing several families. Theor. Appl. Genet. 86:269–280. [ Links ]
Villaseñor–Mir, H. E.; Espitia–Rangel, E. and Marquez–Gutiérrez, C. 2001. Registration of "Cevamex" oat. Crop Sci. 41:266–267. [ Links ]
Villaseñor–Mir, H. E.; Espitia–Rangel, E. y Huerta–Espino, J. 2003. El Campo Experimental Valle de México, estratégico en la producción nacional de avena: Historia y aportaciones. En: 60 años de investigación en el Campo Experimental Valle de México. Instituto Nacional de Investigaciones Forestales, Agrícolas y Pecuarias, Centro de Investigación de la Región Centro, Campo Experimental Valle de México. Chapingo, Estado de México, México. p. 17–30. (Publicación Especial No. 1). [ Links ]
Wilson, W. A.; Harrington, S. E.; Woodman, W. L.; Lee, M.; Sorrells, M. E. and McCouch, S. R. 1999. Inferences on the genome structure of progenitor maize through comparative analysis of rice, maize and the domesticated panicoids. Genetics 153:453–473. [ Links ]
Yaish, M. W. F.; Sáenz de M., L. E. and Pérez de la V., M. 2004. Isolation of a family of resistance gene analogue sequences of the nucleotide binding site (NBS) type from Lens species. Genome 47:650–659. [ Links ]














