Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Agricultura técnica en México
versión impresa ISSN 0568-2517
Agric. Téc. Méx vol.32 no.3 México sep./dic. 2006
Artículos
CARACTERIZACIÓN MOLECULAR Y BIOLÓGICA DE GENES RECOMBINANTES EN MAÍZ CRIOLLO DE OAXACA*
MOLECULAR AND BIOLOGICAL CHARACTERIZATION OF RECOMBINANT GENES IN MAIZE LANDRACES FROM OAXACA
Dora Alicia Landavazo Gamboa1, Karla Guadalupe Calvillo Alba1, Elsa Espinosa Huerta1, Laura González Morelos1, Flavio Aragón Cuevas2, Irineo Torres Pacheco1, Salvador Horacio Guzmán Maldonado1, Victor Montero Tavera1 y María Alejandra Mora Avilés1
1 Unidad de Biotecnología, Campo Experimental Bajío, INIFAP. Km. 6.5 carretera Celaya–San Miguel de Allende. Apartado Postal 112. 38010. Celaya, Guanajuato, México.
2 Campo Experimental Valles Centrales de Oaxaca, INIFAP.
 Autora para correspondencia:
Autora para correspondencia:
mora.alejandra@inifap.gob.mx
* Recibido: Mayo de 2005
Aceptado: Julio de 2006
RESUMEN
Se analizó maíz criollo de tres regiones representativas de producción del estado de Oaxaca (Sierra Norte, Valles Centrales y Costa), con el fin de establecer la presencia de secuencias transgénicas. Las estrategias empleadas fueron: 1) búsqueda del promotor 35S del virus del mosaico de la coliflor por medio de PCR cualitativo, 2) detección inmunológica de la actividad de la enzima fosfinotricin acetil transferasa (PAT) que confiere resistencia a herbicida 3) detección de resistencia a la aplicación de glufosinato en plantas, 4) detección molecular del transgen bar, mismo que codifica la proteína fosfinotricin acetil transferasa, 5) determinación del contenido transgénico de las muestras positivas por PCR en tiempo real y 6) secuenciación de las bandas específicas de amplificación. Como resultado de la primera estrategia se detectó la secuencia del promotor 35 S en plantas de cinco parcelas de maíz de la Sierra Norte, pertenecientes al distrito de Ixtlán. Este promotor no se detectó en el resto de las muestras colectadas de las regiones Valles Centrales y Costa. La detección inmunológica de la proteína PAT fue negativa en todas las muestras analizadas. De 143 5 plantas sometidas a la aplicación de glufosinato, 23 resistieron la actividad del herbicida; sin embargo, ninguna evidenció amplificación del fragmento del transgen bar. Las cinco plantas inicialmente positivas para PCR cualitativo se redujeron a tres muestras positivas por PCR cuantitativo y secuenciación. En conclusión, las muestras amplifi cadas para el promotor 35 S no evidenciaron presencia ni actividad del gen bar; sin embargo, podría ser otro el origen del promotor 3 5 S y la naturaleza de la resistencia al glufosinato en los maíces criollos de la Sierra Norte.
Palabras clave: Zea mays L., fosfinotricin acetil transferasa, glufosinato de amonio, PCR tiempo real, promotor 35S, virus del mosaico de la coliflor.
ABSTRACT
Maize landraces from three regions: Sierra Norte, Valles Centrales and Costa in the state Oaxaca, were sampled looking for transgenic sequences. The strategies to accomplish this goal were: 1) search for 35 S promoter of the caulifl ower mosaic virus by qualitative PCR, 2) immunological detection of phosphinotricin acetyl transferase (PAT) enzyme activity, which confers resistance to the herbicides glufosinate, 3) detection of resistant plants to glufosinate, 4) detection of the transgene bar by PCR, 5) assessment of transgenic content by quantitative PCR and 6) sequencing of specific amplified fragments. The use of the first technique allowed for the detection of the 35S promoter sequence in plants from five plots from the Sierra Norte region, in the district of Ixtlan. The 35S promoter was not detected in samples from the other two regions. Immunological detection for the phosphinotricin acetyl transferase protein was negative for all samples analyzed. Twenty–three plants out of 1435 were resistant to the herbicide glufosinate; however, none showed amplification of the bar transgene. Out of fi ve initial samples only three were confi rmed positive for 35S promoter by quantitative PCR and sequencing. The 35S positive samples did not test positive for the bar transgen; therefore, the detected 35S promoter sequence might have a different origin as well as the herbicide resistance present in these maize landraces from the region Sierra Norte.
Key words: Zea mays L., cauliflower mosaic virus, glufosinate–ammonium, phosphinothricin acetyl transferase, real time PCR, 35S promoter.
INTRODUCCIÓN
México es reconocido como el centro de origen del maíz (Zea mays L.). La arqueología demuestra que los restos más antiguos provienen de los estados de Oaxaca y Puebla, donde existe una gran variedad de especies criollas y parientes silvestres (Benz, 2001; Piperno y Flannery, 2001). La diversidad genética del maíz es patrimonio nacional y mundial, y una fuente importante para los programas de mejoramiento genético en este cultivo (FAO, 1995).
La biotecnología ha producido cultivos de maíz modificado genéticamente con la introducción de genes que codifican para resistir a diferentes factores: por ej emplo, los genes cry (cristal de proteína insecticida, de Bacillus thuringiensis) le confieren resistencia al ataque por lepidópteros; los genes pat (fosfinotricin N–acetiltransferasa, de Streptomyces viridochromogenes) y bar (resistencia a bialafos, de Streptomyces hygroscopicus) proveen resistencia a los herbicidas glufosinato, bialofos y fosfinotricina; el gen cp4 epsps (3–enol piruvil siquimato 5–fosfato sintasa, de Agrobacterium tumefaciens) proporciona resistencia al glifosato (James, 2005). De los materiales de maíz comerciales patentados, 69% poseen resistencia a insectos y a herbicidas simultáneamente: predomina el resistente a glufosinato, en 60% de los tipos de resistencia a herbicidas y los resistentes a glifosato representan 24% (James, 2005).
Estos maíces modificados se han aprobado y liberado comercialmente en Estados Unidos de América, China, Canadá y Argentina. Sin embargo, en muchos otros países aún existen dudas e inquietudes respecto al riesgo sobre la salud de los seres humanos y animales. Asimismo, preocupan las posibles modificaciones de los ecosistemas al introducir estos organismos, aun cuando no se ha reportado algún efecto dañino. Por lo tanto, es importante establecer si existen maíces modificados en áreas de cultivo en México, para aclarar los temores de su posible riesgo ambiental y de consumo.
En el marco de la moratoria de facto al maíz transgénico, se publicó en el año 2001 un reporte que muestra la presencia de dos secuencias de ADN en la construcción de organismos transgénicos, inexistentes en forma natural en el maíz convencional (Quist y Chapela, 2001). Este reporte provocó mucha incertidumbre y preocupación a nivel internacional, tanto en la comunidad científica como en la sociedad civil, debido al flujo de genes del maíz transgénico hacia los parientes silvestres y maíces criollos. Preocupa, principalmente, que los transgenes del maíz resistente a herbicidas se desplacen hacia la maleza, situación naturalmente indeseable (Altieri, 2002).
Ante este escenario, se analizaron las secuencias del promotor 35 S del CaMV y del gen bar, así como la expresión y actividad de la proteína PAT, en plantas de maíz tomadas de un número mayor de parcelas de las que se han estudiado a la fecha. Además, se utilizó un mayor número de estrategias de detección, con el objetivo de analizar globalmente la situación actual de la presencia de los genes pat/bar y del promotor 35 S del CaMV.
MATERIALES Y MÉTODOS
Zona de Muestreo
Las muestras de maíz criollo se colectaron en las regiones Sierra Norte (Distrito de Ixtlán), Valles Centrales (Distritos de Tlacolula, Ocotlán, Ejutla de Crespo y Etla) y Costa (Distritos de Pochutla, Juquila y Jamiltepec) del estado de Oaxaca, abarcando todas las formas y colores de grano (Cuadro 1).
Tipo de Muestreo.
En cada región se seleccionaron al menos ocho localidades y en cada una de éstas al menos cuatro parcelas, de las que se colectaron de 10 a 20 mazorcas de maíz. De cada mazorca se desgranó 50% y el grano se concentró por parcela, cuyo tamaño de muestra consistió en la agrupación de 400 semillas seleccionadas arbitrariamente.
Sensibilidad del tamaño de muestra
El grado de sensibilidad del tamaño de muestra se definió con la ecuación:
r= (1–(p/100))n x 100
donde:
r= Probabilidad de que exista un porcentaje máximo de semillas transgénicas igual a p, aún sin detectar transgenes.
p= Porcentaje de GMO en el lote (0.9%, European Parliament and Council Regulation 1829/2003).
n= Tamaño de muestra [Número de semillas (Dodd, 2002)].
Dado que se seleccionaron 400 granos de maíz por parcela, la ecuación anterior quedó de la siguiente forma: r= (1–(0.9/100))400 x 100. Como resultado se obtuvo un valor de r= 2.7%.
La probabilidad de que exista un porcentaje máximo de semillas transgénicas igual a 0.9% aun si no se detectan transgenes, fue de 2.7%. En otras palabras, existe un nivel de confi anza del 97.3% de encontrar 0.9% de semillas transgénicas en las muestras analizadas.
Germinación de semillas y procesamiento de muestras
Las 400 semillas de cada parcela se trataron con una solución de hipoclorito de sodio comercial (Cloralex ®) al 5% por 10 min, y se lavaron, al menos tres veces, con agua destilada–estéril; para su germinación se colocaron en papel de estraza previamente humedecido con agua destilada–estéril y se depositaron dentro de una bandeja cubierta con plástico en una incubadora a 20–25 °C por tres a cuatro días en oscuridad, hasta el desarrollo del epicótilo.
Los epicótilos se disectaron de los granos germinados y se colocaron en bolsas de malla, distribuidos en tres submuestras de cada parcela. Las bolsas se colocaron sobre hielo, para congelarse posteriormente con nitrógeno líquido. Finalmente, se conservaron en un ultracongelador REVCO a –41 °C hasta su liofilización.
Las muestras congeladas se liofi lizaron a 20 milibares de presión y a –48 a –50 °C de temperatura por tres días; después se molieron y se les extrajo el ADN, de acuerdo con la técnica modifi cada de Dellaporta et al. (1983).
Detección molecular del promotor 35S del CaMV
Las mezclas del PCR se prepararon en un volumen de 25 µL, combinando el ADN blanco (150–300 ng/(µl), iniciadores (0.2 µM; sentido: 5'–gCACCTACAAATgCCATCA–3', antisentido: 5'–gATAgTgggATTgTgCgTCA–3', de PROMEGA, Corp.); dNTP's (0.25 mM), Taq Polimerasa ( 1.5 U), cloruro de magnesio (2mM), solución salina reguladora (buffer; 1X), y agua destilada estéril. El fragmento esperado fue de 195 pb.
Como control interno se amplificó la secuencia 16S ribosomal con los iniciadores, sentido: 5'–TgAgAATggATAAgAggCTC–3' y antisentido: 5'–TgTTgTTCCCCTCCCAAggg–3' (Rivera–Bustamante1, comunicación personal). Las concentraciones de los componentes para cada reacción de PCR fueron las mismas que las del promotor 35S. El fragmento esperado fue de 315 pb.
Las reacciones anteriores se amplificaron en un termociclador (Techne, TC–412) bajo las mismas condiciones: desnaturalización a 95 °C por 4 min durante un ciclo y 30 ciclos de desnaturalización a 95 °C por 1 min, alineamiento a 54 °C por 1 min, y extensión a 72 °C por 1 min; y una extensión final a 72 °C por 7 min durante un ciclo. Los fragmentos amplificados se separaron en geles de agarosa al 2% y teñidos con bromuro de etidio (5 ng/100 ml), para visualizar el producto de la amplificación.
Bioensayo para la detección de resistencia a fosfinotricina
La selección de muestras para este experimento se basó en los resultados de la detección por PCR del promotor 35S, por lo cual se trabajó con muestras que revelaron indicios de contener dicho promotor.
Se probaron un total de 1435 plantas de cinco parcelas, cuyas semillas se obtuvieron de las mazorcas desgranadas de origen, y 50 controles positivos que consistieron en 50 semillas T3, producto de dos retrocruzas parentales transformadas con el promotor 35S y el transgen bar. Además, se probaron 20 controles negativos de maíz híbrido de Michoacán y 20 controles negativos de maíz de la Sierra Norte.
Las semillas se colocaron en una cámara de crecimiento sobre bandejas germinadoras, usando, para germinación, el sustrato Sunshine No. 3® a 25±1 °C; las plántulas se sometieron a un fotoperíodo de 12 h luz–12 h oscuridad e iluminación de 20 000 luxes, después de 15 días, se trasplantaron a vasos de 355 ml, y se trasladaron a un invernadero con medidas de bioseguridad de nivel 1 (Traynor et al., 2001) y mantenidas a 30–32 °C, hasta obtener plantas con cuatro hojas verdaderas. La solución de fosfinotricina (BASTA®) se aplicó a una hoja por planta, mediante un hisopo que absorbió un volumen aproximado de 130 µL de una solución, con una concentración de 0.75% (De la Fuente2, comunicación personal). El hisopo se frotó tres veces a lo largo de la hoja, dentro de un área de 5 cm entre dos líneas delimitadas con un marcador. Los resultados se evaluaron diariamente durante 15 días. Se analizó el área marcada con base en seis niveles de daño (0, 5, 25, 50, 75 y 100%), en función del grado de avance de toxicidad en el tejido del control negativo susceptible a fosfinotricina. Después 24 h de la inoculación, se inició una observación (considerada día 1) del nivel de daño sobre las hojas inoculadas con fosfinotricina. A partir de la aplicación del herbicida se realizaron tres mediciones cada tres días, por un período de 15 días.
Detección del transgen bar por PCR
Las muestras sometidas a este análisis provinieron de las plantas resistentes a la fosfinitosina en el bioensayo. La secuencia del transgen bar se detectó con iniciadores bar1 (sentido: 5'–TgCACCATCgTCAACCACTA–3') y bar2 (antisentido: 5'–CACgCgACCACgCTCTTgAA–3') (Spangenberg et al., 1994), y los iniciadores patjv1 (Sentido: 5 '–CAggAACCGCAggAgTggA–3') y patjv2 (antisentido: 5'–CCAgAAACCCACgTCATgCC–3') (Vickers et al. 1996). Las concentraciones del resto de los componentes de la reacción de amplificación del gen bar fueron similares a las empleadas para amplificar el promotor 35S. El tamaño de fragmento esperado para cada par de iniciadores fue de 310 pb y 372 pb, respectivamente. Las condiciones de amplificación para ambos pares de iniciadores fueron: desnaturalización inicial a 95 °C por 2 min durante 1 ciclo; 30 ciclos de desnaturalización a 95 °C por 45 seg, alineamiento a 58 °C por 30 seg y extensión a 72 °C por 1 min y una extensión final de un ciclo a 72 °C por 7 min. Los productos de las reacciones de amplificación fueron corridas en geles de agarosa a 2% con TBE 1X teñidos con bromuro de etidio.
Detección inmunológica de la actividad de la enzima PAT
Se empleó el paquete comercial Trait LL Lateral Flow (Strategic Diagnostics Inc.), para detectar la expresión de la proteína PAT producida por los genes bar y pat derivados de S. hygroscopicus y S. viridochromogenes, respectivamente. Se molieron 400 semillas por muestra y se tomaron 10 g de polvo. Cada muestra se colocó en pequeños recipientes y se agregaron 10 ml de buffer del kit; se introdujo una tira reactiva y se incubó a temperatura ambiente por 10–15 min. Transcurrido el tiempo, se verificó la presencia de la banda de prueba que indica el resultado positivo.
Contenido transgénico por PCR en tiempo real
Muestras, iniciadores y sondas.
Se utilizó el mismo ADN empleado para los análisis por PCR cualitativo. Los iniciadores y sonda del gen 35S se diseñaron en el laboratorio de Cultivo de Tejidos Vegetales de la Unidad de Biotecnología del Bajío del Instituto Nacional de Investigaciones Forestales, Agrícolas y Pecuarias (INIFAP). Los iniciadores sentido fueron: MORASEQ SF (5'–gACAgTggTCCCAAAgATggA–3') y antisentido MORASEQ SR (5 '–TgCTTTgAAgACgTggTTggAA–3'), y la sonda MORASEQ SM1 FAM 5'–CCCACgAggAgCATC–3' 5NFQ marcada con el fl uoróforo FAM en su extremo 3' y el apagador NFQ (non fluorescent quencher) en su extremo 5'.
El control interno (18S) pertenece a un kit, prediseñado por Applied Biosystems (Applied Biosystems Lincoln, USA) marcado con el fluoróforo VIC y el terminador NFQ. Las reacciones se amplificaron en un volumen total de 25µl (1 (2.5 µl de sonda, 0.25µM) y oligos (2.0 µl, 0.9 µM), 12.5 µl de Master Mix (Taq DNApolimerasa, dNTP's y buffer; Applied Biosystems Lincoln, USA) y de ADN (25 ng). Cada reacción de amplificación para el promotor 35S y el gen 18S se desarrolló por separado (single) y en forma conjunta (multiplex).
Se generó una curva estándar de calibración para determinar el umbral crítico Ct de detección, utilizando concentraciones de ADN conocido, correspondiente al maíz MON 810 en concentraciones de 0, 0.1, 0.5, 1, 2 y 5% de contenido transgénico (IRMM 413; Institute for Reference Materials and Measurements, Fluka). Las condiciones de amplificación se desnaturalizaron inicialmente a 94 °C por 9 min, posteriormente a 95 °C durante 20 seg, alineación a 60 °C por 1 min y extensión a 72 °C por 30 seg durante 40 ciclos (PCR tiempo real Applied Biosystems 7000). El promedio de los valores  Ct se calcularon para determinar el porcentaje de contenido transgénico de cada muestra. Los valores comparativos de Ct (
Ct se calcularon para determinar el porcentaje de contenido transgénico de cada muestra. Los valores comparativos de Ct (
 Ct) se calcularon restando los valores
Ct) se calcularon restando los valores  Ct del gen endógeno marcado con el fl uoróforo VIC a los valores
Ct del gen endógeno marcado con el fl uoróforo VIC a los valores  Ct del transgen marcado con el fl uoróforo FAM. Los datos se analizaron calculando el logaritmo de la tendencia de los valores
Ct del transgen marcado con el fl uoróforo FAM. Los datos se analizaron calculando el logaritmo de la tendencia de los valores 
 Ct (calculada con el método de mínimos cuadrados) con respecto a los valores de los estándares definidos y analizados previamente.
Ct (calculada con el método de mínimos cuadrados) con respecto a los valores de los estándares definidos y analizados previamente.
Clonación y secuenciación de fragmento amplificado
Se purificaron tres fragmentos amplificados a partir de las bandas aisladas de agarosa, con el kit de extracción QUIAquick (QIAGEN Inc. CA, USA). La concentración de estos productos se cuantificó mediante fluorómetro (Minifluorometer, TBS–380 TURNER Biosystems, Inc.), utilizando el kit de medición PicoGreen (Molecular Probes ds DNA Quantitation Reagent and Kits, USA).
El ADN purificado fue precipitado (NaOAc 3M y etanol absoluto) para eliminar trazas de dNTP's e iniciadores. Los controles de amplificación fueron los plásmidos pCAMBIA y pUC. El fragmento amplificado se clonó en el vector pGEM®–T Easy Vector Systems (PROMEGA, Madison, WI, USA) siguiendo las instrucciones del fabricante.
Los posibles transformantes se seleccionaron en medio LB, adicionado con ampicilina (100mg/l), IPTG (0.5 mM), X–Gal (40 µg/ml). En un proceso aleatorio se seleccionó un total de 158 colonias, en su mayoría bacterias blancas, que indican una transformación positiva.
La transformación se verificó mediante amplificación del fragmento insertado en el vector. Se realizó una lisis bacteriana en 50 (µl de agua desionizada estéril, se administró choque térmico en termobaño a 95° C por 10 min y posteriormente se colocó la muestra en hielo por otros 10 min. Se tomaron 3 µ1 de muestra para la verificación por amplificación, empleando los iniciadores de PROMEGA.
De la amplificación se definieron las colonias que dieron positivo para el fragmento de interés, de ellas se extrajo plásmido mediante la técnica de minipreps (Qiagen Spin Miniprep QIAGEN, USA), siguiendo el protocolo de extracción. La calidad de los plásmidos se visualizó en un gel de agarosa al 1 % con buffer TAE 1X y se cuantificaron con el fluorómetro.
Se seleccionaron tres muestras para secuenciarse (dos controles positivos pCAMBIA y pUC y un producto de PCR purificado conteniendo el fragmento 35S) mediante la reacción de PCR, en base a los protocolos de iniciadores marcados de Sequi Therm EXCELTM (Epicentro Technologies, Madison, WI, USA). Los productos del PCR anterior se amplificaron a partir de 400 fmoles del vector pGEM®–T, conteniendo el fragmento 35S de 195 pb, utilizando 1.5 pmoles de iniciadores universales M13 (–29) sentido (5'–CACgACgTTgTAAAAcgAC–3') y M13 antisentido (5–ggATAACAATTTCACACAgg–3') marcados con fluoróforos infrarojos por los canales de 795 nm (IRD800 heptametino alanina) y 685 nm (IRD 700 pentametino carbocianina), respectivamente, desarrollados por Li–cor (Li–cor Lincoln, USA). El programa de amplificación consistió en un ciclo inicial de desnaturalización de 92 °C por 2 min, seguido de 30 ciclos de 92 °C de desnaturalización por 30 seg, 54 °C por 30 seg de alineamiento específi co de los iniciadores y 70 °C por 1 min de extensión. Finalmente, las reacciones se mantuvieron a 4 °C para conservar estable la reacción.
La reacción de secuenciación se analizó directamente con el secuenciador automático de ADN Li–cor Global Edition IR2 System Modelo 4200 (Li–cor Inc., Lincoln, USA), a través de la secuenciación simultánea bidireccional previamente desnaturalizada a 95 °C por 3 min, con 3 µl de amortiguador de carga a base de formamida, y colocados en el sistema de gel vertical de 41 cm de poliacrilamida KB plus 5.5% y 1X TBE KB plus [0.89 M Tris base, 0.89 M ácido bórico, 0.02 M EDTA, agua desionizada (18 MΩ) pH 8.3] de Li–cor a 55 °C y 1200 voltios.
Las secuencias obtenidas se analizaron con el programa e–Seq 3.0.9 (DNA Sequencing and Analysis Software, Copyright 1999–2003, Li–cor Inc., Michigan Technology University), las cuales finalmente se compararon con secuencias conocidas y reportadas en la base de datos NCBI mediante Blast (Altschul et al., 1997) y Blast 2 sequences (Tatusova y Madden, 1999).
RESULTADOS Y DISCUSIÓN
Resultados
Detección del promotor 35S del CaMV por PCR. En cinco parcelas de la Sierra Norte (SN–18, SN–28, SN–30, SN–70 y SN–153) se identificaron plantas que mostraron evidencia de amplificación del promotor 35S, mediante la técnica de PCR, obteniéndose un fragmento esperado de 195 pb (Figura 1). Por otro lado, en ninguna de las parcelas de las regiones Valles Centrales ni Costa se amplificó la secuencia del promotor de las plantas analizadas. Las muestras positivas representan 3.1 del número total de parcelas analizadas de la región Sierra Norte. Asimismo, representan 1.62% de las regiones Sierra Norte, Valles Centrales y Costa con un nivel de confi anza de 97.3%.
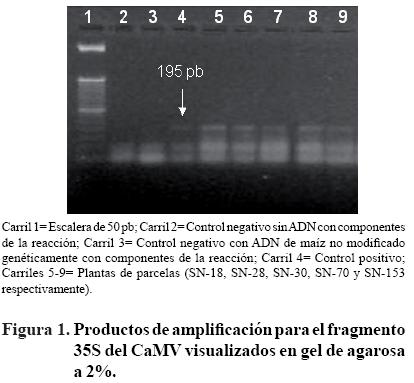
El fragmento de 315 pb de la secuencia 16S ribosomal, usado como control interno, se amplificó con plantas de todas las parcelas, demostrando que el protocolo de amplificación se realizó efi cientemente (datos no mostrados).
Detección inmunológica de la actividad de la enzima PAT. La detección de la expresión de la proteína fosfinotricin acetil transferasa (PAT) por medio de tiras de flujo lateral, resultó negativa para las plantas de todas las parcelas de las tres regiones sometidas a esta prueba (datos no mostrados).
Detección de resistencia a fosfinotricina: bioensayo. La observación de daño de las plantas indicó que al tercer día entre 65 y 93% de las plantas presentaron 25% de daño (Figura 2). Al sexto día, del 89 al 98% de las plantas estaban totalmente cloróticas en el área marcada en las hojas; para el noveno día, del 93 al 99.4% de las plantas mostraban daño en 100% de la superficie de la hoja inoculada; el día 12, 98.4% las plantas mostraron toxicidad en 100% de la planta y para el día 15, habían muerto todas las plantas que mostraron alto nivel de toxicidad (98.4%).
El nivel de daño ocasionado por el herbicida indica que la muestra SN–18 presentó una incipiente resistencia al herbicida durante los primeros tres días del bioensayo; sin embargo, a partir del día seis se aproximó a la media de respuesta del resto de las muestras (Figura 3). Sólo 23 plantas que representan a las cinco parcelas, resistieron la acción del herbicida sin ningún síntoma de toxicidad.

Los controles negativos exhibieron daño en la misma proporción que las plantas bajo estudio. Los daños y número de plantas afectadas cayeron dentro del margen esperado. En el caso de los controles positivos, el número de plantas dañadas también estuvo dentro del rango de los controles negativos y de las muestras. Ninguna de las plantas utilizadas como control positivo sobrevivió, lo cual impidió compararlas con las plantas resistentes.
Detección del transgen bar por PCR. Se analizó el ADN extraído de las 23 plantas que resistieron el efecto del herbicida fosfinotricina en el experimento anterior. Los resultados de la amplificación con los iniciadores patjv1–patjv2 y bar1–bar2, fueron negativos para todas las plantas; ninguna de las 23 presentó las bandas esperadas de 375 pb y 310 pb, respectivamente, las que se observaron en el control positivo (Cuadro 2).
PCR en tiempo real. Se realizó un rango dinámico de la concentración del control positivo para determinar la curva estándar y definir el rango de detección confiable y consistente. El rango dinámico consistió en cuatro diluciones seriales de ADN, manejadas al 50% en cada etapa. El rango dinámico para el gen 35S y el gen 18S muestran una alta efi ciencia, de acuerdo a las curvas de cada concentración en las que en puntos equidistantes y paralelos se duplica la concentración de ADN de una dilución (Figura 4). La curva estándar define el umbral de detección (Ct), donde se observa una relación lineal entre los productos de PCR y la concentración de ADN durante la fase exponencial (Figura 5). La correlación del 98.5%, proporcionada en este análisis, indica que es posible detectar organismos genéticamente modificados en un rango entre 100 ng y 12.5 ng de ADN inicial.
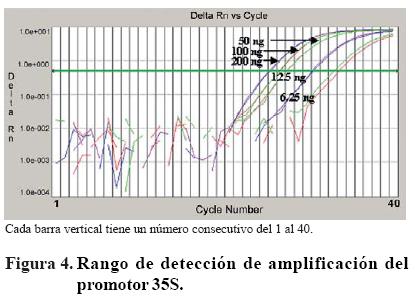
Los estándares de referencia se ajustaron a 12.5 ng. Los estándares de contenido transgénico de 0.1 y 0.5% fueron inconsistentes en su amplificación, por lo que se desecharon en los siguientes análisis.
Únicamente se analizaron los estándares representados por las plantas que contenían 1, 2 y 5% de contenido transgénico.
El patrón de comportamiento de las curvas de amplificación en cada caso corresponde a una distancia equivalente a un ciclo, definiéndose como el doble de contenido transgénico en cada caso (2n). En todos los casos, el control interno amplificó (datos no mostrados).
Las plantas de tres parcelas del distrito de Ixtlán (Figura 6 y Cuadro 2), amplificaron el fragmento 35 S, correspondientes a SN–153 de la localidad de Santiago Xiacui, SN–30 de la localidad de Latuvi y SN–70 de la localidad de SantiagoComaltepec. Todas las muestras amplificaron el gen 18S (datos no mostrados).
Secuenciación de fragmento amplificado. De las tres secuencias analizadas, dos coincidieron con la secuencia del vector pGem7ZF, una correspondió en 100% a la secuencia del promotor 35S del CaMV en ambas secuenciaciones (700 y 800 nm). La secuencia correspondiente al 35S tuvo alta similitud con la p35S–GFP (NCBI U28417) en 95%, evidenciando la presencia del promotor 35S en el maíz de la muestra SN–30, correspondiente a la Sierra Norte de Oaxaca.
De 354 nucleótidos de la secuencia (NCBI X04879) del promotor reportada por Sanderset al. (1987), hubo similitud al 100% en la secuencia amplificada en el material de maíz SN–30 (Figura 7).
Discusión
Todas las muestras evidentes del promotor 35S del CaMV provienen del distrito de Ixtlán de la región Sierra Norte del estado de Oaxaca, resultado concordante con el estudio realizado por Quist y Chapela (2001), quienes reportaron la presencia del promotor 35S en el mismo estado. El muestreo en cada una de las parcelas es un sistema eficiente para detectar introgresiones del promotor 35S a los materiales criollos de maíz. El grado de sensibilidad en el muestreo, indica que el tamaño de muestra fue suficiente tanto para manipular apropiadamente un número sustancial de muestras, como para asegurar un nivel de confianza hasta ahora sin reportar en los análisis publicados.
En conjunto, las muestras SN–18, SN–28, SN–30, SN–70 y SN–153 representan 3.1 del número total de las analizadas en la región Sierra Norte, evidencia contrastante con los datos del reporte preliminar de Herrera et al. (2002), donde se revelan 13 parcelas positivas (44%) con el promotor 35S, de un análisis de 29 parcelas en el estado de Oaxaca.
Es importante resaltar que las frecuencias transgénicas (el número de muestras positivas y el contenido transgénico) son bajas, ya que el porcentaje de parcelas positivas es reducido, sin detectar inmigración en parcelas vecinas. La baja incidencia del promotor 35S puede deberse a varias causas, entre las que destacan: a) las plantas híbridas (con introgresión) tienen baja adaptación a las condiciones ambientales de la región, b) una posible asincronía en los períodos de floración, limitando así la posibilidad del cruzamiento entre la planta transgénica y el criollo, o c) la introgresión puede encontrarse en etapa temprana, por lo que su distribución aún sería limitada.
Las evidencias en los análisis por PCR en tiempo real y secuenciación del promotor 35S, sugieren que la introducción de transgenes proviene de la mezcla de grano a las parcelas y no por polinización libre, en cuyo caso se esperaría un número mayor de parcelas positivas en las localidades a las cuales pertenecen. El promotor 35S resultó inexistente en todas las parcelas de las localidades pertenecientes a las regiones Valles Centrales y Costa.
La ausencia de amplificación de la secuencia del gen bar en las plantas resistentes a fosfinotricina, y de la actividad proteica de la enzima PAT, descarta la hipótesis de la resistencia de las plantas por el transgen 'bar. Esta prueba puede eliminar las teorías del silenciamiento del gen bar (Matzke y Matzke, 1995; Baulcombe, 1996; Depicker y Van Montangu, 1997) o de la baja expresión de la proteína que impidiera detectar su actividad. Una posible explicación de la resistencia a fosfinotricina en las 23 plantas, es la resistencia natural, tomando en cuenta la amplia diversidad genética del maíz en el estado de Oaxaca.
Con este estudio se descarta la presencia del transgen bar; sin embargo, puede haber otros transgenes diferentes como los de la delta–endotoxina de Bacillus thuringiensis (cry), para la resistencia a insectos. Otro evento a detectar en maíz es la presencia del gen cp4 epsps que confiere resistencia al herbicida glifosato; aunque con mucha menor incidencia en los productos biotecnológicos comerciales de maíz. Además, en el análisis realizado por Herrera et al. (2002), se reportó sin ningún evento cp4 epsps.
Los resultados de PCR cuantitativo reducen a tres las muestras positivas de las cinco originalmente obtenidas por PCR. La inconsistencia en resultados de PCR cualitativo y PCR cuantitativo demuestran que, la detección por la técnica de PCR cualitativo no necesariamente proporciona resultados fiables respecto a las muestras positivas. Es claro que amplicones no específicos pueden reducirse eventualmente; sin embargo, también representa una fuente de falsos positivos indeseables en detecciones puntuales. La ventaja del análisis por PCR en tiempo real es la inclusión de una sonda específica, la cual incrementa la precisión en la detección.
La verificación de las muestras positivas mediante secuenciación, valida los resultados de amplificación por PCR y PCR en tiempo real en las muestras SN–30, SN–70 y SN–153. La elevada identidad de las secuencias obtenidas y comparadas con bases de datos de secuencias reportadas para el promotor 35S, confiere la presencia de este promotor en el genoma de maíz en dos parcelas a niveles de introgresión menor al 1% (una semilla de cada 100 tiene el promotor 35S).
Respecto a la posible persistencia de los elementos de introgresión detectados, es importante destacar su permanencia o incremento de su frecuencia en selección positiva, es decir, la asociación de estos genes con otros genes nativos que confieren características seleccionadas por el agricultor, la aplicación de herbicidas o bien la resistencia a plagas. Sin embargo, en la región Sierra Norte no se reporta la aplicación de estos químicos o la presencia de plagas como el gusano barrenador del tallo o algún otro lepidóptero plaga considerado blanco de la toxina Bt, por lo que el material estaría libre a una selección que representara desventaja para los materiales criollos nativos, disminuyendo así la frecuencia del transgen hasta desaparecer. Es importante considerar que, dada la diversidad del maíz criollo, se han detectado genes que de manera natural codifican para la resistencia a glufosinato, como lo demuestran los resultados del bioensayo. Probablemente existen maíces criollos con resistencia natural, sin detectar por falta de selección. Por lo tanto, si hubiera un maíz transgénico resistente a herbicidas en la misma zona donde se encuentra un maíz con resistencia natural a herbicidas, ambos podrían coexistir sin ninguna alteración a la diversidad genética.
Respecto a la preocupación de la seguridad para el consumo, se han desarrollado protocolos de evaluación previos a la aprobación del cultivo comercial de maíz genéticamente modificado. Los protocolos contemplan análisis de alergenicidad y toxicidad de la nueva proteína sintetizada. En ninguno de los casos estudiados hasta ahora, relacionados con las proteínas PAT, se ha reportado un efecto tóxico. Tampoco se ha presentado algún cuadro alergénico en consumidores humanos (Ash et al., 2000; Khumnirdpetch et al., 2001; Klotz et al., 2002). Por lo tanto, con respecto a la inocuidad alimentaria, como se reportó, es improbable el riesgo en el consumo de maíz con la proteína PAT.
Finalmente, queda abierta la posibilidad de encontrar otros eventos transgénicos; sin embargo, la pregunta más importante es: ¿Cuál es la posibilidad de que esto ocurra y sus consecuencias? posteriormente, ¿Estas consecuencias son un riesgo real y científicamente avalado? Los esfuerzos deben orientarse en el análisis de posibles riesgos de la introgresión en especies de países de origen.
CONCLUSIONES
Existe evidencia de la presencia del promotor 35S del virus del mosaico de la coliflor en plantas de maíz de tres parcelas de la región Sierra Norte de Oaxaca. La base para detectarlo fue la vinculación del promotor 35S con construcciones transgénicas, ya que lo contienen la mayoría de los materiales comerciales de maíz genéticamente modificados. El número de parcelas positivas, así como el porcentaje de contenido transgénico (en relación a las referencias internacionales) fue bajo, por lo que se infiere que el tiempo en el que ocurrió la introgresión es reciente. Si bien se desconoce con toda certeza la existencia de transgenes específicos, la evidencia para encontrar el gen bar, que confiere resistencia al herbicida fosfinotricina, está ausente en las muestras obtenidas o no fue posible detectarlo con los diferentes métodos ensayados.
COMUNICACION PERSONAL
1 Rivera–Bustamante, R. CINVESTAV–Campus Irapuato.
2 De la Fuente, J. M., Monsanto–México
LITERATURA CITADA
Altieri, M. A. 2002. Aspectos socioculturales de la diversidad del maíz nativo. Departamento de Ciencias Políticas y Gestión del Medio Ambiente, Universidad de California. www.cec.org/files/pdf//altieri-s.pdf [ Links ]
Altschul, S. F.; Madden, T. L.; Schaffer, A. A.; Zhang, J.; Zhang, Z.; Miller, W. and Lipman, D. J. 1997. Gapped BLAST and PSI–BLAST: a new generation of protein database search programs. Nucl. Acids Res. 25:3389–3402. [ Links ]
Ash, J. A.; Sceideler, E. and Novak, C. L. 2000. The fate of genetically modified protein from Roundup Ready soybeans in the laying hen. Poultry Sci. 79 (Suppl.1), 26. [ Links ]
Baulcombe, D. C. 1996. RNA as a target and an initiator of post–transcriptional gene silencing in transgenic plants. Plant Mol. Biol. 32:79–88. [ Links ]
Benz, B. F. 2001. Archeological evidence of teosinte domestication from Guilá Naquitz, Oaxaca. Proc. Natl. Acad. Sci. 98:2104–2106. [ Links ]
Dellaporta, S. L.; Wood, J. and Hicks, J. B. 1983. A plant DNA minipreparation. Version II. Plant. Mol. Biol. Reptr. 1:19–21. [ Links ]
Depicker, A. and Van Montangu, M. 1997. Post–transcriptional gene silencing in plants. Curr. Opin. Cell Biol. 9:373–382. [ Links ]
Dodd, D. 2002. When zero is not zero. www.psrcorn.com/zeronotzero.htm [ Links ]
European Parliament and Council Regulation 1829/2003. REGULATION (EC) No 1829/2003 of the european parliament and of the council of 22 September 2003 on genetically modified food and feed. Official Journal of the European Union 18.10.2003. [ Links ]
Organización de las Naciones Unidas para la Agricultura y la Alimentación (FAO). 1995. Conferencia técnica internacional de la FAO sobre los recursos fitogenéticos. Conservación y utilización sostenible de los recursos fitogenéticos en América Central y México. Informe síntesis regional. San José, Costa Rica, 57 p. [ Links ]
Herrera L. A.; Joffre y G., A.; Álvarez, M. A.; Huerta, E.; Arriaga, L.; Soberón, J.; Ortiz, S.; Aldama, A.; Cotero, M. A.; Serratos, J. A. 2002. Primer informe sobre análisis de la presencia de maíz transgénico en Oaxaca y Puebla. Secretaría de Agricultura, Ganadería, Desarrollo Rural, Pesca y Alimentación/ Dirección General de Sanidad Vegetal/Comité Intersecretarial de Bioseguridad de Organismos Genéticamente modificados. No publicado, 20 p. [ Links ]
James, C. 2005. Global status of commercialized biotech/ GM Crops: 2005. ISAAA Briefs No. 34. ISAAA: Ithaca, N. Y., USA., 11 p. [ Links ]
Khumnirdpetch, V.; Intarachote, U.; Treemance, S.; Tragoonroong, S. and Thummabood, S. 2001. Detection of GMOs in the broilers that utilized genetically modified soybean meals as a feed ingredient. Plant & Animal Genome IX Conference, January 2001, San Diego, USA. (Poster 585). [ Links ]
Klotz, A.; Mayer, J. and Einspanier, R. 2002. Degradation and possible carry over of feed DNA monitored in pigs and poultry. Eur. Food Res. Technol. 214: 271–275. [ Links ]
Matzke, M. A. and Matzke, A. J. M. 1995. How and why do plants inactivate homologous (Trans) genes? Plant Physiol. 107:679–685. [ Links ]
Piperno, D. R. and Flannery, K. V. 2001. The earliest archeological maize (Zea mays L.) from highland Mexico: New accelerator mass spectrometry dates and their implications. Proc. Natl. Acad. Sci. 98:2101–2103. [ Links ]
Quist, D. and Chapela, I. 2001. Transgenic DNA introgressed into tradicional maize landraces in Oaxaca, Mexico. Nature 29:541–543. [ Links ]
Sanders, P. R.; Winter, J. A.; Barnason, A. R.; Rogers, S. G. and Fraley, R. T. 1987. Comparison of caulifl ower mosaic virus 35S and nopaline synthase promoters in transgenic plants. Nucl. Acids Res. 15:1543–1558. [ Links ]
Spangenberg, G.; Wang, Z.Y.; Nagel, J., and Potrykus, I. 1994. Protoplast culture and generation of transgenic plants in red fescue (Festuca rubra L.). Plant Sci. 97:83–94. [ Links ]
Tatusova, T. A. and Madden, T. L. 1999. BLAST2 Sequences, a new tool for comparing protein and nucleotide sequences. FEMS Microbiol. Lett. 174:247–250. [ Links ]
Traynor, P. L., Adair, D. and Irwin, R. 2001. Greenhouse research with transgenic plants and microbes: A practical guide to containment. Information Systems for Biotechnology. Virginia Tech. Blacksburg VA, USA. 59 p. [ Links ]
Vickers, J. E.; Graham, G. C. and Henry, R. J. 1996. A protocol for the efficient screening of putatively transformed plants for bar, the selectable marker gene, using the polymerase chain reaction. Plant Molecular Biology Reporter 14:363–368. [ Links ]














