Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Acessos
Acessos
Links relacionados
-
 Similares em
SciELO
Similares em
SciELO
Compartilhar
Agricultura técnica en México
versão impressa ISSN 0568-2517
Agric. Téc. Méx vol.32 no.2 México Mai./Ago. 2006
Artículos
Caracterización molecular de genotipos comerciales y elite de papa (Solanum tuberosum L.) en México*
Molecular characterization of commercial and elite genotypes of potato (Solanum tuberosum L.) in Mexico
Fermín Orona–Castro1, Víctor Pecina–Quintero2, Mario Alberto Rocha–Peña3, Mateo Armando Cadena–Hinojosa4, Octavio Martínez de la Vega5 e Isidro Humberto Almeyda–León3,
1 Campo Experimental Edzná, INIFAP. Km 17 carretera Cayal–Edzná, Nhoyaxxhe, Campeche.
2 Campo Experimental Río Bravo, INIFAP.
3 Campo Experimental General Terán, INIFAP.
4 Campo Experimental Valle de México, INIFAP.
5 Centro de Investigación y Estudios Avanzados del Instituto Politécnico Nacional, Unidad–Irapuato.
* Recibido: Junio de 2005
Aceptado: Mayo de 2006
 Autor para correspondencia:
Autor para correspondencia:
e–mail: halmeida@fcb.uanl.mx
RESUMEN
Durante el 2003, se caracterizaron 13 variedades comerciales y tres líneas elite de papa mediante las técnicas de RAPD y SSR. Con la técnica de RAPD se evaluaron 40 iniciadores y se seleccionaron tres (Alpha ADN2, Alpha ADN23 y Alpha ADN30); Alpha ADN2 se usó individualmente, mientras que Alpha ADN23 y Alpha ADN30 se usaron de manera combinada. Con la técnica de SSR se evaluaron seis pares de iniciadores y se seleccionaron cuatro (M1F/M2R, M3F/M4R, M5F/M6R y M7F/M8R). Las relaciones genéticas entre genotipos se calcularon por el método de Similitud Genética. La matriz de distancias generada se utilizó para producir un dendrograma por medio de análisis de conglomerados (UPGMA). Todos los materiales fueron diferenciados por el patrón de fragmentos obtenidos con las dos técnicas utilizadas; con la técnica de RAPD se observaron un total de 26 fragmentos, de los cuales 69.23% fueron polimórficos y en SSR se obtuvieron un total de 49 fragmentos, de los cuales 83.67% fueron polimórficos. Se observó una gran similitud genética entre los materiales ya que la máxima distancia genética entre los diferentes grupos fue de 0.19. Las relaciones genéticas más cercanas fueron entre las variedades Sancal y NAU–6 con un valor de 88% en los intervalos de confianza de Felsenstein. Se identificaron ambos grupos de genotipos, unos estrechamente relacionados y otros, como la variedad Monserrat, genéticamente distintos. Esta información permitirá diseñar los cruzamientos adecuados en el programa de mejoramiento.
Palabras clave: Análisis molecular, germoplasma, relaciones genéticas.
ABSTRACT
During 2003, thirteen commercial cultivars and three elite potato clones were characterized by RAPD and SSR molecular techniques. With the RAPD technique forty primers were initially tested and three selected (Alpha DNA2, Alpha DNA23 y Alpha DNA30) for the characterization; Alpha DNA2 was used alone, while Alpha DNA23 and Alpha DNA30 were combined. With the SSR technique six primer pairs were tested and four selected (M1F/M2R, M3F/M4R, M5F/M6R and M7F/M8R). The genetic relationship among genotypes was calculated by the Genetic Similarity method. The distance matrix generated was used to produce a dendrogram by means of the cluster analysis (UPGMA). All genotypes were distinguished by the banding patterns obtained with both techniques used. With RAPD's, 26 bands were produced of which 69.23% were polymorphic, while with SSR's, 49 bands were observed, of which 83.67% were polymorphic. High genetic similarity was observed, among the potato cultivars as the maximum genetic distance between the groups was 0.19. The highest genetic relationship was observed between the cultivars Sancal and NAU–6 with a value of 88% in the Felsenstein confidence intervals. Groups of genotypes were of two kinds, some with genotypes closely related and other such as cv. Monserrat, geneticly distinct. This information will allow for the design of appropiate parental combinations in the breeding program.
Key words: Genetic relationships, germplasm, molecular analysis.
INTRODUCCIÓN
En México, anualmente se siembran alrededor de 63 762 ha de papa (Solanum tuberosum L.), con una producción de 1 500 000 t ha–1 (SAGARPA, 2003). La mayor colección de germoplasma de papa en el mundo se encuentra en Perú, que incluye más de 5000 materiales entre domesticados y silvestres (CIP, 2004). Sin embargo, se considera que existen bases genéticas reducidas, sobre todo en países tropicales y subtropicales, cuyos programas de mejoramiento genético utilizan cultivares europeos obsoletos como progenitores y que generalmente están muy relacionados (Buso, 1990). La estrecha relación genética entre los cultivares de papa y la dificultad para obtener semilla biológica de muchos cruzamientos, sugiere que es necesario el cruzamiento entre genotipos que tengan la mayor divergencia genética (Barbosa y Bered, 1998).
A pesar de que México no forma parte de la zona de origen de la papa cultivada, existe una gran diversidad de germoplasma de este cultivo. Ferroni (1981) distingue tres grupos de variedades: genotipos de Holanda, de EE. UU. y las variedades mejoradas generadas por el Instituto Nacional de Investigaciones Forestales, Agrícolas y Pecuarias (INIFAP). El primer grupo representa alrededor del 50% del cultivo a nivel nacional (variedad Alpha), 38% el segundo y el último alrededor del 8%. Los dos principales centros de diversidad y producción de papas criollas en México son los valles aledaños al Nevado de Toluca y al Pico de Orizaba. El material genético de ambas regiones ha dado origen a 17 cultivares de S. tuberosum, sin embargo, solo seis de los genotipos clonales presentan amplia adaptación. La presencia de cultivares endémicos, así como la gran diversidad morfológica en el Valle de Toluca y alrededor del Pico de Orizaba, sugieren que estas regiones son los primeros centros de variabilidad genética de papa en México (Ugent, 1968).
Históricamente, los análisis de diversidad o variabilidad genética han estado relacionados con estudios de anatomía comparativa, morfología, embriología y fisiología. Sin embargo, la influencia ambiental y el reducido número de características involucradas en este proceso han limitado el uso de estos marcadores (González y Simpson, 1997). Actualmente, los avances de la biología molecular han permitido el uso de técnicas para detectar cambios en el genotipo mediante el diagnóstico del ADN (González y Simpson, 1997). También se ha generado un gran número de métodos para identificar y analizar marcadores moleculares entre los que destacan los basados en la Reacción en Cadena de la Polimerasa (PCR), que permiten amplificar secuencias específicas o al azar del ADN y requieren de pequeñas cantidades iniciales del mismo. Entre estos marcadores están los denominados polimorfismos del ADN amplificados al azar RAPD (Random Amplified Polymorphic DNA) y los SSR (Simple Sequence Repeats) o microsatélites. Los RAPDs han sido utilizados para determinar la variación intra e interespecífica en híbridos somáticos de papa y para obtener huellas genéticas (Baird et al., 1992; Gorg et al., 1992; Demek y Adams, 1994; Carrasco et al., 1998). Los SSR's o microsatélites también han sido utilizados para establecer las relaciones genéticas entre cultivares de papa (Provan et al., 1996; Milbourne et al., 1997; Ashkenazi et al., 2000) y de otras especies como soya y cebada (Akkaya et al., 1992; Saghai et al., 1994).
Actualmente existe un gran número de colecciones de germoplasma con genotipos de un alto valor agronómico que podrían ser usados como progenitores en los programas de mejoramiento genético; sin embargo, en muchas ocasiones se desconoce su grado de diversidad y la relación existente entre materiales, lo que dificulta su utilización (Becerra y Paredes, 2000). La selección de progenitores y la caracterización de la variabilidad genética existente es decisiva en la eficiencia de los programas de mejoramiento genético (Barbosa y Bered, 1998).
El objetivo de este trabajo fue la caracterización molecular de tres líneas elite y 13 variedades comerciales de papa y determinar sus relaciones genéticas utilizando dos tipos distintos de marcadores moleculares. Esta caracterización permitirá a los mejoradores la identificación y utilización de progenitores no relacionados y complementarios en sus características.
MATERIALES Y MÉTODOS
Material genético y extracción de ADN
El trabajo se realizó durante el 2003, en la Unidad de Investigación en Biología Celular y Molecular del Instituto Nacional de Investigaciones Forestales, Agrícolas y Pecuarias (INIFAP), ubicada en la Facultad de Ciencias Biológicas de la Universidad Autónoma de Nuevo León. El material biológico utilizado en este estudio fue de 13 variedades comerciales y tres líneas elite de papa (Cuadro 1). Para asegurar la pureza de los materiales evaluados, éstos se obtuvieron del banco de germoplasma del Instituto Nacional de Investigaciones Forestales, Agrícolas y Pecuarias, ubicado en el Campo Experimental Valle de México, en Texcoco, Estado de México. En el presente trabajo se utilizaron tres plantas por variedad y se corrieron tres y cuatro ensayos diferentes en forma simultánea para cada tipo de marcador, respectivamente. La extracción de ADN genómico se realizó utilizando la metodología descrita por Almeyda (1997).

Análisis RAPD
Se evaluaron 40 iniciadores y con base en el polimorfismo generado se seleccionaron tres con diferente contenido de guanina–citosina (Cuadro 2). El iniciador Alpha ADN2 se utilizó de manera individual y los iniciadores Alpha ADN23 y Alpha ADN30 se usaron combinados. Las condiciones de reacción utilizadas fueron las reportadas por Pecina et al. (2001), con algunas modificaciones: en un volumen final de 25 µl se mezcló la solución amortiguadora al 1X, 2 mM de MgCl2, 200 µM de dNTP's, 0.5 µM de cada iniciador, 2.5 unidades de Taq polimerasa, 2 µl de ADN (50 ng) y 12.5 µl de agua estéril. Cuando se utilizó la combinación de dos iniciadores se agregaron 0.5 µM de cada iniciador. Las condiciones de la Reacción en Cadena de la Polimerasa (PCR), fueron las siguientes: a) 3 min a 94 °C; b) 35 ciclos de 1 min a 94 °C; 1 min a 36 °C y 1.5 min a 72 °C, con una extensión final de 7 min a 72 °C. Los productos amplificados se fraccionaron en geles de poliacrilamida al 6% y se tiñeron con bromuro de etidio para ser visualizados en una fuente de luz ultravioleta y ser fotografiados
Análisis SSR
En el análisis de microsatélites inicialmente se evaluaron seis pares de iniciadores reportados por Ashkenazi et al. (2000) y Ghislain et al. (2000) y con base en el polimorfismo generado se seleccionaron cuatro (Cuadro 2). Las condiciones de reacción utilizadas fueron: en un volumen final de 25 µl se mezcló solución amortiguadora al 1X, 2 mM de MgCl2, 200 µM de dNTP's, 0.5 µM de cada iniciador, 2.5 unidades de Taq polimerasa, 2 µl de ADN (50 ng) y 12.5 µl de agua estéril. Las condiciones de la PCR fueron las siguientes: a) 3 min a 94 °C; b) 35 ciclos de 1 min a 94 °C; 2 min a 47 °C y 1.5 min a 72 °C; con una extensión final de 5 min a 72 °C. Los productos de amplificación se fraccionaron en geles de poliacrilamida al 6% y se tiñeron con bromuro de etidio para ser visualizados en una fuente de luz ultravioleta y ser fotografiados.
Análisis de datos
Todos los geles se analizaron y las bandas polimórficas fueron contabilizadas. Para crear la matriz binaria se asumió que bandas que migran en el gel a la misma altura son similares. La presencia de una banda fue indicada por un número uno (1) y la ausencia como cero (0). Las relaciones genéticas entre genotipos se calcularon por el método de Similitud Genética propuesto por Nei y Li (1979), usando el paquete de software S–Plus Versión 4.0. La matriz de distancias generada se utilizó para construir un dendrograma con el algoritmo UPGMA (Unweighted Pair Group Method with Arithmetic Averages). Para obtener el intervalo de confianza para los grupos formados en cada nodo del dendrograma se uso el método reportado por Felsenstein (1985), para lo cual se realizaron 1000 repeticiones con muestreo con reemplazo de los datos originales. Los intervalos de confianza Felsenstein se refieren al porcentaje de veces que cada nodo del dendrograma es repetido en las 1000 muestras con reemplazo, lo que a su vez indica lo robusto de los grupos formados en cada nodo. Un porcentaje cercano a 100% sugiere que el grupo refleja la estructura de la población y que el error de muestreo es mínimo, mientras que bajos porcentajes (30% o menos) sugieren que existe variación entre los genotipos de algún nodo en particular, cada vez que realiza un análisis, por lo que es necesario aumentar el tamaño de muestra. Para estimar la utilidad de los sistemas de marcadores en papa, se calcularon algunos parámetros propuestos por Powell et al. (1996), como el índice de diversidad mediante la ecuación: 
donde:
Pi= Frecuencia de una banda dada en una población y S= Número total de bandas.
La proporción múltiple efectiva (PME), es el producto de la división entre el número de loci polimorficos y el número total de loci amplificados y multiplicado por el número de loci polimórficos, para cada iniciador utilizado en cualquier sistema de marcadores. El índice de marcador (IM), cuya estimación es usada para evaluar la utilidad total de cada sistema de marcadores y se define como el producto de los promedios del índice de diversidad para los fragmentos polimórficos y el PME para cada análisis.
RESULTADOS Y DISCUSIÓN
Resultados
Análisis RAPD. Las tres reacciones de amplificación de RAPDs y las cuatro de SSR amplificaron un total de 75 productos lo que permitió caracterizar el germoplasma evaluado. En general, el análisis RAPD amplificó un total de 26 fragmentos (en promedio 9.0 fragmentos por iniciador), de los cuales 18 (69.23%) fueron polimórficos. El tamaño de los fragmentos analizados fue de 200 a 2000 pb aproximadamente, aunque se observaron fragmentos de mayor y menor tamaño (Figuras 1 y 2). No se detectaron diferencias significativas en el número de fragmentos amplificados entre los iniciadores utilizados (datos no mostrados). En las mismas figuras se muestran dos repeticiones por cada genotipo evaluado para estar seguros que los resultados obtenidos son confiables y reproducibles, además se demostró que no hubo variación dentro de las líneas y variedades de papa.
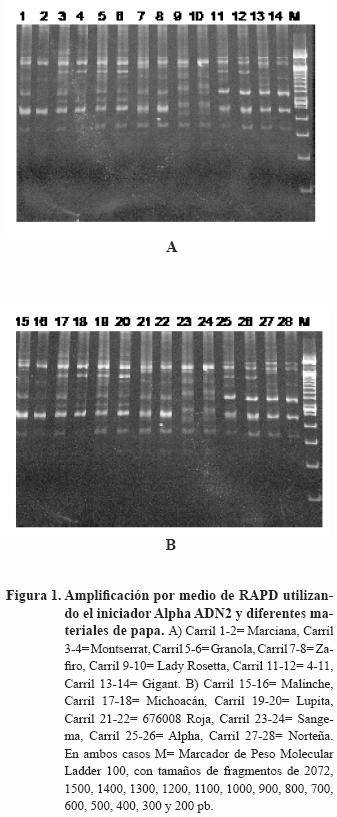
Análisis SSR. Con la técnica de SSR se amplificó un total 49 fragmentos diferentes (12.25 fragmentos en promedio), 41 (83.67%) de ellos fueron polimórficos. El tamaño de los fragmentos observados estuvo en el ámbito de 150 a 850 pares de bases (Figura 3).
Relaciones genéticas. Se formaron seis grupos diferenciales de genotipos (Figura 4). El A fue homogéneo e incluyó las variedades Sancal y NAU–6 cuya distancia genética fue de 0.15; el B solo incluyó la variedad Montserrat; el C fue heterogéneo y se incluyeron las variedades Alpha, Michoacán, Lupita, Malinche y Norteña cuyas distancias genéticas fluctuaron entre 0.18 y 0.19; el D fue homogéneo e incluyó las variedades Lady Rosetta y Gigant donde las distancias genéticas fueron de 0.18 aproximadamente; el E solo incluyó la variedad Marciana y el F fue muy heterogéneo e incluyó, por un lado, las líneas 4–11 y 676008 Roja, y por otro, las variedades Zafiro, Granola y Sangema, donde las distancias genéticas oscilaron entre 0.17 a 0.18.
Con los grupos formados y con base en las genealogías de los genotipos evaluados, se determinó que los materiales con distancias genéticas muy cercanas fueron las variedades Montserrat y Marciana que tienen una distancia genética de 0.05, que a la vez fueron los genotipos más divergentes del resto de los materiales analizados (Figura 4).
Con relación a los análisis de remuestreo se observó que únicamente cuatro subgrupos tuvieron un valor superior al 30%. Sancal y NAU–6 con 88%, Lupita y Michoacán con 66%, Alpha, Lupita y Michoacán con 39%, y Lady Rosetta y Gigant con 71% (Figura 4).
Lo anterior indica que los materiales que conforman cada uno de estos subgrupos están estrechamente relacionados entre sí. El resto de los subgrupos mostraron porcentajes bajos en los intervalos de confianza de Felsenstein.
Comparación de la utilidad de los tipos de marcadores. Con el fin de estimar la utilidad de cada sistema de marcadores se calcularon algunos parámetros reportados por Powell et al. (1996), como el Índice de Diversidad (ID), la Proporción Múltiple Efectiva (PME) y el Índice de Marcador (IM) para cada iniciador o par de iniciadores evaluados por sistema de marcadores moleculares. Los altos o bajos niveles de polimorfismo determinan el ID genética presente en un grupo de individuos. En este caso se observó un ID de 44.7 y 62.7% en los genotipos evaluados para RAPD y SSR, respectivamente. En el Cuadro 3 se muestra un resumen de las medias aritméticas para la PME de cada sistema de marcadores. El promedio de PME en RAPD fue de 6.32, mientras que para los SSR fue de 1. En el caso de los SSR esto fue debido a que este sistema de marcador sólo revela un locus simple. En cuanto al IM fue de 2.91 y 0.627 para RAPD y SSR, respectivamente.
Discusión
Los iniciadores utilizados en esta investigación se seleccionaron en una evaluación previa de genotipos de papa (Orona et al., 2003; Orona et al., 2004). Se utilizaron tres clonas por variedad y se corrieron tres ensayos de RAPDs en forma simultánea con resultados idénticos, lo que indica la reproducibilidad de los resultados y sugiere que existe un manejo adecuado de las clonas en el incremento de semilla y almacenamiento, ya que no se detectó variación dentro de la misma variedad. Lo mismo ocurrió con los microsatélites como lo cita Ghislain et al. (2000). Además, el número de bandas y porcentajes de polimorfismos observados en ambas técnicas fueron similares a los reportados en otros estudios con otros cultivares (Milbourne et al., 1997; Isenegger et al., 2001, Silveira et al., 2004), por lo que estos marcadores pueden ser usados como una herramienta para detectar diferencias genéticas entre genotipos relativamente cercanos (Ashkenazi et al., 2000; Raker y Spooner, 2002).
Las técnicas de SSR y RAPD utilizadas en el presente estudio diferenciaron los 16 genotipos evaluados y permitieron establecer las relaciones genéticas entre variedades a pesar de que la papa es un cultivo propagado clonalmente, por lo que la variación dentro de la especie es muy bajo comparado con especies de reproducción sexual (Barbosa y Bered, 1998). El análisis de agrupamiento observado en la Figura 4, refleja una correlación entre los grupos definidos por Ferroni (1981), ya que los subgrupos en su mayoría estuvieron formados por separado con materiales mexicanos y con materiales de origen europeo. Otros subgrupos estuvieron formados por materiales mexicanos y materiales de origen europeo mezclados, lo anterior no es nada sorprendente y puede deberse a que existe correlación entre sus coancestros y refleja afinidad en su pedigrí (Demek et al., 1996). Los valores de 88, 66 y 71% respectivamente, observados en el remuestreo con reemplazo (Felsenstein, 1985), en los subgrupos formados por los cultivares Sancal y NAU–6; Michoacán y Lupita, Lady Roseta y Gigant, confirman la estrecha relación genética entre estos pares de materiales y los cruzamientos entre ellos no son recomendables.
El agrupamiento de unidades de distancias mínimas y promedios aritméticos propuesto por Nei y Li (1979), permitieron distinguir las diferencias genéticas existentes entre los 16 genotipos, lo que concuerda con los resultados obtenidos en otros trabajos (Miller y Spooner, 1999) (Figuras 1, 2 y 3). Así mismo, el desarrollo de microsatélites en Solanum tuberosum analizados con el modelo propuesto por Nei y Li (1979) (Figura 4), soportan las diferencias genéticas de la mayoría de los genotipos, como ha sucedido en estudios reportados por Raker y Spooner (2002).
Los bajos niveles de confiabilidad observados en algunos subgrupos por el análisis de remuestreo con reemplazo (Felsenstein, 1985), se atribuye al bajo número de ensayos realizados tanto en RAPD como en SSR, sin embargo, esto no influyó en la caracterización genética de los materiales como ha sido reportado en papa (Isenegger et al., 2001; Bornet et al., 2002; Silveira et al., 2004). Los valores de confiabilidad se pueden mejorar, incrementando el número de ensayos en ambas técnicas. Al comparar las relaciones genéticas entre los dos sistemas de marcadores aunque la correlación entre ambos fue intermedia (r = 0.56) esta fue superior a la reportada en otros estudios (Milbourne et al., 1997). Con ambas técnicas se observó una estrecha relación entre los grupos formados por las variedades Sancal y NAU–6 así como Sangema, Granola y Zafiro, aún cuando no fue en el mismo orden. Con la técnica de SSR se logró una mejor discriminación entre genotipos, lo que coincide con lo reportado por varios investigadores, que le atribuyen a esta técnica mayor eficiencia para identificar variación en sitios determinados (Powell et al., 1996; Milbourne et al., 1997; Ashkenazi et al., 2000; Raker y Spooner, 2002). El hecho de que la variedad Montserrat fuera la más divergente y se desligara de los otros materiales tanto en RAPD como en SSR, es un indicador de la distancia genética de este cultivar con el resto y que pudiera ser explotada en programas de mejoramiento donde se incluya esta variedad como progenitor.
Los porcentajes de diversidad genética obtenida en los análisis RAPD y SSR (44.7 y 62.7 %), reflejan la variación observada por cada sistema de marcador, y son comparables a los valores obtenidos por Milbourne et al. (1997). Mientras que para PME, los marcadores RAPD presentaron valores mayores que los SSR (6.32 y 1), lo cual es normal debido a que éstos revelan un solo locus, mientras que en el caso de los RAPD se asume que cada banda pertenece a un locus distinto. Por otra parte, si se considera que la papa es una especie tetraploide, se puede asumir que en un locus pueden existir cuatro alelos distintos, por lo tanto, en este caso es contradictorio señalar que en los análisis SSR sólo se está amplificando un solo locus. Por lo tanto, el valor de 1 asignado en PME para SSR incidió en bajos valores de IM, aún cuando pudieran utilizarse combinaciones de oligonucleotidos iniciadores que puedan revelar más de un locus (Milbourne et al., 1997).
CONCLUSIONES
Las técnicas de RAPD y SSR, fueron eficientes para caracterizar las variedades y clones mejorados de papa incluidos en el estudio; con SSR se logró una mejor discriminación entre genotipos.
Se identificaron ambos grupos de genotipos, unos estrechamente relacionados y otros, como la variedad Monserrat, genéticamente distintos. Esto permitirá diseñar los cruzamientos adecuados en el programa de mejoramiento.
LITERATURA CITADA
Akkaya, M. S.; Bhagwat, A. A. and Cregan, P. B. 1992. Length polymorphisms of simple sequence repeat DNA in soybean. Genetics 132:1131–1139. [ Links ]
Almeyda L., I. H. 1997. Detección molecular de fitoplasmas y su uso en el diagnóstico del amarillamiento letal del cocotero. Tesis de Doctor en Ciencias. Universidad Autónoma de Nuevo León, División de Estudios de Postgrado. San Nicolás de los Garza, N. L., México. 88 p. [ Links ]
Ashkenazi, V.; Chani, E.; Lavi, U.; Levy, D.; Hillel, J. and Veilleux, R. E. 2000. Development of microsatellite markers in potato and their use in phylogenetic and fingerprinting analysis. Genome 44:50–62. [ Links ]
Baird, E.; Coope, B. S.; Waugh, R.; De Maine, M. and Powell, W. 1992. Molecular characterization of inter–specific and intra–specific somatic hybrids of potato using randomly amplified polymorphic DNA (RAPD) markers. Mol. Gen. Genetic. 233:469–475. [ Links ]
Barbosa N., J. F. and Bered, F. 1998. Marcadores moleculares e diversidade genética no melhoramento de plantas. In: Milack, S. (ed.). Marcadores moleculares en plantas. Porto alegre UFRGS. p. 29–41. [ Links ]
Becerra V., V. y Paredes C., M. 2000. Uso de marcadores bioquímicos y moleculares en estudios de diversidad genética. Agric. Téc. (Chile) 60(3):270–281. [ Links ]
Bornet, B.; Goraguer, F.; Joly, G. and Branchard, M. 2002. Genetic diversity in european and argentinian cultivated potatoes (Solanum tuberosum subsp. Tuberosum detected by inter–simple sequence repeats (ISSRs). Genome 45:481–484. [ Links ]
Buso, J. A. 1990. Os programas de melhoramento genético de batata no Brasil. In: Hidalgo, O. A. and Rincón, H. R. (eds.). Avances en el mejoramiento genético de la papa en los países del Cono Sur. Centro Internacional de la Papa. Lima; Perú. p. 31–34. [ Links ]
Carrasco, A.; Ruiz de G, J. I. and Ritter, E. 1998. Caracterización morfológica, cariotípica y molecular de tres somaclones de Solanum tuberosum L. obtenidos mediante cultivo de protoplastos. Invest. Agr. Prod. Prot. Veg. 13(3):385–391. [ Links ]
Centro Internacional de la Papa. (CIP). 2004. About potatoes. Lima, Perú. [ Links ]
Demek, T. and Adams, R. P. 1994. The use of PCR–RAPD analysis in plant taxonomic and evolution. In: Griffing, H. G. and Griffing, A. M. (eds.) PCT Technology Current Innovations. CRC Press. Norwich, U. K. p. 179–214. [ Links ]
Demek, T.; Lynch, D. R.; Kawchuk, L. M.; Kosub, G. C. and Armstrong, J. D. 1996. Genetic diversity of potato determined by random amplified polymorfic DNA analysis. Plant Cell Rep. 15:662–667. [ Links ]
Felsenstein, J. 1985. Confidence limits on phylogenies: an approach using the bootstrap. Evolution 39: 783–791. [ Links ]
Ferroni, M. A. 1981. El potencial de la papa como recurso alimenticio y como fuente de ingreso. Instituto Nacional de Investigaciones Agrícolas, Programa Regional Cooperativo de Papa. Toluca, Edo. de México, México. 30 p. (Publicación Especial Núm. 1.). [ Links ]
Ghislain, M.; Rodríguez, F.; Villamón, F.; Núñez, J.; Waugh, R. and Bonierbale, M. 2000. Establishment of microsatellite assays for potato genetic identification. Centro Internacional de la Papa. Program Report. 1999–2000. Lima, Perú. p. 167–174. [ Links ]
González Ch., M. y Simpson, J. 1997. Diversidad genética en hongos (origen y análisis). In: Martínez S., J. P. y Martínez E., A. D. (eds.) Tópicos Selectos de Fitopatología: Genética Molecular. Centro de Investigación y Estudios Avanzados del Instituto Politécnico Nacional–Unidad Irapuato. Guanajuato, México. p. 4–23. [ Links ]
Gorg, R.; Schachtschabel, U.; Ritter, E.; Salamini, F. and Gebhardt, C. 1992. Discrimination among 136 tetraploid potato varieties by fingerprints using highly polymorphic DNA markers. Crop Sci. 32:815–819. [ Links ]
Isenegger, D. A.; Taylor, P. W. J.; Ford, R.; Franz, P.; McGregor, G. R. and Hutchinson, J. F. 2001. DNA fingerprinting and genetic relationships of potato cultivars (Solanum tuberosum L.) commercially grown in Australia. Aust. J. Agr. Res. 52(9):911–918. [ Links ]
Milbourne, D.; Meyer, R.; Bradshaw, J. E.; Baird, E.; Bonar, N.; Provan, J.; Powell, W. and Waugh, R. 1997. Comparison of PCR–based marker systems for the analysis of genetics relationships in cultivated potato. Mol. Breed. 3:127–136. [ Links ]
Miller, L.T. and Spooner, D. M. 1999. Collapse of species boundaries in the wild potato Solanum brevicaule complex (Solanaceae, S. sect. Petota): Molecular data. Plant Syst. Evol. 214:103–130. [ Links ]
Nei, M. and Li, W. 1979. Mathematical model for studying genetic variation in terms of restriction endonucleases. Proc. Natl. Acad. Sci. USA. 76:5269–5273. [ Links ]
Orona C., F.; Pecina Q., V.; Rocha P., M. A.; Parga T., V. M. y Almeyda L., I. H. 2003. Caracterización molecular de 15 genotipos de papa (Solanum tuberosum L.) mediante RAPD's Y SSR's. In: Décima Sexta Reunión Científica Tecnológica Forestal y Agropecuaria, Veracruz 2003 y Primer Foro Regional de Discusión y Análisis de la Investigación Agropecuaria, Forestal y Pesquera. Resumen de Memorias. Veracruz, México. p. 19. [ Links ]
Orona C., F.; Pecina Q., V.; Rocha P., M. A.; Parga T., V. M.; Martínez de la V., O. y Almeyda L., I. H. 2004. Caracterización de variedades y líneas elite de papa (Solanum tuberosum L.) en México utilizando marcadores RAPD y SSR. Phyton 2004:289–300. [ Links ]
Pecina Q., V.; Martínez de la V., O.; Alvarado B., M. de J.; Vandemark, J. G. and Williams A., H. 2001. Comparación de dos sistemas de marcadores moleculares en el análisis de las relaciones genéticas de Macrophomina phaseolina (Tassi) Goid. Rev. Mex. Fitopatol. 19(2):128–139. [ Links ]
Powell, W.; Morgante, M.; Andre, C.; Hanaffey, M.; Vogel, J.; Tingey, S. and Rafalski, A. 1996. The comparisons of RFLP, RAPD, AFLP and SSR (microsatellite) markers for germoplasm analysis. Mol. Breed. 2:225–238. [ Links ]
Provan, J.; Powell, W. and Waugh, R. 1996. Microsatellite analysis of relationships within cultivated potato (Solanum tuberosum). Theor. Appl. Genet. 92:1078–1084. [ Links ]
Raker, M. C. and Spooner, D. M. 2002. Chilean tetraploid cultivated potato, Solanum tuberosum, is distinct from the Andean populations: microsatellite data. Crop Sci. 42:1451–1458. [ Links ]
Secretaría de Agricultura, Ganadería, Desarrollo Rural, Pesca y Alimentación (SAGARPA). 2003. Servicio de información y estadística agroalimentaria y pesquera. Cultivo de papa. México, D. F. p. 550–563. [ Links ]
Saghai, M. M. A.; Biyasher, R. M.; Yang, G. P.; Zhang, Q. and Allard, R. W. 1994. Extraordinarily polymorphic microsatellite DNA in barley species diversity, chromosomal locations and populations dynamics. Proc. Natl. Acad. Sci. USA 91:5466–5470. [ Links ]
Silveira C., E. A.; Choer, E. and da Silva, P. A. 2004. Characterization of potato genotypes using molecular markers. Pesq. Agropec. Bras. 39(9):871–878. [ Links ]
Ugent, D. 1968. The potato in Mexico: Geography and primitive culture. Econ. Bot. 22:108–123. [ Links ]














